养殖红鳍东方鲀的肠和胃转录组比较分析
2022-01-11周金旭王盛南胡子文余云登仇雪梅王秀利
周金旭,王盛南,胡子文,余云登,仇雪梅,王秀利
(大连海洋大学水产与生命学院,辽宁大连 116023)
红鳍东方鲀Takifugu rubripes 作为一种海洋洄游性鱼类,虽然其内脏器官中含有地球上最致命的河鲀毒素(tetrodotoxin,TTX)[1],但其鱼肉却作为一种昂贵的美食被许多地方的人们所喜爱。红鳍东方鲀的许多器官中都含有TTX,其在肝脏和卵巢中含量最高,其次是肠道,但在皮肤、睾丸、肌肉中却几乎不存在TTX。由于红鳍东方鲀的养殖和食用逐渐开放,红鳍东方鲀也慢慢成为中国的餐桌美食和出口创汇的主要海产品。基于红鳍东方鲀人工繁育技术取得了成功[2],且在已有研究中发现了养殖红鳍东方鲀的营养品质要优于野生的红鳍东方鲀[3],所以提高养殖红鳍东方鲀对饵料的利用效率也成为新的研究目标,而对肠和胃的研究则是饵料研究的基础。
转录组技术是一种特殊测序技术,既能揭示给定时刻生物样品中RNA 的表达量,也能揭示基因表达调控的机制[4]。由于转录组能够从整体水平上研究基因的功能和基因的结构,并能揭示某些特定的生物学过程和分子机理,所以转录组测序技术已经被广泛的应用于植物、动物、微生物的研究中[5-6]。由于红鳍东方鲀的基因组非常小,使其成为继人类基因组公开发布后的第一个脊椎动物基因组,虽然其基因组仅为人类基因组大小的八分之一,但它含有与人类基因组相似的基因库[7],所以对红鳍东方鲀的转录组进行研究具体重要意义。
目前,已有一些对红鳍东方鲀生长、摄食、消化生理等不同方面的研究[8-10],但对红鳍东方鲀消化组织转录组方面的研究较少,本研究利用转录组测序技术对一龄的红鳍东方鲀的肠和胃进行测序。根据差异基因分析,简单探究红鳍东方鲀肠和胃之间的功能差异,同时也为其他生物肠道基因的研究提供参考,此外对红鳍东方鲀肠和胃进行研究也可为今后养殖红鳍东方鲀饵料选择和提高其饵料系数提供思路。
1 材料和方法
1.1 实验材料
红鳍东方鲀的养殖实验在大连天正公司的大黑石养殖场进行。实验先将1 龄90 尾红鳍东方鲀分为3组,每组30 尾,在溶氧7~8 mg·L-1,pH 7~8,温度17~18 ℃的条件下饲养7 d,每天上午8 点和下午2 点进行定点投喂,在采样前一天停止投喂。在正式实验中,从每组红鳍东方鲀中随机选取10 尾,将每尾红鳍东方鲀的肠和胃全部取出,使用PBS 缓冲液将其中内容物清洗干净,取1 cm 的肠和胃,按分组置于6 个不同的10 mL 无酶离心管,每组织间的3 个混合样本作为3 个生物学重复。胃混合样本分别标号SA1、SA2和SA3,肠的混合样本分别标号IA1、IA2 和IA3。每个离心管中均加入超过样品5 倍体积的RNAlater 保存备用。
1.2 总RNA 的提取、cDNA 文库的构建与测序
将每组红鳍东方鲀的肠和胃组织分别置于液氮中进行研磨,研磨后的样品使用Trizol 试剂(宝生物公司)进行总RNA 提取,利用1%琼脂凝胶电泳分析RNA 完整性和RNA 是否存在DNA 的污染,再使用HAgilent 2100 仪器对样品的RNA 纯度、浓度和完整性进行检测。在红鳍东方鲀的样本质检合格后委托诺禾致源公司进行测序。按照NEB 普通建库方式进行cDNA 文库构建,建库试剂盒为Illumina 的NEBNextUltraTM RNA Library Prep Kit[11]。先通过磁珠富集带有polyA 尾的mRNA,其在逆转录酶体系中合成单链cDNA,再合成双链,双链cDNA 经过末端修复和加polyA 尾,用AMPure XP beads 筛选250~300 bp 左右的cDNA,最终获得文库。
1.3 数据处理
利用高通量测序仪将测序片段转化为序列数据(reads)。为了使序列数据分析具有较高的质量及更高的可靠性,实验对原始数据进行过滤,过滤标准:1 去除带接头(adapter)的reads;2 去除含N(N 表示无法确定碱基信息)的reads;3 去除低质量reads(Qphred<=20 的碱基数占整个read 长度的50%以上的reads)。使后续分析均是基于过滤数据的高质量分析。此外还对过滤数据进行相应的Q20、Q30 和GC 含量计算。
1.4 数据分析
使用HISAT2v2.0.5 注释和构建参考基因组的索引,将过滤数据与红鳍东方鲀参照基因组进行比对[12]。再利用DESeq2 对原始的read count 进行标准化和检验校正,得到一个FDR 值,以padj<0.05 为标准对差异基因进行筛选[13-14]。将筛选出的差异基因通过clusterProfiler(3.4.4)软件与GO 数据库进行比对分析,再利用clusterProfiler(3.4.4)软件分析KEGG 通路中差异表达基因的富集情况。
1.5 RT-qPCR 验证
为了验证转录组实验结果的准确性,选择以β-actin 为内参基因[15],筛选具有代表性的6 个基因进行RT-qPCR 验证,每个样品设3 个生物重复,并进行3 次技术重复。采用Primer Premier 5 软件设计引物,引物信息见表(表1)。使用ABI 公司的StepOne plus 定量仪进行实验,其反应体系为:cDNA 1 μL,2×TransStart PCR SuperMix 10 μL,DyeⅠ0.4 μL,上、下游引物各0.4 μL,RNase free H2O 7.8 μL。最后相对表达量采用2-ΔΔCt法计算。

表1 RT-qPCR 引物信息Tab.1 RT-qPCR primer information
2 结果与分析
2.1 测序数据质控分析
本研究对红鳍东方鲀肠和胃进行了转录组测序,测序片段经CASAVA 碱基识别转化为序列数据(reads),后对原始数据进行过滤并进行相应的Q20、Q30 和GC 含量计算(表2)。数据结果显示,每个样本序列数据在4.89×107~6.37×107bp 之间,过滤后的序列数据(clean reads)在4.82×107~6.28×107bp 之间,所测数据的碱基长度在7.23~9.42 G 之间,Q20 在97.55%~97.21%之间,Q30 在92.55%~93.24%之间,GC 含量在49.86%~51.06%之间。以上数据表明测序数据真实可靠,且具有优质的建库质量,数据可用于之后的分析。
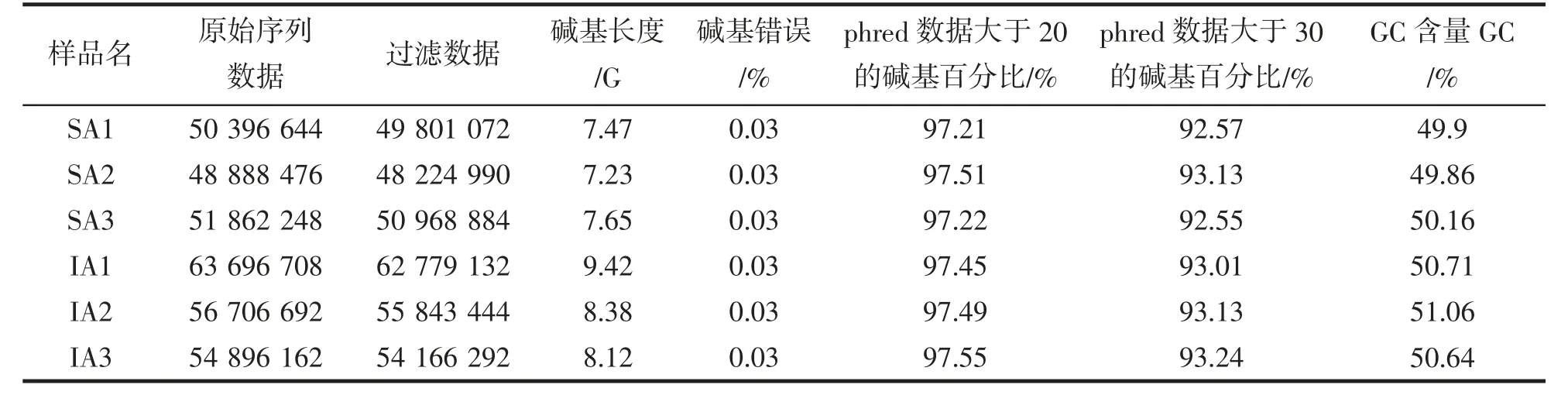
表2 红鳍东方鲀肠和胃测序数据质量Tab.2 Quality of T.rubripes intestine and stomach sequencing data
2.2 转录组与参考基因组的比对分析
将质控后的过滤数据与红鳍东方鲀的参考基因组(fTakRub1.2(GCF 901000725.2))进行比对(表3)。结果表明6 个样本的过滤数据与参考基因组的mapping 率在94.24%~86.17%,单一位置的mapping 率为90.1%~78.82%,多位置mapping 率都小于7.35%。实验中各样本mapping 率的比对效率都相对较高,这说明参考基因组的组装较好。样本与参考基因组的亲缘关系近,样本的外源污染较少、样本整体测序质量较好。
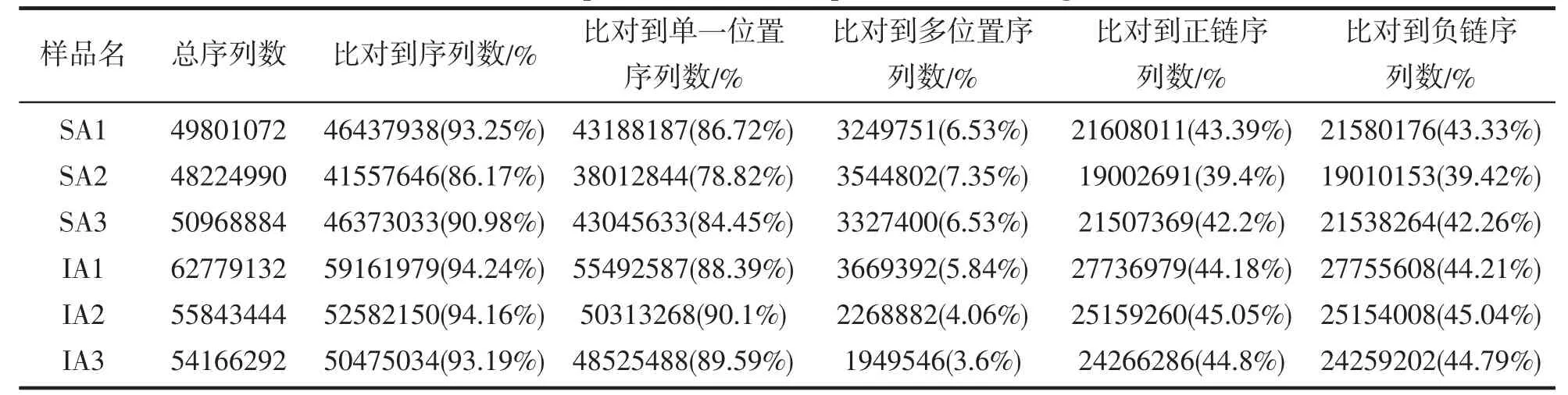
表3 转录组数据与基因组比对Tab.3 Comparison of transcriptome data with genome
2.3 差异基因分析
实验将1 龄红鳍东方鲀肠和胃(SA13vsIA13)的表达基因进行比对,再使用DESeq2 软件,以|log2(FoldChange)|>0 或padj<0.05 作为标准筛选差异基因,如果差异基因在胃表达量高于肠表达量(即log2(FoldChange)>0)则为上调,反之则为下调(图1)。本研究在比较分析中共获得3 564 个差异表达基因,其中包括个2 167 上调表达的差异基因,1 397 个下调表达的差异基因。由此分析可以看出肠和胃(SA13vsIA13)的差异基因多数呈上调表达,少数呈下调表达。此外经数据分析发现了许多与有意义的差异基因,其中包括回肠型脂肪酸结合蛋白基因(fabp6)表达差异最大(log2FC=-14.14262182),fabp6 在胃中几乎不表达,但在肠中却大量表达,脂肪酸转位酶基因(cd36)差异最显著(FDR=1.60×10-42),cd36 在胃中表达较低,但在肠中却大量表达。此外还有胆固醇酰基转移酶(soat2 或acat2)、载脂蛋白(apoA-IV2)也呈下调表达。反之则筛选到了紧密连接蛋白(cldn3a)和钙黏着蛋白(cdh2)在肠和胃(SA13vsIA13)比较中呈上调表达。
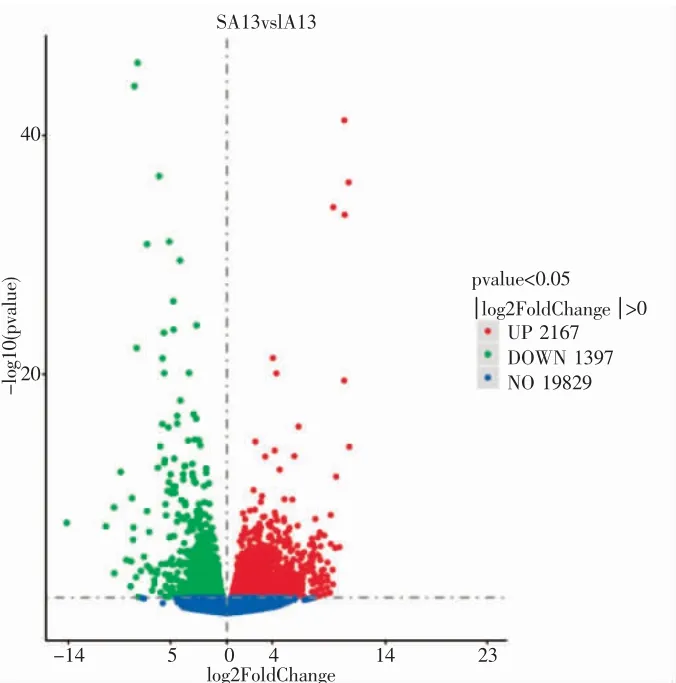
图1 红鳍东方鲀肠和胃差异基因Fig.1 Differential genes of intestine and stomach of T.rubripes
2.4 GO 功能富集分析
实验将红鳍东方鲀肠和胃(SA13vsIA13)比较中筛选到差异基因注释到GO(gene ontology,基因本体论)上,通过注释将这些差异基因分为3 类包括:(1)生物过程(biological processes BP)、(2)细胞组成(cell composition CC)、(3)分子功能(Molecule Function MF)(图2)。GO 富集结果表明:本实验将1652 差异基因注释到915 个GO 术语上,其中发现18 个具显著富集(padj<0.05)的GO 术语,在生物过程有5 个GO 术语(term),细胞组成有1 个GO 术语,分子功能有12 个GO 术语。在显著富集的GO 术语中:在细胞组成中胞膜外区(extracellular region)注释到了最多的差异基因共77 个。此外在分子功能中酶调节活动(enzyme regulator activity)注释到45 个差异基因;细胞骨架结合(cytoskeletal protein binding)注释到44 个差异基因;跨膜转运蛋白活性(active transmembrane transporter activity)注释到37 个差异基因等。
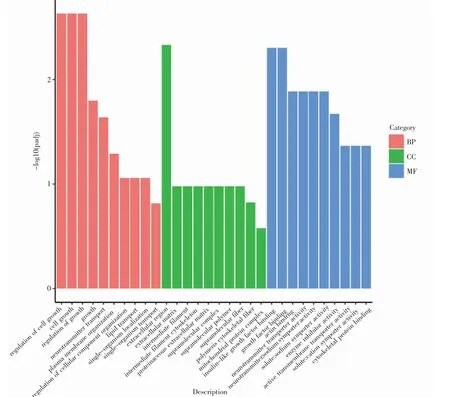
图2 红鳍东方鲀肠和胃GO 富集分析Fig.2 GO enrichment analysis of intestine and stomach of T.rubripes
2.5 KEGG 功能富集分析
实验将红鳍东方鲀肠和胃(SA13vsIA13)比较中筛选到差异基因注释到KEGG(kyoto encyclopedia of genes and genomes),京都基因与基因组百科全书)上,以此系统分析这些差异基因的关联性(图3)。KEGG 富集结果表明,本实验将989 个差异基因,富集到151 条通路上,其中有15 条通路显著富集(padj<0.05),这些显著富集的通路中花生四烯酸代谢(arachidonic acid metabolism)为差异最显著的通路且富集到21 个差异基因。按富集到差异基因数量由高到低进行通路排序前三个通路是粘着连接(focal adhesion)共富集到61 个基因,其次是紧密连接(tight junction)富集到了56 个基因,最后是细胞粘附分子(cell adhesion molecules,CAMs)富集到了50 个基因。此外还富集到大量与代谢有关的通路存在显著富集:甘油酯代谢(glycerolipid metabolism)、鞘脂代谢(sphingolipid metabolism)、胆固醇代谢(cholesterol metabolism)等通路。
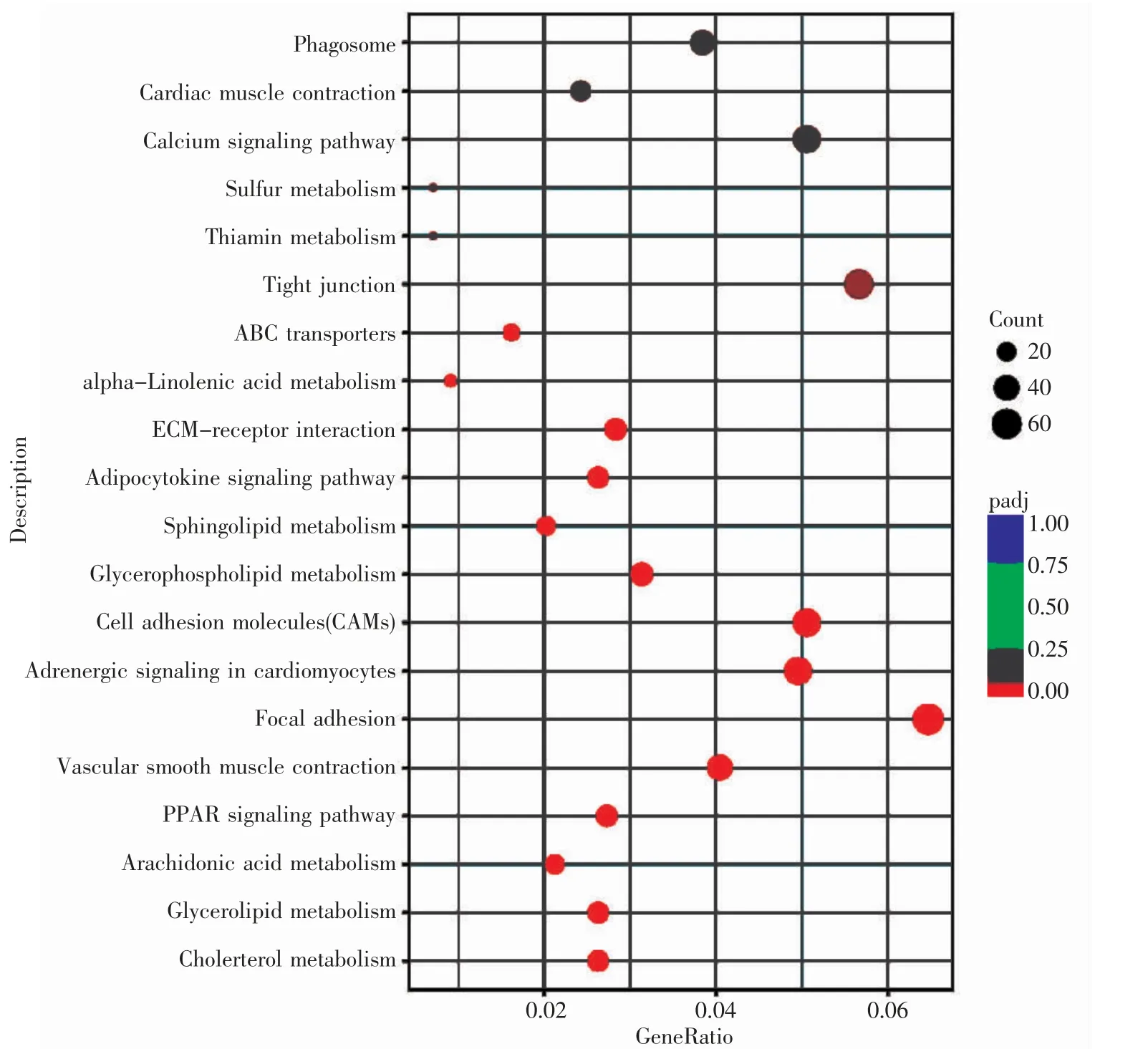
图3 红鳍东方鲀肠和胃KEGG 富集分析Fig.3 KEGG enrichment analysis of intestine and stomach of T.rubripes
2.6 RT-qPCR 验证结果
采用染料法(SYBR 法)对筛选到的6 个基因:回肠型脂肪酸结合蛋白(fabp6)、脂肪酸转位酶(cd36)、胆固醇酰基转移酶(soat2)、载脂蛋白(apoA-IV2)、紧密连接蛋白(cldn3a)、钙黏着蛋白(cdh2)进行实时荧光定量PCR 实验,结果显示:虽然转录组与RT-qPCR 在表达量和差异倍数存在一些差异,但各基因所表达的趋势是一致的,这进一步的表明转录组数据是较准确可靠的。
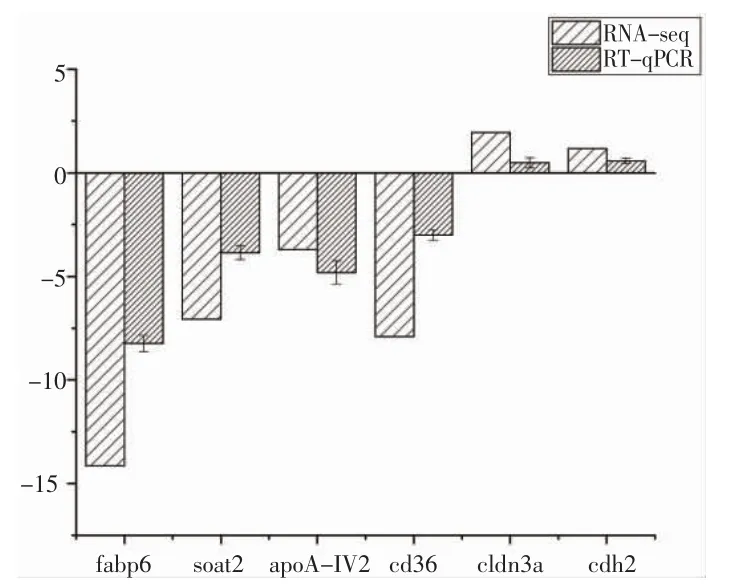
图4 红鳍东方鲀6 个基因的RT-qPCR 验证结果Fig.4 RT-qPCR validation results of six genes of T.rubripes
3 讨论
消化系统是生物食物消化和生长的基础,对于鱼类来说消化系统不仅具有消化吸收的功能,同时也具有免疫的功能,能防止有害物质进入体内。研究消化系统的基因表达是了解鱼类的营养需求和提高饵料系数的基础。肠和胃既是消化系统中重要的组成部分,也是鱼体中主要食物的消化和吸收场所。关于红鳍东方鲀有无胃的问题一直存在争论,有研究有认为红鳍东方没有胃,理由是其没有胃腺的存在[16]。另外也有研究认为红鳍东方鲀有胃,理由是其存在明显的分隔[17]。本研究的红鳍东方鲀胃组织中没有发现胃蛋白酶原的表达,这与黑川等[18]的研究一致。由以上研究分析发现红鳍东方鲀具有胃的结构,但缺少部分胃的功能。
本研究通过转录组测序方法分析肠和胃中的差异基因,发现了许多与代谢和消化相关的基因如回肠型脂肪酸结合蛋白(fabp6)、脂肪酸转位酶(cd36)、胆固醇酰基转移酶(soat2 或acat2)、载脂蛋白(apoA-IV2)、紧密连接蛋白(cldn3a)、钙黏着蛋白(cdh2)。fabp6 是一种脂肪酸载体蛋白,与脂肪酸的吸收、转运和代谢密切相关。此外还有研究发现脂肪酸结合蛋白与肌肉的生长密切相关[19]。在已有研究中发现绿河鲀Tetraodon nigroviridis 的fabp6 在肠中高表达[20];在小鼠的肠道中可以检测到fabp6 的表达,在胃中检测不到[21],这与本研究结果相一致。fabp6 在肠中高表达可能是因为胆中一些成分可以调控肠上皮细胞中fabp6 基因的表达,参与肠中胆汁盐的转运和类固醇激素的代谢[22];在胃中表达量很低可能是因为红鳍东方鲀具有胃的结构但是缺少胃的功能,缺少可以调控fabp6 基因表达的物质。cd36 是一种在脊椎动物中发现的完整膜蛋白,其参与了脂肪酸代谢[23]和肠道中的脂肪加工[24]。soat2 通过参与小肠胆固醇的吸收和肝脏中脂蛋白的分泌来调节胆固醇的代谢[25]。apoA-IV2 可以结合和运输血脂到机体各组织进行代谢和利用,对于维持体内平衡起重要作用[26]。本研究发现以上的基因均在肠和胃(SA13vsIA13)比较中也均呈下调表达,这表明了在红鳍东方鲀肠中这些基因的表达量更高,发挥了更多的作用,所以推测其肠代谢和消化的功能更强。但在斑马鱼消化功能研究中则发现,斑马鱼的前肠(胃)、中肠的消化功能更强[27],这样消化功能的不同可能与两种鱼的食性不同有关。cldn3a 是保护肠道的屏障、防止有害大分子物质进入生物体内[28]。cdh2 通过介导细胞间的粘附,以此促进粘附结构的形成和稳定[29]。cldn3a 和cdh2 在本研究中均呈上调表达。根据以上研究推测红鳍东方鲀胃的免疫和保护功能更发达。
在KEGG 的富集中发现大量与代谢有关的通路存在显著富集:花生四烯酸代谢、甘油酯代谢、胆固醇代谢等,这些代谢通路中大多数的差异基因在肠中表达量高于胃呈下调表达。但在粘着连接、紧密连接、细胞粘附分子等通路中大多数的基因在肠中表达量低于胃呈上调表达。通过KEGG 富集分析了差异基因之间的关联性,进一步证明本研究推测的红鳍东方鲀胃的消化功能可能不太发达,其消化和吸收可能主要是在肠道中进行。已有的研究表明,pH 影响消化道蛋白酶的活性[30],且在微颗粒饲料中添加外源性酶对鱼的生长有明显的促进作用[31]。以此推测由于胃中pH 偏酸性,肠中pH 偏碱性[32],而红鳍东方鲀消化可能主要是在肠中进行,选择在红鳍东方鲀的饲料中添加适量偏碱性环境的外源性酶,可能更能提高其饲料的利用率。
本文通过对红鳍东方鲀肠和胃的转录组比较分析,研究2 种组织在基因表达上的差异性。在肠相对胃的比较中共获得3 564 个差异表达基因,其中2 167 个上调表达,1 397 个下调表达。在GO 富集结果中酶调节活动、细胞骨架结合、跨膜转运蛋白活性等功能存在显著富集。在KEGG 的富集分析中发现大量与代谢有关的通路存在显著富集,这些代谢通路中大多数的差异基因在肠中表达量高于胃呈下调表达。在粘着连接、紧密连接、细胞粘附分子等通路中也存在显著富集,其中大多数的基因在肠中表达量低于胃呈上调表达。通过对相关差异基因和信号通路的研究,为今后养殖红鳍东方鲀饵料选择和提高饵料系数奠定了基础。
