结合互信息的因子分析对患癌因素的分类仿真
2021-11-17孙士保赵鹏程李玉祥李元颖
孙士保,赵鹏程,李玉祥,李元颖
(1. 河南科技大学信息工程学院,河南 洛阳 471023;2. 河南科技大学临床医学院,河南 洛阳 471023)
1 引言
随着“人工智能+医疗”技术的发展,智慧医疗越来越被人们所熟知和接受。人工智能(AI)正在越来越多地被开发用于治疗和诊断以及对患病风险进行评估和分类[1]。Manogaran Gunasekara等人测量跨基因组DNA高维数据集来诊断癌症,使用贝叶斯隐马尔可夫模型(HMM)与高斯混合(GM)聚类方法进行处理[2]。廖志军等人利用随机森林分类算法提取mRNA特征应用于六种癌症的诊断[3]。夏春秋通过低秩表示从高维基因数据中找到具有判别力的特征再对癌症进行分类[4]。Moloud Abdar则是利用置信度加权投票方法和增强集合技术对早期乳腺癌进行诊断[5]。Subhashis Banerjee 等人在选择重要特征的同时利用自适应神经模糊分类器对脑瘤分类,达到85.83%的分类正确率[6]。
综上所述的研究都是对患者进行诊断,但是在早期阶段的大多数癌症均没有明显症状,当诊断出癌症时,早期治疗的延误会增加病患的致死率,导致无法挽回的后果。因此,在智慧医疗领域中迫切需要准确对早期患癌风险进行筛查,尽早的发现癌症并进行治疗,最大限度的延长患者的生命。研究者康桂霞使用ReliefF算法分析癌症的最具辨别力的特征,通过决策树来预测癌症的风险[7]。Reedy Jill采用因子分析和指数分析比较3种膳食模式方法导致结直肠癌的风险[8]。王云溪应用因子分析和Logistics回归模型分析胃溃疡癌变的潜在预测因子[9]。面对早期癌症数据这类高维复杂性数据集,在处理过程中,采取因子分析的主要是将具有错综复杂关系的变量(或特征)综合为若干个因子,以解释原始数据与因子之间的相互关系,达到特征选择和降维的目的,以便算法模型的预测和分类效果有效,降低计算复杂度[10]。但是因子分析在计算因子得分时用到最小二乘法、极大似然法,在面对非线性关系时容易失效,使得分类效果欠佳。因子分析更倾向于描述原始变量之间的相关关系。
本文为解决传统的因子分析特征选择算法中(相关性矩阵)协方差矩阵只能够衡量具有相关关系的特征。将互信息引入到因子分析中进行特征选择,由于互信息能够利用信息熵衡量特征与类别或者两个特征之间依赖程度的强弱,展现出两个特征间拥有共同信息的含量,并且不局限于线性关系[11]。从而更有效地对高维数据进行特征选择,用以提高算法的分类精度。因此,提出一种结合互信息的因子分析对患癌因素的分类方法。人们可对早期癌症风险因素进行分类,避免延误最佳治疗时机。
2 因子分析
因子分析是当前特征选择中应用最为广泛的方法之一[12]。在高维数据中,因子分析通过多个特征间协方差矩阵的内部依赖性关系,找到能够反映出所有特征主要信息的公因子。
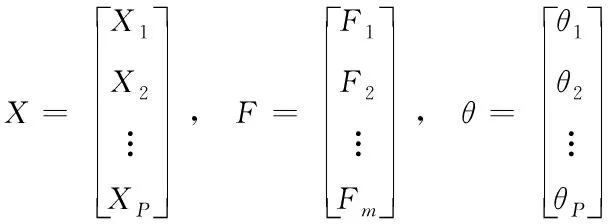
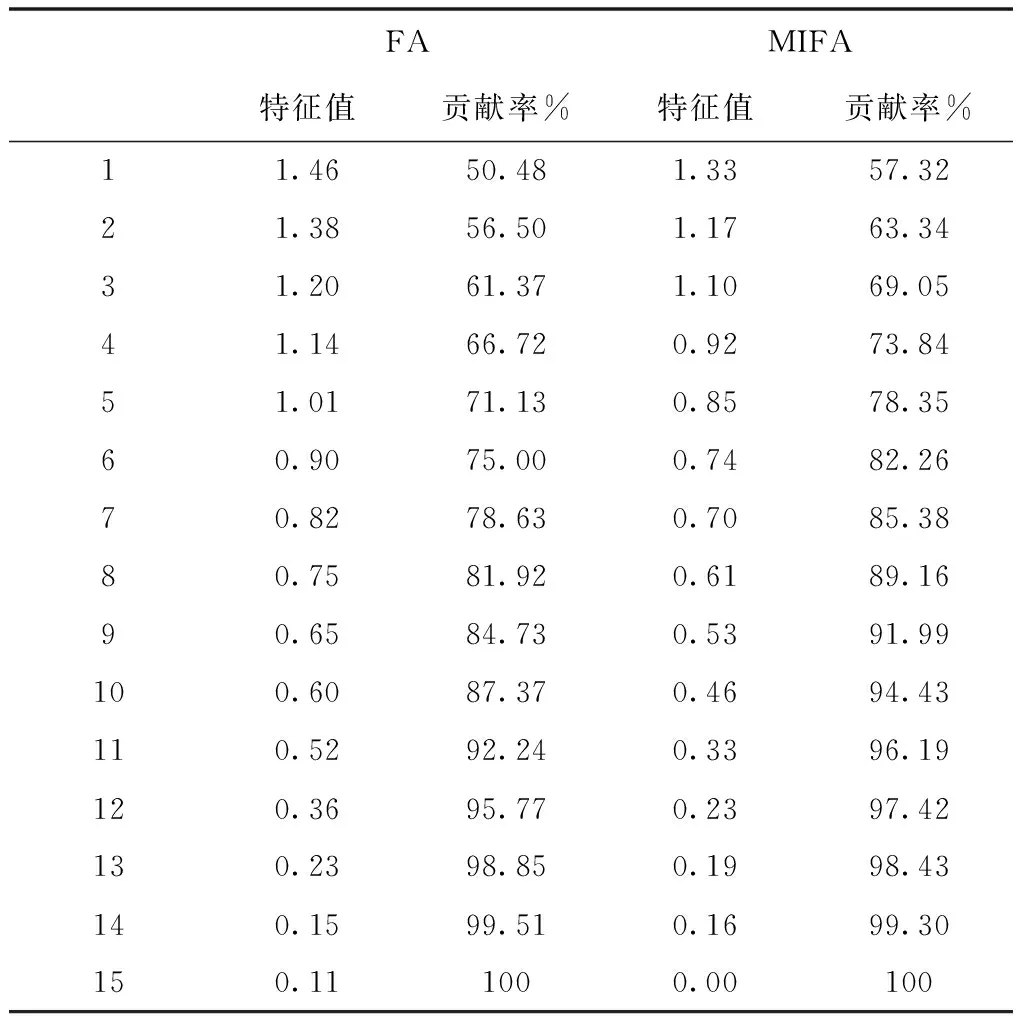
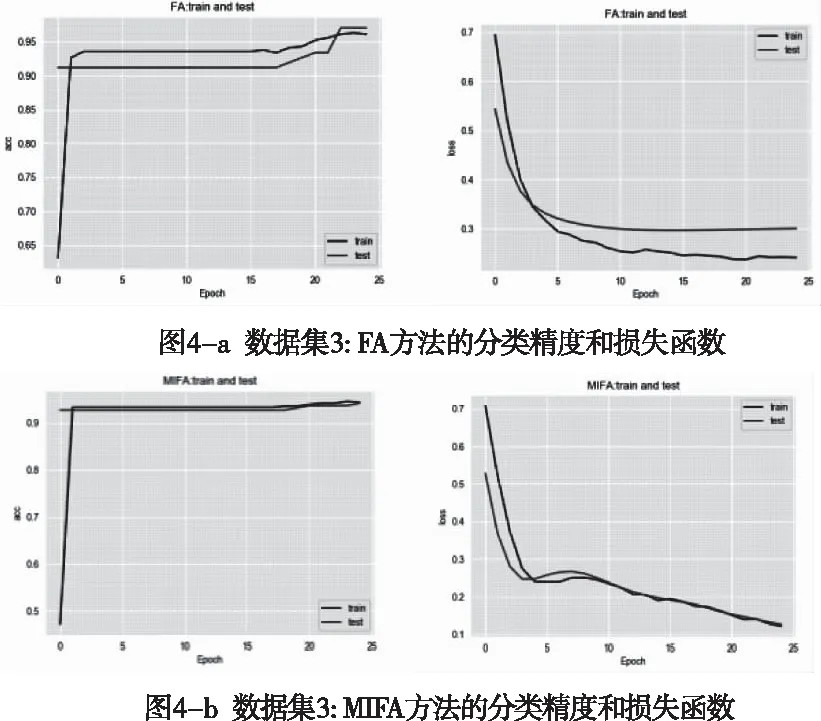
假设有n个样本量,p个指标(特征),X=(X1,X2,…Xp)T为随机特征,其中协方差矩阵cov(x)=Z;可得出本假设的公共因子为F=(F1,F2,…Fm,)T(m (1) 其中θp为特殊因子,则A=(aij) 称为因子载荷矩阵,aij为因子载荷(loading),实质表示Xi依赖因子Fj的程度[13]。该因子分析模型满足正交,矩阵形式具体表示为 (2) (3) 因子模型简单表示 X=AF+θ (4) 式中cov(F,θ)=0, 公共因子和特殊因子满足不相关的条件。针对高维数据集,使用因子分析进行降维,特征之间需具有线性相关性,得出的各个公共因子应具有可解释性。因子分析算法如下所示。 Input: 原数据集N 特征X={X1,X2…Xn} output: 因子模型X=AF+θ 1)标准化原数据集N; 2)计算高维数据集N中特征X间协方差阵; 3)若KMO∈[0,1] 并KMO≥0.5则输出F=[F1,F2…Fn]; 4)通过因子旋转,使得公共因子F更具有解释性; 5)对因子F计算得分,转换为因子载荷A=(A1,A2,…,Am),并得出特殊因子θ=[θ1,θ2…θn]; 6)输出因子模型:X=AF+θ. 在信息和概率论中,两个随机变量的互信息(Mutual Information, MI)衡量它们之间相互依赖的程度,解释为两个特征之间共同拥有信息的含量[14]。互信息具有两个显著的优点: 1)能够对随机变量间复杂的关系进行处理,包括处理非线性关系,保证特征与类别间在未知关系情况下依然有效; 2)不局限于实值随机变量,并在特征空间的变换情况下互信息的值不会改变,保证了在任意阶段都可以准确度量特征间的关系。 一般地,两个变量X和Y的互信息[15]可以定义为 (5) 其中p(x,y)是X和Y的联合概率分布函数,p(x)和p(y)分别是它们的边缘概率分布函数。若I(X;Y)=0当且仅当X和Y是独立的随机变量,可知 p(x,y)=p(x)p(y) (6) 因此 (7) 综上所述,互信息具有非负性I(X;Y)≥0和对称性I(X;Y)=I(Y;X)。X和Y两者依赖程度越高,I(X;Y)的值就越大,类别与特征间包含的共有信息也就越多,反之,则类别与特征相互独立,不存在任何共同信息。 结合互信息的因子分析算法框架如图1所示。因子分析中使用协方差矩阵只能反映出特征间的相关性,即为线性关系,无法有效的评价特征间的非线性关系,而算法的思想就是引入互信息,利用其评估特征间的共有信息这样的特异性来优化特征选择的处理过程,使得算法不局限于线性关系。 图1 结合互信息的因子分析算法框架图 首先,对原数据的p个指标标准化处理,消除特征在量纲上的影响。根据标准化后的数据矩阵求出协方差矩阵Z。定义ZI(X;Y)为原数据的互信息阵,使用拉格朗日因子法得到协方差阵对应的特征值的特征向量。原数据特征值为λ1,λ2,…λp,单位特征向量U为 (8) (9) 因此,在实际因子分析过程中采用互信息来替代协方差阵,本文提出一种结合互信息的因子分析(MIFA)特征选择算法用于患癌风险因素的分类。得到因子模型为 (10) ψ是Z的特征值构成的对角阵。由非负性I(X;Y)≥0和互信息对称性I(X;Y)=I(Y;X)可知,不论是互信息(非对角线元素表示两个特征间的互信息)或自信息(信息熵,对角线元素表示的变量)均为实数,ZI(X;Y)为非负实数对称矩阵。 (11) 表示因子分析中公因子对特征的Xi总方差所做出的贡献,取值在0~100%之间,数值越大,说明该特征能被公因子解释的信息量越大。最终,选择贡献率为85%以上的前M个公因子作为原数据新特征,统计学上指标达到85%即可认为这些因子包含了全部特征的原始主要信息。 Input: 数据集D output:M个新特征 1)标准化数据集D; 2)计算求得协方差矩阵Z; 3)Z转换为ZI(X;Y)互信息阵,并求得特征值λ={λ1,λ2,…λp}; 本章节首先给出实验数据集的信息、实验相关设定和算法性能评价指标,然后分为3组实验对结果分析,并与文献[7]中所采用ReliefF进行特征选择的多个分类算法进行比较。 仿真数据来自于开源的机器学习数据库UCI,选取了高维度的宫颈癌(Cervical)、乳腺癌(Breast)和肝癌(HCC)数据集,均属于可预防的癌症类型,早期发现患癌风险并进行治疗即可完全治愈。因此数据集作为对患癌风险因素的分类具有很好的借鉴作用,且高维特征符合实验要求,数据集信息如表1所示。仿真使用Python语言在Ubuntu系统(CPU Intel Corei5/8GRAM)中运行。 表1 实验数据集信息 实验组中神经网络分类算法评估学习模型选择常用的两个参数:分类正确率accuracy和损失函数loss。其中损失函数loss的表达式为 Loss=-ln(Pz)=-ln(Pcorrect) (12) Pz是将样本分配到类别Z的概率,即正确分类概率Pcorrect。当对于一个迭代(Epoch)中含有无穷多个样本时: Loss=E(-ln(accuracy)) (13) 即 accuracy=e-Loss (14) 在神经网络中通常可知accuracy数值越高,loss越小,模型性能越好。 实验组1:本实验组在用于患癌风险因素分类时,对三个数据集进行公因子提取。比较了通过FA与MIFA从原数据集提取15个(累积贡献率>50%)的因子特征值和累积方差贡献率,如表2、表3和表4所示。 表2 数据集1:FA和MIFA求得的因子信息 通过表2可以得出,以因子累积贡献率等于85%作为指标,以FA作为特征选择算法需要9个新特征才能解释原数据全部信息,而采用MIFA则只需要5个新特征即可包含原来的所以特征信息。从可解释性方面可以看出,在相同维度下,FA的因子贡献率低于MIFA,例如同样在公因子5的情况下,FA的贡献率为70.39%,而MIFA的贡献率为85.47%。 同样地,由表3可以发现采用FA进行特征公因子选择需要8个公因子数量,累积贡献率大于85%,而使用MIFA方法仅需要5个公因子即可解释原始数据集实际意义。同理,通过表4可以清楚看出对于因子的可解释性,相同维度下,例如在公因子8的情况下,因子分析的贡献率为81.92%,而结合互信息的因子分析的贡献率则为89.16%。 表3 数据集2:FA和MIFA求得的因子信息 表4 数据集3:FA和MIFA求得的因子信息 综上所述,即可证明MIFA降低的数据维度量和公因子解释性高于传统的因子分析方法,有利于模型的分类正确性。实验组2将采用常用的分类算法进一步验证。 实验组2:以实验组1降维之后的数据集作为分类算法的输入进行仿真。本组实验以常用的神经网络(ANN)作为分类器来验证因子分析(FA)与结合互信息的因子分析(MIFA)得到的公因子对宫颈癌活检进行预测。神经网络分类器包含有输入层(Input layer)、隐藏层(Hidden layer)、输出层(Output layer),激励函数设置为sigmoid;采用十折交叉验证法。 通过图2-a看到FA训练集和检测集产生很大的过拟合现象,可以看到检测集的精确度只有89%左右。而通过图2-b可以明显看出MIFA的模型过拟合现象被解决,检测集的精确度逐渐达到95%,有较大的提升。通过图3-a和3-b明显看出FA的损失函数并未趋于收敛,而MIFA的损失函数在完成10次迭代后快速收敛,最终的损失函数值只有0.1138,说明算法达到实验效果。 同样可以看出对于数据集2和3所得出的分类精度和损失函数。如图3和4所示,数据集2、3在迭代15-20次时精确度出现很大波动,产生较大过拟合,检测集精度过低,相对的损失函数也未趋于收敛,特别地,对于数据集3检测集的损失函数过大,算法性能较差。而MIFA则在处理高维度癌症数据集产生较好的分类效果,神经网络循环迭代10次后损失函数趋近于收敛,分类精度分别为95.96%和96.13%,损失函数为0.1341和0.1216。 实验组3 为了客观展示实验结果,通过10次十字交叉法验证,如图5所示。清楚地看出针对高维数据集采用MIFA计算出的公因子作为分类器输入项所得到的分类正确率高于FA方法。 图5 数据集交叉验证分类精度对比 综上所述,证明了MIFA特征选择较传统FA方法性能更加优越。而在文献[7]中康桂霞采用ReliefF特征选择对早期癌症风险因素进行分类,通过决策树DT和支持向量机SVM以及BP神经网络构建模型得出的分类精确度如表5所示。可以清楚地发现本文提出的MIFA算法均值高于文献[7]所示的ReliefF特征选择算法,证明了算法的有效性。 表5 分类精确度对比 本文提出了结合互信息的因子分析对患癌因素的分类算法,并进行仿真。算法在进行特征选择的时利用互信息处理非线性关系的优点,使用协方差阵转换为互信息阵从而确定公因子特征达到降维目的。采用神经网络作为分类器,三组数据分类精度分别达到96.51%、95.96%和96.13%。仿真结果表明在处理具有复杂性和高维度的癌症数据效果显著。今后的研究工作将主要集中在如何结合条件互信息与因子分析对高维的医学数据集进行处理。

3 结合互信息的因子分析对患癌因素的分类算法
3.1 互信息相关知识
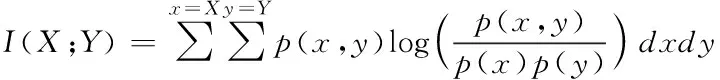

3.2 算法描述与分析



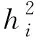
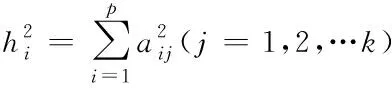
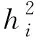

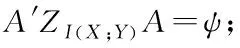



4 实验分析
4.1 实验信息

4.2 实验结果与分析






5 结束语
