番茄?拟南芥 PREs 及水稻 ILIs 基因生物信息学分析
2021-09-27郭鹏宇杨志杰
郭鹏宇 杨志杰
摘要 为进一步了解常见模式植物番茄、拟南芥 PREs 以及水稻 ILIs 共 18 个基因的生物信息学数据,为基因功能的研究奠定理论基础,结合 NCBI 等数据库,运用 MG2C 等工具,对上述基因结构、蛋白理化性质等生物信息学数据作出预测与分析。除 OsILI6 外,其余基因均只含1个内含子,且 CDS 序列均较短。蛋白理化性质分析表明这些蛋白质稳定性较低,二级结构分析表明 α 螺旋与无规则卷曲构成蛋白质的主体部分。三维结构模拟表明这些蛋白质以二聚化的形式发挥功能,在结构上相对保守,分析数据可为后续基因功能研究提供支持。
关键词 番茄;拟南芥;水稻;PREs;生物信息学分析
中图分类号 Q 812 文献标识码 A
文章编号 0517-6611(2021)18-0099-06
doi:10.3969/j.issn.0517-6611.2021.18.025
开放科学(资源服务)标识码(OSID):
Bioinformatics Analysis of Tomato,Arabidopsis PREs and Rice ILIs Genes
GUO Peng-yu,YANG Zhi-jie (Bioengineering College,Chongqing University,Chongqing 400044)
Abstract In order to further understand the bioinformatics data of 18 genes in common model plants tomato,Arabidopsis PREs and rice ILIs,and lay a theoretical foundation for the study of gene function,combined with NCBI and other databases,using MG2C and other tools,bioinformatics data such as the above gene structure and protein physical and chemical properties were predicted and analyzed .Except for OsILI6,all other genes contain only one intron,and the CDS sequence was relatively short.The analysis of physicochemical properties showed that these proteins had low stability and the analysis of the secondary structure showed that α helix and random coils constitute the main part of the protein.Three-dimensional structural simulations indicated that these proteins function in the form of dimerization.These proteins were relatively conservative in structure,and the analytical data could provide support for subsequent gene function studies.
Key words Tomato;Arabidopsis thaliana;Rice;PREs;Bioinformatics analysis
作者简介 郭鹏宇(1995—),男,山西吕梁人,硕士研究生,研究方向:生物化学与分子生物学。
收稿日期 2021-02-04
番茄是管状花目的一年生或多年生草本植物。据考证,番茄最早起源于南美的秘鲁、厄瓜多尔,于 16 世纪末或 17世纪初的明万历年间传入我国[1]。1753年,Linnaeus 根据雄蕊数目和雌雄蕊的着生习性将番茄划归于茄科茄属中。目前,番茄已成为研究果实发育的重要模式植物[2]。作为一种兼具营养价值和商业价值的园艺作物,近年来世界范围内的番茄产量不断提升[3]。 拟南芥是十字花科的一年生细弱草本植物,分布广泛,植株虽小却结子较多。早在 2000 年,其基因组测序就已全部完成,其遗传背景较为简单,且作为一种自花授粉植物,基因高度纯合,是公认的基因组学研究的模式植物[4]。
水稻属禾本科稻属作物,是世界公认的三大粮食作物之一。2002 年,我国宣布完成水稻基因组精细图的绘制。凭借其较为简单的遗传背景及遗传转化的潜力,水稻成为谷类作物育种及基因功能研究的模式植物之一[5]。研究发现,拟南芥中存在一类能够编码对赤霉素合成抑制剂多效唑产生拮抗作用蛋白质的基因,称为多效唑抗性基因(PACLOBUTRAZOL RESISTANCE GENE),简称 PREs,该类蛋白质为非典型的bHLH 转录因子。拟南芥 PRE 基因家族共有 6 个成员,即 AtPRE1-AtPRE6,在调控拟南芥赤霉素[6]、油菜素内酯[7]、生长素响应[8]以及调节细胞长度[9]、介导光信号[10]响应等方面发挥重要作用。番茄中共有 5 个PRE基因,即SlPRE1~SlPRE5[11]。Slstyle2.1,即 SlPRE1 参与调控花柱长度,并且可以促进栽培品种自花授粉的进化[12]。SlPRE2 的超表达会影响番茄植株形态的变化[11],而SlPRE2-RNAi 会影响胎座以及果皮细胞的大小,改变植株对外源赤霉素的响应[13]。水稻共有7个PRE基因,即OsILI1~OsILI7。AtPRE1 同源基因 ILI1(INCREASED LAMINA INCLINATION1) 的过表达会导致水稻叶片倾斜角度增加,通过与水稻 IBH1 的相互作用参与油菜素内酯信号转导来调控细胞的伸长[9]。ILI4 的过量表达增加了水稻籽粒的大小以及对油菜素内酯合成抑制剂的抗性[14]。该研究旨在利用生物信息学的方法对拟南芥、番茄 PRE家族、水稻 ILI 家族基因結构、染色体定位、蛋白质理化性质分析、进化关系等方面进行分析与预测,结合已有的功能研究报,为尚未进行功能研究的基因提供理论基础。
1 材料与方法
1.1 鉴定与染色体定位
从茄科基因组数据库(https:∥solgenomics.net/)、拟南芥基因组数据库(https:∥www.arabidopsis.org/)及水稻基因组数据库(http:∥rice.plantbiology.msu.edu/)下载得到全部蛋白质序列、cDNA 序列、CDS(coding-sequence)序列以及各基因在基因组上的序列。运行在线软件MG2C(http:∥mg2c.iask.in/mg2c_v2.1/)绘制染色体定位图谱。
1.2 基因结构分析
从各物种基因组数据库下载各基因的 CDS 序列以及该基因在基因组上 DNA 序列的 FASTA 格式文件。运行在线工具 Genes Structure Display Server 2.0(http:∥gsds.gao-lab.org/index.php)绘制各基因编码区及非编码区序列分布图。
1.3 蛋白质保守基序分析
根据各物种蛋白质序列,运行在线软件 MEME(https:∥meme-suite.org/meme/),分析各物种蛋白质序列中的保守基序,motif 个数设置为 10,其他参数为默认值。
1.4 蛋白保守结构域序列比对和系统进化树构建
利用 DNAMAN 6.0 软件完成所有蛋白质序列的比对分析,利用软件 MEGA 6.0 完成系统进化树的构建。
1.5 蛋白理化性质与亚细胞定位预测构建
运行在线软件 ExPasy (https:∥www.expasy.org/)分析各编码蛋白质的理化性质,运行在线数据库 GenScript(https:∥www.genscript.com/psort.html?src=leftbar),对各蛋白质进行亚细胞定位预测。
1.6 蛋白二级结构分析
运行在线软件 SOPMA (https:∥npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_sopma.html)分析各蛋白质的二级结构组成。
1.7 蛋白三维结构模拟
根据同源建模法,运行 Swiss-Model 软件(https:∥swissmodel.expasy.org/interactive)对各蛋白质的三维结构进行模拟。
1.8 启动子区域顺式作用元件分析
从各物种基因组数据库下载得到各基因上游 3 000 bp 序列的 FASTA 文件,利用在线网站 Plant CARE (http:∥bioinformatics.psb.ugent.be/webtools/plantcare/html/)进行顺式作用元件分析,利用 TBtools 1.072 绘制顺式作用元件分布图。
1.9 蛋白互作网络预测分析
根据 STRING 11.0 (https:∥string-db.org/cgi/input?sessionId=bABx2NvfkwQD&input_page_active_form=single_sequence)在线数据库进行各蛋白质互作网络的预测。
2 结果与分析
2.1 鉴定与染色体定位
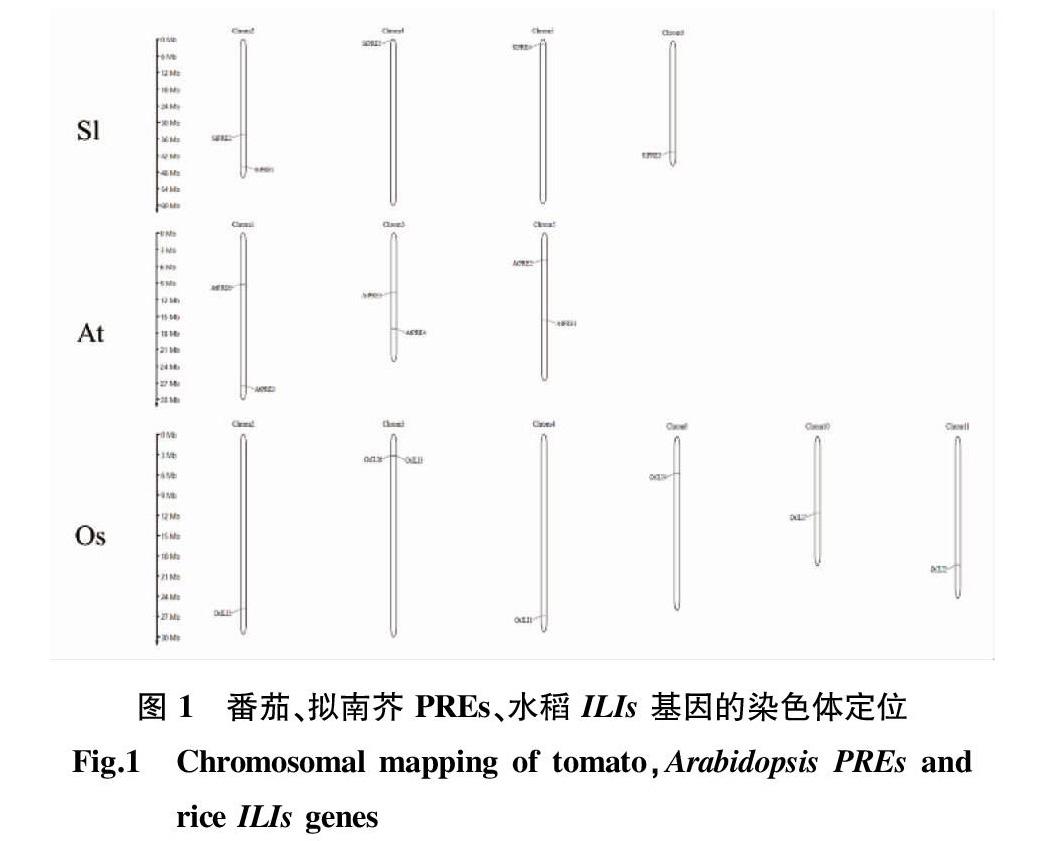
根据已有的文献报道,结合 SGN (茄科基因组数据库Solanaceae Genomics Network)、TAIR (拟南芥数据库The Arabidopsis Information Resource)、RGAP (水稻基因组数据库Rice Genome Annotation Project) 以及 NCBI (National Center for Biotechnology Information Search database)数据库检索分析,分别找到番茄 5 个 PREs、拟南芥 6个 PREs、水稻 7 个 ILIs 基因共 18 个相关基因。在各物种基因组数据库中查找得到各基因在基因组染色体上的位置(图1),结果表明,番茄的 5 个PRE 基因分布于第 2、4、5、6 染色体上,拟南芥的 PRE 基因分布于第 1、3、5 染色体上,而水稻的 ILIs 基因分布于第 2、3、4、6、10、11 染色体上。
2.2 基因结构
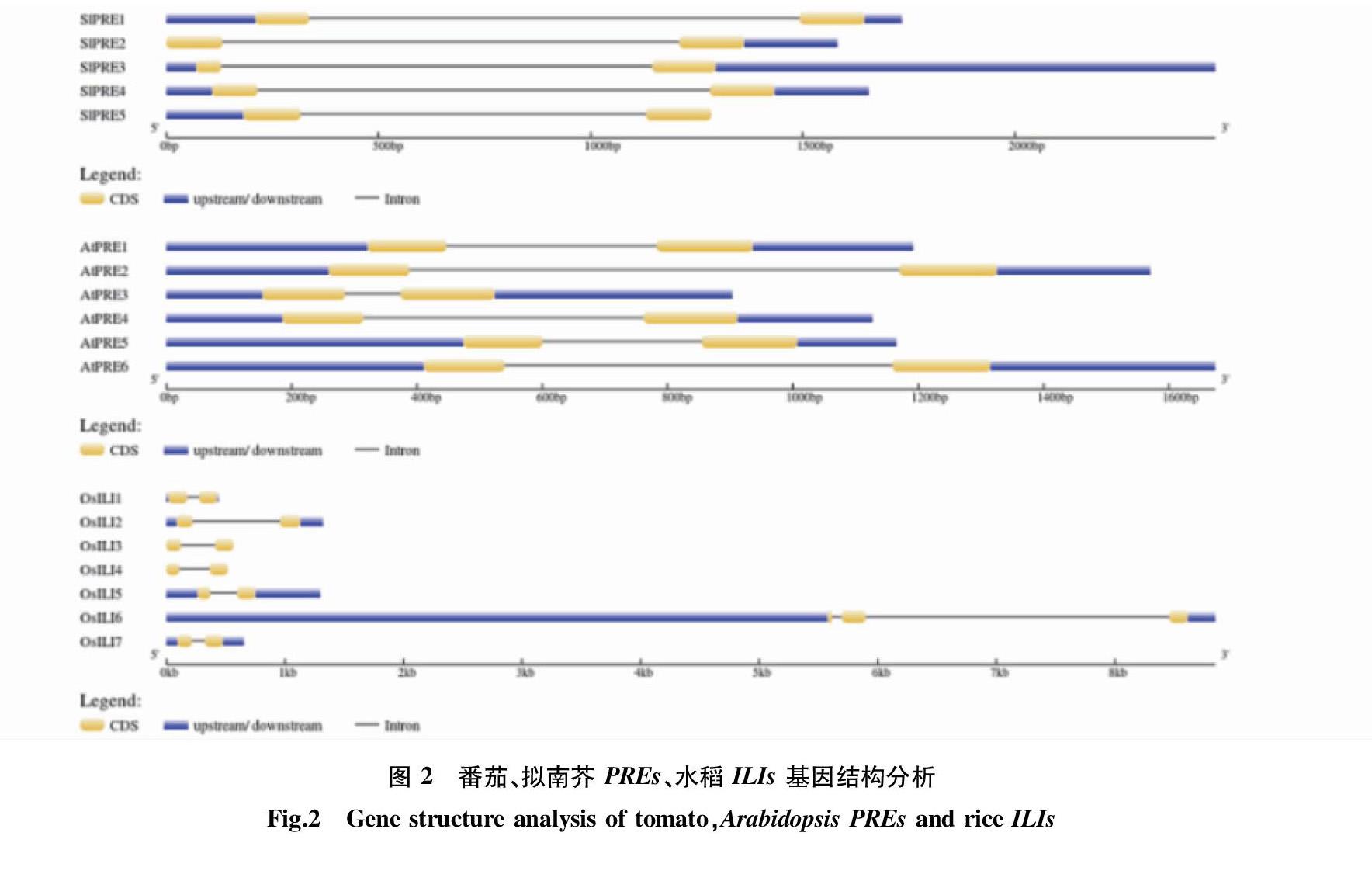
对番茄、拟南芥 PREs、水稻 ILIs 基因进行基因组序列与編码序列分析(图2),结果表明,除 OsILI6 的编码序列含 3 个外显子,其余基因外显子数量均为 2;此外,OsILI3 与 OsILI4 只含有外显子与内含子,而其他基因均包含 5′与 3′非编码序列,且个别基因非编码序列较长,如 SlPRE3 的 3′端以及 OsILI6 的 5′端。这表明该家族基因结构相对简单。
2.3 蛋白质保守基序
对番茄、拟南芥 PREs 与水稻 ILIs 家族的蛋白质序列进行保守基序的预测分析(图3),结果表明,这些蛋白质包含 3~4 个保守基序;所有序列均包含 Motif 1,而 OsILI6 无 Motif 2,SlPRE3、AtPRE3、AtPRE4 无 Motif 3,而 SlPRE4、OsILI4、OsILI5、OsILI7 则无 Motif 4。保守基序中字母越高,表明对应氨基酸残基在该位点出现的频率越大,相对保守程度也越高。综合来看,4 个 Motif 的保守程度相对较高。这些保守基序的分析有利于深入分析蛋白质结构。
49卷18期 郭鹏宇等 番茄·拟南芥 PREs 及水稻 ILIs 基因生物信息学分析
2.4 蛋白保守结构域序列比对和系统进化树分析
通过对番茄、拟南芥 PREs、水稻 ILIs基因家族蛋白质进行氨基酸组成的分析(图 4A),结果表明,这些蛋白质存在有相对的氨基酸序列组成的保守结构域,即 HLH 结构域,而无典型的 Bas-ic 结构域,而在进化过程中,这些基因的功能高度保守与上述保守结构域的构成密切相关。
为了进一步明确这些基因之间的进化关系,利用氨基酸序列对上述 18 个基因构建系统进化树(图4B) 。目前番茄 SlPRE1 与 SlPRE2 已有功能研究,且与 AtPRE1 及 AtPRE5 亲缘关系较近。总体来看,番茄与拟南芥亲缘关系较近,而二者与水稻的亲缘关系相对较远。
2.5 蛋白理化性质与亚细胞定位预测分析
利用 ExPasy 在线软件对上述 18 个基因的编码蛋白质进行理化性质分析(表 1)。结果表明,18 个蛋白质的氨基酸残基数为86~130,其中 SlPRE4 最长,OsILI6 最短;蛋白质分子量为9.746~14.215;理论等电点为4.43~9.61,其中 SlPRE1、AtPRE2 最大,而 OsILI6 最小;脂溶指数为81.08~106.26,其中 OsILI6 最小,OsILI7 最大;不稳定系数为60.29~93.81;蛋白质的疏水性在 -0.766~-0.218,表明这些蛋白质均属亲水性蛋白;亚细胞定位预测结果表明这些蛋白均定位于细胞核内。
2.6 蛋白二级结构分析
运行 SOPMA 在线软件对 18 个基因所编码的蛋白质进行二级结构解析(表 2)。结果表明,在上述 18 个蛋白质的二级结构中,α 螺旋和无规则卷曲占主体地位;部分蛋白质无 β转角及延伸链(如SlPRE4、AtPRE1、AtPRE2、AtPRE3、AtPRE4、AtPRE6、OsILI1、OsILI7);除α螺旋与无规则卷曲外,个别蛋白质只含 β 转角与延伸链之中的一种结构,如SlPRE2、SlPRE5、AtPRE5、OsILI5 无延伸链,而OsILI2、OsILI3、OsILI4 则无 β 转角。总体来看,α 螺旋和无规则卷曲是这些蛋白质的主要组成成分。
2.7 蛋白三维结构模拟 为了进一步了解上述 18 个蛋白质的三维空间结构,根据同源建模法运行 Swiss-Model 在线软件进行同源建模(图 5),结果表明,这些蛋白质均以二聚化的形式完成建模。
2.8 基因启动子区域顺式作用元件分析
通过 SGN数据库、TAIR 数据库、RGAP 数据库获得各基因 ATG 上游 3 000 bp 的序列,运行在线数据库 Plant CARE 网站对序列进行顺式作用元件分析。结果显示(图6),这些基因上游存在大量的光响应元件 (G-box、GT1-motif、ACE),除此之外,激素响应元件(GARE-motif、ABRE、CGTCA-motif、AuxRR-core、P-box)、逆境胁迫响应元件(WUN-motif、TC-rich repeats、LTR、MBS、DRE)、分生组织表达元件(CAT-box)、胚乳表达元件(GCN4_motif)及种子特异性调控元件(RY-element)也存在与上游启动子区域。SlPRE3、AtPRE4~AtPRE6、OsILI2~OsILI7 启动子区域均含有干旱响应元件;除 SlPRE3、SlPRE4、AtPRE1、AtPRE2、OsILI1、OsILI2、OsILI7 外,其余基因启动子区域均含有低温响应元件,部分基因启动子区域含有类黄酮物质合成基因调控元件(SlPRE3、AtPRE4),推测这2个基因有可能参与类黄酮物质的合成。
2.9 基因蛋白互作网络预测分析
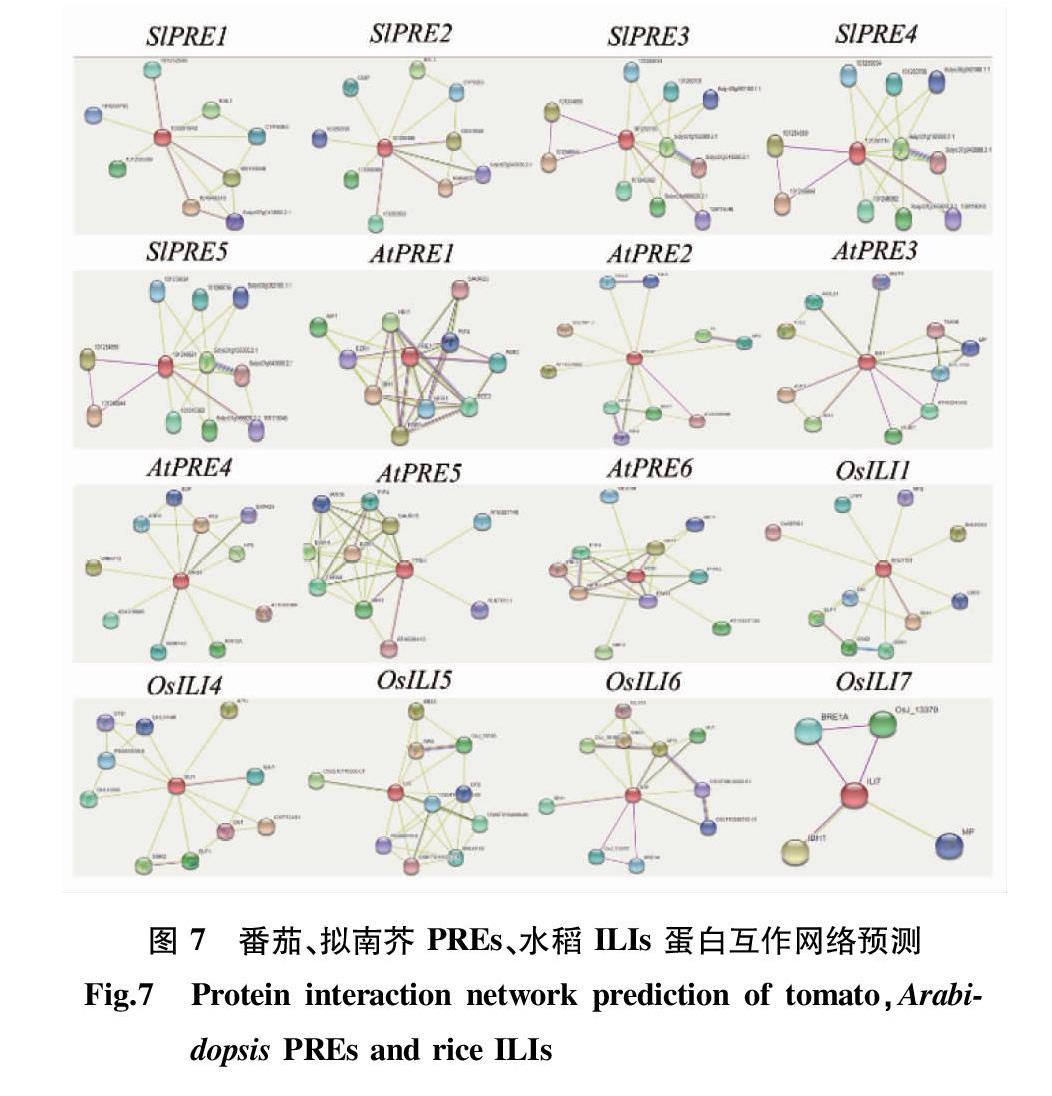
为进一步了解上述基因可能的互作蛋白,该部分参考 STRING 在线数据库,对这些基因可能的互作网络进行了预测。结果表明,除 OsILI2 与 OsILI3 外,其余蛋白质均有预测的蛋白质网络互作图。这表明大部分基因可以通过蛋白质的相互作用参与生长过程的调控(图7)。
3 讨论与结论
截至目前,已有功能研究报道的 PREs 以及水稻 ILIs 均属于非典型的 bHLH 转录因子[8,14-15]。这些基因通过赤霉素、油菜素内酯[16]以及生长素参与植物营养生长与生殖生长[17]的调控。目前,拟南芥 AtPRE1~AtPRE6、SlPRE1~SlPRE2 以及 OsILI1、OsILI4 均有功能研究报道,而其余基因暂无功能研究。该研究通过生物信息学的方法,结合已有的功能研究报道,对番茄、拟南芥 PREs 以及水稻 ILIs 基因进行生物信息学分析,为这些基因功能的进一步研究奠定理论基础。
該研究对3个物种共 18 个蛋白质的理化性质进行了初步分析,发现这些蛋白质均属于疏水性蛋白质,且稳定性较差;此外,α 螺旋和无规则卷曲是这些蛋白质主要的二级结构组分,仅有较少的 β 转角与延伸链存在其中,从三维结构模拟可以更直观地发现这一点。蛋白质的三维空间模拟结果表明,这些蛋白质可以通过二聚化的形式参与调控植株生长。与其他蛋白质的互作网络预测表明,这些蛋白通过与其他蛋白质的相互作用发挥调控作用。蛋白质保守基序分析与蛋白质多序列比对及进化关系分析表明这些蛋白质在调控植物生长发育方面存在功能相似性。
通过对3个物种共 18 个基因进行生物信息学分析,从染色体定位、基因结构、蛋白质理化性质以及二级、三级结构分析等方面做出分析预测,为进一步的功能研究提供理论基础与数据支撑。
参考文献
[1]
赵凌侠,李景富.番茄起源、传播及分类的回顾[J].作物品种资源,1999(3):29-31.
[2] QUINET M,ANGOSTO T,YUSTE-LISBONA F J,et al.Tomato fruit development and metabolism[J].Frontiers in plant science,2019,10:1-23.
[3] CUI J,JIANG N,ZHOU X X,et al.Tomato MYB49 enhances resistance to Phytophthora infestans and tolerance to water deficit and salt stress[J].Planta,2018,248(6):1487-1503.
[4] MEINKE D W,CHERRY J M,DEAN C,et al.Arabidopsis thaliana:A model plant for genome analysis[J].Science,1998,282(5389):662-682.
[5] LI X X,DUAN X P,JIANG H X,et al.Genome-wide analysis of basic/helix-loop-helix transcription factor family in rice and Arabidopsis[J].Plant physiology,2006,141(4):1167-1184.
[6] LEE S,LEE S,YANG K Y,et al.Overexpression of PRE1 and its homologous genes activates gibberellin-dependent responses in Arabidopsis thaliana[J].Plant & cell physiology,2006,47(5):591-600.
[7] BAI M Y,SHANG J X,OH E,et al.Brassinosteroid,gibberellin and phytochrome impinge on a common transcription module in Arabidopsis[J].Nature cell biology,2012,14(8):810-817.
[8] CASTELAIN M,LE HIR R,BELLINI C.The non-DNA-binding bHLH transcription factor PRE3/bHLH135/ATBS1/TMO7 is involved in the regulation of light signaling pathway in Arabidopsis[J].Physiologia plantarum,2012,145(3):450-460.
[9] ZHANG L Y,BAI M Y,WU J,et al.Antagonistic HLH/bHLH transcription factors mediate brassinosteroid regulation of cell elongation and plant development in rice and Arabidopsis[J].Plant cell,2009,21(12):3767-3780.
[10] HYUN Y,LEE I.KIDARI,encoding a non-DNA Binding bHLH protein,represses light signal transduction in Arabidopsis thaliana[J].Plant molecular biology,2006,61(1/2):283-296.
[11] ZHU Z G,CHEN G P,GUO X H,et al.Overexpression of SlPRE2,an atypical bHLH transcription factor,affects plant morphology and fruit pigment accumulation in tomato[J].Scientific reports,2017,7:1-11.
[12] CHEN K Y,CONG B,WING R,et al.Changes in regulation of a transcription factor lead to autogamy in cultivated tomatoes[J].Science,2007,318(5850):643-645.
[13] ZHU Z G,LIANG H L,CHEN G P,et al.The bHLH transcription factor SlPRE2 regulates tomato fruit development and modulates plant response to gibberellin[J].Plant cell reports,2019,38(9):1053-1064.
[14] TANAKA A,NAKAGAWA H,TOMITA C,et al.BRASSINOSTEROID UPREGULATED1,encoding a helix-loop-helix protein,is a novel gene involved in brassinosteroid signaling and controls bending of the lamina joint in rice[J].Plant physiology,2009,151(2):669-680.
[15] IKEDA M,FUJIWARA S,MITSUDA N,et al.A triantagonistic basic helix-loop-helix system regulates cell elongation in Arabidopsis[J].Plant cell,2012,24(11):4483-4497.
[16] WANG H,ZHU Y Y,FUJIOKA S,et al.Regulation of Arabidopsis brassinosteroid signaling by atypical basic helix-loop-helix proteins[J].The plant cell 2009,21(12):3781-3791.
[17] MARA C D,HUANG T B,IRISH V F.The Arabidopsis floral homeotic proteins APETALA3 and PISTILLATA negatively regulate the BANQUO genes implicated in light signaling[J].Plant cell,2010,22(3):690-702.
