菌株WYT降解还原蓝4的关键基因
2021-08-09李海红王洋涛西安工程大学环境与化学工程学院陕西西安710600
马 倩,李海红,王洋涛 (西安工程大学环境与化学工程学院,陕西 西安 710600)
蒽醌染料是纺织工业中普遍存在的合成着色剂[1].由于蒽醌染料价格低廉,容易获取且染色效果好,是继偶氮染料之后应用量最大的一类染料.蒽醌染料结构复杂并稳定,对微生物和人体细胞的毒性甚至大于偶氮染料[2].若将未彻底处理的印染废水排放到环境中,会对水、土壤造成严重的污染.目前,处理印染废水方法主要有物理、化学和生物处理法,包含吸附、絮凝、光催化、离子交换及生化氧化等方法[3-5].生物法处理蒽醌染料具有效率高、成本低且环保的优点,近年来成为研究热点.还原蓝4(VB4)作为一种蒽醌类染料,不易被降解,具有良好的色牢度[6-7].微生物主要通过生物降解和生物吸附将 VB4脱色.生物降解过程中,原始染料结构被微生物的酶分解成小分子化合物,芳香族聚合物被转化为CO2、H2O和一些无机盐[8].在生物吸附过程中,作为污染物的染料被吸收到微生物细胞质中或者表面,而不是结构被破坏[9].由于细胞表面富含多糖成分,被含有官能团的蛋白质和脂质包围,染料可以与菌体表面的活性基团相互作用从而被吸附固定[10].池振明等[11]从印染厂排出水中分离出的邻单胞菌Plesiomonas sp.X13脱色率可达75%以上.Velayutham等[12]筛选出一株Staphylococcus sp.K2204菌株,在12h内可完全降解活性蓝 KN-R.Yang等[13]利用混合菌群降解活性蓝19,可在 48h内脱色 89.2%的染料.虽然目前已筛选出一些高效降解脱色蒽醌染料的菌株,但对其降解机理研究较少,尤其缺乏对降解的关键基因和酶的研究.
本文对前期筛选出的一株高效降解 VB4的菌株 WYT进行研究,通过全基因组测序,确定该菌株中降解脱色的关键基因 DyP,通过基因敲除回补验证DyP基因为降解VB4的关键基因.以期为印染废水的微生物处理技术提供参考.
1 材料与方法
1.1 材料
1.1.1 菌株 Pseudomonas aeruginosa(铜绿假单胞菌)WYT,是本课题组前期从印染厂活性污泥中筛选出的一株可高效降解VB4的革兰氏阴性菌.
1.1.2 培养基及培养条件 培养基:无机盐培养基(ISM):KH2PO41.0g, Na2HPO4⋅12H2O 2.0g, MnSO4⋅7H2O 0.02g,FeSO4⋅7H2O 0.01g,MgSO4⋅7H2O 0.2g,CaCl20.02g,NH4Cl 0.5g,2.0mL/L微量盐溶液,用1mol/L NaOH与1mol/L HCl调节初始pH 7.0.121℃,20min高温高压灭菌备用.微量盐溶液(mg/L):CoCl2⋅6H2O 4.0,MnSO4⋅H2On2.8,H3BO34.0, CuSO4⋅5H2O 0.02,ZnSO4⋅7H2O 28.0,MoO34.0.0.24μm 滤膜过滤备用.用于培养菌株时,按需要加入VB4染料及其他底物.LB培养基(g/L)、LB固体培养基.培养条件:Pseudomonas aeruginosa WYT菌株在ISM或LB中活化培养,大肠杆菌在 37℃恒温培养,抗生素添加的浓度为:庆大霉素(Gm)50µg/mL,四环霉素(Tc)10µg/mL.异丙醇-β-D 硫代半乳糖苷(IPTG)添加至所需浓度,2,6-二氨基庚二酸(2,6-DAP)终浓度为0.3mmol/L.
1.1.3 试剂 基因组提取、质粒提取、胶回收、核酸纯化采用 Omega公司的试剂盒(E.Z.N.A.TM).其他常规的化合物购于阿拉丁、国药集团或美国Sigma-Aldrich科技有限公司,且均为分析纯以上.
1.2 基因组文库构建与测序分析方法
首先对菌株WYT处理后的样品进行检测,将合格的样品用于文库构建,用g-tubes将DNA基因组随机打断,产生 DNA 片段,利用 PacbioSMRTBELL建库试剂盒构建,将大片段的连接产物用 BluPippin回收,最后对合格的文库进行测序.
利用 SMRTAnalysisv2.3软件对原始测序数据进行处理,计算得到有效数据.用三代数据拼接算法,对测序数据进行组装和纠错,将处理后的优质数据优化,得到基因组拼接序列.分析基因组进行组分,得到基因成分,参考 GO、KEGG 和 Swiss-Prot,以及NR和COG数据库,将预测获得的ORF区域进行功能注释.
基因组提取、质粒提取、割胶回收及核酸纯化等操作均按照试剂盒说明书进行.
1.3 关键目的基因分析比对
为探索影响 VB4降解脱色的关键基因,将测得的全基因组序列与主要基因库中的所有数据进行比对,并未发现与VB4降解相关的基因.分析其他染料降解的过氧化物酶,根据 PeroxiBase数据库,将DyP-type过氧化物酶分为4个类型,分别为A型、B型、C型以及D型.下载4个分组中的基因序列,用Bioedit软件与原基因序列比对,然后利用MEGA进行系统发育树分析,在WYT基因组的hits找到目的基因.
1.4 基因敲除与回补
1.4.1 抗生素敏感性测试 参考文献[14],将WYT菌液分别接种到含有庆大霉素(GM)、四环霉素(Tc)青霉素(P)、氯霉素(C)、卡那霉素(Kan)等抗生素的培养基中培养,每种抗生素设不同浓度,庆大霉素(Gm)50µg/mL,四环霉素(Tc)10µg/mL.异丙醇-β-D 硫代半乳糖苷(IPTG)添加至所需浓度,2,6-二氨基庚二酸(2,6-DAP)终浓度为0.3mmol/L,观察菌株的生长情况.
1.4.2 线性化质粒 PEX18Tc线性化:利用含有10µLTc的液体LB活化培养PEX18Tc菌株,提取2管质粒备用.选择双酶切位点:BamHI与 HindIII,质粒添加量需根据提取的浓度计算.酶切温度选择 2个内切酶温度比较低的参数,酶切时间为 4h,然后跑胶验证是否酶切完全.将酶切成功的质粒进行纯化,用Nanodrop确定纯化后的线性质粒浓度,浓度不可过低,过低导致后续连接效率变低.酶切反应的体系(50µL) 如下 :5µL Buffer10×K,1µL BamHI,1µL HindIII,40µL PEX18Tc质粒,3µL 无菌水.
1.4.3 构建敲除载体 利用 VectorNTI软件构建敲除质粒[15],为保证敲除目的基因不受两端碱基的影响,选择扩增目的基因的上下游长度为1000bp的同源臂序列,模拟构建敲除载体[16-18].敲除质粒的构建顺序为:质粒序列-上游1000bp上游同源臂-替代目的基因的抗性基因-1000bp下游同源臂-质粒序列,保证以上序列 ORF方向与待敲除的目的基因序列的方向一致.
1.4.4 扩增基因片段 设计 3对引物,设计规则根据无缝克隆设计[19-20].基因片段连接处重复 15bp,加18~22bp待扩增基因序列,总长为33~37bp且一对上下游引物设计的Tm值差距较小(无需考虑15bp的重复序列),将设计好的引物用于扩增目的基因的上游同源臂、下游同源臂及抗性基因序列.用高保真酶(fastPfu)对各个基因片段进行扩增,PCR扩增[21-22]的体系及程序如下:扩增体系:正向引物(10µmol/L)1µL,反向引物(10µmol/L)1µL,5×高保真酶缓冲液 10µL,2.5mmol/L 底物4uL,高保真酶DNA聚合物1uL,蒸馏水适量.扩增程序;95℃ 20s,33个循环为95℃ 20s,55℃ 20s,72℃ 20s,72℃ 5min,16℃ 1min.等待 3 个基因片段扩增完成后,跑胶验证条带大小是否正确,若准确则开始纯化,用核酸纯化试剂盒纯化后测序,若未发现突变点,进行下一步的研究.
1.4.5 无缝克隆 无缝克隆[23-25]参考ⓇHDCloning Kit的操作说明书进行,主要是上游同源臂、GM抗性基因片段、下游同源臂、线性化的PEX18Tc等4个片段的连接.无缝克隆的体系为:5×无缝克隆HD复合酶制剂2µL,上游同源臂50ng,下游同源臂50ng,线性化后质粒 50ng,基因组片段 50ng,加至10µ超纯水.
研究需在冰上进行,完成后在 50℃的金属浴中孵育 10min,迅速取出冰浴 2~3min,将无缝克隆后的产物导入 DH5α感受态细胞,涂抗性平板进行后续研究.在 DH5α平板上挑取单克隆,验证各个片段的连接是否成功.重新设计 2对引物,最外端的引物需包含所有的片段,即200bp质粒上游-1000bp上游同源臂-GM 抗性基因序列-1000bp下游同源臂序列-200bp质粒下游片段.将PCR扩增产物跑胶,验证条带大小后测序.用VectorNTI软件将测序结果与模拟序列比对,若无突变点,可进行下一步.
1.4.6 基因敲除 提取测序后的 DH5α质粒,用热激法[26-27]导入 E.coli WM3064感受态细胞中,将含有E.coliWM3064菌液涂布到抗生素平板中,挑取单克隆后PCR扩增,跑胶验证条带大小.分别在LB(需添加 DAP 及抗生素)培养液中活化含有敲除载体的E.coli WM3064及待敲除菌株(WYT).收集生长至对数期的菌体,用 LB培养液清洗 3遍,以E.coliWM3064与WYT菌株的体积比为2:1均匀混合,涂布到含有DAP的LB平板,放置在30℃下培养,直到结合菌液周围出现晕圈,用 LB清洗下来,涂含有抗生素(GM)的平板.在同源臂之外需再次设计引物,进行PCR扩增,然后测序验证,若菌液中扩增出了完整的1000bp上游同源臂-GM序列-1000bp下游同源臂,则获得敲除菌株.
1.4.7 基因回补 当菌株的 DyP基因被敲除且该菌株不具备降解VB4能力时,应设计回补实验,将敲除的基因回补,验证其降解能力.选取革兰氏阴性菌质粒pRK415作为载体进行回补实验.将pRK415质粒活化培养后,用HindIII与EcoRI作双酶切,导入完整的 DyP基因序列,保证启动子下游的翻译起始框和DyP基因序列同框.扩增后的DyP基因与线性质粒无缝克隆连接,导入DH5α中后涂Tc抗性平板,挑取单克隆,将新设计的引物用于验证,跑胶验证和测序结果正确后,将质粒导入E.coliWM3064,再涂抗性平板,挑取单克隆,条带大小准确则进行后续实验.将带有DyP基因序列的E.coliWM3064和WYT菌株过夜结合,洗脱结合菌体后涂抗性平板筛选,挑取单克隆做验证实验,验证是否产生了双交换,如果发生了改变就证明已经获得敲除株△DyP.
1.5 敲除回补表型验证
准备50mg/L的VB4培养液,将野生株、敲除株及回补株接入进行降解脱色实验,每隔 4h提取0.5mL培养液,以8000r/min的转速离心10min去除菌体,测其在特征波长615nm下的吸光度,计算脱色率率.公式如下:
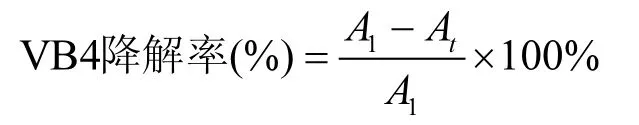
式中:A1为初始的吸光度,At为培养 t h的吸光度值[28].
2 结果与讨论
2.1 全基因测序结果及统计分析
对基因组数据进行测序后对菌种的碱基修饰类型进行分析,预测该基因组的编码区及非编码RNA结构、串联重复序列、微卫星 DNA序列、CRISPR结构组件,最后对预测的编码蛋白质的基因进行数据库比对的功能注释.利用软件得到有效数据后,统计其中的数量、数据产量、序列的平均长度等参数,见表1.

表1 测序数据统计Table 1 Sequencing data of WYT
通过基因预测、重复序列预测、非编码 RNA预测等方法,可获取测序基因组的组成情况,见表2.
由表 2可知,基于测序数据组装得到基因组的大小为6.94Mb,GC含量65.86%.基因组组分分析后发现,基因组含有6357个基因,总长度为6190347bp,平均长度 974bp,占基因组全长的 89.19%.简单重复序列共 59个,总长为 11017bp,占基因组全长的0.16%.tRNA 67个,rRNA 12个,其他ncRNA 126个.对预测出的基因蛋白序列,通过 blastp与 GO、KEGG、Swiss-Prot、NR、COG等通用的数据库进行注释.

表2 基因组组分分析Table 2 Genomic component analysis
2.2 疑似目的基因的查找与确定
在通用的数据库中未发现相关的基因注释,分析菌株WYT降解VB4的代谢途径可以确定,该催化过程为氧化还原反应,结合前人已经报道的脱色酶及其特异性底物[29],推测是在水解酶、单加氧酶或过氧化物酶的共同作用下,使蒽醌环开环从而降解脱色,因此关注主要基因组中所有过氧化物酶的基因注释.过氧化物酶是氧化还原酶的一种,且大多是血红素过氧化物酶,其在生命合成以及污染物降解中发挥重要的作用[30].血红素过氧化物酶可分为 3类:原核起源的过氧化物酶;真菌分泌的过氧化物酶;植物起源的过氧化物酶[31].近年来,随着数据的不断更新,在 PeroxiBase数据库中将血红素过氧化物酶分为 5个家族,分别为:过氧化氢酶超家族、环氧酶超家族、卤素过氧化物酶超家族、双铁色素过氧化物酶超家族以及染料过氧化物酶.DyP-type过氧化物酶最早是在真菌的水稻纹枯病菌(Thanatephorus cucumeris Dec1)中首次发现[32].
分析全基因数据发现,6个CDS区已经注释为过氧化酶基因序列,将这 6个片段与各类型的目的基因比对,发现在CDS区中的1713643~1714542有900bp的疑似染料脱色相关基因,用 MEGA(7.0版)软进行比对,发现其属于染料过氧化物酶基因中的B型,命名为DyP,其系统发育树如图1所示.

图1 DyP的系统进化分析Fig.1 Phylogenetic analysis of DyP gene
2.3 菌株的抗生素抗性
抗生素抗性测试可以得出菌株对不同抗生素的耐药性[33].在LB固体平板上测试8种常用抗生素的抗性,生长状态如图2.

图2 8种抗生素抗性测试Fig.2 Resistance test of 8antibiotic
由图2可知,菌株与多粘菌素、四环素、庆大霉素等没有抗性.在后续实验中需多次使用LB进行活化培养,且菌株在不同培养条件下抗性程度存在差异,因此需验证菌株在 LB液体中的生长情况,结果表明,WYT菌株对GM和Tc抗生素不具抗性,因此在后续实验中使用GM和Tc抗生素进行突变株的筛选实验.
2.4 DyP基因敲除
本研究利用高保真酶,从菌株中扩增待敲除基因(DyP)的上下同源臂,并在 pBBR1McS5中扩增GM 的抗性基因,其中上下游同源臂各 1000bp,GM为 900bp左右,对其进行 1%琼脂糖凝胶电泳分析,结果如图3.

图3 凝胶电泳图及构建的PEX18TcSacB-GM载体Fig.3 Gel-electrophoresis diagram and constructed PEX18TCSACB-GM vector
由图3中a可知,由于扩增条带中存在较多杂质,需要割胶纯化,将3个片段与线性化的PEX18Tc连接,随后扩增,结果如图3中b,待敲除基因(DyP)的上下同源臂和 GM 抗性基因扩增成功.测序验证碱基无突变,构载成功.
载体构建成功后,利用同源重组法对目的基因进行敲除.通过同源重组法对目的基因进行敲除是一种经典的基因编辑技术,在动物、植物、真菌、细菌等各类生物的遗传操作研究中被广泛应用[34].林丹枫等[35]以自杀型质粒 pSVP202为载体,成功敲除类球红细菌的八氢番茄素合成基因.韩武洋等[36]利用自杀载体 pKB18mobsacB,采用无抗性标记的同源重组法,成功敲除了野生型菌株谷氨酸棒杆菌中关键转运蛋白基因和葡萄糖转运系统关键转运蛋白基因.图4(a)为将所构的PEX18TcSacB-GM质粒导入到 DH5α中,转导效率较高,隔夜可明显的观察到单克隆.然后将测序正确的样品提质粒,导入到自杀质粒 WM3064中,凝胶电泳验证结果图 4(b)所示.WYT菌株与构建的敲除载体发生了双交换,凝胶电泳如图 4(f)所示,测序验证正确,敲除成功.第 3个样品为用扩增 DyP基因序列的引物进行扩增,并没有条带,再次验证敲除成功.敲除的一段DyP基因组所在的基因簇如图4(e)所示.

图4 同源重组法进行基因敲除Fig.4 Homologous recombination was used for gene knockout
将敲除成功的 WYT△DyP转接至降解脱色培养液中进行表型验证,具体结果如图5所示.
由图5可知,在24h后,在敲除株的作用下VB4浓度从50mg/L下降到34.24mg/L.对比前期的研究发现,加热致死菌体在24h后的浓度为39.753mg/L,所以推测敲除株WYT△DyP几乎失去了降解VB4染料的功能.在野生株WYT降解脱色VB4染料的实验组中,24h后染料的浓度仅为 1.65mg/L,脱色率率达 96.7%,野生株 WYT可高效降解 VB4,敲除株WYT△DyP失去了降解VB4染料的功能.可见DyP基因是降解脱色VB4染料的关键基因.

图5 WYT△DyP表型验证Fig.5 Phenotype verification of WYT△DyP
2.5 DyP基因回补实验
将敲除的关键基因回补,验证基因敲除是否成功.从WYT菌株基因组中扩增 DyP基因序列,跑凝胶电泳图并测序,结果如图6.
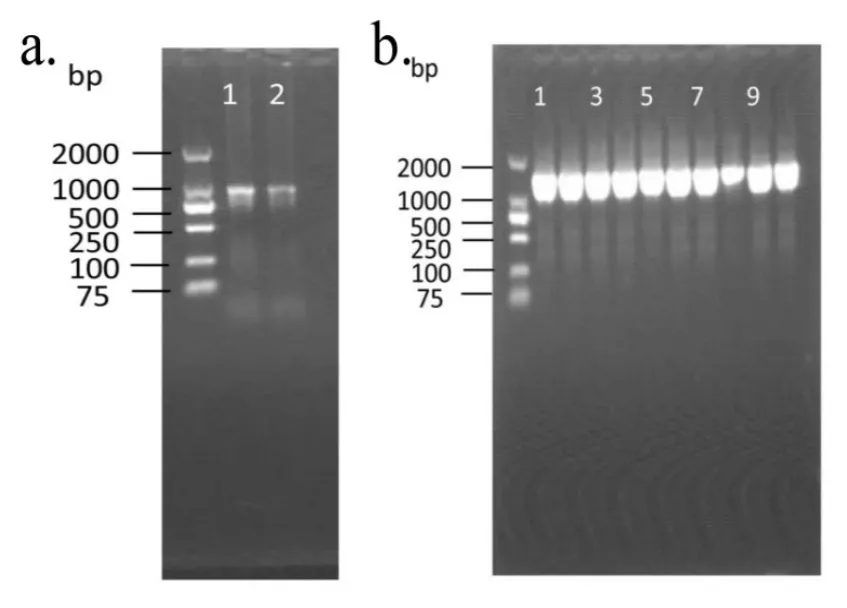
图6 基因回补Fig.6 Genes to cover
由图6a可知,回补质粒并未发生突变,跑胶条带大小为1000bp左右.随后用无缝克隆进行连接后导入E.coli.DH5α中,验证是否连接成功,凝胶电泳结果如图6b示,跑胶条带大小均为1000~2000bp,测序后与模拟序列对比,无突变点,提质粒导入 E.coli.WM3064并构建载体.
载体构建成功后,将 WYT ΔDyP与 E.coli.pRK415-DyP以1:2的菌体量滴在含有0.3mmol/L DAP 的 LB平板上(图 7a),直到菌落周围有明显的晕圈(图 7b).由于结合的效率较低,所以在结合筛选时,LB 平板中添加较高浓度的 Tc(50µg/mL)进行筛选,3d后长出明显的单克隆(7c).挑取结合成功的单克隆,并进行凝胶电泳验证(图 7d)后测序无突变点,获得了WYTΔDyP::DyP回补株.

图7 WYT△DyP::DyP回补株Fig.7 WYTΔDyP::DyP complementary strain
2.6 野生株、敲除株、回补株表型验证
VB4的降解脱色是由于蒽醌基团的开环,导致发色基团裂解.在相同条件下测试野生株、敲除株及回补株对 VB4染料的降解效果,记录菌株降解过程VB4浓度的变化,得到菌株的脱色率,如图8所示.

图8 野生菌株WYT、突变株WYT△DyP及回补株WYT△DyP::DyP对VB4的脱色率Fig.8 Growth and degradation curves of VB4 by DyP strains WYT△DyP and WYT△DyP
由图8可知,敲除株24h后对VB4的脱色率为22.17%,而在前期实验中,通过活细胞和加热至死细胞对VB降解研究中对比发现,加热致死菌24h后染料脱色率为20.49%,与敲除株相似,推测敲除株的少量脱色效果是由菌体的生物吸附引起的.Walker等[37]将菌株Bacillus gordonae,Bacillus benzeovorans和Pseudomonas putida在110℃高温下加热15min,测试对酸性蓝277的脱色性能,结果3株菌分别可去除 13%、19%和 18%的染料.基因回补后,回补株的脱色率为96%,与野生株相近,基因回补成功.
3 结论
3.1 通过全基因组测序分析推测了关键基因,WYT菌株的关键基因属于染料过氧化物酶基因中的B型.
3.2 通过基因敲除和回补验证推测的基因是 VB4降解脱色的关键基因.
3.3 测试野生株、敲除株、回补株降解VB染料的效果,发现敲除株几乎失去降解染料的功能,而回补株恢复了降解能力.因此,DyP基因是VB4降解脱色的关键基因.
致谢:感谢论文完成过程中给予我意见的安凤秋老师和李海红老师,同时感谢课题组的同学,谢谢你们!
