不同凡纳滨对虾养殖群体的微卫星遗传多样性分析
2021-08-03唐芳温贝妮刘红
唐芳 温贝妮 刘红


摘要:【目的】明確不同凡纳滨对虾(Litopenaeus vannamei)养殖群体间的遗传信息丰富度和遗传分化程度,为构建适应上海独特气候对虾品种繁育计划中的交配系谱分析提供参考依据。【方法】选用13对微卫星引物对来自厄瓜多尔恒兴对虾养殖公司2个养殖场(Pesquera和San Alfonso)共9个凡纳滨对虾养殖群体进行遗传多样性分析,探究不同群体间的遗传信息丰富度和遗传分化程度。【结果】13个微卫星位点在9个凡纳滨对虾养殖群体中检测到的等位基因数(Na)为2~6个,共有37个等位基因,多态信息含量(PIC)为0.1292~0.6799,平均为0.3326。在13个微卫星位点中,仅有1个微卫星位点(TUMXLV9.116)呈高度多态性,有4个微卫星位点(TUMXLV10.147、TUMXLV5.45c、TUMXLV10.191c和TUMXLV10.96)呈低度多态性,其余8个微卫星位点呈中度多态性。9个凡纳滨对虾养殖群体的观测杂合度(Ho)为0.2225~0.3662,平均为0.2915;期望杂合度(He)为0.3317~0.4539,平均为0.3974;Hardy-Weinberg平衡指数(D)为0.0214~0.4214,其中San Alfonso P23群体和Pesquera P23群体的D相对更接近于0,其基因型分布接近于Hardy-Weinberg平衡状态。9个凡纳滨对虾养殖群体的Fst平均值为0.1259,说明有12.59%的遗传分化来源于群体间,而87.41%的遗传分化来自群体内部;群体间的平均基因流(Nm)为1.7356,表明遗传漂变未能主导种群遗传结构的变化。在9个凡纳滨对虾养殖群体间,以Pesquera 29群体与Pesquera 15群体的遗传距离最大(0.2426),San Alfonso 23群体与Pesquera 23群体的遗传距离最小(0.0215);基于遗传距离的UPGMA聚类分析结果表明,9个凡纳滨对虾养殖群体可分为两大类,其中Pesquera 29群体和San Alfonso 12群体独立聚为一类。【结论】在9个凡纳滨对虾养殖群体中存在观测等位基因丢失现象,且遗传多样性较低,群体间分化程度为中等水平。因此,可通过引进不同地区拥有不同遗传背景且亲缘关系较远的群体作为亲本,以丰富子代群体的遗传多样性。
关键词: 凡纳滨对虾;养殖群体;微卫星分子标记;遗传多样性;遗传分化
中图分类号: S917.4 文献标志码: A 文章编号:2095-1191(2021)04-1108-08
Microsatellite genetic diversity in different Litopenaeus
vannamei breeding populations
TANG Fang, WEN Bei-ni, LIU Hong*
(College of Fisheries and Life Science, Shanghai Ocean University/Key Laboratory of Aquatic Genetic Resources Exploration and Utilization of Ministry of Education, Shanghai 201306, China)
Abstract:【Objective】To provide reference for mating and pedigree analysis in the breeding plan that the species in this research group could adapt to Shanghais unique climate, genetic information abundance and the degree of genetic differentiation among different Litopenaeus vannamei breeding populations were clarified. 【Method】Thirteen pairs of microsatellite primers were used to analyze the genetic diversity of nine L. vannamei breeding populations,which came from two farms(Pesquera and San Alfonso) of the L. vannamei breeding company in Ecuador. It could also explore the richness of genetic information and differentiation among different populations. 【Result】The number of alleles(Na) detected among nine L. vannamei breeding populations by 13 microsatellite loci ranged from 2 to 6, there were 37 alleles. Polymorphism information content index(PIC) was between 0.1292 and 0.6799, and the average PIC was 0.3326. Among the 13 microsatellite loci,only 1 microsatellite loci(TUMXLV9.116) had highly polymorphic,another 4 microsatellite loci(TUMXLV10.147, TUMXLV5.45c, TUMXLV10.191c and TUMXLV10.96) showed low polymorphism,and the remaining 8 microsatellite loci were moderately polymorphism. Otherwise, the observed heterozygosity(Ho) of 9 populations ranged from 0.2225 to 0.3662, and the average Ho was 0.2915. Expected heterozygosity(He)ranged from 0.3317 to 0.4539, and the average He was 0.3974. Hardy-Weinberg balance index(D) was ranging from 0.0214 to 0.4214. Among which the D of the San Alfonso P23 population and the Pesquera P23 population were relatively closer to 0, and their geno-type distribution was close to the Hardy-Weinberg balance state. The numerical valueof mean differentiation index(Fst) was 0.1259, indicating that there was 12.59% of genetic differentiation took place between the populations, and 87.41% of genetic differentiation occurred within the populations. Besides, the mean gene flow(Nm) between populations was 1.7356, indicating that genetic drift had not been able to dominate the change of population genetic structure. Among the 9 L. vannamei breeding populations, the genetic distance between Pesquera 29 and Pesquera 15 was the largest (0.2426), and the genetic distance between San Alfonso 23 and Pesquera 23 was the smallest(0.0215). According to the UPGMA clustering based on genetic distance, the 9 L. vannamei populations could be divided into two major groups, among which the Pesquera 29 population and the San Alfonso 12 population were grouped independently into one group. 【Conclusion】In the 9 breeding populations, lost observed allelic genes occurs, and they have low genetic diversity and mode-rate degree of differentiation among populations. It is possible to enrich the diversity of off spring populations by introdu-cing populations from different regions with different genetic backgrounds and long genetic distance as parents.
Key words: Litopenaeus vannamei; breeding populations; microsatellite molecular marker; genetic diversity; genetic differentiation
Foundation item: Shanghai Science and Technology Promoting Agriculture Project(2019-02-08-00-F01111)
0 引言
【研究意義】凡纳滨对虾(Litopenaeus vannamei)又称南美白对虾,是我国重要的养殖对虾品种,但近年来其经济性状明显衰退且病害频发(蒋葛等,2019),严重限制了凡纳滨对虾养殖产业的持续健康发展(包秀凤,2014;彭敏等,2020)。培育抗逆性强且生长快的优良对虾养殖品种,是确保凡纳滨对虾养殖业健康发展的重要保障(杨铭等,2017)。国外学者针对凡纳滨对虾的存活率、生长性能、抗病毒等性状已开展了一系列良种选育工作(Benzie et al.,1997;Argue et al.,2002;Gitterle et al.,2005);我国也选育出中兴1号、壬海1号、海兴农2号等生长速度快、存活率高、可高密度养殖的凡纳滨对虾品种(唐扬等,2018),但能大规模应用到实际生产的品种不多。本课题组多年来一直致力于开展耐低盐、能适应上海当地气候条件的凡纳滨对虾良种品系选育工作,现已完成F3代选育工作。为了更合理地利用这一种质资源,高质量开展良种选育工作,因此非常有必要对其种质资源进行遗传背景分析。【前人研究进展】微卫星(Microsatellites)又称为简单重复序列(Simple sequence repeats,SSR),是由2~6个碱基组成基序(motif)串联重复而形成的DNA序列(马宁和曾地刚,2013),具有共显性和多态性,且稳定性较好,现已广泛应用于生物群体间的遗传关系研究。Jerry等(2004)通过模拟和控制交配以开发日本对虾微卫星分子标记,并证实其在后代指定亲本方面的潜力,即微卫星分子标记可作为亲缘关系鉴定中的保留系谱信息。刘海情等(2012)利用微卫星分子标记分析广东省化州光辉养殖场有限公司新选育的吉富罗非鱼第16代群体(F16)的遗传多样性,证实该吉富罗非鱼群体具有较高的遗传异质性和较大的选育空间,可作为下一步选育工作的基础选育群体。鲁翠云等(2015)通过构建镜鲤全同胞家系的微卫星遗传图谱,最终筛选出83个优势基因型。孙成飞等(2015)基于微卫星分子标记比较分析中国罗氏沼虾和泰国罗氏沼虾养殖群体的遗传多样性及遗传结构,发现二者均具有较高的遗传多样性,但彼此亲缘关系较远。黄小芳等(2020)选取8个微卫星分子标记对广西不同地区的克氏原螯虾群体进行遗传多样性分析,结果显示广西不同地区的克氏原螯虾群体杂合子缺失现象普遍存在,应通过适当引种及加强不同地区群体间的遗传交流。近年来,凡纳滨对虾微卫星分子标记的开发和应用得到迅速发展(于洋,2014)。Meehan等(2003)最先公布了251个凡纳滨对虾微卫星序列,并筛选出93个具有多态性的微卫星引物,为凡纳滨对虾微卫星分子标记的开发奠定了基础。Cruz等(2004)利用微卫星分子标记(Pvan1578和Pvan1815)监测2代凡纳滨对虾的遗传多样性,结果显示在育种计划中无明显的遗传变异性损失,但等位基因的可变性降低,建议通过不同品系杂交以增加种群的遗传变异。谢丽等(2009)采用9对微卫星引物分析凡纳滨对虾4个选育群体(Molokai、OI、SIS和Kona Bay)的遗传多样性,结果显示OI群体与Kona Bay群体的亲缘关系最近,而SIS群体与Molokai群体的亲缘关系最远。李东宇等(2016)建立了一套微卫星多重PCR扩增体系并将其应用于凡纳滨对虾家系亲权鉴定,其模拟配对率和亲权鉴定准确率均为100%,尤其是全同胞和半同胞家系的鉴别效果良好。此外,王鸿霞等(2006)、刘洪涛等(2018)、方振朋等(2019)、黄小帅等(2019)、李强勇等(2020)也先后将微卫星分子标记应用于凡纳滨对虾的多态性分析及家系鉴定研究领域。【本研究切入点】微卫星分子标记具有多态性高、稳定性好、经济实惠、便于大批量分析等优点(杨铭等,2017),可用于监测育种方案中的群体遗传力及遗传变异水平。本课题组前期已培育出累计世代的自交和杂交群体,为了更好地继续开展育种工作,亟需引进不同地区拥有不同遗传背景且亲缘关系较远的群体作为亲本。【拟解决的关键问题】利用微卫星分子标记对来自厄瓜多尔恒兴对虾养殖公司2个养殖场(Pesquera和San Alfonso)共9个凡纳滨对虾养殖群体的遗传背景进行分析,明确9个群体间的遗传信息丰富度和遗传分化程度,以期为构建适应上海独特气候对虾品种繁育计划中的交配系谱分析提供参考依据。
1 材料与方法
1. 1 试验材料
凡纳滨对虾样品来自恒兴对虾养殖公司在厄瓜多尔的2个养殖场(Pesquera和San Alfonso),共9个养殖群体,其幼体来源于Texcumar公司,亲本均为泥塘养殖群体,每个群体选取30尾活虾,采集其肌肉组织置于无水乙醇中,-20 ℃保存备用。9个凡纳滨对虾养殖群体的亲本信息见表1。
1. 2 总DNA提取
采用海洋动物组织基因组DNA提取试剂盒[天根生化科技(北京)有限公司]提取凡纳滨对虾组织DNA,以Eppendorf分光光度计测定DNA浓度及OD值,并使用1.0%琼脂糖凝胶电泳检测DNA条带完整性;最后用TE溶液稀释至100 ng/μL,4 ℃保存备用。
1. 3 微卫星引物筛选与合成
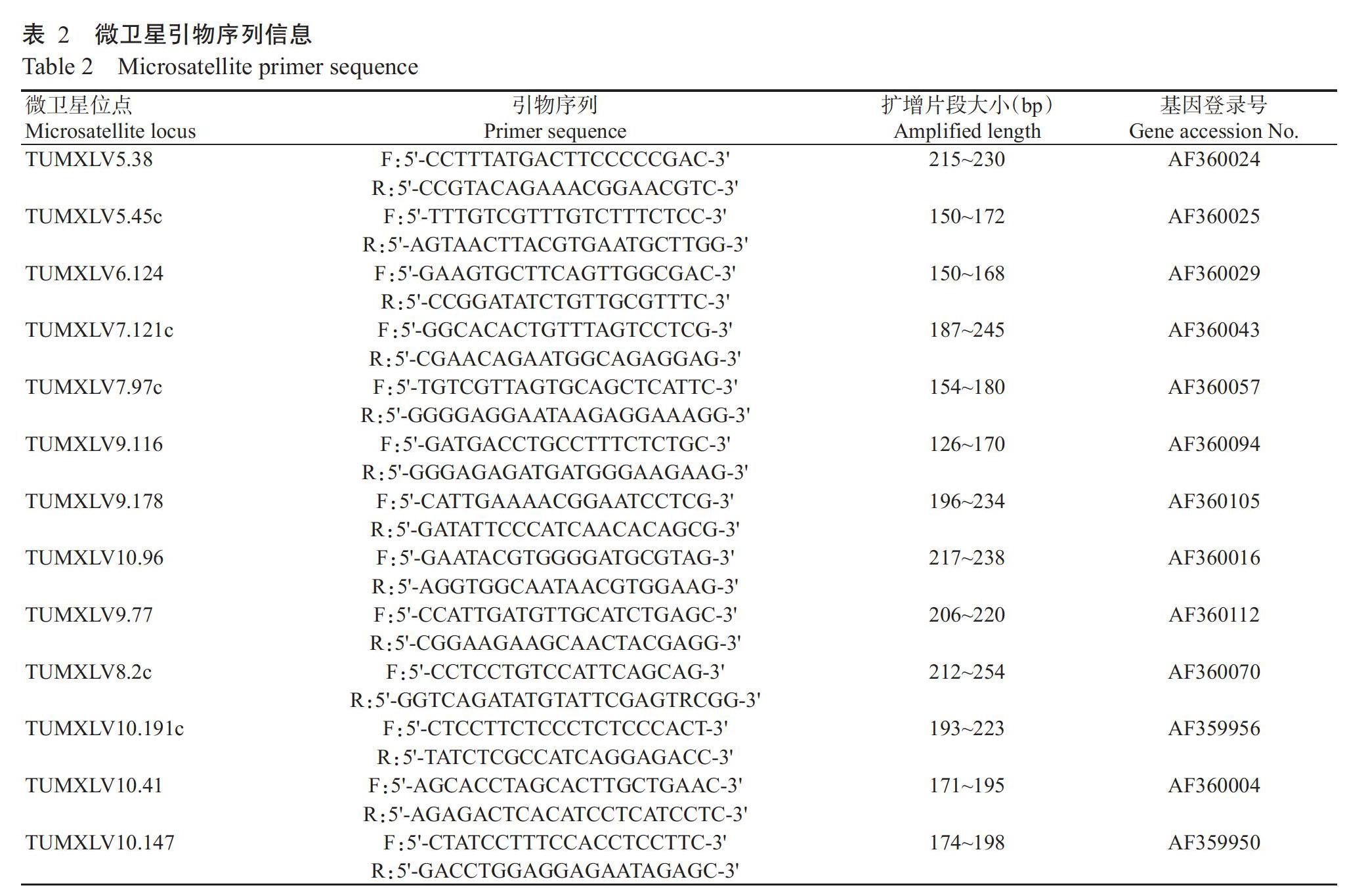
选用的13对微卫星引物由本课题组成员张海强于2015年筛选获得,委托生工生物工程(上海)股份有限公司合成。微卫星引物序列见表2。
1. 4 PCR扩增及电泳分析
PCR反应体系20.0 μL:2×Taq PCR MasterMix[生工生物工程(上海)股份有限公司]10.0 μL,DNA模板终浓度为10 ng/μL,上、下游引物各1.0 μL,加ddH2O补足至20.0 μL。扩增程序:95 ℃预变性4 min;94 ℃ 30 s,50~60 ℃ 30 s,72 ℃ 30 s,进行38个循环;94 ℃ 40 s,45 ℃ 40 s,72 ℃ 40 s,进行13个循环;72 ℃延伸5 min。采用8% SDS-PAGE进行分型,硝酸银染色,观察拍照。
1. 5 统计分析
获得9个凡纳滨对虾养殖群体的基因分型数据后,采用PopGene32计算群体的等位基因数(Na)、有效等位基因数(Ne)、观测杂合度(Ho)、期望杂合度(He)、Nei?s多样性指数(H)及Hardy-Weinberg平衡指数(D)等群体遗传学参数(Yeh and Boyle,1997);使用Cervus 3.0计算微卫星位点的多态信息含量(PIC);参照Nei(1978)的无偏向性方法(Unbiased measures)计算群体间的遗传距离和遗传相似系数;并根据9个凡纳滨对虾养殖群体的遗传距离,采用MEGA 5.0进行UPGMA聚类分析。
2 结果与分析
2. 1 凡纳滨对虾微卫星位点扩增结果
13对微卫星引物均能从凡纳滨对虾组织中稳定扩增出相应的条带,从9个凡纳滨对虾养殖群体中共检测獲得37个等位基因,且等位基因大小主要集中在150~260 bp。微卫星引物TUMXLV10.41在Pesquera 23群体中的PCR扩增产物经8% SDS-PAGE分型,可观察到单一(纯合)或2条(杂合)目的扩增条带(图1)。
2. 2 凡纳滨对虾微卫星位点遗传多样性分析结果
13个微卫星位点在9个凡纳滨对虾养殖群体中的遗传参数见表3。其中,Na为2~6个,平均为2.8462个;Ne的范围在1.2074~4.6310个,平均为2.0269个,Ho的范围在0.0171~0.5977,平均为0.2922;He的范围在0.1453~0.7856,平均为0.4412;PIC的范围在0.1292~0.6799,平均为0.3326。依据Botstein等(1980)的判断标准:PIC<0.25代表该位点呈低度多态性,0.25≤PIC≤0.50代表该位点呈中度多态性,PIC>0.50代表该位点呈高度多态性。在13个微卫星位点中,仅有1个微卫星位点(TUMXLV9.116)呈高度多态性,有4个微卫星位点(TUMXLV10.147、TUMXLV5.45c、TUMXLV10.191c和TUMXLV10.96)呈低度多态性,其余8个微卫星位点则呈中度多态性。
9个凡纳滨对虾养殖群体在13个微卫星位点上的遗传参数见表4。其中,Ne的范围在1.6341~1.9992个,Ho的范围在0.2225~0.3662,He的范围在0.3317~0.4539。Pesquera 5群体的PIC最小(0.2772),San Alfonso 32群体的PIC最大(0.3573),9个凡纳滨对虾养殖群体的PIC均在0.25~0.50范围内,表明这9个养殖群体的遗传多态性均为中等水平。此外,凡纳滨对虾养殖群体的D范围在0.0214~0.4214,均大于0,表明这9个凡纳滨对虾养殖群体的杂合子过剩,不同程度地偏离于Hardy-Weinberg平衡状态。
2. 3 凡纳滨对虾群体遗传分化分析结果
物种群体间的遗传分化等级通常采用遗传分化指数(Fst)和基因流(Nm)进行表示。由表5可知,有3个微卫星位点的Fst在0~0.05范围内,分别是TUMXLV10.147、TUMXLV8.2c和TUMXLV6.124,表明凡纳滨对虾群体在这3个微卫星位点上的遗传分化很小,可忽略不计;有2个微卫星位点(TUMXLV 10.191c和TUMXLV10.96)的Fst均大于0.25,表明凡纳滨对虾群体在这2个微卫星位点上存在明显的遗传分化;其余8个微卫星位点的Fst在0.05~0.15范围内,则表明凡纳滨对虾群体在这8个微卫星位点上存在中等程度的遗传分化。9个凡纳滨对虾养殖群体的Fst平均值为0.1259,说明有12.59%的遗传分化来源于群体间,而87.41%的遗传分化来自群体内部。由于Nm与Fst呈负相关,因此二者反映的群体遗传分化结果具有趋同性。在13个微卫星位点中,有3个微卫星位点(TUMXLV10.147、TUMXLV8.2c和TUMXLV6.124)的Nm>4.000,有2个微卫星位点(TUMXLV10.191c和TUMXLV10.96)的Nm<1.000,其余8个微卫星位点的Nm则介于1.000~4.000。
由表6可知,在9个凡纳滨对虾养殖群体间,以Pesquera 29群体与Pesquera 15群体的遗传距离最大(0.2426),而San Alfonso 23群体与Pesquera 23群体的遗传距离最小(0.0215)。在遗传相似性系数方面则表现为:Pesquera 29群体与Pesquera 15群体的遗传相似系数最小(0.7846),San Alfonso 23群体与Pesquera 23群体的遗传相似系数最大(0.9787)。可见,凡纳滨对虾养殖群体间的遗传相似性系数与遗传距离呈线性负相关。基于9个凡纳滨对虾养殖群体的遗传距离,采用MEGA 5.0进行UPGMA聚类分析,以直观呈现9个凡纳滨对虾养殖群体间的亲缘关系。如图2所示,9个凡纳滨对虾养殖群体可分为两大类(I类和II类),其中Pesquera 29群体和San Alfonso 12群体独立聚为I类。在II类中,Pesquera 15群体单独作为一支,San Alfonso 32群体和Pesquera 5群体聚为一支,其余4个养殖群体则聚为另一支。
3 讨论
3. 1 凡纳滨对虾的遗传多样性
本研究结果表明,从9个凡纳滨对虾养殖群体的13个微卫星位点共检测到37个等位基因,Na为2~6个,与张海强(2015)检测到Na为3~5个的结论存在差异,可能是近交繁殖导致这9个凡纳滨对虾养殖群体连续世代中存在观测等位基因丢失现象。等位基因较多的位点倾向于具有较高的等位基因分布值,因此所含信息量更大,更适用于养殖对虾遗传变异检测。PIC反映某个遗传标记多态性可提供的信息量。在本研究中,13个微卫星位点的PIC为0.1292~0.6799,平均为0.3326,略低于Meehan等(2003)研究得到的PIC(0.195~0.873),可能是在凡纳滨对虾育种过程中存在复杂的人为干扰,而对其种群的遗传信息量产生影响。此外,9个凡纳滨对虾养殖群体在13个微卫星位点上的Ho为0.2225~0.3622,平均为0.2915,低于Perez-Enriquez等(2009)、de Lima等(2010)检测凡纳滨对虾养殖群体获得的Ho(0.48~0.60和0.464~0.498)。说明本研究中的9个凡纳滨对虾养殖群体遗传多样性较低,究其原因可能是引进亲本数量较少,零等位基因的存在减少了样本量,或其亲本使用了起源不确定的杂交种。因此,今后应尽可能在较大范围内引进不同地区的亲本来繁育后代,并完善亲本遗传背景分析,以保证子代处于较高的杂合水平。
Hardy-Weinberg平衡指数(D)表征Ho与He的平衡关系。D为正值表示杂合子过剩,D为负值则表示处于杂合子缺失状态;D越接近于0,表明其基因型分布越接近于Hardy-Weinberg平衡狀态(谢丽等,2009)。在本研究的9个凡纳滨对虾养殖群体中,San Alfonso P23群体和Pesquera P23群体的D分别为0.0214和0.0376,相对更接近于0,说明其基因型分布接近于Hardy-Weinberg平衡状态,另外7个群体则明显偏离于Hardy-Weinberg平衡状态。究其原因可能是人工选择或组合交配育种过程中,凡纳滨对虾亲本数量偏少,即子代群体是由少量亲本组合杂交获得,子代群体等位基因数量也相对减少,进而导致偏离Hardy-Weinberg平衡状态及遗传漂变(赵志英等,2018)。
3. 2 凡纳滨对虾的遗传距离和遗传分化
Fst是反映群体间遗传分化程度的一个重要参数。本研究中,9个凡纳滨对虾养殖群体的平均Fst为0.1259,说明有12.59%的遗传分化来源于群体间,而87.41%的遗传分化来自群体内部,群体间的分化程度为中等水平。Hamrick等(1995)研究认为,Nm>1.000时能抵消遗传漂变作用,阻止种群间发生分化;Nm<1.000时,遗传漂变能主导种群遗传结构变化。本研究结果表明,9个凡纳滨对虾养殖群体间的平均Nm为1.7356,即遗传漂变未能主导种群遗传结构的变化,各群体间的遗传性趋向同一化,群体间的分化指数变小。Crawford和Littlepohn(1998)指出分化时间是影响群体间变异的主要决定因素,而遗传距离能客观反映群体间的分化时间及遗传变异。在9个凡纳滨对虾养殖群体中检测到的最大遗传距离为0.2426、最小遗传距离为0.0215,与Andriantahina等(2015)报道的最大遗传距离(0.481)和最小遗传距离(0.098)存在明显差异。现代杂种优势理论认为,子代的杂种优势与亲本间的遗传变异呈正相关(冯娜娜等,2012),因此在凡纳滨对虾人工繁育过程中,应选择遗传距离较远群体中具有优良性状的个体作为亲本,进行良种选育和亲本扩繁。
4 结论
在9个凡纳滨对虾养殖群体中存在观测等位基因丢失现象,且遗传多样性较低,群体间分化程度为中等水平。因此,可通过引进不同地区拥有不同遗传背景且亲缘关系较远的群体作为亲本,以丰富子代群体的遗传多样性。
参考文献:
包秀凤. 2014. 凡纳滨对虾选育群体遗传多样性分析[D]. 湛江:广东海洋大学. doi:10.7666/d.D522247. [Bao X F. 2014. Analysis of genetic diversity of selected stocks of Litopenaeus vannamei[D]. Zhanjiang:Guangdong Ocean University.]
方振朋,孟宪红,李旭鹏,栾生,曹家旺,陈宝龙,孔杰,闫茂仓,胡利华. 2019. 基于微卫星分子标记的凡纳滨对虾商业苗种遗传多样性分析[J]. 渔业科学进展,41(5):101-109. doi:10.19663/j.issn2095-9869.20190527004. [Fang Z P,Meng X H,Li X P,Luan S,Cao J W,Chen B L,Kong J,Yan M C,Hu L H. 2019. Genetic diversity analysis of domestic commercial brands seedlings of Litopenaeus vannamei based on microsatellite molecular mar-kers[J]. Progress in Fishery Sciences,41(5):101-109.]
冯娜娜,徐真,马洪雨,马春艳,乔振国,马凌波. 2012. 凡纳滨对虾7个不同家系遗传差异的微卫星标记分析[J]. 生物技术通报,(11):133-138. doi:10.13560/j.cnki.biotech.bull.1985.2012.11.015. [Feng N N,Xu Z,Ma H Y,Ma C Y,Qiao Z G,Ma L B. 2012. Genetic variances in seven families of Litopenaeus vannamei revealed by microsatellite markers[J]. Biotechnology Bulletin,(11):133-138.]
黃小芳,唐章生,刘俊丹,张宏燕,钟一治,卢智发,侯树鉴,王大鹏,陆专灵. 2020. 广西不同地区克氏原螯虾群体遗传多样性微卫星分析[J]. 南方农业学报,51(2):437-444. doi:10.3969/j.issn.2095-1191.2020.02.025. [Huang X F,Tang Z S,Liu J D,Zhang H Y,Zhong Y Z,Lu Z F,Hou S J,Wang D P,Lu Z L. 2020. Genetic diversity microsa-tellite analysis of Procambarus clarkii populations in different regions of Guangxi[J]. Journal of Southern Agriculture,51(2):437-444.]
黄小帅,徐煜,胡晓娟,徐武杰,苏浩昌,文国樑,杨铿,曹煜成. 2019. 利用微卫星标记分析7个凡纳滨对虾引进群体一代的遗传多样性[J]. 南方水产科学,15(1):54-62. doi:10.12131/20180135. [Huang X S,Xu Y,Hu X J,Xu W J,Su H C,Wen G L,Yang K,Cao Y C. 2019. Genetic diversity analysis of first filial generation of seven introduced Litopenaeus vannamei populations using microsa-tellite DNA markers[J]. South China Fisheries Science,15(1):54-62.]
蒋葛,沈辉,万夕和,乔毅,黎慧,王李宝,史文军. 2019. 凡纳滨对虾急性肝胰腺坏死综合症病虾与健康虾肠道优势菌群比较分析[J]. 江苏农业学报,35(1):142-148. doi:10.3969/j.issn.1000-4440.2019.01.021. [Jiang G,Shen H,Wan X H,Qiao Y,Li H,Wang L B,Shi W J. 2019. Di-fference analysis of intestinal flora between healthy Litopenaeus vannamei and acute hepatopancreatic necrosis syndrome L. vannamei[J]. Jiangsu Journal of Agricultu-ral Sciences,35(1):142-148.]
李东宇,孔杰,孟宪红,栾生,罗坤,卢霞,曹宝祥. 2016. 凡纳滨对虾(Litopenaeus vannamei)微卫星多重PCR体系的建立及其在家系亲权鉴定中的应用[J]. 渔业科学进展,37(3):58-67. doi:10.11758/yykxjz.20151125002. [Li D Y,Kong J,Meng X H,Luan S,Luo K,Lu X,Cao B X. 2016. Development of multiplex PCR systems of microsatellite markers for Pacific white shrimp(Litopenaeus vannamei) and its application for parentage identification[J]. Progress in Fishery Science,37(3):58-67.]
李强勇,李旻,曾地刚,朱威霖,彭敏,杨春玲,刘青云,赵永贞,陈秀荔,陈晓汉. 2020. 凡纳滨对虾微卫星分子标记的开发及不同养殖家系遗传多态性分析[J]. 南方农业学报,51(2):429-436. doi:10.3969/j.issn.2095-1191. 2020.02.024. [Li Q Y,Li M,Zeng D G,Zhu W L,Peng M,Yang C L,Liu Q Y,Zhao Y Z,Chen X L,Chen X H. 2020. Development of microsatellite markers of Litopenaeus vannamei and genetic polymorphism analysis of different cultured families[J]. Journal of Southern Agriculture,51(2):429-436.]
刘海情,郭昱嵩,王中铎,刘丽,刘楚吾. 2012. 利用微卫星分析吉富罗非鱼群体的遗传多样性[J]. 南方农业学报,43(1):94-98. doi:10.3969/j:issn.2095-1191.2012.01.94. [Liu H Q,Guo Y S,Wang Z D,Liu L,Liu C W. 2012. Genetic diversity analysis amongst GIFT strains of Oreochromis niloticus using microsatellites[J]. Journal of Southern Agri-culture,43(1):94-98.]
刘洪涛,杨明秋,何玉贵,唐贤明. 2018. 凡纳滨对虾八个地理群体遗传多样性的微卫星分析[J]. 海南大学学报(自然科学版),36(2):146-152. doi:10.15886/j.cnki.hdxbzkb.2018.0019. [Liu H T,Yang M Q,He Y G,Tang X M. 2018. Microsatellite analysis of genetic diversity in eight geographical populations of Litopenaeus vannamei[J]. Natural Science Journal of Hainan University,36(2):146-152.]
魯翠云,顾颖,李超,郑先虎,程磊,曹顶臣,俞菊华,何立川,孙效文. 2015. 镜鲤与建鲤生长性状共享QTL标记及优势基因型[J]. 中国水产科学,22(3):371-386. doi:10. 3724/SP.J.1118.2015.14293. [Lu C Y,Gu Y,Li C,Zheng X H,Cheng L,Cao D C,Yu J H,He L C,Sun X W. 2015. Analysis of shared QTLs and preferred genotypes between mirror carp and Jian carp[J]. Journal of Fishery Sciences of China,22(3):371-386.]
马宁,曾地刚. 2013. 凡纳滨对虾微卫星序列的筛选[J]. 西南农业学报,26(6):2629-2633. doi:10.16213/j.cnki.scjas. 2013.06.094. [Ma N,Zeng D G. 2013. Isolation of microsatellite sequences from Litopenaeus vannamei[J]. Southwest China Journal of Agricultural Sciences,26(6):2629-2633.]
彭敏,陈慧芳,李强勇,杨春玲,曾地刚,刘青云,赵永贞,陈晓汉,林勇,陈秀荔. 2020. 凡纳滨对虾连续3个世代选育群体的遗传多样性分析[J]. 南方农业学报,51(6):1442-1450. doi:10.3969/j.issn.2095-1191.2020.06.026. [Peng M,Chen H F,Li Q Y,Yang C L,Zeng D G,Liu Q Y,Zhao Y Z,Chen X H,Lin Y,Chen X L. 2020. Genetic diversity of three consecutive generations of Litopenaeus vannamei[J]. Journal of Southern Agriculture,51(6):1442-1450.]
孙成飞,叶星,董浚键,田园园,梁健辉. 2015. 罗氏沼虾6个养殖群体遗传多样性的微卫星分析[J]. 南方水产科学,11(2):20-26. doi:10.3969/j.issn.2095-0780.2015.02.003. [Sun C F,Ye X,Dong J J,Tian Y Y,Liang J H. 2015. Genetic diversity analysis of six cultured populations of Macrobrachium rosenbergii using microsatellite markers[J]. South China Fisherise Science,11(2):20-26.]
唐扬,孟小菲,沈瑞福,黄永春. 2018. 凡纳滨对虾家系选育的研究与应用[J]. 水产科学,37(4):555-563. doi:10.16378/j.cnki.1003-1111.2018.04.020. [Tang Y,Meng X F,Shen R F,Huang Y C. 2018. Research and application of family selective breeding in culture of Pacific white shrimp Litopenaeus vannamei[J]. Fisheries Science,37(4):555-563.]
王鸿霞,吴长功,张留所,相建海. 2006. 微卫星标记应用于凡纳滨对虾家系鉴别的研究[J]. 遗传,28(2):179-183. doi:10.16288/j.yczz.2006.02.010. [Wang H X,Wu C G,Zhang L S,Xiang J H. 2006. The application of microsa-tellite markers for parentage determination in selective breeding of Pacific white shrimp(Litopenaeus vannamei)[J]. Hereditas(Beijing),28(2):179-183.]
谢丽,陈国良,叶富良,栗志民. 2009. 凡纳滨对虾4个选育群体遗传多样性的SSR分析[J]. 广东海洋大学学报,29(4):5-9. doi:10.3969/j.issn.1673-9159.2009.04.002. [Xie L,Chen G L,Ye F L,Li Z M. 2009. Genetic diversity of four selected stocks of Litopenaeus vannamei as revealed by SSR marker[J]. Journal of the Guangdong Ocean University,29(4):5-9.]
杨铭,于洋,张晓军,王全超,刘敬文,李富花,相建海. 2017. 基于转录组数据的凡纳滨对虾微卫星标记开发[J]. 海洋科学,41(2):96-102. doi:10.11759//hykx20160322002. [Yang M,Yu Y,Zhang X J,Wang Q C,Liu J W,Li F H,Xiang J H. 2017. Development of microsatellite markers from the transcriptome sequences of Pacific white shrimp (Litopenaus vannamei)[J]. Marine Science,41(2):96-102.]
于洋. 2014. 凡纳滨对虾分子标记的开发及其在遗传育种中的应用[D]. 北京:中国科学院大学. [Yu Y. 2014. Deve-lopment of molecular markers and their application in selective breeding of the Pacific white shrimp,Litopenaeus vannamei[D]. Beijing:The University of Chinese Academy of Sciences.]
張海强. 2015. 不同来源的凡纳滨对虾群体遗传多样性的初步分析[D]. 上海:上海海洋大学. [Zhang H Q. 2015. Preliminary analysis of genetic diversity of different po-pulations of Litopenaeus vannamei[D]. Shanghai:Shanghai Ocean University.]
赵志英,梁丽运,白丽蓉. 2018. 斑节对虾3个野生群体遗传多样性的微卫星标记分析[J]. 热带海洋学报,37(3):65-72. doi:10.11978/2017084. [Zhao Z Y,Liang L Y,Bai L R. 2018. Analysis of genetic diversity among three wild populations of Penaeus monodon using microsatellite mar-ker[J]. Journal of Tropical Oceanography,37(3):65-72.]
Andriantahina F,Liu X L,Huang H. 2015. Using microsatellite markers to identify heritability of Pacific whiteleg shrimp Litopenaeus vannamei[J]. Acta Oceanologica Sinica,34(6):59-65. doi:10.1007/s13131-015-0688-6.
Argue B J,Arce S M,Lotz J M,Moss S M. 2002. Selective breeding of Pacific white shrimp(Litopenaeus vannamei) for growth and resistance to Taura Syndrome Virus[J]. Aquaculture,204(3-4):447-460. doi:10.1016/S0044-8486(01)00830-4.
Benzie J A H,Kenway M,Trott L. 1997. Estimates for the heritability of size in juvenile Penaeus monodon prawns from half-sib matings[J]. Aquaculture,152(1-4):49-53. doi:10.1016/S0044-8486(96)01528-1.
Botstein D,White R L,Skolnick M,Davis R W. 1980. Construction genetic linkage map in man using restriction fragment length polymorphisms[J]. American Journal of Human Genetics,32(3):314-331.
Crawford A M,Littlepohn R P. 1998. The use of DNA marker in deciding conservation priorities in sheep and other livestock[J]. Animal Genetic Resources Information,23:21-26. doi:10.1017/S1014233900004909.
Cruz P,Ibarra A M,Mejia-Ruiz H,Gaffney P M,Pérez-Enríquez R. 2004. Genetic variability assessed by microsatellites in a breeding program of Pacific white shrimp (Litopenaeus vannamei)[J]. Marine Biotechnology,6(2):157-164. doi:10.1007/s10126-003-0017-5.
de Lima A P S,da Silva S M B C,O1iveira K K C,Maggioni R,Coimbra M R M. 2010. Genetics of two marine shrimp hatcheries of the Pacific white shrimp Litopenaeus vannamei (Boone,1931) in Pernambuco,Brazil[J]. Ciência Rural,40(2):325-331. doi:10.1590/S0103-8478 2010005000008.
Gitterle T,Salte R,Gjerde B,Cock J,Johansen H,Salazar M,Lozano C,Rye M. 2005. Genetic(co) variation in resistance to White Spot Syndrome Virus(WSSV) and harvest weight in Penaeus(Litopenaeus) vannamei[J]. Aquaculture,246(1-4):139-149. doi:10.1016/j.aquaculture. 2005.02.011.
Hamrick J L,Godt M J W,Sherman-Broyles S L. 1995. Gene flow among plant populations:Evidence from genetic markers. In Experimental and molecular approaches to plant biosystematics[M]//Hoch P G, Stephenson A G. Experimental and molecular to plant biosystematics. St Louis:Missouri Botanical Garden Press:215–232.
Jerry D R,Preston N P,Crocos P J,Keys S,Meadows J R S,Li Y T. 2004. Parentage determination of Kuruma shrimp Penaeus(Marsupenaeus) japonicus using microsatellite markers(Bate)[J]. Aquaculture,235(1-4):237-247. doi: 10.1016/j.aquaculture.2004.01.019.
Meehan D,Xu Z K,Gladys Z,Alcivar-Warren A. 2003. High frequency and large number of polymorphic microsatellites in cultured shrimp,Penaeus(Litopenaeus) vannamei[Crustacea:Decapoda][J]. Marine Biotechnology,5(4):311-330. doi:10.1007/s10126-002-0092-z.
Nei M. 1978. Estimation of average hetemzygosity and gene-tic distance from a small number of individuals[J]. Genetics,89(3):583-590.
Perez-Enriquez R,Hernández-Martínez F,Cruz P. 2009. Genetic diversity status of White shrimp Penoeus(Litopenaeus) vannamei broodstock in Mexico[J]. Aquaculture,297(1-4):44-50. doi:10.1016/j.aquaculture.2009.08.038.
Yeh F C,Boyle T J. 1997. Population genetic analysis of co-dominant and dominant markers and quantitative traits[J]. Belgian Journal of Botany,129(2):157.
(責任编辑 兰宗宝)
