水产品腐败希瓦氏菌冷激蛋白的结构与功能分析
2021-05-15李秋莹徐瑾秀朱金帅励建荣
李秋莹,徐瑾秀,朱金帅,林 洪,孙 彤,励建荣*
(1 渤海大学食品科学与工程学院 辽宁锦州121013 2 生鲜农产品贮藏加工及安全控制技术国家地方联合工程研究中心 辽宁锦州121013 3 中国海洋大学食品科学与工程学院 山东青岛266100)
水产品口感鲜美、营养丰富,深受消费者的喜爱,然而,水产品极易腐败变质,造成巨大的经济损失。由于微生物的生长和入侵是导致水产品腐败的主要因素[1],因此水产品的保鲜过程中对微生物的抑制十分重要。低温保鲜技术是使用最早,也是应用最广的水产品保鲜技术[2]。低温会使微生物的生理机能发生重要的变化,比如膜流动性降低,核酸二级结构稳定性增强,转录、翻译及蛋白折叠的效率降低等[3]。在低温下有些微生物仍能以一定的生长速率生长,即使生长较慢。腐败希瓦氏菌(Shewanella putrefaciens)是一种耐冷菌,在0 ℃下也能生长、繁殖,其在有氧冷冻或冷藏状态下是多种水产品的特定腐败菌[4],引起冷藏水产品的腐败变质,这严重降低了水产品的品质,极有可能导致食品安全问题。鉴于水产品保鲜的重要经济价值,深入了解腐败希瓦氏菌低温适应的分子机制,可以辅助预测腐败菌在冷藏食品中的行为,便于进行更准确的风险预测和防范。
研究发现,微生物在低温作用下会产生冷应激反应,生成一系列的抗低温蛋白,称为冷激蛋白(Cold shock protein,CSPs)[5]。细菌冷激蛋白是一类包含核酸结合冷休克结构域的高度保守的小分子蛋白(~7.4 ku),可以作为分子伴侣与单链DNA或单链RNA 结合,阻止DNA 或RNA 在低温下形成二级结构,从而促进转录和翻译[6]。此外,冷激蛋白还参与细菌的多个生理生化过程,如:细胞膜的流动性,细菌正常生长与分化,营养胁迫,细菌的毒力等。冷激蛋白对细菌在低温时恢复生长和发挥细胞各种功能是非常重要的[7-9],是解析微生物低温适应机制的关键蛋白。然而,目前关于腐败希瓦氏菌冷激蛋白的研究鲜有报道。
细菌通常具有多个冷激蛋白,且冷激蛋白的作用规律十分复杂,并非所有冷激蛋白都受低温诱导表达[10]。通过对NCBI(National center for biotechnology information)数据库中腐败希瓦氏菌模式菌株的全基因组序列检索,发现腐败希瓦氏菌中存在3 个典型冷激蛋白,目前尚未发现有关其主要冷激蛋白及相关功能的研究。本文以水产品的腐败希瓦氏菌为研究对象,通过PCR 扩增获得水产品腐败希瓦氏菌的3 个冷激蛋白基因序列,在此基础上通过生物信息学分析腐败希瓦氏菌冷激蛋白的理化性质、结构和功能特性等,旨在为冷激蛋白在腐败希瓦氏菌低温适应过程中的功能研究,以及对水产品中腐败希瓦氏菌的抑制提供参考。
1 材料与方法
1.1 材料与试剂
腐败希瓦氏菌为本实验室自腐败的冷藏大菱鲆中分离所得,于-80 ℃保藏。聚合酶链式反应(Polemerase chain reaction,PCR)引物委托生工生物工程(上海)技术服务有限公司合成。EasyTaqRDNA Polymerase,北京全是金生物技术有限公司;细菌基因组DNA 提取试剂盒、GeneRed 核酸染料,天根生化科技(北京)有限公司;Biowest 琼脂糖、LB 营养琼脂、LB 营养肉汤,青岛高科园海博生物技术有限公司。
1.2 仪器与设备
防蒸发梯度PCR,美国Eppendrof 公司;凝胶电泳及配套设备,美国Bio-Bad 公司;微量冷冻离心机,美国Thermo 公司。
1.3 菌种鉴定及冷激蛋白基因的克隆与测序
活化本实验室保藏的腐败希瓦氏菌,按照基因组DNA 提取试剂盒的操作步骤提取基因组DNA,利用细菌16S rRNA 基因通用引物27F 和1492R 进行PCR 扩增,获得腐败希瓦氏菌的16S rRNA 基因(表1)[11]。采用NCBI(https://www.ncbi.nlm.nih.gov/) 检索近缘腐败希瓦氏菌菌株基因组信息(GenBank:CP002457.1),依据注释冷激蛋白基因及其上、下游序列,设计引物(表1),进行冷激蛋白的Csp1589,Csp1376 和Csp2252 基因的扩增。PCR 扩增条件为94 ℃5 min;94 ℃30 s,Tm℃30 s(详见表1),72 ℃45 s,循环30 次。PCR 产物经2%的琼脂糖凝胶电泳检测后送至上海生工测序。
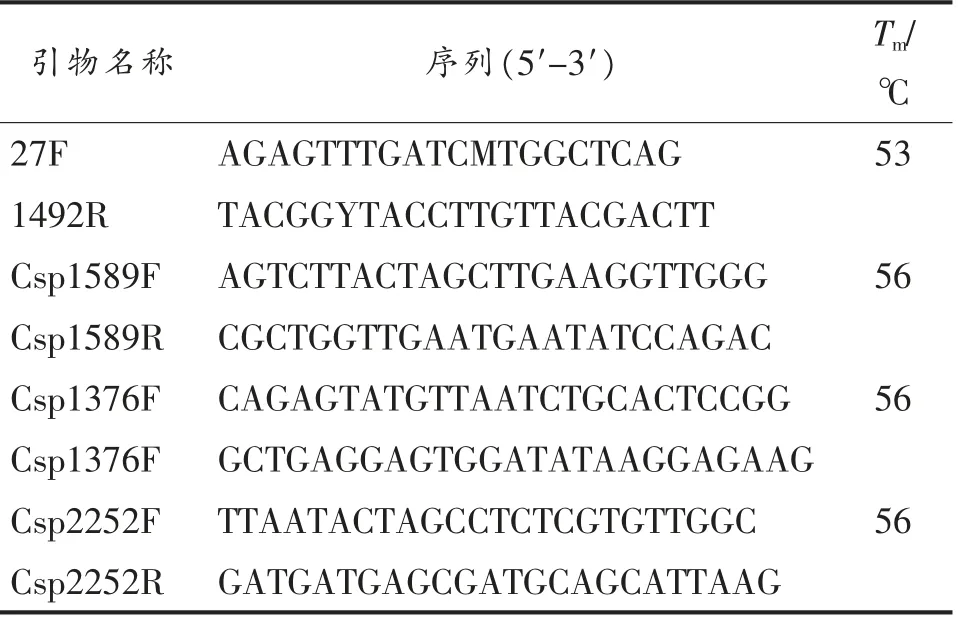
表1 菌种鉴定及冷激蛋白基因扩增的引物Table 1 Primers of strain identification and cold shock protein gene amplification
1.4 系统发生树的构建
自NCBI 数据库获得大肠杆菌(NC_000913.3)的9 个冷激蛋白(CspA~I),以FASTA file 形式保存。通过软件Clustal X 对测序所得腐败希瓦氏菌冷激蛋白和大肠杆菌的冷激蛋白进行序列比对,并用Mega7.0 中的邻接法 (Neighbor-jioning method,NJ)为其构建系统发生树,步长值(Bootstrap values)为1 000[12]。
1.5 冷激蛋白的理化性质及结构域的预测
用ExPASy 网站中的一个工具ProtParam(http://web.expasy.org/protparam/)来预测腐败希瓦氏菌冷激蛋白的基本生理、生化特征,用PSORT www Server 中的PSORT Prediction 工具预测冷激蛋白细胞定位,用TMHMM Server 2.0 预测冷激蛋白跨膜区域,用SignalIP 5.0 在线预测冷激蛋白信号肽,用SMART(http://smart.embl-heidelberg.de/)预测冷激蛋白结构域的构成及其功能,用在线软件NCBI Conserved Domain Search 进行其氨基酸保守序列分析。
1.6 结构预测与模型质量评估
使 用PSIPRED (http://bioinf.cs.ucl.ac.uk/psipred/) 预测腐败希瓦氏菌冷激蛋白的二级结构。将腐败希瓦氏菌冷激蛋白的氨基酸序列提交到SWISS-MODEL(https://swissmodel.expasy.org)网站进行三级结构的预测[13]。将构建的蛋白三级模型用PROCHECK 和Verify3D 进行质量评估[14-16]。PROCHECK 和Verify3D 是SAVES v5.0(http://servicesn.mbi.ucla.edu/SAVES/)中的结构评估服务。
1.7 互作蛋白及配体结合位点的预测
用STRING(https://string-db.org/)工具确定在活体内与腐败希瓦氏菌冷激蛋白存在相互作用的蛋白。这种相互作用既包括蛋白质之间直接的物理相互作用,也包括蛋白质之间间接的功能相关性[17]。将从SWISS-MODEL 得到的模型文件输入COACH (https://zhanglab.ccmb.med.umich.edu/COACH/),以预测腐败希瓦氏菌冷激蛋白潜在的配体结合位点[18-19]。
2 结果与分析
2.1 冷激蛋白基因的克隆
通过16S 测序结果的比对分析确定该菌为腐败希瓦氏菌。以这株水产品腐败希瓦氏菌的基因组DNA 为模板,用相应引物对冷激蛋白基因进行PCR 扩增,然后用2%的琼脂糖凝胶电泳检测PCR 扩增产物[19],结果如图1所示。PCR 扩增产物的长度在500 bp 左右,与预期的产物大小相符。将PCR 产物送至上海生工测序,并将测序序列用ORF Finder 软件和Blast 分析。使用ORF Finder软件的结果表明,克隆的基因片段都有完整的开放阅读框 (ORF),3 个冷激蛋白基因Csp1589,Csp1376 和Csp2252 的ORF 长度分别为210,210,207 bp,起始密码子为ATG,终止密码子为TAA,分别编码69,69,68 个氨基酸。Blast 结果表明,水产品腐败希瓦氏菌的3 个冷激蛋白基因与腐败希瓦氏菌模式菌株的3 个冷激蛋白基因的序列相似性均达100%,说明冷激蛋白高度保守。
2.2 系统发生树的构建
使用MEGA 7.0 软件构建腐败希瓦氏菌和大肠杆菌的冷激蛋白的系统发生树,结果如图2所示。水产品腐败希瓦氏菌的冷激蛋白基因Csp1589 和Csp1376 与大肠杆菌的冷激蛋白基因CspA 聚类在同一类群,表明它们之间的有较高的相似性和较近的亲缘关系。大肠杆菌CspA 为主要的冷诱导蛋白,低温下大量表达[7,20-21]。初步推测Csp1589 和Csp1376 在腐败希瓦氏菌对低温胁迫的适应中起关键作用。腐败希瓦氏菌的冷激蛋白基因Csp2252 与大肠杆菌的冷激蛋白基因CspD位于同一分枝,大肠杆菌CspD 在营养缺乏时大量表达并能结合单链DNA,从而抑制染色体的复制[22],因此腐败希瓦氏菌的冷激蛋白基因Csp2252可能与细胞生长分化有关。
2.3 冷激蛋白的理化特点
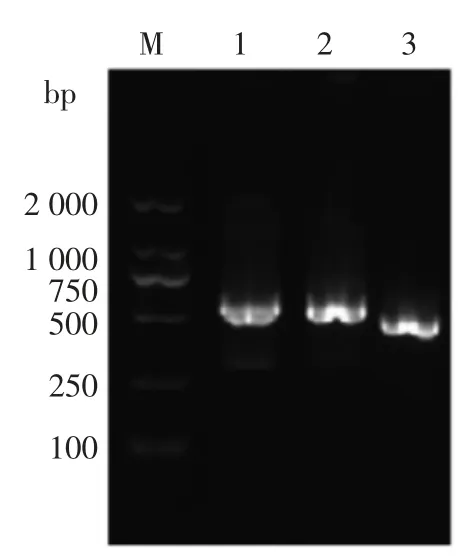
图1 冷激蛋白基因PCR 扩增产物的凝胶电泳图Fig.1 Gel electrophoresis of PCR amplification products of CSPs genes
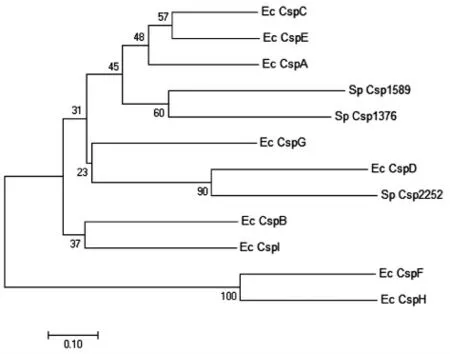
图2 冷激蛋白系统发生树Fig.2 Phylogenetic analysis of the CSPs
利用ProtParam 预测腐败希瓦氏菌冷激蛋白的基本生理、生化特征,结果见表2。预测的腐败希瓦氏菌冷激蛋白是由68~69 个氨基酸残基构成,分子质量在7.4 ku 左右的小分子蛋白,且其带负电荷的氨基酸的量大于带正电荷氨基酸的量,即酸性氨基酸的量大于碱性氨基酸的量,PI 小于7,说明这些蛋白质都是酸性蛋白质,符合CSPs 的特点[23-24]。蛋白质的总平均亲水性为负数,说明测定的蛋白皆为亲水性蛋白[25]。由不稳定系数可知,除Csp2252 外,蛋白质在体外的稳定性都较好,在大肠杆菌体内的半衰期大于10 h。PSORT 预测腐败希瓦氏菌冷激蛋白皆定位于细胞质。TMHMM预测结果显示腐败希瓦氏菌冷激蛋白均不存在α螺旋跨膜,是非跨膜蛋白。SignalIP 预测结果表明腐败希瓦氏菌冷激蛋白不存在信号肽,不能分泌到胞外。推测腐败希瓦氏菌的3 个冷激蛋白均为非分泌型、非跨膜蛋白,冷激蛋白在腐败希瓦氏菌细胞质中合成后不进行蛋白转运,而是保留在细胞基质中发挥作用。

表2 冷激蛋白的理化性质分析Table 2 Analysis of the psysicochemical property of the CSPs
2.4 冷激蛋白的氨基酸保守序列及结构域分析
将腐败希瓦氏菌冷激蛋白的氨基酸序列提交至CD service,检测结果显示:腐败希瓦氏菌的冷激蛋白Csp1589、Csp1376 和Csp2252 都具有DNA 结合位点和RNA 结合基序,且同属于类S1总科。类S1 总科是一类包含与核糖体蛋白S1 的RNA 结合结构域相似的结构域的蛋白合集[26]。这种结构最初在S1 核糖体蛋白中被发现,目前已在多种RNA 相关蛋白中被发现。SMART 预测结果表明:腐败希瓦氏菌的3 个冷激蛋白都包含冷休克结构域(Cold shock domain,CSD),冷休克结构域属于类S1 总科这个超级家族,且其从原核生物到高等真核生物高度保守,具有RNA 分子伴侣功能[27-28]。包含冷休克结构域的蛋白可以参与多种细胞生理生化过程,包括对低温的适应、营养胁迫、细胞生长和发育过程等[29]。此外,由图3可知腐败希瓦氏菌的冷激蛋白Csp1589 和Csp1376 属于保守蛋白结构域家族CspC,是冷诱导蛋白,因此推测Csp1589 和Csp1376 与细菌对低温的适应性有关;而腐败希瓦氏菌Csp2252 不属于CspC 家族,其可能参与其它的细胞过程,这个结果与上述系统发生树的结果一致。
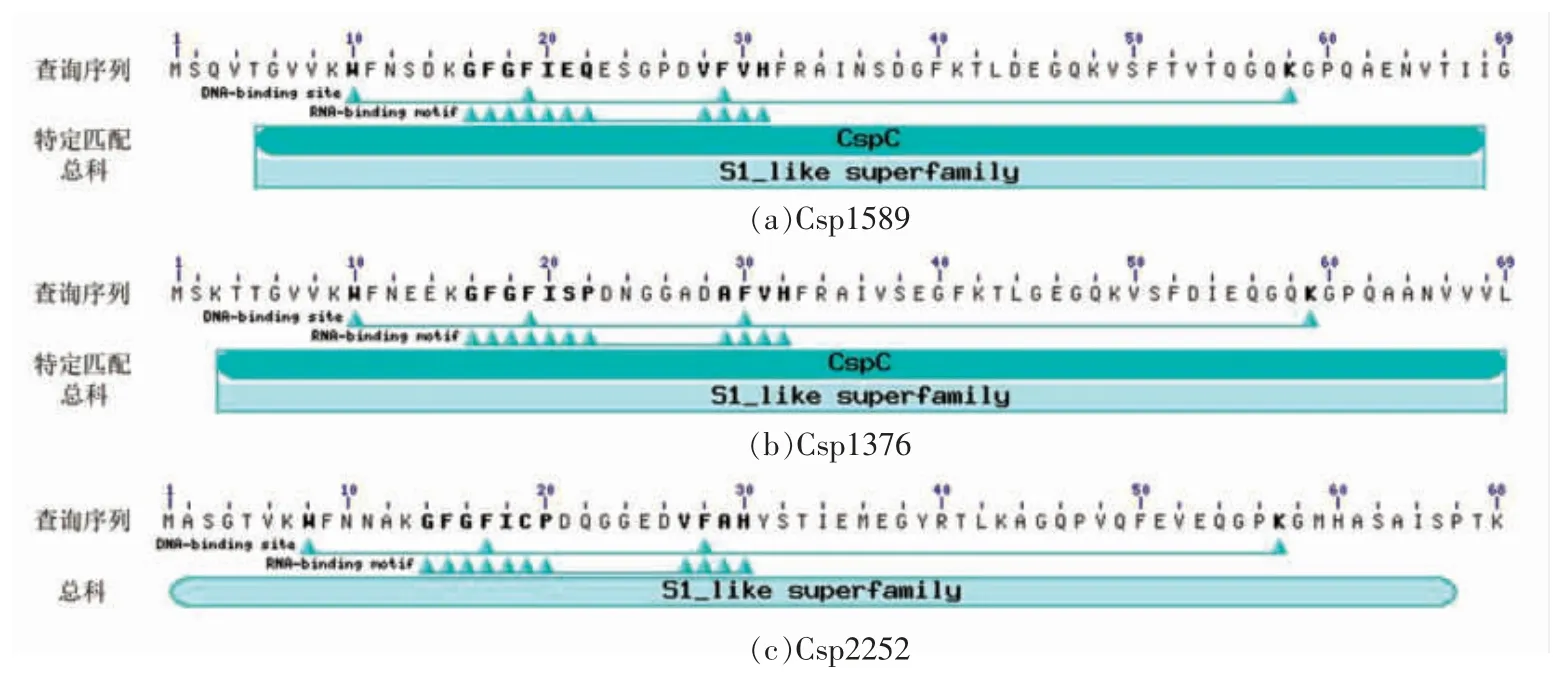
图3 腐败希瓦氏菌冷激蛋白的氨基酸保守序列Fig.3 Amino acid conserved sequence of S.putrefaciens CSPs
2.5 冷激蛋白的结构分析
为更好了解冷激蛋白潜在的生物功能,分析其蛋白的空间结构。将水产品腐败希瓦氏菌冷激蛋白的氨基酸序列提交到PSIPRED 预测其二级结构,结果见图4。典型的冷激蛋白的二级结构包含5 个β 折叠[24],预测得到的二级结构与这个结果大致相符,Csp1589 和Csp1376 二级结构的最后一个β 折叠出现断层,这可能是天冬酰胺预测可信度太低所致。
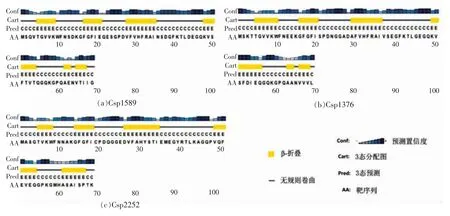
图4 腐败希瓦氏菌冷激蛋白的二级结构Fig.4 Secondary structure of S.putrefaciens CSPs
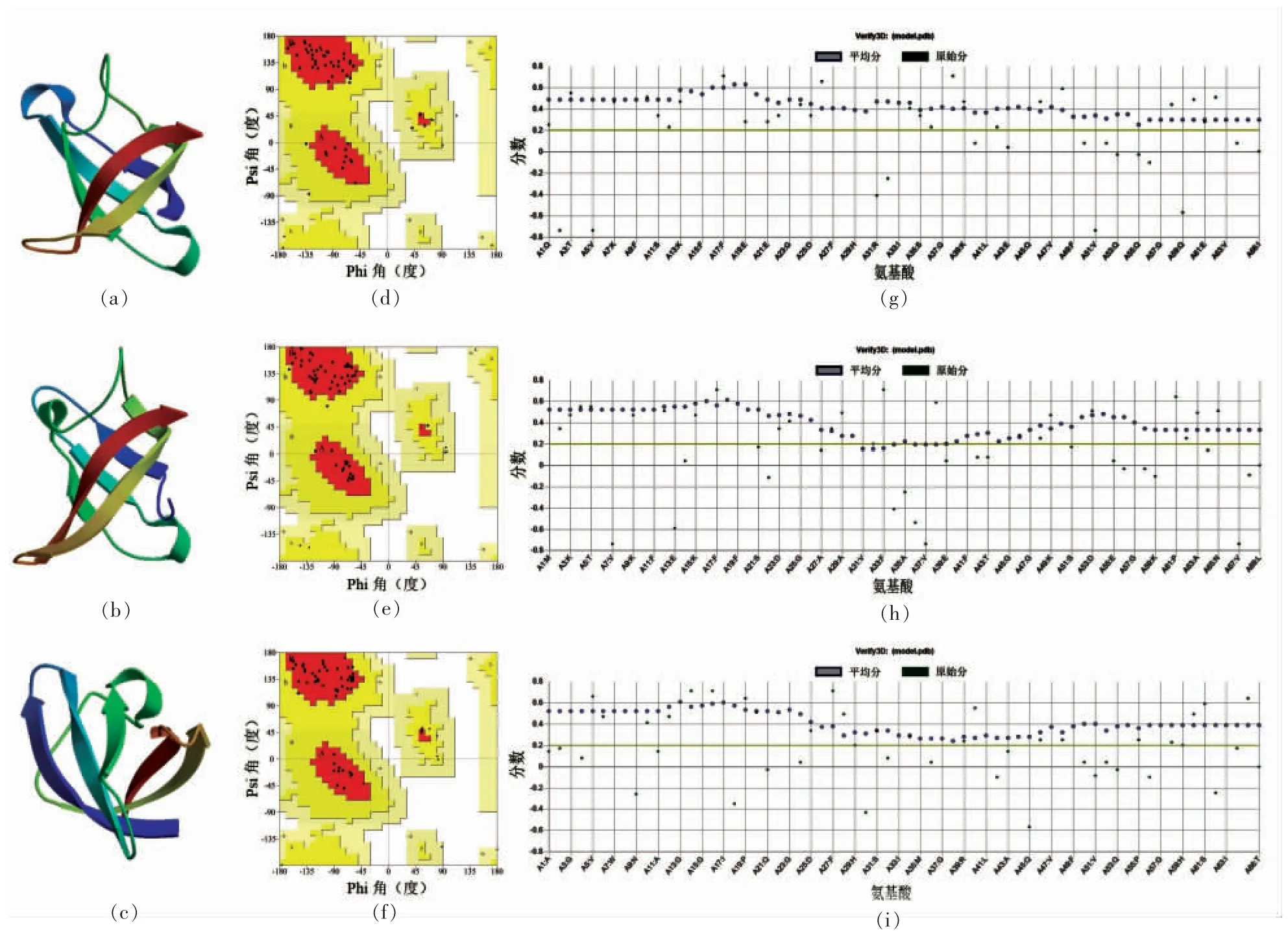
图5 腐败希瓦氏菌冷激蛋白的三级结构及结构评估Fig.5 Tertiary structure and structure assessment of S.putrefaciens CSPs
将冷激蛋白的氨基酸序列提交SWISS-MODEL 网站,根据序列相似性及模型质量分别选取1mjc 的A 链、5xv9 的A 链、3i2z 的B 链作为Csp1589、Csp1376 和Csp2252 的模板建模。然后,用PROCHECK 和verify3D 对所建三级模型进行质量评估。构建的腐败希瓦氏菌冷激蛋白三级结构都包含5 个反向平行的β 链,其形成了一个桶状结构,符合冷激蛋白的已知特征[24]。由拉氏构象图可看出,对于Csp1589 蛋白模型,氨基酸处于有利区域、额外允许区域、一般允许区域以及不允许区域的量分别为88.9%,11.1%,0%和0%。对于Csp1376 型蛋白模型,对应值分别为92.7%,7.3%,0.0%和0.0%。对于Csp2252 型蛋白模型,对应值分别为96.1%,3.9%,0.0%和0.0%,氨基酸全部处于有利区域和额外允许区域,模型质量较好。在Verify3D 结果中,对于Csp1589、Csp1376 和Csp2252 模型,平均分≥0.2 的氨基酸的量分别是和100%,89.86%和100%,平均分≥0.2 的氨基酸的量达到80%就认为蛋白三级模型是合格的,因此,以verify3D 结果来看,这3 个模型均具有较好的质量。
2.6 冷激蛋白的互作蛋白及配体结合位点分析
利用STRING 和COACH 网站对腐败希瓦氏菌冷激蛋白进行分析,得到如图6所示与其紧密作用的蛋白质和潜在的配体结合位点。对于腐败希瓦氏菌冷激蛋白 Csp1589、Csp1376 和Csp2252,STRING 分别检测到5,4,10 个与其相互作用的蛋白(图6)。与冷激蛋白Csp1589 存在相互作用的蛋白是组氨酸激酶(ABP76854.1)、甲基转移酶(ABP75296.1)、DNA 聚合酶1(polA)、多功能融合蛋白(trpC)、伴侣蛋白(htpG)。与冷激蛋白Csp1376 存在相互作用的蛋白是组氨酸激酶(ABP76854.1)、DNA 聚合酶1(polA)、多功能融合蛋 白(trpC)、伴 侣 蛋 白(htpG)。与 冷 激 蛋 白Csp2252 存在相互作用的蛋白是依赖ATP 的Clp蛋白酶(ABP75948.1)、依赖ATP 的Clp 蛋白酶适配蛋白(ClpS)、组氨酸激酶(ABP76854.1)、DNA聚合酶1(polA)、多功能融合蛋白(trpC)、组氨酸-磷酸氨基转移酶(hisC)、磷酸庚糖异构酶(gmhA)、含有CBS 结构域的蛋白(ABP76571.1)、氮调控蛋白PtsN(ABP74443.1)、伴侣蛋白(htpG)。其中组氨酸激酶是一个磷酸化组氨酸保守残基的信号传导酶家族,参与构成双组分信号传导系统。双组分信号传导系统可以感应环境变化,应对环境变化引起的负面效应[30],研究表明一些双组分信号转导系统参与了细菌的低温适应过程[10],推测腐败希瓦氏菌冷激蛋白与细胞对逆境的适应有关。DNA聚合酶是DNA 复制的重要酶,冷激蛋白可能与DNA 复制有关。冷激蛋白作为一种伴侣蛋白,可能与伴侣蛋白htpG 存在竞争关系。从分析结果可推测相较Csp1589 和Csp1376,腐败希瓦氏菌冷激蛋白Csp2252 的功能可能更为复杂。在许多重要的生命活动中,蛋白质功能的执行依赖于蛋白质与配体的相互作用,对于蛋白质配体结合位点的预测具有重要的意义[31]。例如:把小分子放到目的蛋白的配体结合位点,优化配体构象和位置,使之和目的蛋白有最佳的结合作用,就有可能发现抑制该蛋白的化合物。对于腐败希瓦氏菌冷激蛋白Csp1589、Csp1376 和Csp2252,其分别预测到2,2,1 个可能性较高的配体结合位点(图6)。

图6 腐败希瓦氏菌冷激蛋白的互作蛋白及其潜在的配体结合位点Fig.6 Protein and protein interaction and potential ligand binding site of S.putrefaciens CSPs
3 结论
冷激蛋白是解析细菌低温适应的关键蛋白之一。本研究成功扩增了水产品腐败希瓦氏菌的3个冷激蛋白基因,并对其进行生物信息学分析,获得蛋白理化性质、结构和功能方面的相关进展。腐败希瓦氏菌冷激蛋白均为亲水的小分子酸性蛋白质,皆位于细胞质,为非分泌型、非跨膜蛋白,都存在冷激蛋白特有的冷休克结构域,存在DNA 结合位点和RNA 结合基序(motif),能与单链DNA 和单链RNA 结合。腐败希瓦氏菌冷激蛋白Csp1589和Csp1376 与大肠杆菌冷激蛋白CspA 亲缘关系较近,可能在腐败希瓦氏菌低温适应过程中起重要作用。冷激蛋白的二级结构和三级结构均符合冷激蛋白家族的特征,其可靠的三级模型可用于各种对接和药物靶向研究。通过蛋白互作和配体结合位点的分析,对其功能有更深的了解,为后续验证冷激蛋白在腐败希瓦氏菌低温适应过程中的功能,以及水产品中腐败希瓦氏菌冷激蛋白抑制剂的筛选奠定基础。
