利用东乡普通野生稻染色体片段置换系定位产量相关性状QTL
2021-04-27雷丽霞张瑞华金桂秀黎毛毛马小定赵正武韩龙植
罗 兰 雷丽霞 刘 进 张瑞华 金桂秀 崔 迪 黎毛毛 马小定,* 赵正武 韩龙植,*
研究简报
利用东乡普通野生稻染色体片段置换系定位产量相关性状QTL
罗 兰1,**雷丽霞1,**刘 进2,3张瑞华4金桂秀4崔 迪2黎毛毛3马小定2,*赵正武1,*韩龙植2,*
1重庆师范大学, 重庆 401331;2中国农业科学院作物科学研究所/ 农作物基因资源与基因改良国家重大科学工程, 北京 100081;3江西省农业科学院水稻研究所, 江西南昌 330200;4临沂市农业科学院, 山东临沂 276000
以东乡普通野生稻和日本晴为亲本构建的染色体片段置换系为研究材料, 2019年分别在北京、山东临沂和江西南昌对分蘖数、穗粒数和粒形等11个产量相关性状进行多环境鉴定, 结合染色体片段置换系基因型数据定位水稻产量相关性状QTL。3个环境共检测到68个QTL, 包括株高4个、穗长5个、分蘖数2个、一次枝梗数7个、一次枝梗粒数8个、二次枝梗数8个、二次枝梗粒数10个、每穗粒数6个、千粒重7个、粒长8个和粒宽3个; LOD值介于2.50~12.66之间, 贡献率变幅为4.67%~27.79%, 15个QTL的贡献率大于15%; 24个QTL与已报道位点/基因位置重叠, 44个QTL为新发现位点; 6个QTL在2个环境能被检测到, 1个QTL能在3个环境检测到, 且是还未报道的新位点。最后, 利用BSA法验证了、和三个QTL的可靠性。本研究将为后续产量相关性状基因克隆以及进一步解析其遗传基础和分子调控机制奠定基础。
普通野生稻; 染色体片段置换系; 产量相关性状; QTL分析
水稻(L)是世界最重要的粮食作物之一, 全球一半以上的人口以稻米为主食[1]。品种改良对水稻增产具有非常重要的作用, 我国水稻品种改良经历了矮化育种、杂种优势利用和绿色超级稻培育3次飞跃, 产量从160千克增长到1000多千克[2], 但高产仍然是当前和今后相当一段时间内水稻育种的主要目标之一。水稻产量相关性状, 包括株高、有效分蘖数、每穗粒数和千粒重等。株高是株型的重要组成部分, 间接影响水稻产量; 分蘖数直接决定了每株穗数, 是产量的直接影响因子; 穗长、一二次枝梗数以及枝梗着粒数等与每穗粒数密切相关, 是影响产量的直接因素; 粒长、粒宽、粒厚以及籽粒灌浆程度又直接决定了千粒重[3]。
水稻株高、有效分蘖数、穗长、穗粒数和粒形等产量相关性状为多基因控制的数量性状, 易受种植环境影响。由于其重要性, 产量性状的遗传规律解析一直是研究的重点和热点, 特别是近年来分子生物学的发展, 部分性状的分子调控机制得到了很好的解析。例如矮化育种的株高性状由“绿色革命”基因控制,基因突变导致植株变矮[4-5], 从而解决了水稻的倒伏问题。分蘖数主要由调控, 该基因突变的植株只有1个主茎, 没有任何分蘖[6]。此外,[7-8]、[9]、[10]等基因也与水稻分蘖相关。是调控水稻穗粒数的关键基因, 功能获得性突变的基因能引起稻穗变短、直立、着粒密集[11]。此外, 与水稻每穗粒数相关的基因, 还包括[12]、[13]、[14]、[15]等。是水稻粒长和粒重主效QTL, 通过负调控颖壳细胞数目控制水稻粒长[16];是粒宽和粒重主效QTL, 该基因突变能显著增加粒宽和粒重[17];控制水稻千粒重, 来自Kasalath的近等基因系NIL ()能使日本晴增产15%[18]; 此外, 目前已克隆的粒形相关基因, 还包括[19]、[20]、[21]、[22]等。
综上所述, 虽然产量相关性状的遗传规律和分子调控机制研究取得了一些进展, 然而产量相关性状的QTL发掘仍然是重要的研究内容, 而且每隔一段时间依然有新发现。因此, 要想完全解析产量性状的遗传规律, 亟需利用新资源和新群体发掘更多新基因以完善水稻产量性状的调控网络。本研究利用东乡普通野生稻(Griff.)为供体亲本, 日本晴为受体亲本构建的染色体片段置换系为研究材料, 多环境考察, 对株高、分蘖数等产量相关性状进行连锁分析, 以期发掘新位点, 为进一步解析产量相关性状的遗传基础、阐明产量性状的分子调控机制及遗传调控网络奠定基础。
1 材料与方法
1.1 试验材料
以江西东乡普通野生稻为供体亲本、日本晴为受体亲本, 构建的染色体片段置换系(chromosome segment substitution lines, CSSLs)为研究材料, 包括104个家系[23]。2019年将该套材料分别播种于北京、山东临沂和江西南昌3个地点进行多环境表型鉴定。北京于4月26日播种, 临沂于5月20播种, 南昌于5月14日播种, 成苗后按照当地大田种植密度单本插秧, 2行区, 每行15株; 其他按照水稻大田常规方法进行管理。由于东乡普通野生稻生育期长, 3个地点仅南昌可以抽穗。本文仅以日本晴为对照进行表型考察。
1.2 产量相关性状考察
产量相关性状考察在水稻成熟期进行, 为消除边际效应, 每个置换系从第1行的第3株开始连续取5个植株, 考察株高和分蘖数, 从取样的5个植株上, 每株取4个穗,对穗长、一次枝梗数、一次枝梗粒数、二次枝梗数、二次枝梗粒数、每穗粒数、千粒重、粒长和粒宽进行考察, 参照崔国庆等[24]报道的方法测定。
1.3 QTL定位
利用QTL IciMapping (V4.1)[25]软件的RSTEP-LRT- ADD方法进行加性QTL分析, 基因型来自本实验室前期发表的染色体片段置换系的基因型数据[23,26], LOD阈值设定为2.5, 其他参数设置为默认值。
1.4 次级群体构建及BSA分析
QTL验证采用分离群体分组分析法(BSA)。CSSLs与日本晴杂交, 构建F2次级分离群体。2019年冬季种植于海南基地, 表型考察和统计分析后, 将分离群体中30~50个极端表型的单株叶片等量混合, 提取基因组DNA, 组成DNA混池, 然后进行Illumina PE150测序, 测序深度为50×。经过滤, 将有效测序数据通过BWA软件[27]比对到参考基因组(fttp://fttp.ensemblgenomes.org/pub/plants/ release-28/fasta/oryza_sativa/dna/)。采用GATK3.8软件[28]进行多样本SNP检测, 以参考基因组为参考, 分别计算混池的SNP频率(SNP-index), 去掉SNP-index混池中都小于0.3且SNP深度都小于7的位点, 以提高定位效率。为了直观体现混池间差异区域, 计算2个混池SNP-index的差值:D(SNP-index) = SNP-index (混池1)-SNP-index (混池2), 选择1 Mb为窗口, 1 kb为步长, 计算每个窗口中D(SNP-index)的平均值来反应D(SNP-index)分布, 置换检验1000次[29], 最后画出曼哈顿图(Manhattan Plot)显示D(All-index)在染色体上的分布。
2 结果与分析
2.1 CSSL和日本晴产量相关性状表型分析
CSSL群体和日本晴的产量相关性状, 在北京、南昌和临沂3个地点的表型分布如图1和表1所示。3个环境中, 产量相关性状在CSSL群体中均呈现连续分布, 并存在双向超亲分离现象, 说明产量相关性状属于受多基因控制的数量性状。在考察的11个性状中, 变异系数最大的是二次枝梗粒数, 北京、临沂和南昌分别为35.46%、41.80%和36.72%; 变异系数最小的是粒宽, 3个地点分别为3.09%、3.34%和4.64%。11个性状在3个地点表型差异明显, 而且不同地点的变异又各不相同, 综合分析临沂和南昌2个地点的变异要高于北京(表1), 说明产量相关性状的表现易受种植环境的影响。
2.2 产量相关性状QTL定位
3个环境下共检测到68个产量相关性状QTL, 分布于水稻的12条染色体上, LOD值在2.50~12.66之间, QTL贡献率为4.67%~27.79%, 其中15个QTL的贡献率大于15%, 8个QTL贡献率小于8%。6个QTL能在2个环境下同时被检测到, 分别是、、、、和; 1个千粒重QTL-在3个环境下被重复检测到(表2和图2)。具体分析如下:
2.2.1 株高 共检测到4个株高QTL, LOD值在2.63~5.87之间, 贡献率变幅为8.99%~20.74%。位于2号染色体DX-C2-13标记附近, 在北京和南昌2个环境中都能被检测到, 贡献率分别为20.74%和10.90%;和分别位于5号染色体DX-C5-10和8号染色体DX-C8-12标记, 2个QTL仅在临沂环境中被检测到, 贡献率分别为11.80%和8.99%;位于7号染色体indel-c7-1标记处, 仅在北京环境中被检测到, 贡献率为9.86%。
2.2.2 穗长 共检测到穗长QTL 5个, LOD值在2.66~5.48之间, 贡献率变幅为8.06%~17.36%。位于2号染色体02-067标记处, 在临沂和南昌2个环境下均能被检测到, 贡献率分别为12.87%和11.88%;位于1号染色体01-009处,位于2号染色体DX-C2-13标记处,位于10号染色体DX-S10-1标记处, 3个QTL仅在北京环境下被检测到, 贡献率分别为9.17%、17.36%和8.06%;位于5号染色体05-028标记处, 仅在南昌环境中被检测到, 贡献率为10.42%。
2.2.3 分蘖数 检测到的有效分蘖数QTL 2个, LOD值在3.03~3.89之间。和分别位于1号染色体DX-C1-2和010-060标记处, 2个QTL均仅在临沂环境下被检测到, 贡献率分别为10.96%和13.32%。
2.2.4 一次枝梗数 共检测到7个一次枝梗数QTL, LOD值介于2.68~7.98之间, QTL贡献率在6.79%~18.54%范围内。和位于1号染色体DX-C1-6和DX-C1-10标记处, 仅在临沂环境下被检测到, 贡献率分别为9.25%和8.34%;、和分别位于3号染色体DX-C3-8、5号染色体DX-C5-3和8号染色体DX-S8-14标记处, 仅在北京环境下被检测到, 贡献率分别为9.86%、11.23%和18.54%;和分别位于8号染色体DX-C8-11和DX-C8-12标记处, 仅在南昌环境下被检测到, 贡献率分别为6.79%和13.99%。
2.2.5 一次枝梗粒数 共检测到8个一次枝梗粒数QTL, LOD值在2.61~9.27之间, 贡献率变幅为8.18%~17.32%。、、、和分别位于1号染色体DX-C1-3、DX-C1-10、3号染色体DX-C3-28、8号染色体DX-S8-14和11号染色体DX-S11-10标记处, 仅在北京环境下能检测到, 贡献率分别为9.85%、12.29%、8.18%、9.33%和9.86%;仅在临沂环境下能被检测到, 位于1号染色体01-046标记处, 贡献率为17.32%; 仅在南昌环境下被检测到的有和, 分别位于8号染色体DX-C8-11和DX-C8-12标记处, 贡献率分别为7.45%和16.35%。
2.2.6 二次枝梗数 共检测到8个二次枝梗数QTL, LOD值在3.09~9.46之间, 贡献率范围为6.71%~23.99%。、和分别位于1号染色体DX-C1-7、2号染色体DX-C2-1和4号染色体S4-12-2标记处, 仅在临沂环境下能检测到, 贡献率分别为8.86%、15.83%和15.75%;、、和分别位于2号染色体DX-C2-2、02-057、4号染色体DX-C4-12和6号染色体S6-9-1标记处, 仅在北京环境下能检测到, 贡献率分别为23.99%、12.01%、8.10%和6.71%;仅在南昌环境下能被检测到, 位于6号染色体DX-C6-2标记处, 贡献率为13.08%。
2.2.7 二次枝梗粒数 共检测到二次枝梗粒数QTL 10个, LOD值在2.68~12.66之间, 贡献率变幅为4.67%~27.79%。、、和分别位于2号染色体DX-C2-1、3号染色体DX-S3-16、4号染色体DX-C4-2和DX-C4-8标记处, 仅在临沂环境下能检测到, 贡献率分别为27.79%、6.99%、9.03%和4.67%;、、和分别位于2号染色体DX-C2-2、2号染色体02-057、4号染色体DX-C4-12和6号染色体S6-9-1标记处, 仅在北京环境下能检测到, 贡献率分别为21.39%、9.79%、10.55%和9.47%;和仅在南昌环境下能被检测到, 分别位于3号染色体DX-S3-17和6号染色体DX-C6-2标记处, 贡献率分别为15.34%和10.56%。
2.2.8 每穗粒数 共检测到6个每穗粒数QTL, LOD值在3.08~5.14之间, 贡献率变幅为9.96%~17.43%。、和分别位于1号染色体01-046、2号染色体DX-C2-1和4号染色体S4-12-2标记处, 仅在临沂环境下能检测到, 贡献率分别为13.92%、14.43%和12.58%;位于6号染色体06-013标记处, 仅在南昌环境下能检测到, 贡献率分别为13.45%;和仅在北京环境下能被检测到, 分别位于6号染色体06-037和11号染色体DX-S11-10标记处, 贡献率分别为17.43%和9.96%。
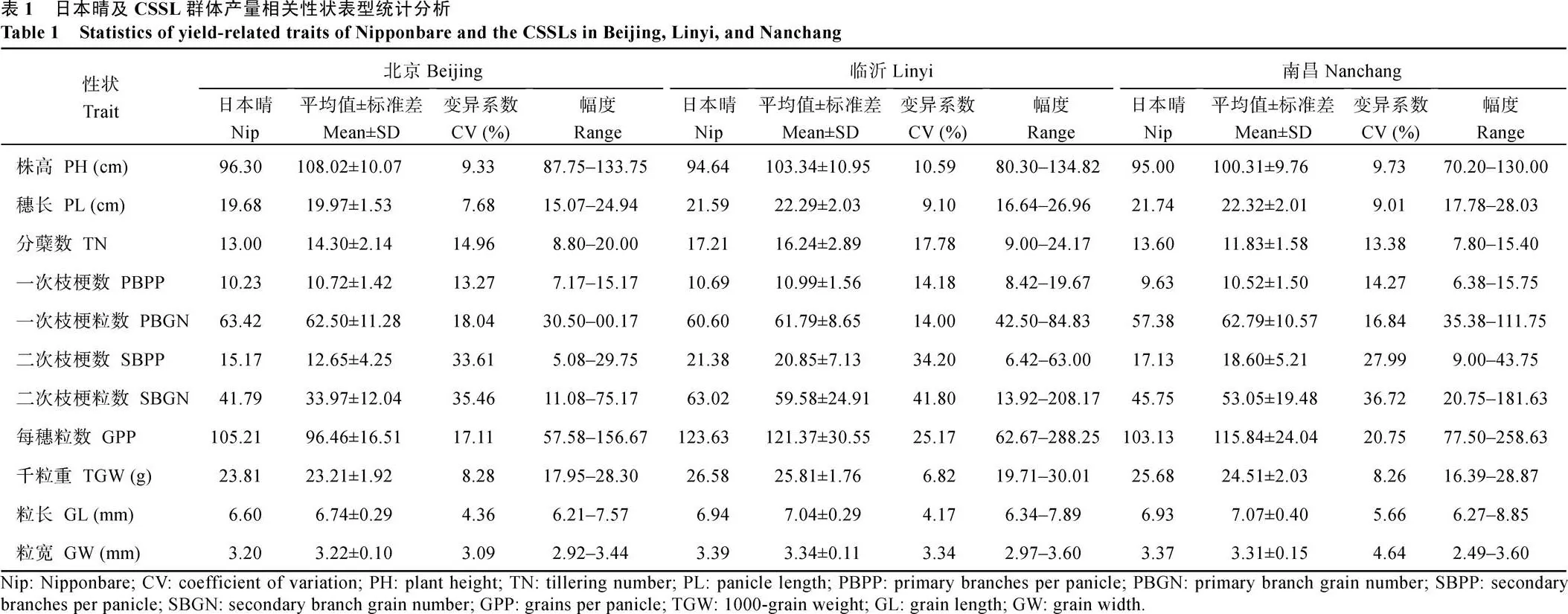
图1 CSSL群体中11个产量相关性状的频率分布
Fig. 1 Frequency distribution of 11 yield-related traits in the CSSLs
纵坐标表示CSSL数量。蓝色、红色和绿色长柱分别代表CSSL中11个性状在北京、临沂和南昌的频率分布。蓝色、红色和绿色箭头表示日本晴的11个性状在北京、临沂和南昌的表现。
The vertical axis of each figure represents the number of CSSL individuals. The blue, red and green rectangles represent the distribution of eleven yield-related traits in the CSSLs at the Beijing, Linyi and Nanchang locations, respectively. The blue, red, and green triangles represent the positions of the means of Nipponbare in Beijing, Linyi, and Nanchang, respectively.
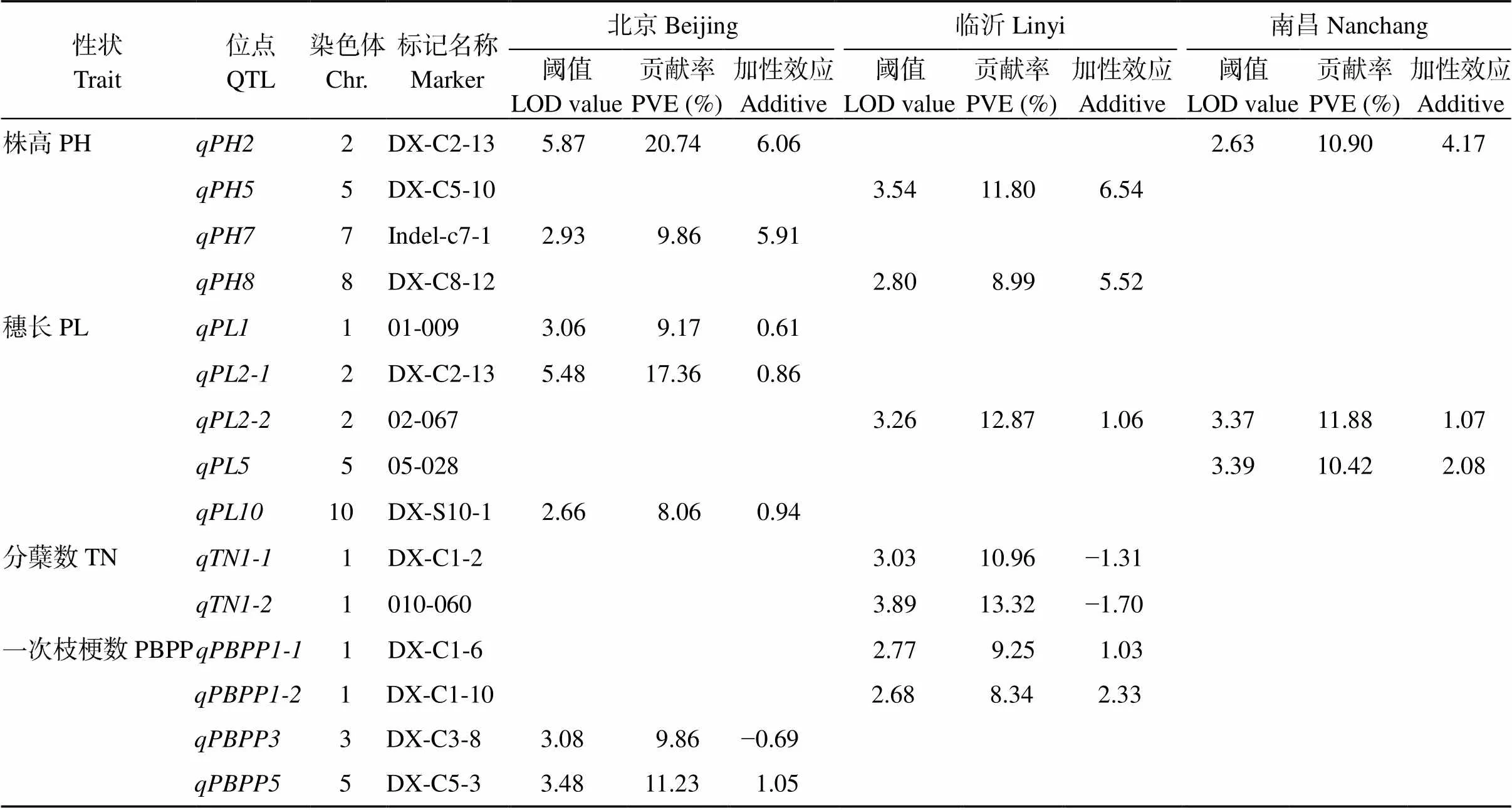
表2 北京、临沂和南昌3地产量相关性状QTL分析

(续表2)
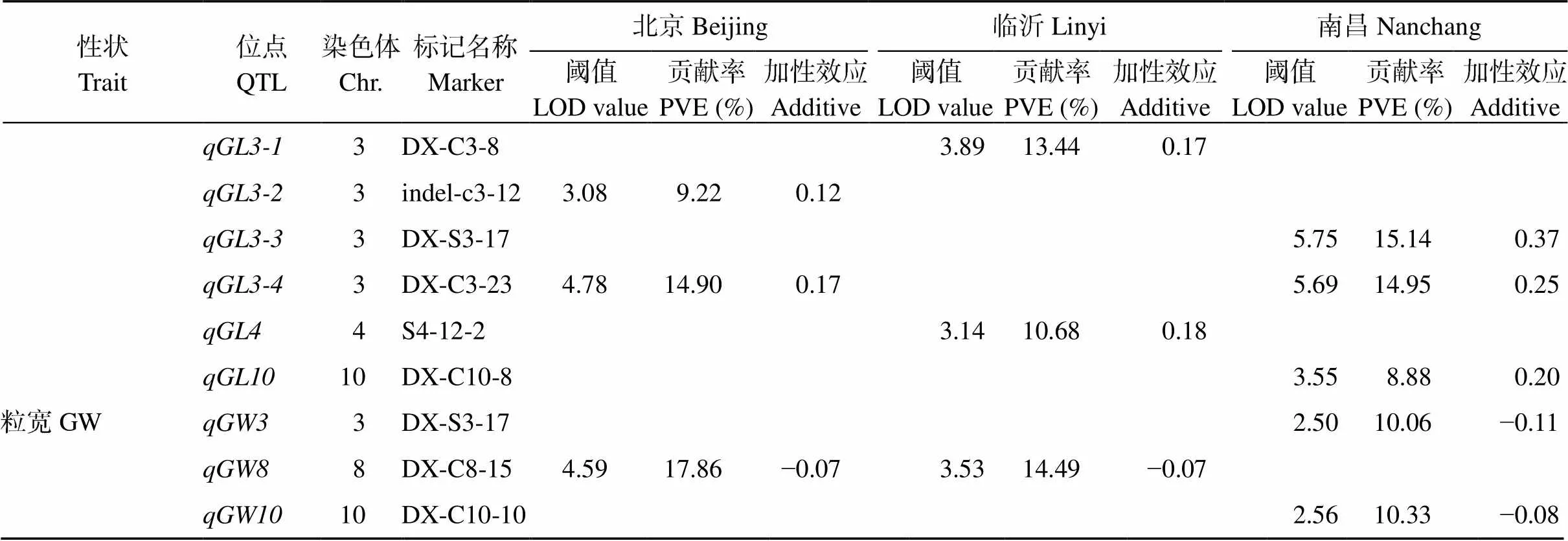
(续表2)
正值表示增效等位基因来自东乡普通野生稻, 负值表示增效等位基因来自日本晴。各性状缩写同表1。
Positive values indicate that the positive alleles come from Dongxiang common wild rice. Negative values indicate that the positive alleles come from Nipponbare. Abbreviations of traits are the same as those given in Table 1. PVE: percentage of total phenotypic variance explained by the QTLs.
检测到的QTL具体信息如表2所示。每个标记的物理位置显示在染色体的左侧, 标记名称显示在染色体的右侧。绿色字符表示在2个地点都能检测到的QTL, 蓝色表示在3个地点都能检测到的QTL。
The definitions of the abbreviations for the symbols representing the QTLs are listed in Table 2. The position of each marker is based on the physical distance shown to the left of each chromosome, and the molecular marker is shown on the right. The green characters represent the names of the QTLs detected at two sites, and the blue characters represent the name of the QTL detected at three sites.
2.2.9 千粒重 共检测到7个千粒重QTL, LOD值在2.90~5.28之间, 贡献率变幅为7.81%~14.36%。位于2号染色体02-008标记处, 在北京、临沂和南昌3个环境中都能被检测到, 贡献率分别为14.36%、9.19%和10.96%;位于8号染色体DX-S8-14标记处, 在临沂和南昌2个环境中能被同时检测到, 贡献率分别为8.54%和7.86%;位于3号染色体DX-C3-1标记处, 仅在北京环境下检测到, 贡献率为10.64%;和分别位于8号染色体DX-C8-3和9号染色体DX-C9-4标记处, 仅在临沂环境中被检测到, 贡献率分别为11.68%和8.11%;和分别位于9号染色体DX-C9-10和12号染色体S12-6-3标记处, 仅在南昌环境中被检测到, 贡献率分别为7.81%和14.11%。
2.2.10 粒长 共检测到8个粒长QTL, LOD值在2.86~5.75之间, 贡献率变幅为7.12%~16.65%。位于2号染色体02-067标记处, 在临沂和南昌2个环境中都能被检测到, 贡献率分别为10.71%和7.12%;位于3号染色体DX-C3-23标记处, 在北京和南昌2个环境中都能被检测到, 贡献率分别为14.90%和14.95%;和分别位于1号染色体DX-C1-18和3号染色体indel-c3-12标记处, 仅在北京环境中被检测到, 贡献率分别为16.65%和9.22%;和仅在临沂环境中被检测到, 分别位于3号染色体DX-C3-8和4号染色体S4-12-2标记处, 贡献率分别为13.44%和10.68%; 在南昌环境下能够被检测到QTL有和, 分别位于3号染色体DX-S3-17和10号染色体DX-C10-8标记处, 贡献率分别为15.14%和8.88%。
2.2.11 粒宽 共检测到3个粒宽QTL, LOD值在2.50~4.59之间, 贡献率介于10.06%~17.86%之间。位于8号染色体DX-C8-15标记处, 在北京和临沂2个环境中都能被检测到, 贡献率分别为17.86%和14.49%;3和分别位于3号染色体DX-S3-17和10号染色体DX-C10-10标记处, 仅在南昌环境下能够被检测到, 贡献率为10.06%和10.33%
2.3 QTL验证
利用已构建的次级分离群体, 对株高QTL、一次枝梗数QTL和粒宽QTL三个位点进行了验证。
将含有置换系L13、置换系L78和置换系L90分别与日本晴杂交, 构建F2次级分离群体。分别考察了相应群体530个单株的株高、402个单株的一次枝梗数和198个单株的粒宽, 发现3个性状在分离群体中均呈连续分布(图3-A, C, E)。通过高通量测序, 极端矮株混池获得188,787,238个Reads, 极端高株混池获得190,156,356个Reads, 检测到602,546个SNP,D(All-index)在染色体上的分布如图3-B; 在5号染色体的23,053,001~25,260,000区间、6号染色体的7,388,001~20,383,000区间和7号染色体的1,727,001~3,832,000区间有3个明显的QTL,处于7号染色体区间内。极端多一次枝梗混池获得217,405,862个Reads, 极端少一次枝梗混池获得230,822,574个Reads, 检测到221,953个SNP,D(All-index)在染色体上的分布如图3-D; 在7号染色体的1~1,552,000区间、8号染色体的201,001~2,764,000区间以及18,259,001~25,357,000区间有3个明显的QTL,在8号染色体18,259,001~25,357,000区间内。极端窄粒混池获得200,257,918个Reads, 极端宽粒混池获得205,590,678个Reads, 检测到166,301个SNP,D(All-index)在染色体上的分布如图3-F; 在1号染色体的4,069,001~4,803,000区间、5号染色体的2,584,001~5,802,000区间以及10号染色体17,095,001~23,206,000区间有3个明显的QTL,在10号染色体区间内。因此,、和三个QTL是真实存在的。
3 讨论
普通野生稻是亚洲栽培稻的近缘野生种, 在长期的进化过程中, 保留了栽培稻丢失的高产优质、抗病虫、耐逆和营养高效利用等有利基因和优异单倍型, 但一些不利性状往往和有利性状紧密连锁, 限制了野生稻的利用效率, 构建普通野生稻染色体片段置换系在一定程度上可以解决这个难题。近年来, 我国水稻、特别是北方粳稻产量一直徘徊不前, 利用普通野生稻丰富的遗传多样性, 拓宽育种亲本的遗传基础, 是改良现有品种、提高水稻产量的有效途径。本实验室前期针对粳稻育种材料, 利用东乡普通野生稻为供体亲本, 日本晴为受体亲本, 构建了一套CSSL[23]。本研究在此基础上, 2019年分别在北京、临沂和南昌种植该CSSL, 考察了11个与产量相关的性状。统计分析发现, 11个性状都存在超亲分离现象(图1和表1), 而且, 有些如落粒、长芒、高植株等野生稻不利性状与有利性状连锁已打破(数据没有提供), 可以直接选择一些家系供改良粳稻育种亲本利用。
CSSL不仅可以为拓宽育种亲本的遗传基础提供基因资源, 而且还是定位与克隆重要数量性状基因的理想群体, 特别是对微效QTL的发掘具有良好的效果。本研究在3地对11个产量相关性状进行了表型鉴定, 结合CSSL分子标记数据, 共检测到68个QTL, QTL贡献率介于4.67%~27.79%, 贡献率最大的是二次枝梗粒数QTL, 贡献率为27.79%; 贡献率最小的是二次枝梗粒数QTL, 贡献率仅有4.69% (表2)。检测到的一二次枝梗数、枝梗粒数、每穗粒数及粒形等驯化相关性状QTL数量较多, 也说明普通野生稻在这些性状上具有更为丰富的遗传多样性, 可以为这些性状相关基因的克隆和性状改良提供多样的基因资源。
A:位点次级分离群体中株高的频率分布; B: 株高极端混池∆(SNP-index)曼哈顿图; C:位点次级分离群体中一次枝梗数的频率分布; D: 一次枝梗数极端混池∆(SNP-index)曼哈顿图; E:位点次级分离群体中粒宽的频率分布; F: 粒宽极端混池∆(SNP-index)曼哈顿图。蓝色和红色箭头分别表示日本晴和对应染色体片段置换系3个性状的表现。
A: frequency distribution of plant height in secondary segregated population related of; B: ∆(SNP-index) graph of plant height extreme mixed pools; C: frequency distribution of primary branches per panicle in secondary segregated population related of; D: ∆(SNP-index) graph of primary branches per panicle extreme mixed pools; E: frequency distribution of grain width in secondary segregated population related of; F: ∆(SNP-index) graph of grain width extreme mixed pools. The blue and red triangles represent the positions of the means of Nipponbare and its related CSSL, respectively.
环境显著影响数量性状QTL定位的数量和质量, 特别是产量相关性状的表现受环境影响更大。本研究在北京、临沂和南昌3地种植CSSL群体和亲本日本晴, 各地表型差异较大, 即使是高遗传力的粒长和粒宽性状也差异显著(图1), 不可避免地影响到定位QTL的可重复性。检测到的68个QTL中, 在北京环境下检测到的有29个, 临沂26个, 南昌21个; 6个QTL能在2个环境下被重复检测到, 仅有1个QTL在3个环境下被重复检测到。利用前期已构建的F2次级分离群体采用BSA法分别对株高、一次枝梗数和粒宽三个位点进行了验证, 虽然结果显示相应的3个分离群体的株高、一次枝梗数和粒宽均呈连续分布, 受多个基因控制, 但是本文检测的3个QTL在BSA分析中都能重现。因此, 一些在单环境中检测到的QTL可能也是真实可靠的(图3)。68个QTL中, 有24个QTL与已报道的QTL/基因在染色体上的位置相同或邻近。株高QTL和穗长QTL与邻近[30],与邻近[31-32]。一次枝梗粒数QTL与邻近[12]; 二次枝梗数QTL和二次枝梗粒数QTL与邻近[33]; 每穗粒数QTL1与、和与区间重叠[34-35]。在粒形相关QTL中, 本研究中的相关QTL大多与已克隆的粒形基因位置重叠, 例如与[36]、与/[37-39]、与[40]、和与[16]、与[41]、与[42]以及和与[20]。此外, 穗长QTL、一次枝梗数QTL和一次枝梗粒数QTL二次枝梗数QTL和每穗粒数QTL分别与粒形基因/、和位置相同或者邻近。除了24个QTL与已报道的位点/基因位置相近外, 其余44个为本研究新发现位点。
[1] 郭韬, 余泓, 邱杰, 李家洋, 韩斌, 林鸿宣. 中国水稻遗传学研究进展与分子设计育种. 中国科学: 生命科学, 2019, 49: 1185–1212. Guo T, Yu H, Qiu J, Li J Y, Han B, Lin H X. Advances in rice genetics and breeding by molecular design in China.(Vitae), 2019, 49: 1185–1212 (in Chinese).
[2] 吴比, 胡伟, 邢永忠. 中国水稻遗传育种历程与展望. 遗传, 2018, 40: 841–857. Wu B, Hu W, Xing Y Z. The history and prospect of rice genetic breeding in China., 2018, 40: 841–857 (in Chinese with English abstract).
[3] Yan S, Zou G H, Li S J, Wang H, Liu H Q, Zhai G W, Guo P, Song H M, Yan C J, Tao Y Z. Seed size is determined by the combinations of the genes controlling different seed characteristics in rice., 2011, 123: 1173–1181.
[4] Monna L, Kitazawa N, Yoshino R, Lisa M, Suzuki J, Masuda H, Maehara Y, Tanji M, Sato M, Nasu S, Minobe Y. Positional cloning of rice semidwarfing gene,: rice “green revolution gene” encodes a mutant enzyme involved in gibberellin synthesis., 2002, 9: 11–17.
[5] Spielmeyer W, Ellis M H, Chandler P M. Semidwarf (), “green revolution” rice, contains a defective gibberellin 20-oxidase gene., 2002, 99: 9043–9048.
[6] Li X Y, Qian Q, Fu Z M, Wang Y H, Xiong G S, Zeng D L, Wang X Q, Liu X F, Teng S, Hiroshi F, Yuan M, Luo D, Han B, Li J Y. Control of tillering in rice., 2003, 422: 618–621.
[7] Zhou F, Lin Q B, Zhu L H, Ren Y L, Zhou K N, Shabek N, Wu F Q, Mao H B, Dong W, Gan L, Ma W W, Gao H, Chen J, Yang C, Wang D, Tan J J, Zhang X, Guo X P, Wang J L, Jiang L, Liu X, Chen W Q, Chu J F, Yan C Y, Ueno K, Ito S, Asami T, Cheng Z J, Wang J, Lei C L, Zhai H Q, Wu C Y, Wang H Y, Zheng N, Wan J M. D14–SCFD3-dependent degradation ofregulates strigolactone signaling., 2013, 504: 406–410.
[8] Jiang L, Liu X, Xiong G S, Liu H H, Chen F L, Wang L, Meng X B, Liu G F, Yu H, Yuan Y D, Yi W, Zhao L H, Ma H L, He Y Z, Wu Z S, Melcher K, Qian Q, Xu H. E, Wang Y H, Li J Y. DWARF 53 acts as a repressor of strigolactone signaling in rice., 2013, 504: 401–405.
[9] Ishikawa S, Maekawa M, Arite T, Onishi K, Takamure I, Kyozuka J. Suppression of tiller bud activity in tillering dwarf mutants of rice., 2005, 46: 79–86.
[10] Zou J H, Chen Z X, Zhang S Y, Zhang W P, Jiang G H, Zhao X F, Zhai W X, Pan X B, Zhu L H. Characterizations and fine mapping of a mutant gene for high tillering and dwarf in rice (L.)., 2005, 222: 604–612.
[11] Huang X, Qian Q, Liu Z, Liu Z B, Sun H Y, He S Y, Luo D, Xia G M, Chu C C, Li J Y, Fu X D. Natural variation at thelocus enhances grain yield in rice., 2009, 41: 494–497.
[12] Ashikari M, Sakakibara H, Lin S Y, Yamamoto T, Takashi T, Nishimura A, R. Angeles E, Qian Q, Kitano H, Matsuoka M. Cytokinin oxidase regulates rice grain production., 2005, 309: 741–745.
[13] Wu Y, Wang Y, Mi X F, Shan J X, Li X M, Xu J L, Lin H X. The QTLencodes GA20ox1, which increases grain number and yield by increasing cytokinin activity in rice panicle meristems., 2016, 12: e1006386.
[14] Jin J, Hua L, Zhu Z, Tan L B, Zhao X H, Zhang W F, Liu F X, Fu Y C, Cai H W, Sun X Y, Gua P, Xie D X, Sun C Q.encodes a secreted peptide that regulates grain number, grain length, and awn development in rice domestication., 2016, 28: 2453–2463.
[15] Huo X, Wu S, Zhu Z, Liu F X, Fu Y C, Cai H W, Sun X Y, Gu P, Xie D X, Tan L B, Sun C Q.increases grain production in rice., 2017, 8: 1497.
[16] Fan C C, Yu S B, Wang C R, Xing Y Z. A causal C–A mutation in the second exon ofhighly associated with rice grain length and validated as a functional marker., 2009, 118: 465–472.
[17] Song X J, Huang W, Shi M, Zhu M Z, Lin H X. A QTL for rice grain width and weight encodes a previously unknown RING-type E3 ubiquitin ligase., 2007, 39: 623–630.
[18] Ishimaru K, Hirotsu N, Madoka Y, Murakami N, Hara N, Onodera H, Kashiwagi T, Ujiie K, Shimizu B, Onishi A, Miyagawa H, Katoh E. Loss of function of the IAA-glucose hydrolase geneenhances rice grain weight and increases yield., 2013, 45: 707–711.
[19] Li Y B, Fan C C, Xing Y Z, Jiang Y H, Luo L J, Sun L, Shao D, Xu C J, Li X H, Xiao J H, He Y Q, Zhang Q F. Natural variation inplays an important role in regulating grain size and yield in rice., 2011, 43: 1266–1269.
[20] Wang S K, Wu K, Yuan Q B, Liu X, Liu Z B, Lin X Y, Zeng R Z, Zhu H T, Dong G J, Qian Q Zhang G Q, Fu X D. Control of grain size, shape and quality byin rice., 2012, 44: 950–954.
[21] Wang Y X, Xiong G S, Hu J, Jiang L, Yu H, Xu J, Fang Y X, Zeng L J, Xu E, Xu J, Ye W J, Meng X B, Liu R F, Chen H Q, Jing Y H, Wang Y H, Zhu X D, Li J Y, Qian Q. Copy number variation at thelocus contributes to grain size diversity in rice., 2015, 47: 944–948.
[22] Zhao D S, Li Q F, Zhang C Q, Zhang C, Yang Q Q, Pan L X, Ren X Y, Lu J, Gu M H, Liu Q Q.acts as a transcriptional activator to regulate rice grain shape and appearance quality., 2018, 9: 1240.
[23] Ma X D, Han B, Tang J H, Zhang J N, Cui D, Geng L Y, Zhou H Y, Li M M, Han L Z. Construction of chromosome segment substitution lines of Dongxiang common wild rice (Griff) in the background of the japonica rice cultivar Nipponbare (L.)., 2019, 144: 274–282.
[24] 崔国庆, 王世明, 马福盈, 汪会, 向朝中, 李云峰, 何光华, 张长伟, 杨正林, 凌英华, 赵芳明. 水稻高秆染色体片段代换系Z1377的鉴定及重要农艺性状QTL定位. 作物学报, 2018, 44: 1477–1484. Cui G Q, Wang S M, Ma F Y, Wang H, Xiang C Z, Li Y F, He G H, Zhang C W, Yang Z L, Ling Y H, Zhao F M. Identification of rice chromosome segment substitution line Z1377 with increased plant height and QTL mapping for agronomic important traits., 2018, 44: 1477–1484 (in Chinese with English abstract).
[25] Wang J K, Wan X Y, Crossa J, Crouch J, Weng J F, Zhai H Q, Wan J M. QTL mapping of grain length in rice (L.) using chromosome segment substitution lines., 2006, 88: 93–104.
[26] 马小定, 唐江红, 张佳妮, 崔迪, 李慧, 黎毛毛, 韩龙植. 东乡野生稻与日本晴多态性标记的开发. 作物学报, 2019, 45: 316–321. Ma X D, Tang J H, Zhang J N, Cui D, Li H, Li M M, Han L Z. Development of molecular markers polymorphic between Dongxiang wild rice andrice cultivar ‘Nipponbare’., 2019, 45: 316–321 (in Chinese with English abstract).
[27] Li H, Durbin R. Fast and accurate short read alignment with Burrows-Wheeler transform., 2009, 25: 1754–1760.
[28] McKenna A, Hanna M, Banks E, Sivachenko A, Cibulskis K, Kernytsky A, Garimella K, Altshuler D, Gabriel S, Daly M, Depristo M A. The Genome Analysis Toolkit: a MapReduce framework for analyzing next-generation DNA sequencing data., 2010, 20: 1297–1303.
[29] Takagi H, Abe A, Yoshida K, Kosugi S, Natsume S, Mitsuoka C, Uemura A, Utsushi H, Tamiru M, Takuno S, Innan H, Cano L M, Kamoun S, Terauchi R. QTL-seq: rapid mapping of quantitative trait loci in rice by whole genome resequencing of DNA from two bulked populations., 2013, 74: 174–183.
[30] Liu J H, Shen J Q, Xu Y, Li X H, Xiao J H, Xiong L Z., a CONSTANS-like gene, confers drought sensitivity through regulation of senescence in rice., 2016, 67: 5785–5798.
[31] 王仁晓, 李培金, 陈红旗, 闵绍楷, 李家洋, 朱旭东. 水稻顶节间长度控制基因()的精细定位. 遗传学报, 2005, 32: 955–959.Wang R X, Li P J, Chen H Q, Yan S K, Li J Y, Zhu X D. Fine localization of ricegene controlling elongation of the uppermost internode., 2005, 32: 955–959 (in Chinese with English abstract).
[32] Zhang Y Y, Zhu Y Y, Peng Y, Yan D W, Li Q, Wang J J, Wang L Y, He Z H. Gibberellin homeostasis and plant height control byand a role for gibberellin in root gravity responses in rice., 2008, 18: 412–421.
[33] Zha X J, Luo X J, Qian X Y, He G M, Yang M F, Li Y, Yang J S. Over-expression of the ricegene improves quantitative yield components., 2009, 7: 611–620.
[34] Moncada P, Martínez C P, Borrero J, Chatel M, Gauch Jr H, Guimaraes E, Tohme J, McCouch S R. Quantitative trait loci for yield and yield components in an×BC2F2population evaluated in an upland environment., 2001, 102: 41–52.
[35] Thomson M J, Tai T H, McClung A M, Lai X H, Hinga M E, Lobos K B, Xu Y, Martinez C P, McCouch S R. Mapping quantitative trait loci for yield, yield components and morphological traits in an advanced backcross population betweenand thecultivar Jefferson., 2003, 107: 479–493.
[36] Suh J P, Ahn S N, Cho Y C, Kang K H, Choi I S, Kim Y G, Suh H S, Hwang H G. Mapping of QTLs for yield traits using an advanced backcross population from a cross betweenand., 2005, 37: 214–220.
[37] Che R H, Tong H N, Shi B H, Liu Y Q, Fang S R, Liu D P, Xiao Y H, Hu B, Liu L C, Wang H R, Zhao M F, Chu C C. Control of grain size and rice yield by-mediated Brassinosteroid responses., 2015, 2: 15195.
[38] Duan P G, Ni S, Wang J M, Zhang B L, Xu R, Wang Y X, Chen H Q, Zhu X D, Li Y H. Regulation ofby OsmiR396 controls grain size and yield in rice., 2015, 2: 15203.
[39] Sun P Y, Zhang W H, Wang Y H, He Q, Shu F, Liu H, Wang J, Wang J M, Yuan L P, Deng H F.controls grain shape, panicle length and seed shattering in rice., 2016, 58: 836–847.
[40] Yu J P, Xiong H Y, Zhu X Y, Zhang H L, Li H H, Miao J L, Wang W S, Tang Z S, Zhang Z Y, Yao G X, Zhang Q, Pan Y H, Wang X, Rashid M A. R, Li J J, Gao Y M, Li Z K, Yang W C, Fu X D, Li Z C.contributing to rice grain length and yield was mined by Ho-LAMap., 2017, 15: 28.
[41] Zhang X J, Wang J F, Huang J, Lan H X, Wang C L, Yin C F, Wu Y Y, Tang H J, Qian Q, Li J Y, Zhang H S. Rare allele ofassociated with grain length causes extra-large grain and a significant yield increase in rice., 2012, 109: 21534–21539.
[42] Guo T, Chen K, Dong N Q, Shi C L, Ye W W, Gao J P, Shan J X, Lin H X. GRAIN SIZE AND NUMBER1 negatively regulates the OsMKKK10-OsMKK4-OsMPK6 cascade to coordinate the trade-off between grain number per panicle and grain size in rice., 2018, 30: 871–888.
Mapping QTLs for yield-related traits using chromosome segment substitution lines of Dongxiang common wild rice (Griff.) and Nipponbare (L.)
LUO Lan1,**, LEI Li-Xia1,**, LIU Jin2,3, ZHANG Rui-Hua4, JIN Gui-Xiu4, CUI Di2, LI Mao-Mao3, MA Xiao-Ding2,*, ZHAO Zheng-Wu1,*, and HAN Long-Zhi2,*
1Chongqing Normal University, Chongqing 401331, China;2National Key Facility for Crop Gene Resources and Genetic Improvement / Institute of Crop Sciences, Chinese Academy of Agricultural Sciences, Beijing 100081, China;3Rice Research Institute, Jiangxi Academy of Agricultural Sciences, Nanchang 330200, Jiangxi, China;4Linyi Academy of Agricultural Sciences, Linyi 276000, Shandong, China
In former study, we constructed a chromosome segment substitution lines (CSSLs) of Dongxiang common wild rice (Griff.) in the background of Nipponbare (L.). In this study, in order to investigate 11 yield-related traits, such as tillering number, grains per panicle and grain shapes, the CSSLs were planted in Beijing, Linyi and Nanchang. The results of quantitative trait loci (QTLs) for yield-related traits showed that a total of 68 QTLs were detected, including 4 QTLs for plant height, 5 QTLs for panicle length, 2 QTLs for tillering number, 7 QTLs for primary branch grain number, 8 QTLs for primary branch grain number, 8 QTLs for secondary branch grain number, 10 QTLs for secondary branch grain number, 6 QTLs for grains per panicle, 7 QTLs for 1000-grain weight, 8 QTLs for grain length and 3 QTLs for grain width. LOD score of the detected QTLs ranged from 2.50 to 12.66. The phenotypic variation explained by these QTLs ranged from 4.67% to 27.79%. There were 15 QTLs with a contribution rate more than 15%, 24 QTLs overlapped with the reported loci or gene position, 44 QTLs newly detected loci. In addition, 6 QTLs were stably detected at two sites, and 1 QTL () as a novel QTL was detected at three sites. Finally, the reliability of the three QTLs of,andwas verified by BSA. Our results will be helpful for the subsequent cloning of yield-related trait genes and further analysis of their genetic basis and molecular regulation mechanism.
common wild rice (Griff.); chromosome segment substitution lines (CSSLs); yield-related traits; QTL analysis
10.3724/SP.J.1006.2021.02054
本研究由国家重点研发计划项目(2016YFD0100101, 2016YFD0100301), 国家自然科学基金项目(31671664), 中国农业科学院科技创新工程项目, 国家农作物种质资源保护项目(2018NWB036-01, 2018NWB036-12-2)和国家农作物种质资源平台项目(NICGR2018-001)资助。
This study was supported by the National Key Research and Development Program of China (2016YFD0100101, 2016YFD0100301), the National Natural Science Foundation of China (31671664), the CAAS Science and Technology Innovation Program, Protective Program of Crop Germplasm of China (2018NWB036-01, 2018NWB036-12-2), and the National Infrastructure for Crop Germplasm Resources (NICGR2018-001).
韩龙植, E-mail: hanlongzhi@caas.cn; 赵正武, E-mail: zhaozhengwu513@sina.com; 马小定, E-mail: maxiaoding@caas.cn
**同等贡献(Contributed equally to this work)
罗兰, E-mail: 1366730598@qq.com; 雷丽霞, E-mail: 2873804227@qq.com
2020-08-13;
2020-11-13;
2020-12-29.
URL: https://kns.cnki.net/kcms/detail/11.1809.S.20201229.1139.002.html
