mRNA序列与相应内含子序列匹配的普适性分析
2020-11-02赵小庆薄素玲曹艳娟苏文霞
张 强,赵小庆,薄素玲,曹艳娟 ,苏文霞
(1.内蒙古农业大学 理学院,呼和浩特 010018; 2.内蒙古医科大学 计算机信息学院, 呼和浩特 010110;3.内蒙古自治区农牧业科学院,呼和浩特 010031)
近几年,诸多研究都开始重视内含子对基因表达的影响[1]。大量研究表明,内含子是一类具有生物学功能的序列。许多基因表达调控元件,例如基因转录和mRNA加工(尤其是可变剪接),都属于内含子序列的一部分。各种非编码RNA,例如microRNA和snoRNA,也属于内含子[2]。对内含子序列而言,它的丢失和获得对非编码RNA变异和基因重组有影响,这是关系到真核基因进化的主要方面[3-7]。许多疾病的发生是由于内含子的突变造成的[8-9]。内含子两端和中间序列的突变,都是因为激活隐性切割位点进而导致的疾病。虽然部分基因的表达并不需要内含子序列的参与甚至不存在内含子,然而内含子在许多情况下能够最大限度地增强转基因生物的基因表达[10-11]。内含子序列已经成为对转基因生物中外源基因表达进行改善不可或缺的一部分[12-15]。以上所有情况间接表明内含子序列的存在与否对基因表达有显著差别。同时,在pre-mRNA上,所有剪接过程的发生,都可以看作内含子和外显子序列相互作用后,所产生的必然结果。序列匹配是体现上述相互作用最基本的形式[16-19]。那么必定只能由更加复杂的序列结构才能组成内含子序列并执行多种生物功能。比如,内含子通过与mRNA的序列匹配,调节结合蛋白因子与mRNA的相互作用,才能进行调节[20-22]。在mRNA序列上存在功能区域,例如翻译起始与终止位点,还存在与内含子匹配的特殊形式,这些形式对基因的表达调控具有至关重要的作用[23]。因此,寻找mRNA序列与相应内含子的最佳匹配区域是解决问题的思路。
基于此思路,以13个生物基因的编码序列作为研究样本,目的在于找到mRNA序列上,能够与内含子序列存在相互作用关系的最佳匹配片段,进而分析这些片段的序列特征,讨论在mRNA序列上匹配频率的分布特点,研究该分布在不同物种中的进化规律。
1 数据与方法
1.1 基因序列数据
13个物种1号染色体编码基因序列取自The Exon-Intron Database (EID)数据库。13个物种中包括4个植物,2个无脊椎动物和7个有脊椎动物。在选取样本(见表1)中去掉了含有重复元件和ncRNA等已知非剪接功能的基因。
1.2 比对方法
mRNA序列与相应内含子序列存在碱基互补匹配片段,该片段能够反映它们之间的相互特点。因此它们之间的相互作用可由最佳匹配片段表征出来。首先把内含子进行转化,得到它的互补序列,其次通过软件Smith-Waterman,对其进行局部相似性比对,再次比对互补序列与其所对应的mRNA序列,进而找出最佳相似片段,最后通过转化得到两者最佳匹配片段。
1.3 最佳匹配频数分布
定义1:匹配打分函数
对给定序列的每个碱基位点赋予一个分值,在最佳匹配区域内,则碱基位点赋值为1;如果不在最佳匹配区域内,则赋碱基位点值为0。定义匹配打分函数如下:
(1)
公式中,i代表第i条序列,j是第i条序列中,第j个碱基位点(j=1,2,…,Li);那么Li是第i条序列的长度,则Nis和Nie分别是第i条序列上最佳匹配片段的起始和终止碱基位点。这样一条序列就转换成由0和1组成的数字串,1代表最佳匹配片段位置。

表1 13种真核生物基因Table 1 Protein coding genes of 13 eukaryotes
定义2:匹配频率函数
在所分析的m条序列上,统计第j碱基位点上出现1的次数,除以m就得到该位点的匹配频率。匹配频率函数F(j)定义如下:
(2)
函数F是匹配频率,它代表的是比对序列的第j个碱基位点,与被比对序列两者的匹配强度,或者是它们相互作用的强度。由此可以推出,根据F值的大小,可以对此位点参与最佳匹配的概率进行评价。如果F(j)等于1,那么就能够说明,所有m条序列的第j个碱基位点,应该全都位于最佳匹配区域内。由此得到匹配频率F值在序列上的分布。
定义3:序列长度标准化
实际序列可以是编码序列,或者是内含子序列,针对它们长度各异这一情况,序列的最佳匹配片段要进行相对位置分布比较,为使其比较起来更方便,则需要把序列长度标准化,得到长度为L的目标序列。长度标准化采用如下方法:
(3)
分析以上公式,将其中第i条比对序列长度设置为Li;那么位于第i条比对序列上,第j个碱基位点是Nij;在长度标准化之后,与第i条比对序列相对应,第j个相对位点是Nij。高斯取整函数用方括号代表,要求是,取一个实数的整数部分。如此,就在m条长度各不相同的序列经过转化之后,得到L长度的目标序列。实际上,序列长度标准化的过程就是,按照一定的比例将长度各异的多条序列缩放成等长的序列,便于更好地对最佳匹配区域的相对位置分布进行分析。
2 结 果
分析最佳匹配片段的序列构成,有助于深入研究内含子与相应mRNA序列之间相互作用。通过对mRNA序列和相应内含子序列之间的比对,得到各物种mRNA序列上全部最佳匹配片段集合。要对所有最佳匹配片断进行序列特征分析,那么就要用到最佳匹配片段中的3个特征参数,它们分别为最佳匹配片段长度、最佳配对率及其中的GC含量。
2.1 最佳匹配长度分布
通过mRNA序列与相应内含子序列之间的比对分析,得到了13个物种mRNA序列上的最佳匹配长度分布(见图1)。考虑这13类生物的最佳匹配片段长度分布形式是一样的,与α>1的伽马分布相接近。长度大多在10~80 bp之内分布。无脊椎动物、植物,它们的长度平均为20 bp左右;而有脊椎动物,长度大约平均为30 bp。我们发现,对于高等真核生物,其最佳匹配片段的长度,平均起来要长于低等真核生物。 这就是说生物进化的同时,最佳匹配片段长度,亦随之增加。那么也就是说,物种愈复杂,内含子与其相应mRNA序列二者之间的相互作用模式,也会愈复杂。
2.2 最佳匹配片段的配对率
对配对率给出如下定义:把L设定为最佳匹配片段长度,如果k个碱基可以进行完全配对,也就是存在C-G和U-A,那么,k/L即是该最佳匹配片段的配对率。最佳匹配片段的配对率分布图(见图2)。不同的物种的配对率分布基本无差别,大多分布在60%~85%,甚至极少部分最佳匹配片段的配对率能达到100%,这些片段的长度都很短,低于15 bp。配对率分布曲线上有一些小的峰值出现,比较明显的峰值出现在配对率为0.75, 0.8, 0.85 和0.95处,这些峰值分布对各个物种都是一样的,说明了最佳匹配区域序列匹配是有规律的。最佳匹配片段配对率分布在各个物种中具有普适性,反映了配对率的保守性。这表明不同进化水平的物种其mRNA与相应内含子的相互作用或序列匹配方式遵循同一个匹配机制。
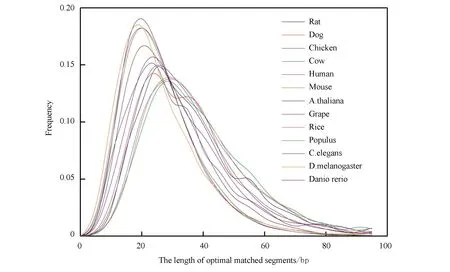
图1 最佳匹配片段长度分布Fig.1 Length distributions of optimal matched segments
2.3 最佳匹配片段GC含量分布
对mRNA的序列特征分析时,发现,GC含量是非常重要的参数。13类生物基因的mRNA序列上的最佳匹配片段的GC含量分布情况(见图3)。四种植物最佳匹配片段的GC含量分布在一个比较窄的范围(0.2~0.6)之内,最概然分布是0.35,两个脊椎动物鸡和斑马鱼的分布范围比植物要宽,在0.1~0.7,最概然GC含量是0.45。线虫的分布与植物相似,果蝇的分布与脊椎动物的分布相似。哺乳动物最佳匹配区域的GC含量分布各不相同,大鼠和小鼠的最概然GC含量更大一些(约为0.55),牛的最概然GC含量很小为0.3。但它们的分布范围很宽,主要在0.1~0.8。人类和狗的分布与其它物种的分布不同,不仅GC含量分布更宽,在0.1~0.9,而且最佳匹配区域的GC含量分布是双峰分布。第一个峰的最概然GC含量值是0.3,第二个峰的最概然GC含量值是0.7。
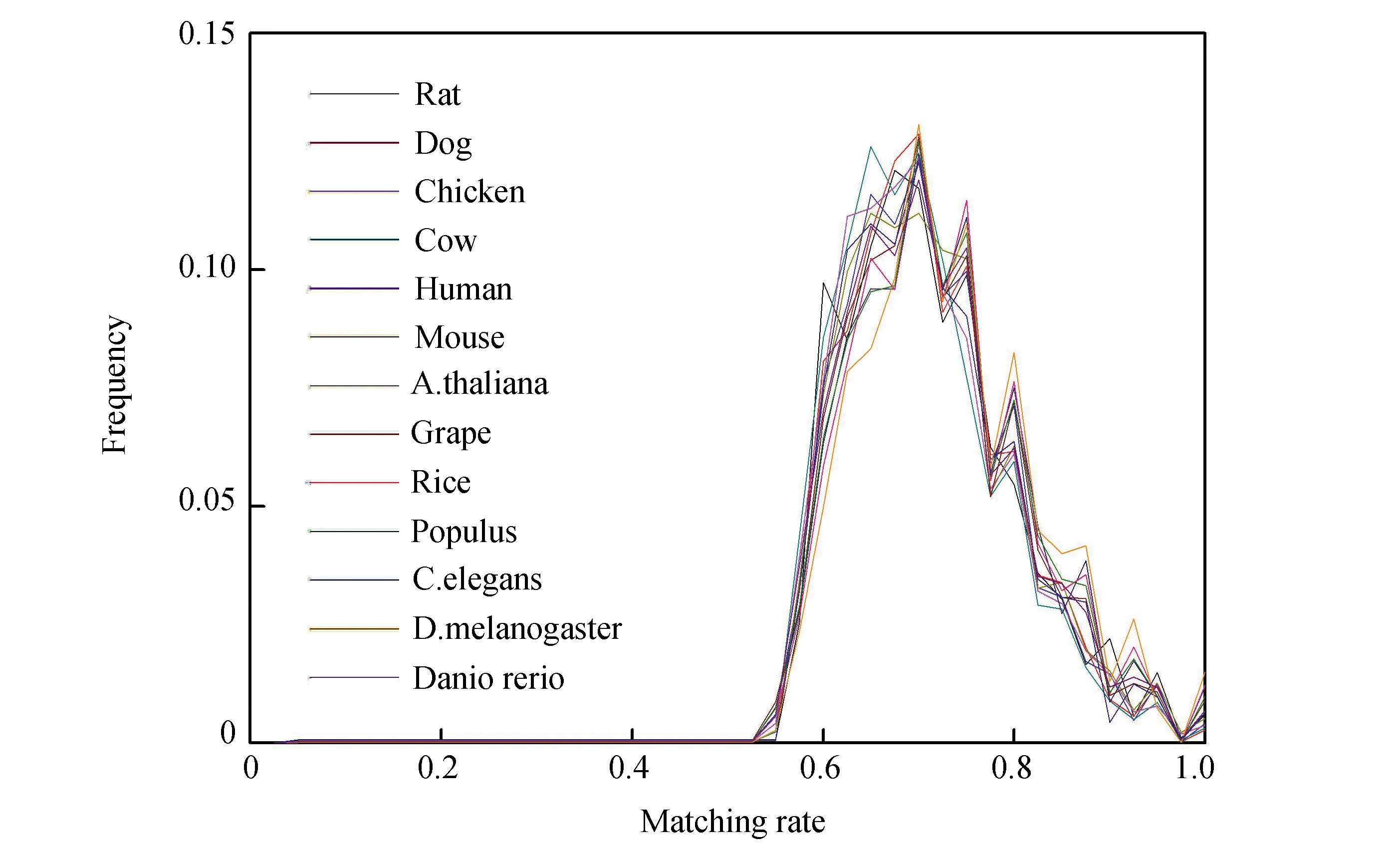
图2 最佳匹配片段配对率分布Fig.2 Matching rate distributions of optimal matched segments
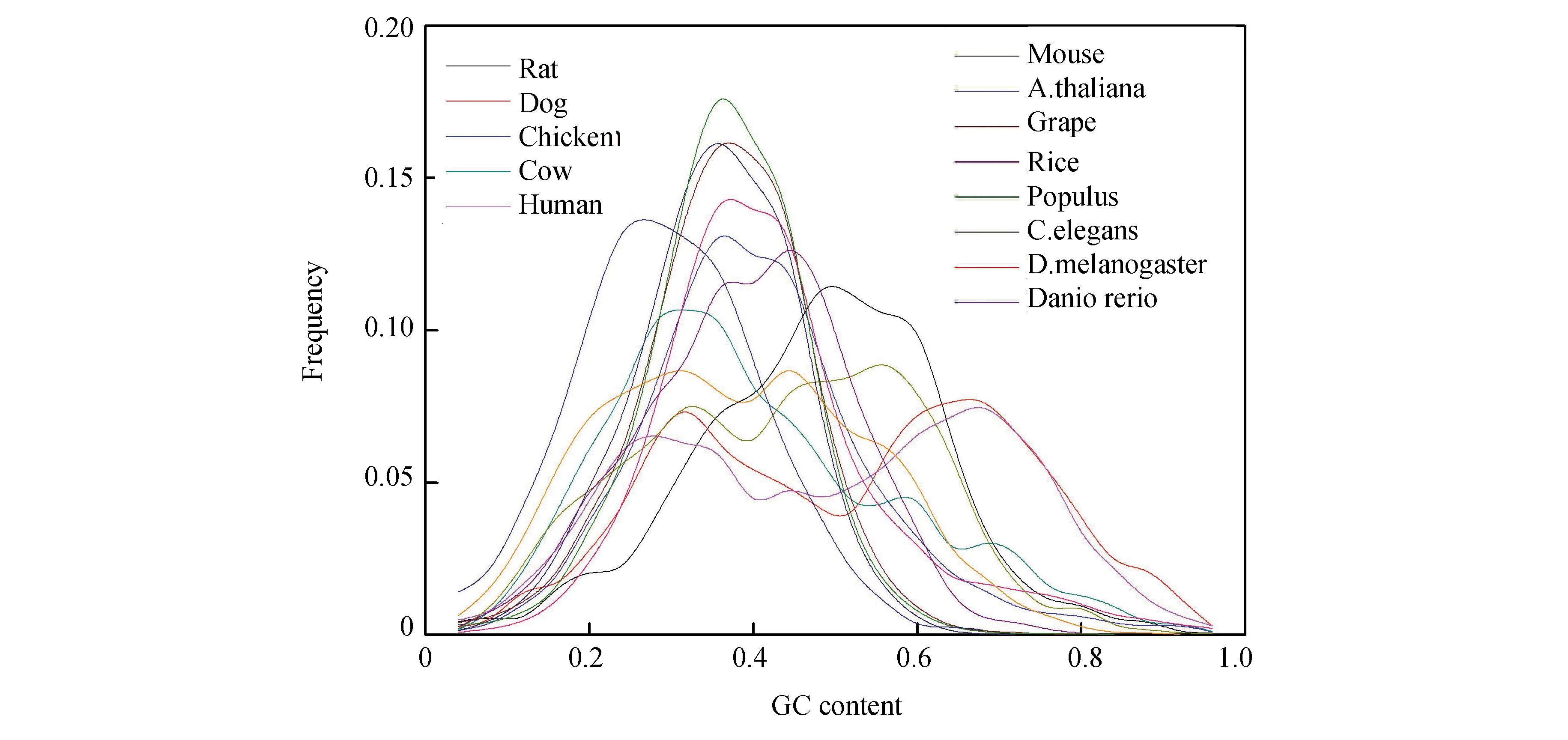
图3 最佳匹配片段的GC含量分布Fig.3 GC content distribution of optimal matched segments
可见最佳匹配片段的最概然GC含量随着物种进化在逐渐增加,这与基因组序列GC含量随物种进化而增加的现象是一致的。值得注意的是最佳匹配片段的GC含量分布范围很广,在0.1~0.9广泛分布。由此说明最佳匹配片段的发生不受GC含量水平的影响,有的GC含量高,有的GC含量低。
对于大多数低等生物,最佳匹配片段中GC含量均偏低,接近内含子的GC含量。这表明基因中mRNA序列上,最佳匹配多发生在低GC区域。而且内含子与mRNA之间存在一种弱相互作用,从而形成弱双链结构。然而对高等生物而言,最佳匹配片段中GC含量多数偏高,与其外显子中GC含量接近。在人类和狗中,最佳匹配片段中GC含量分布特征与线虫和小鼠的分布一致。尽管人类与小鼠基因在进化上很接近,但最佳匹配片段中GC含量分布却有差别。这说明内含子与mRNA序列之间的最佳匹配序列组成在一定程度上反映了表观遗传的进化差异。
2.4 最佳匹配片段在mRNA序列上分布特征
针对13个物种基因序列,把它们的内含子和对应的成熟mRNA放在一起,进行局域比对。在成熟mRNA上,出现最佳匹配区域位置。因成熟mRNA序列长度不一致,为研究mRNA序列上各个相对位置的匹配强度,所以把成熟mRNA序列进行长度标准化,长度标准化到100 bp。发现成熟mRNA上出现相对匹配频率的分布情况(见图4)。结果如下:(1)所有物种的匹配强度分布趋势一致,在序列UTR区(两端)的匹配频率明显高于编码序列(中部),特别是在3’UTR区出现极大值分布。(2)无脊椎动物在3’UTR区的峰值最大,比如果蝇和线虫的极大值大约是它们极小值的7倍(见图4b),植物的匹配频率的极大值略高于有脊椎动物的峰值。(3)在mRNA序列的5’UTR区,有3个有脊椎动物(人类,狗和大鼠)的匹配F值高于它们的CDS区(见图4a),而其它有脊椎物种则低于CDS区。单子叶植物(水稻)的F值低于双子叶植物的F值,也低于CDS区的F值(见图4c)。(4)所有物种在CDS区匹配强度大小较一致,并且F值都比较低。从整体上来说,植物物种在CDS区的F值相对最高,无脊椎动物的F值相对最低,而有脊椎动物F值介于它们之间。
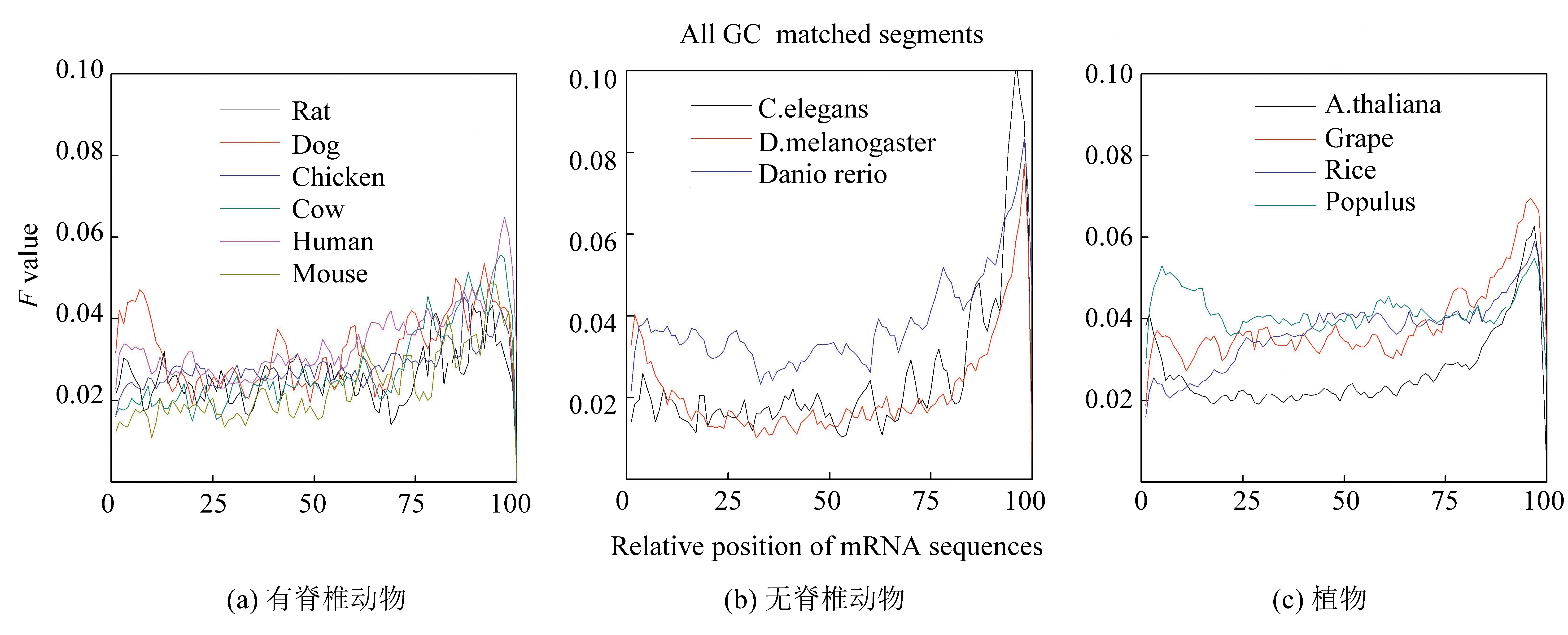
图4 mRNA序列和相应的内含子序列局域比对的相对频率分布Fig.4 Distributions of matching frequency between mRNA sequences and cooresponding intron sequences
图3中,为找到最佳匹配片段的GC含量分布情况,把最佳匹配片段按照GC含量的大小分为2组:GC含量高于0.5的高GC片段和GC含量小于0.3的低GC片段。得到它们在mRNA序列的上分布(见图5、图6)。分析发现,在mRNA序列的UTR区,低GC片段在其匹配强度更高,而在编码区对应的F值则更低。无脊椎动物和植物在3’UTR区有显著的峰值(见图5b,5c),对有脊椎动物来说,3’UTR区出现了多个极值的匹配区域(见图5a)。4个植物物种和果蝇仍在mRNA序列的5’UTR区出现极大值分布,且约为极小值的3倍(见图5c),但是其他物种在该区域的匹配频率极低。
对于高GC片段在mRNA序列上的匹配强度分布,我们发现,无脊椎动物在mRNA序列上的各个位点的F值大小相近(见图6b),然而植物和有脊椎动物在5’UTR匹出现极大值,其中有脊椎动物出现显著的极大值约为极小值的5.5倍(见图6a,6c)。另外在CDS区和3’UTR区的匹配频率一致,且F值均较低。
mRNA序列上存在许多与内含子序列的匹配区域,低GC片段偏好与UTR区作用,特别是在mRNA序列的3’UTR区。高GC片段偏好与mRNA序列的5’UTR区匹配。高GC片段在mRNA序列的3’UTR区和编码区分布没有显著的差别,说明在内含子上还存在一些高GC含量区域,它们与整个mRNA序列都有作用。

图5 GC含量在0-0.3内mRNA序列匹配频率Fig.5 Distribution of F value with GC content between 0.0 and 0.3

图6 GC含量大于0.50的mRNA序列匹配频率Fig.6 Distribution of F value with GC content greater than 0.5
3 结果与讨论
通过序列比对获得了13个物种基因中内含子与mRNA二者的最佳匹配片段。通过这些最佳匹配片段序列特征及匹配频率的分布规律,我们发现,siRNA和miRNA的结合特征与得到的最佳匹配片段的平均长度和配对率分布一致;mRNA上的UTR区偏好与内含子相互作用,而CDS区域与内含子的匹配程度较低。结论表明内含子与成熟mRNA序列存在相互作用。
目前,人们对内含子与mRNA序列二者的最佳匹配片段还缺乏一定的了解。小干扰RNA(siRNA)、微小RNA(miRNA)以及piRNA(Piwi-interactingRNA)这三个非编码序列承担了RNA干涉和RNA抑制的功能。一方面,Dicer可以把长度在21~25 bp之间的siRNA加工形成双链RNA,然后双链RNA与靶mRNA进行严格地互补再指导mRNA沉默[24]。另一方面,要形成单链RNA,Dicer还会在18~25 bp之间,选出miRNA进行加工,单链RNA与目标mRNA这二者之间,在不同程度地进行完成互补,之后对靶mRNA转录和表达进行干涉和抑制[25]。通过统计数据,可以看到miRNA与靶mRNA的匹配率在65%~95%的范围内,那么我们认为miRNA在调控发育过程中发挥着关键作用。piRNA是一类长度为26~31 bp单链的小RNA,大部分集中在29~30 bp之间,而且只有通过小RNA与属于PIWI蛋白的家族成员进行结合,piRNA才能发挥调控作用。piRNA的发现对非编码小分子RNA的研究开拓出新的领域,因此Science把该研究称为2006年十大科技进展之一[26]。
把内含子与mRNA之间相互作用的最佳匹配片段的长度以及配对率的数据区间范围与miRNA和siRNA进行比较,结果显示双方的长度和配对率范围的特性,竟然相当的一致。内含子在经过剪切之后,和对应的mRNA之间,相互作用的程度高,无论是mRNA与内含子作用的最佳匹配片段还是siRNA、miRNA和piRNA,从生物选择的功能片段长度到配对率,它们所遵循的生物学机制是一样的。由此推论,在生物基因组序列上,还存在大量的各种形式的类似miRNA的功能片段,它们在基因表达调控和实现表观遗传多样性方面起着决定性的作用。在不同程度上,通过和mRNA序列之间的互补,一些存在于内含子序列上的区域,才能够对基因调控和表达产生深刻影响。对配对率的分布进行研究之后,我们发现,绝大部分的片段所进行的配对并不严格,然而有极少的片段,严格进行配对,但这些片段的长度很短,远小于20 bp。按照RNA干涉理论,完全匹配对基因表达是致死的,因此在mRNA与内含子之间存在的相互作用片段,显然避开了这类致死的匹配片段。从这个角度来看,mRNA-内含子的相互作用理论是合理的。分析认为,对于真核基因这一基因种群,他们自己就具备基因调控所需的原件。因为内含子是非编码序列,所以它可以完成剪接与可变剪接这两个任务,而且能够实践基因表达调控这一重要功能。内含子序列作为具有功能的一类RNA-RNA相互作用的非编码序列集合,研究人员需要对此重要特征予以关注。
发现内含子与mRNA序列的匹配片段在mRNA序列上有分布,其匹配频率,发生在两端非编码序列区域的值较大,在中间编码序列区域,却有较低值。同时,内含子又出现偏好和mRNA序列上3’UTR区相互作用。在mRNA序列的3’UTR区,GC值比较小的片段,拥有相对高的匹配强度,匹配强度却在编码区较低。那么就说明内含子与相应mRNA之间存在的相互作用主要是弱键,也就是所谓的AU匹配,同时还包括GC值大的匹配。但是,大家有可能提出疑问的是,基因序列进化时,UTR类似于内含子的GC值,很清楚的显示出不同于编码序列的GC值,这一现象的原因是什么呢?想要出现内含子和UTR序列发生较强的相互作用,然后通过该作用对基因进行调控,从而使以上两类序列的进化趋于一致。在UTR和编码区上GC含量较高的最佳匹配片段分布几乎不存在差别,这也表示一些高GC区域存在于内含子序列上。可以看出,要想进一步探究内含子对基因的表达与调控,还必须继续挖掘mRNA序列上匹配频率分布的内涵。
综上所述,所有的研究结论,有力地证明了,内含子和mRNA之间发生的相互作用,是真实存在的。在维持基因组正常运转的过程中,内含子可能起到了比较关键的调控作用[27]。然而尚需进一步对真核生物内含子与相应的mRNA序列的相互作用进行深入探讨, 本文虽然揭示了一些有意思的论点,然而一些结论尚需通过实验进一步验证。
