基于美国高加索人群瘦体重的全基因组关联研究
2020-09-27张宇雪刘宝林
冉 姝, 刘 宇, 张宇雪, 刘宝林
(上海理工大学 医疗器械与食品学院,上海 200093)
肌少症(sarcopenia)是与增龄相关的、进行性的全身肌量减少和(或)肌强度下降或肌肉生理功能减退的综合症[1-2]。肌少症和骨质疏松症相伴出现,致使老年人易于跌倒和骨折,继而成为老年人群致残、致死的主要原因之一[3-4]。中国城市地区高龄男性肌少症的患病率为12.3%,女性为4.8%,农村高龄男性肌少症患病率为6.4%,女性为 11.5%[5]。最近一项研究估计全球肌少症的患病率为10%[6]。预计到2050 年,髋部一个部位的跌倒骨折人数就将达到600 万,造成254 亿美元的直接治疗费用以及更高的术后护理费用[7]。肌少症将是未来面临的主要健康问题之一。
肌少症的核心是骨骼肌数量和质量的丢失,在研究其发病因素及防治方法时应主要考虑这两个方面。使用双能X 线骨密度仪(DXA)测量的瘦体重(LBM)是肌少症的重要性状[8]。LBM 受遗传控制,遗传度在50%~80%之间[9]。与肌少症高度相关的其他性状的研究中发现,握力的遗传度为32%~77%[10-11],肌肉横截面积的遗传度为70%~90%[12]。因此,LBM 显著的遗传度为肌少症遗传学研究提供了良好的理论基础。瘦体重指数(LMI)为瘦体重与身高的平方之比[13]。
全基因组关联研究(genome-wide association study,GWAS)是一种通过扫描整个基因组,鉴定出与复杂疾病或复杂性状相关的遗传因素的研究方法。目前GWAS 研究已经发现许多SNPs(单核苷酸多态性)与瘦体重相关。2009 年,Liu 等[14]对1 000 名白人全身瘦体重进行全基因组关联分析,发 现 位 于 TRHR 基 因 的 SNPs( rs16892496 和rs7832552)与LBM 显著关联。Guo 等[15]以1 627 名中国人作为实验样本、2 286 名欧洲白人作为验证样本进行双变量全基因组关联研究,发现与葡萄糖代谢和能量代谢相关的GLYAT 基因与四肢瘦体重(appendicular lean mass, ALM)关联。Urano 等[16]对1 081 名日本绝经后妇女进行关联研究,发现PRDM16 基因在调节LBM 中有重要作用。2017 年,Zillikens 等[17]对38 292 名欧洲人全身LBM 和28 330名欧洲人ALM 进行大规模关联分析,并分别在63 475和45 090 名不相关的欧洲人群中进行验证,研究发现VCAN,ADAMTSL3,IRS1 和FTO 基因与全身LBM 和ALM 相 关 联。2019 年,Pei 等[18]对来自弗雷明汉心脏研究队列内6 587 名受试者的腿部瘦体重进行了关联分析,发现位于6p21.1 处的两个功能变体(rs524533 和rs571770)与LBM 有关,并通过Cis-eQTL 分析发现这些SNPs 与NFKBIE基因表达相关联。
为了鉴定与肌少症相关联的基因,本研究对2 283 名不相关的美国高加索人样本进行基于基因水平的关联分析,并在1 000 名不相关的美国高加索人样本中验证。
1 材料与方法
1.1 样本
研究参与者是从美国奥马哈市(Omaha)和堪萨斯城(Kansas)及其周边地区招募的健康个体,研究得到了克瑞顿大学和密苏里大学堪萨斯分校伦理委员会的批准。所有研究者在进入研究之前都提供了书面知情同意书,并且完成了包括生活方式、病史、家庭信息等相关问卷调查。研究采用肌少症遗传研究中广泛应用的排除标准对样本进行筛选,避免出现假阳性结果。
研究样本由2 283 名不相关的美国高加索受试者组成,包括556 名男性和1 727 名女性。验证样本由1 000 名不相关的美国高加索人组成,包括501 名男性和499 名女性。研究样本和验证群组之间没有重叠。
使 用Hologic 公 司 的DXA4500(Hologic Inc.,Bedford, MA, USA)骨密度仪对研究者进行瘦体重和脂肪量的测量。
1.2 基因分型和质量控制
a. 研究样本。抽取每位受试者10 mL 的外周静脉血。本研究采用标准的DNA 提取试剂盒进行DNA 提取,提取后采用Beckman 公司的DU530 核酸/蛋白分析仪(Beckmanlnc., Fullerton,CA.UK)进行浓度和纯度的测量。采用Genome-Wide Human SNP Array 6.0 芯片(Affymetrix, Santa Clara, CA, USA)对研究样本进行基因分型,并在个体和SNP 水平上执行严格的质量控制步骤。在个体水平,使用PLINK软件根据X 染色体上的基因型数据推断出个体的性别,并与问卷中记录的性别作比较,将不明确的以及与报告性别不一致的个体删除。在SNP 水平,所挑选的SNP 符合以下条件:最低基因分型率为95%;哈迪温伯格平衡值大于0.001;等位基因频率大于0.01。最后有746 709 个SNPs 保留在研究样本中。
b. 验证样本。基因分型采用Affymetrix 250K Nsp 和Affymetrix 250K Sty,过程按标准步骤进行,共有379,319 个SNPs 用于关联分析。
1.3 统计学处理
研究以瘦体重指数(LMI)作为表型,采用SPSS 19.0 软件对性别、年龄、年龄的平方和脂肪量进行协变量校正。为了避免因群体分层现象而导致出现假阳性或假阴性结果, 采用主成分分析法(principal component analysis, PCA)校正2 283 名美国高加索人样本中潜在的种群分层。在样本中挑选一组无关样本并计算它们的主成分,作为它们的遗传背景信息。再选用前5 个主成分, 利用这5 个主成分值对表型进行校正。使用PLINK 软件( http://zzz.bwh.harvard.edu/plink/summary.shtml) 的加性遗传模式对校正后的表型进行关联分析。荟萃分析采用Fisher 方法[19],对研究样本和验证样本的结果进行合并,得到合并p值。
1.4 功能预测
采用GTEx 数据库(http://gtexportal.org)和Regulome DB 数据库(http://regulomedb.org/)进行功能预测。GTEx 数据库目前收集了来自544 个人体的基因型和基因表达数据,提供了跨器官的表达数量性状基因座(eQTL)研究平台。Regulome DB 数据库集合GEO,ENCODE 等项目的数据集,通过评分对SNPs 是否处于启动子区域、增强子区域、转录因子结合位点以及DNase 敏感区域进行注释。
2 结 果
受试者的基本信息如表1 所示。UQCR 基因上的SNPs rs8697(研究样本p=2.50×10−2,验证样本p=2.33×10−2,合并p=4.94×10−3)以 及rs56122285(研究样本p=9.92×10−3,验证样本p=2.85×10−2,合并p=2.59×10−3)与LMI 关 联。MBD3 基因上的11 个SNPs 以及TCF3 基因上的3 个SNPs 与LMI有关联,具体信息列于表2 中。
通过Regulome DB 查询rs8110543 和rs7252741的DB 得分分别为1b 和1f。得分为1b 的变体对功能具有很高的置信度,rs8110543 不仅位于DNase敏感区域,还是转录因子与eQTL 的结合位点。DNase 敏感区域的DNA 特别活跃而且具有顺式调节的作用,在一定程度上可以说DNase 敏感区域表示转录调节的控制区域。
采用GTEx 对SNPs 进行eQTL 分析,发现16 个SNPs 的eQTL 靶基因都是UQCR,并起到顺式调节的作用(表2)。GTEx 查询得到UQCR 基因在骨骼肌表达图(图1)和 rs8697,rs56122285 的eQTL 图(图2 和图3),N为样本容量。TPM为每百万映射读取的转录本。
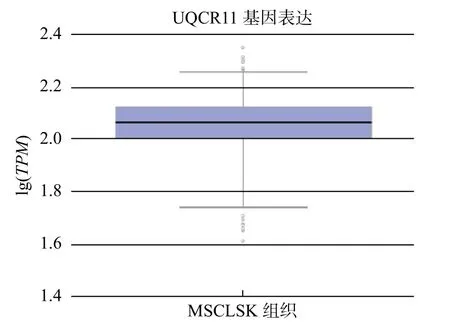
图 1 UQCR11(UQCR)eQTL 基因骨骼肌表达图Fig. 1 Structure of the eQTL gene skeletal muscle expression of UQCR11(UQCR)
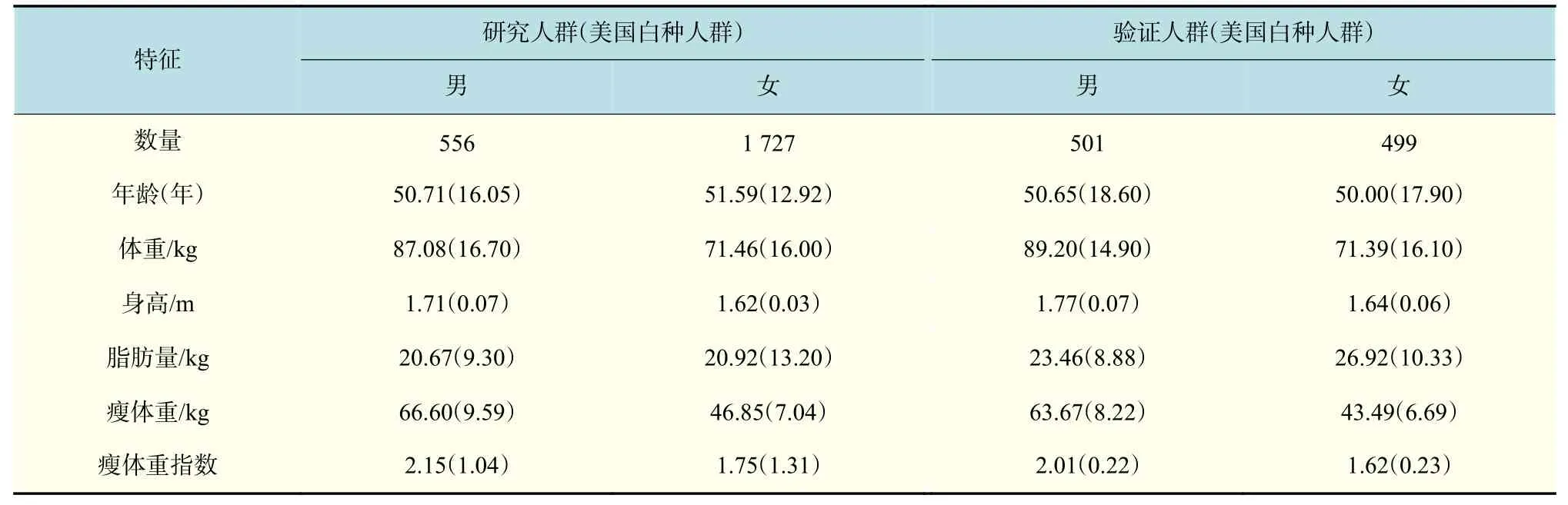
表 1 研究样本和验证样本的基本信息Tab.1 Information of the discovery sample and replication sample
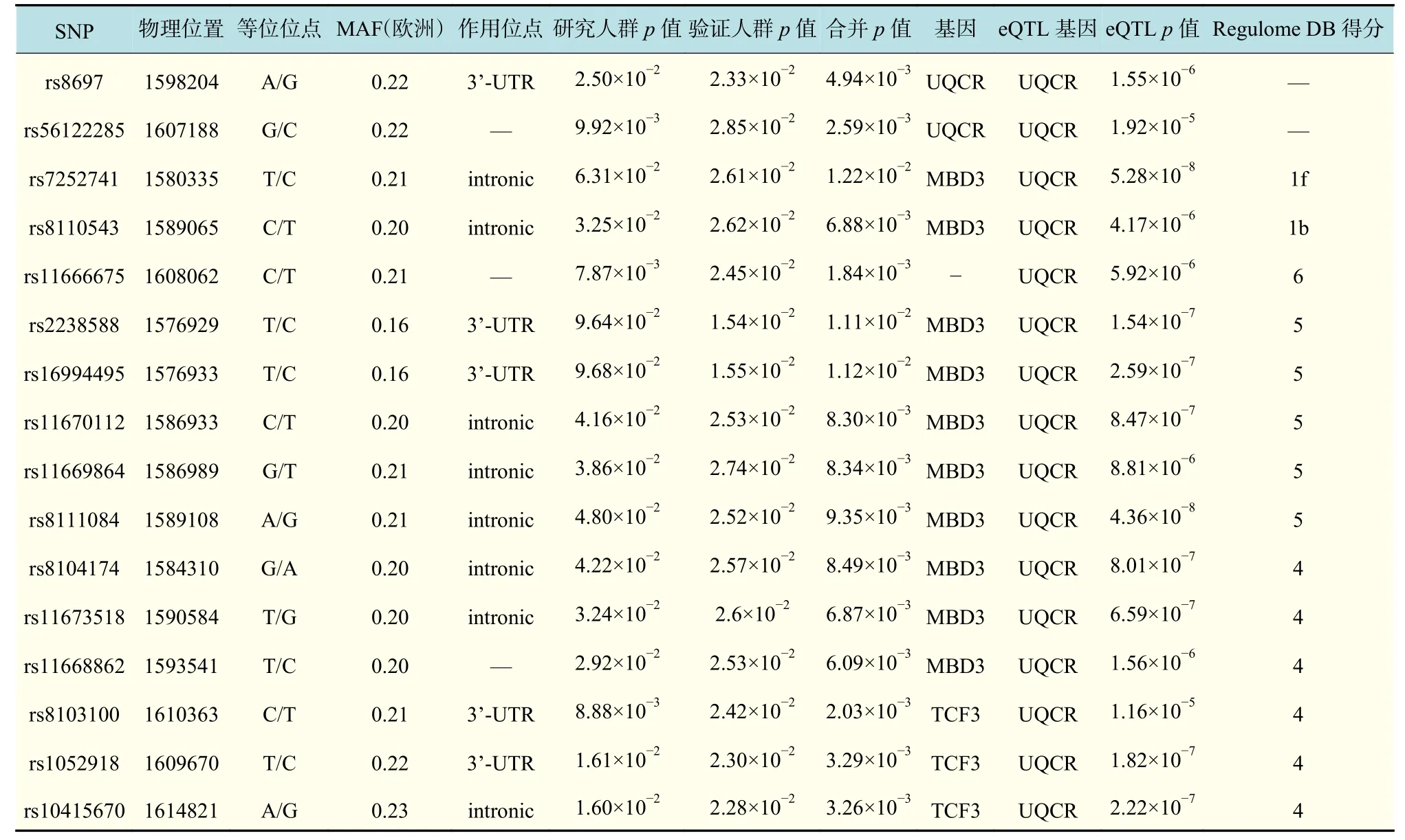
表 2 19p13.3 显著SNP 和相关基因的eQTL 分析结果Tab.2 19p13.3 eQTL analysis results about significant-association SNP and genes
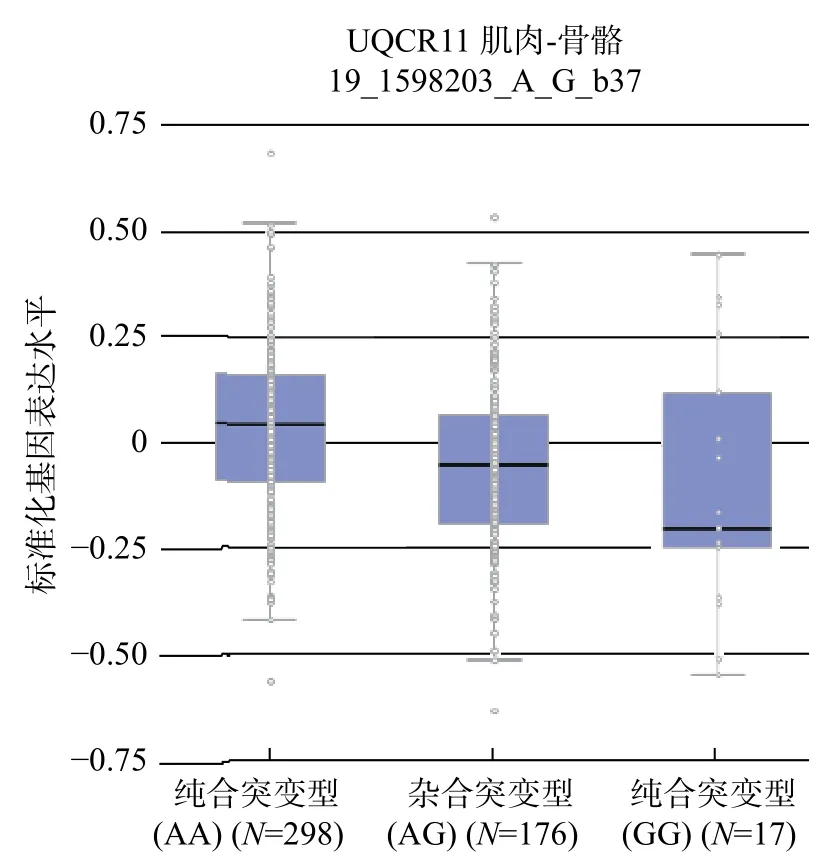
图 2 rs8697 的eQTL 基因表达图Fig. 2 Strueture of the eQTL gene expression of rs8697
图 3 rs56122285 的eQTL 基因表达图Fig. 3 Strueture of the eQTL gene expression of rs56122285
使用STRING 蛋白互作网络对TCF3,MBD3 相关肌肉代谢蛋白进行分析(如图4 所示)。MBD3蛋白是抑制性核小体重塑和去乙酰化酶(NuRD)复合物的核心成分,具有保守的甲基CpG 结合域[20-21]。TCF3 蛋白靶基因的表达限于特定的细胞谱系,通过含有TCF3 和组织限制性螺旋−环−螺旋(bHLH)因子的异二聚体来激活细胞特异性基因表达并促进细胞分化[22]。这种谱系选择机制在骨骼肌的分化中得到了清楚的阐释[23-24]。
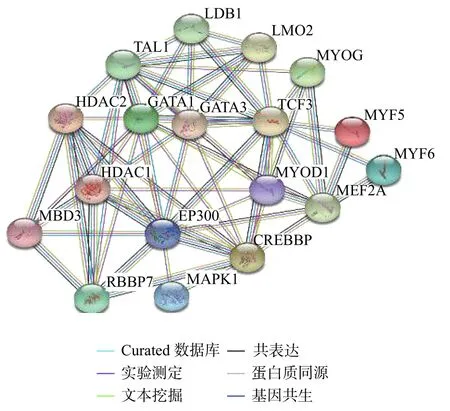
图 4 TCF3 及MBD3 与肌肉代谢蛋白的STRING 蛋白质互作网络Fig. 4 STRING protein interaction network between TCF3 and MBD3 and muscle metabolic proteins
3 讨 论
UQCR(ubiquinolcytochrome c reductase)泛醌细胞色素c 还原酶、产物线粒体复合物III 是电子传递链上的核心元素,在辅酶Q 氧化和细胞色素c 还原的过程中起到介导的作用[25]。复合物III 在高等哺乳动物中由11 个亚基组成,其中,有3 个是催化亚基,分别是细胞色素b、细胞色素c1 和Rieske 铁硫蛋白[26]。在三磷酸腺苷(ATP)的生化合成过程中可以催化电子使其转移到细胞色素b,还能利用能量在线粒体膜上转移质子。在儿童和成年人的神经肌肉和非神经肌肉疾病患者中发现线粒体复合物III 缺乏[27]。
MBD3(methyl-CpG binding domain protein 3)甲基CpG 结合域蛋白3 是组成NuRD 复合物的一个亚基。此外,NuRD 还是由HDAC1(组蛋白脱乙酰酶1)、HDAC2、MTA2 和RBBP7 等组成的多亚基复合物[28],其在DNA 的损伤修复过程以及DNA转录调节中发挥重要功能。HDAC1 可以使生肌决定因子(MYOD)去乙酰化并阻止MYOD 在分化条件下启动肌源性程序[29]。在肌细胞分化时,HDAC1水平的降低与MYOD 乙酰化水平的增加有关,后者会刺激转录[30]。
TCF3(transcription factor 3)转录因子3 可以编码螺旋−环−螺旋(helix-loop-helix)结构的转录因子E12(免疫球蛋白转录因子−1)和E47(转录因子−3)[31]。E12 和E47 两种因子可以与生肌决定因子(MYOD)和 肌细胞生成素(MYOG)相互作用,参与肌细胞特异性bHLH 蛋白的二聚化[24]。bHLH 转录因子家族包括MYOD,MYOG,生肌因子5(MYF5),生肌因子6(MYF6)和生肌调节因子4(MRF4),它们可以调节肌源性细胞分化,然后再融合成肌纤维[32]。
采用全基因组关联分析方法,在2 283 名不相关的美国高加索人中发现位于基因UQCR,MBD3和TCF3 的16 个SNPs 与LMI 有关。这16 个SNPs都具有eQTL 效应,可能会影响或者导致肌少症。
