多组学分析在解淀粉芽孢杆菌相关功能机制研究中的应用进展
2020-08-28王世伟王卿惠翟丽萍刘军于志丹向文胜
王世伟,王卿惠,翟丽萍,刘军,于志丹,向文胜*
多组学分析在解淀粉芽孢杆菌相关功能机制研究中的应用进展
王世伟1,2,王卿惠3,翟丽萍1,2,刘军1,2,于志丹1,2,向文胜3*
(1.齐齐哈尔大学生命与农林学院,黑龙江 齐齐哈尔 161005;2.抗性基因工程与寒地生物多样性保护重点实验室,黑龙江 齐齐哈尔 161005;3.东北农业大学生命科学学院,黑龙江 哈尔滨 150030)
解淀粉芽孢杆菌()具有多种有益生物学特性,可产生多种抗菌活性物质,具有重要的生理生化功能,如生物膜形成与定殖、促进植物生长、诱导植物产生耐盐性等。综述了基因组、转录组和蛋白质组等多组学分析方法在解淀粉芽孢杆菌的抗菌活性、定殖、促生、植物耐盐性诱导等方面的研究进展。
解淀粉芽孢杆菌;多组学分析;鉴定;功能;分子机制
解淀粉芽孢杆菌()属革兰阳性菌,能产生多种酶类和抗菌活性物质,具有生物膜形成、定殖和促生等重要生理功能;因此,在种植业、养殖业、果蔬采后病害防治、环保、食品安全等领域具有广泛的应用前景[1–2]。随着基因组测序的进行,发现尽管解淀粉芽孢杆菌基因序列比较保守,但其基因结构仍存在差异[3]。笔者归纳了近年来利用多组学分析对解淀粉芽孢杆菌进行的生理生化特性及相关功能的研究,旨在将成果更好地应用于实践。
1 解淀粉芽孢杆菌基因组学研究
基因组学分析是系统生物学研究的重要方法,它包括以全基因组测序为目标的结构基因组学和以基因功能鉴定为目标的功能基因组学。解淀粉芽孢杆菌是芽孢杆菌属的1个种。为鉴定该菌种,常通过其形态学,如细胞大小、芽孢形态、位置和菌落等特征加以研究;在此基础上再根据其生理生化特征进行进一步鉴定。该种与枯草芽孢杆菌()在形态学上十分相似,单凭上述方法难以准确鉴定;因此,需要通过16 SrRNA基因序列比对和比较DNA回旋酶A亚基基因()或B亚基基因()等分子方法才能更准确地鉴定。随着解淀粉芽孢杆菌全基因组测序技术的不断完善,可通过全基因组测序技术对该菌进行鉴定。
1.1 解淀粉芽孢杆菌的结构基因组学研究
自从2007年完成解淀粉芽孢杆菌FZB42全基因组测序,迄今已对50多株菌株的全基因组进行了测序。通过测序,发现其结构有以下特点(表1):①染色体一般呈环状结构;②已测序基因组大小平均为3 990.462 kb,已测序并发表的15株解淀粉芽孢杆菌平均编码蛋白质基因3 927.86个;③包含的RNA基因的数目不一,如有的菌有63或87个tRNA基因,有的菌有7、16或27个rRNA基因,有的菌有6个sRNA基因,有的菌有86、69、80、109、117个不同RNA基因。
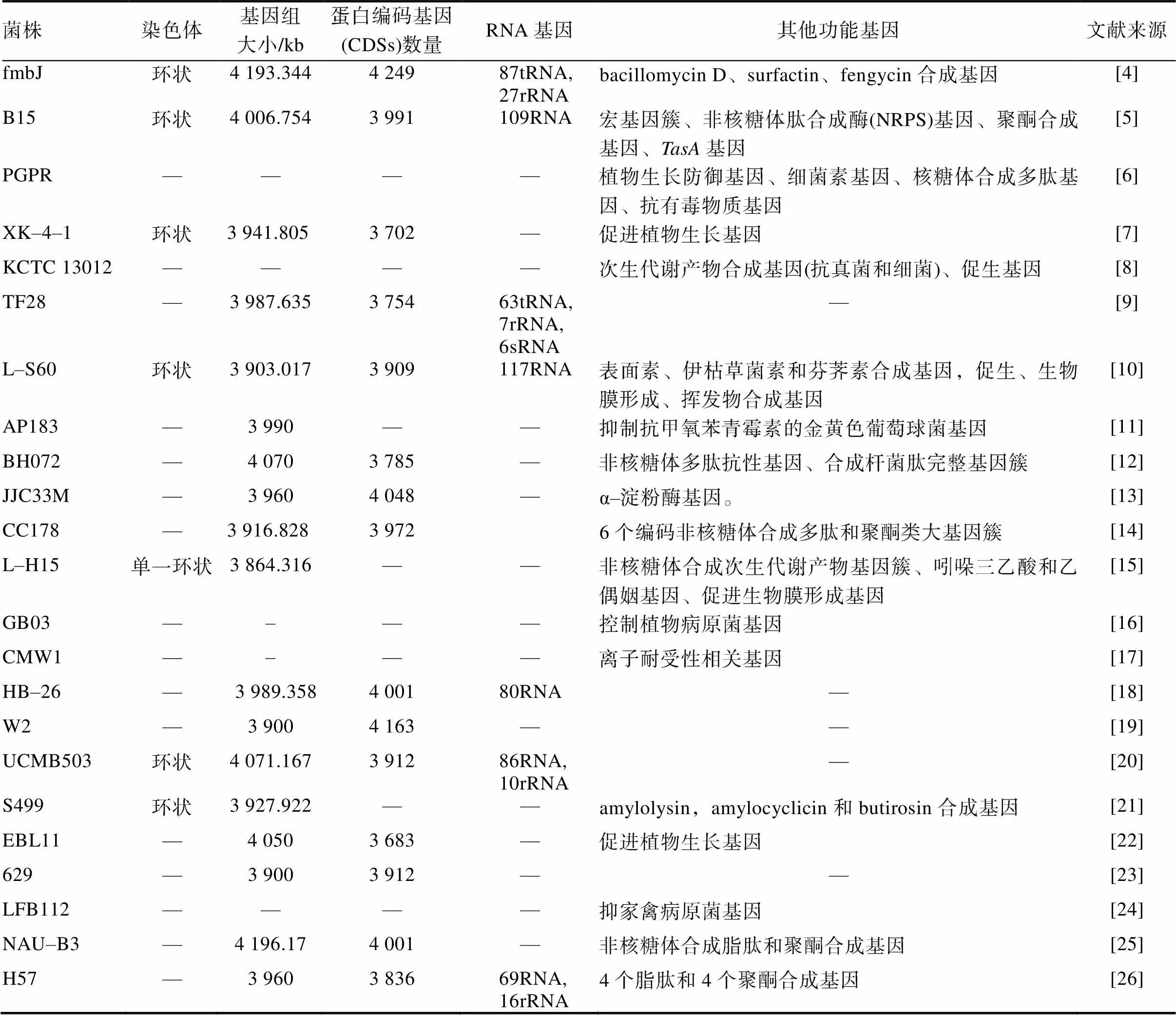
表1 部分解淀粉芽孢杆菌的全基因组测序结果及基因的结构和功能
从表1还可以看出,解淀粉芽孢杆菌功能基因组有如下几个特征:①含质粒或无质粒,G+C高(分别为46.50%、46.45%);②有串联重复序、重叠群(contigs);③含有微卫星或大卫星DNA;④一些菌基因组中心部分倒置,可能是前噬菌体或CRISPR结构域;⑤含有核糖体合成多肽基因(如细菌素);⑥含有非核糖体肽合成酶(NRPS)基因,如表面素(surfactin)、芬荠素(fengycin),伊枯草菌素(iturin)、杆菌肽的合成基因,以及聚酮类大基因簇;⑦含有产酶基因,如α–淀粉酶基因;⑧含有植物生长、防御、促生、生物膜形成基因,如吲哚三乙酸和乙偶姻相关基因;⑨含有抗毒、耐受性和挥发物合成基因;⑩含有基因、调节基因()等。可见,解淀粉芽孢杆菌基因结构决定了其生理功能。
1.2 解淀粉芽孢杆菌基因组的总体特征
从不同角度对解淀粉芽孢杆菌基因组进行研究,发现解淀粉芽孢杆菌基因组的相关基因具有多样性、保守型、可塑性和进化性等特征。
1) 多样性。NIAZI等[27]对解淀粉芽孢杆菌UCMB5113基因组进行了测序,发现其染色体由3 889 532 bp组成,能编码3 656种蛋白,含有能促进植物生长的基因(如吲哚–3–乙酸、乙酰胆碱酯酶合成基因及铁载体生产基因)、负责非核糖体次级代谢产物合成的基因、与菌株环境适应性相关的抗药物合成基因及抗金属基因,以及编码一系列分泌蛋白(降解根际大分子)、初级和次级代谢酶、碳水化合物活性酶和膜转运体基因。可见,就某个菌株而言,基因组相关功能基因比较丰富,具有多样性。
2) 相对保守性。HOSSAIN等[28]对12个芽孢杆菌基因组进行了测序。研究发现,尽管芽孢杆菌基因家族基因组存在比较宽泛的相似度(32%~90%),但其基因组存在高度保守性。对其核心基因组中2 839个基因进行研究发现,其中73个基因与生物控制以及根、叶定殖有关;尽管编码次生代谢产物合成基因簇不同,但在亚种内聚酮生物合成、编码地非西丁和麦克劳汀的基因簇高度保守。可见,对大多数解淀粉芽孢杆菌菌株而言,其基因组特定功能基因具有相对的保守性。
3) 可塑性。BELBAHRI 等[29]以所有已测序的解淀粉芽孢杆菌基因组数据为基础进行研究,发现不同菌株促进植物生长的策略不同,其次级代谢产物具有多样性。推测正是因为这种基因组的可塑性区域,塑造了基因组结构和功能的差异,并控制菌株适应不同生态位。
4) 进化性。解淀粉芽孢杆菌在进化过程中受到植物宿主和植物病原菌的影响,其基因组基因进化源于多种因素。ZHANG等[30]分析了31株在进化地位上比较接近的枯草芽孢杆菌和解淀粉芽孢杆菌全基因组序列,这些菌株包括来源于植物生境菌株(PA)和非植物生境菌株(NPA)。研究发现,与NPA菌株相比,PA菌株核心基因组中间代谢产物、次级代谢产物合成相关基因的丰度更高,并存在额外植物源底物利用和抗生素合成专门基因。这些“特化”基因只有少数是在最初分化过程中由PA菌株获得,而大多数是由不同PA菌株亚群在进化过程中依次获得。可见,来源于不同生境的芽孢杆菌基因组的变化很大程度受植物生境的影响,不同生境对其基因组的影响是逐步发生的,从而导致不同菌株生理功能的变化。解淀粉芽孢杆菌对尖孢镰刀菌(sp.)的抑制作用较强,然而镰刀菌也能产生多种重要的酶类,包括纤维酶、果胶酶和木聚糖水解酶等[31];因此,在研究解淀粉芽孢杆菌基因组变化时,不但要考虑生境对其进化的影响,还要考虑其所抑制的植物病原真菌对其进化的影响。
1.3 解淀粉芽孢杆菌基因的调控研究
解淀粉芽孢杆菌基因调节受多种原件控制,其中启动子、σ因子D、胶原蛋白基因、和基因以及DegU的调节作用尤为明显。
1.3.1有关解淀粉芽孢杆菌启动子的研究
启动子(promoter)的强弱直接影响转录率,因此,对解淀粉芽孢杆菌强启动子的筛选具有重要意义。LIAO等[32]构建了启动子探针载体pBE–,鉴定出266个含活性启动子成分菌,其中启动子P41在大肠埃希菌和解淀粉芽孢杆菌中表现出较强β–gal活性。核糖体结合位点从p41变成p382的优化,使β–gal活性提高200%。可见,获得强启动子对该菌基因表达具有重要意义。为解除嘌呤合成途径中嘌呤操纵子的调控,优化呼吸链能量生产,提高解淀粉芽孢杆菌XH7鸟苷生产量,LIAO等[33]在启动子取代突变子的研究中发现,与XH7相比,XH7purE::P41获得了最高鸟苷生产量(16.3 g/L),在XH7purE::P41突变菌株中,嘌呤操纵子基因(,和) 相对表达水平被上调,嘌呤代谢途径中的主要中间产物(次黄苷–磷酸,IMP)浓度大幅度增加,XH7pure::p41突变体最终鸟苷产量比XH7突变体增加41%。可见,要提高某种产物的产量,可以选用强启动子,也可以在此基础上,破坏5非翻译区的某些原件、结合启动子突变和修饰呼吸链上的某些酶来提高中间产物产量,最终提高目的产物的产量。
1.3.2有关解淀粉芽孢杆菌σ因子D的研究
σ因子作为一种非专一性蛋白,是DNA依赖的RNA聚合酶固有组分,能识别启动子共有序列并与全酶结合。在杆菌中,σ因子D负责鞭毛蛋白、甲基接受趋化因子和自溶素的合成。解淀粉芽孢杆菌某些菌株的σ因子对功能基因的调节也起着重要的作用。FAN等[34]研究发现,在解淀粉芽孢杆菌FZB42突变子中,敲除σ因子D,将使一整套基因下调;在转录水平上,该菌株的σ因子D能控制8个基因(、、、、、、和),其中4个基因功能未知,2个基因在模式菌株168中不存在(为解淀粉芽孢杆菌特有的基因),这8个基因参与解淀粉芽孢杆菌孢子形成、生物膜形成以及代谢物转运等功能。可见,对解淀粉芽孢杆菌σ因子D的研究有助于揭示解淀粉芽孢杆菌许多特殊的重要功能和相关功能基因的调控机制。
1.3.3有关解淀粉芽孢杆菌胶原蛋白基因及和基因功能的研究
胶原蛋白(CLPs)基因簇对解淀粉芽孢杆菌生理功能的发挥具有重要作用。ZHAO等[35]研究了解淀粉芽孢杆菌FZB42中推断的CLPs,运用扫描电镜和疏水值检测技术,研究了CLPs突变细胞的形态和表面结构。结果发现,CLPs对细菌自聚集和根系粘附具有一定作用;在生物膜形成和细菌–植物相互作用中,位于细菌细胞外的CLPs发挥了重要作用,敲除任何一个基因都将产生独特的形态表型。表明解淀粉芽孢杆菌生物膜形成与特定基因有关,并受其他调控因子调控。
解淀粉芽孢杆菌和基因对定殖和促生具有调控作用。BUDIHARJO 等[36]以转座子突变为基础,建立转座子文库,筛选参与植物多细胞行为和根系生物膜形成的基因,并通过救援克隆和DNA测序确定了转座子的插入位点。结果发现,FZB42中的A和基因产物对细菌定殖于植物具有重要作用;A突变体的生长、生物膜形成以及在琼脂平板上群集运动均受到限制;基因在枯草芽孢杆菌类群中出现的频率很低,直接参与促进植物生长。
2 多组学分析运用于解淀粉芽孢杆菌功能的研究
转录组学分析是细胞表型和功能研究的重要手段。与基因组不同,转录组定义中包含时间和空间限定。同一细胞在不同生长时期及生长环境下,其基因表达不完全相同。解淀粉芽孢杆菌转录组分析能揭示其重要功能基因表达的时空性。蛋白质组学是指利用各种技术研究蛋白质组的一门新学科,其主要目的是研究生物体内所有蛋白质种类、表达水平和修饰状态,以了解蛋白质之间的相互联系,揭示蛋白质的功能。
2.1 解淀粉芽孢杆菌对植物病原菌的抗性的研究
2.1.1利用转录组学分析研究解淀粉芽孢杆菌对植物抗病原菌的抗性
虽然控制特定代谢物基因簇转录的起始蛋白已经确定,但影响转录延伸的蛋白质还未被广泛研究。GOODSON等[37]分析了转录延伸蛋白NusG家族的系统发育,发现其包括1个连贯的旁系外群(LoaP),通常位于代谢产物基因簇之间或内部;解淀粉芽孢杆菌LoaP作为此蛋白亚群的一个范例,调节位于2种不同抗生素生物合成操纵子终止位点的转录读取。LIU等[38]筛选了2株对大豆赤霉菌有抑制作用的芽孢杆菌(淀粉样芽孢杆菌JDF3和枯草芽孢杆菌RSS–1),应用比较转录组分析法研究了大豆赤霉菌在解淀粉芽孢杆菌和枯草芽孢杆菌胁迫下的反应。结果表明,大豆赤霉菌表达基因发生了显著变化,共检测到1 616个差异表达基因(DEGs);解淀粉芽孢杆菌参与2种主要类型的监管,即“殊性监管”和“共性监管”,通过抑制大豆赤霉菌核糖体活性来抑制其生长;同时解淀粉芽孢杆菌发酵液可诱导活性氧迸发(Active oxygen burst),产生NO,使愈伤组织沉积和木质化,从而对植物起到保护作用。
2.1.2利用多组学分析研究解淀粉芽孢杆菌抗植物病原菌、抗肿瘤的特性
解淀粉芽孢杆菌对多种植物病原菌具有抗性。TANG等[39]筛选了生防剂淀粉芽孢杆菌GJ1,用于苗圃植物柑橘黄龙病(HLB) 的控制。转录组和等压标记RNA序列相对和绝对定量蛋白质组分析显示,经解淀粉芽孢杆菌GJ1处理和未经处理的亚洲鼠李菌感染柑橘的解毒反应存在差异。蛋白质组和转录组相关分析显示,蛋白质表达显著不同,在GO和KEGG 通路中基因表达也显著不同。解淀粉芽孢杆菌Q–426能产生具有抗真菌活性脂肽类化合物,起始pH对脂肽化合物生产影响很大。ZHAO等[40]采用比较蛋白质组学、二维电泳技术以及质谱技术对解淀粉芽孢杆菌 Q–426 进行了研究,发现pH 5.0时,一共鉴定出24个差异表达蛋白点;某些蛋白专门用于调节细菌素和芬荠素的合成,其中包括3种与压力反应相关的诱导性蛋白质(焦磷酸硫胺素依赖乙酰胆碱酯酶、丁二醇脱氢酶、2ABC型寡肽运输系统蛋白)和2个组成反应调节剂(DegU、分支酸变位酶PheB)。说明在不同pH条件下,解淀粉芽孢杆菌Q–426抗性存在天然差异,其原因是解淀粉芽孢杆菌产生了相关的诱导性蛋白质。
LU等[41]从解淀粉芽孢杆菌X030中分离到脂肽–杆菌霉素 Lb,发现其对多种肿瘤细胞(如 SMMC–7721和MDA–MB–231)具有抑制活性;全基因组和生长蛋白组学分析表明,杆状霉素 Lb 生物合成基因簇、关键酶基因、调控蛋白基因(、、和)以及其初级代谢相关基因与杆状霉素Lb合成之间具有相关性;添加氨基酸(如谷氨酸)和蔗糖的发酵上清液具有显著的抗肿瘤活性,说明其在代谢调控中起着关键作用。
2.2 多组学分析运用于解淀粉芽孢杆菌与植物共生分子机制的研究
2.2.1转录组学分析运用于解淀粉芽孢杆菌生物膜形成及植物促生机理的研究
解淀粉芽孢杆菌与植物可形成共生关系,促进植物生长。FAN等[42]为了解细菌–植物共生分子机制,在根际模拟生长条件下,利用差分RNA测序(drna–seq)系统分析了FZB42菌株初级转录组,发现有4 877个编码蛋白质基因的转录起始位点,确定了不同生长条件下的差异表达基因,并纠正了许多以前错误注释的基因;研究还发现,大量核糖开关和顺式编码反义RNA,以及可能在芽孢杆菌基因调控中起重要作用的反式小非编码RNA,提高了根杆菌与宿主的相互作用。KRÖBER等[43]在转录水平上研究了解淀粉芽孢杆菌FZB42生物膜的形成机制,并在生物膜形成和浮游生长条件下,对菌株整个转录组cDNA文库进行了高通量测序。通过比较转录组,发现了1组与基本细胞功能有关的常见高度转录基因;在2种生长条件下,菌体细胞中转录最快的基因为编码抗菌肽(AMP)基因;相比之下,在生物膜形成细胞中,编码抗菌特性次级代谢产物合成基因簇仅被适度转录,而不被诱导;转录组中有331个基因明显上调,230个基因下调;在最高上调基因中,出现操纵子,它编码与乳链菌肽(class I bacteriocin)抗性有关的产物,在果糖胺代谢中起作用的操纵子产物得到增强;在FZB42生物膜中,参与细胞外生物膜基质生产基因、胞外多糖基因()和编码纤维合成的操纵子在其转录过程中被上调;而鞭毛器合成、组装和调节基因以及芳香化合物降解基因和铜分泌等相关基因被强烈下调。
IRIZARRY等[44]利用基因芯片和RT–qPCR 检测了接种解淀粉芽孢杆菌的棉花幼苗根系中基因表达的变化,发现接种解淀粉芽孢杆菌的棉花幼苗根系中有252个转录体差异表达,139个转录体上调,113个转录体下调;部分上调的转录体与硝酸盐同化、细胞生长、激素分泌和转运、转录因子产生以及抗氧化作用有关;接种解淀粉芽孢杆菌的棉花幼苗根系中活性氧和木质素积累量较大,同时出现了多种功能基因的差异表达。说明解淀粉芽孢杆菌能引起幼苗根系复杂的遗传反应。GAMEZ等[45]对威廉姆斯香蕉中芽孢杆菌属和假单胞菌属的植物根际促生菌(PGPR)进行了评估,研究发现,植物体接种了解淀粉芽孢杆菌菌株Bs006和荧光假单胞菌Ps006之后,22个威廉姆香蕉基因中,大部分有差异表达。说明解淀粉芽孢杆菌Bs006 和荧光假单胞菌Ps006在不同时间点刺激不同基因群的定殖过程和水平受单独细菌的影响。
2.2.2蛋白质组学分析运用于解淀粉芽孢杆菌的定殖、促生的研究
SHAHZAD等[46]研究发现,在培养基中添加2%和3.5%甲醇的内生解淀粉芽孢杆菌RWL–1显著生长;在甲醇存在下,RWL–1产生的IAA水平明显提高,但ABA水平降低;酶抗氧化剂和功能性氨基酸(谷氨酸和丙氨酸)明显上调,并表达出8种不同类型蛋白质(包括解毒蛋白、抗氧化特性酶、蛋白酶、代谢酶、核糖体蛋白、抗氧化蛋白、伴侣蛋白和热休克蛋白);RWL–1在补充甲醇的培养基中生长时,可产生植物激素,特异性蛋白和不同生物化学物质的表达增加。QIU等[47]为了鉴定用于根系定殖和生物膜形成的关键蛋白,比较研究了浮游和根系定殖的解淀粉芽孢杆菌SQR9蛋白质谱系。研究表明,当SQR9菌株定殖于根部时,共鉴定出了755种蛋白质,其中有78种蛋白质显著增加,95种蛋白质显著减少,它们多是与根部定殖密切相关的功能性蛋白,包括生物控制、脱毒、生物膜形成、细胞运动和趋化、运输和植物多糖降解等功能蛋白;与浮游状态相比,双组分系统蛋白ResE增加了100倍;基因损伤可通过spooa–sini–yqxm途径调节双组分系统蛋白,推迟了细胞生物膜形成,降低了根系定殖能力。可见,SQR9蛋白组学数据为筛选植物根杆菌中的关键蛋白提供了有价值的线索。
2.3 转录组学分析运用于解淀粉芽孢杆菌诱导植物耐盐性的研究
解淀粉芽孢杆菌FZB42能诱导拟南芥产生系统耐盐性。LIU等[48]利用Illumina测序技术对盐胁迫下生长的拟南芥幼苗组织进行了大规模转录组测序。结果表明:FZB42接种后,在0 mmol/L NaCl处,有1 461个基因差异表达,其中953个基因上调,508个基因下调;在100 mmol/L NaCl处,有1 288个基因差异表达,其中1 024个基因上调,264个基因下调;FZB42接种后,盐敏感性降低,盐适应性增强; FZB42通过激活植物ET/JA信号而非ABA依赖途径诱导植物耐盐性。CHAUHAN等[49]选择解淀粉芽孢杆菌–SN13和水稻()研究了盐胁迫下植物与PGPR相互作用的复杂调控网络。研究发现,胁迫期间,接种SN13能显著提高水稻生物量、相对含水量、脯氨酸和可溶性总糖含量,降低脂质过氧化和电解质渗漏;在SN13 胁迫下,水稻根系转录组基因表达也发生了广泛变化,与盐胁迫或单独接种SN13 相比,根际细菌在盐胁迫下诱导了大量与光合作用、激素和胁迫反应相关基因以及细胞壁和脂质代谢相关基因的表达,表明根际细菌SN13 能减少盐胁迫的有害影响。为了验证RNA–seq数据,对不同功能类别(包括代谢、调节、应激反应和转运体)的表达基因进行了qRT–PCR。结果表明,在应力和非应力条件下,根系对SN13的响应存在质和量的差异,表明SN13在逆境中发挥了重要作用。
亚精胺诱导植物产生耐盐性的机理正在不断被揭示。CHEN等[50]研究发现,接种淀粉芽孢杆菌SQR9能提高拟南芥和玉米的耐盐性;利用不同截留分子量透析袋对SQR9分泌的分子进行了分离,发现亚精胺有助于提高植物耐盐性,SQR9精胺缺乏突变体无法诱导植物耐盐性;而与谷胱甘肽(GSH)生物合成途径相关基因的突变,能破坏植物耐盐性诱导。实时定量PCR证实,由SQR9产生的精胺可导致谷氨酰胺合成酶和谷胱甘肽还原酶基因表达增加,从而导致谷胱甘肽水平升高,这对清除活性氧物质至关重要。由SQR9衍生的精胺也上调了和的表达,后者将Na+隔离到液泡中,并将Na+从细胞中排出,从而降低离子毒性。可见,亚精胺有助于诱导植物产生耐盐性,其原因是它能提高谷胱甘肽(GSH)水平,产生一系列连锁反应,并提高相关基因表达,最后使植物产生耐盐性。
根据转录组研究,解淀粉芽孢杆菌某一功能发挥作用是多种基因上调和下调联合效应引起的。由此推断,解淀粉芽孢杆菌某种生理功能呈现是多基因共同协调作用的结果,通过转录组变化研究解淀粉芽孢杆菌–植物–病原菌之间作用是一条有效途径。
3 解淀粉芽孢杆菌基因洗牌与功能变化的比较蛋白组学分析
基因组洗牌是基因组工程的重要手段,比较蛋白组学分析运用于解淀粉芽孢杆菌基因洗牌菌株功能变化的研究是当前的一个热点。ZHAO等[51]运用比较蛋白质组学分析了检测亲本菌株和基因组洗牌菌株FMB38蛋白表达情况,通过二维电泳和质谱分析了蛋白质组。通过凝胶成像,检测到解淀粉芽孢杆菌基因洗牌菌株FMB38有51个差异表达蛋白点,具有2倍高光斑密度;通过银染和进一步质谱分析,检测到另外的46个蛋白质点。结果说明,在基因组洗牌突变子中,引起特别表达的46个蛋白质点中有15个与代谢相关,5个与DNA复制、重组和修复有关,6个与转录和后转录修饰有关,1个与细胞分泌和信号传导机制有关,3个与表面活性素合成相关,2个与能量生产和转换相关,14个与其他方面的功能相关。说明通过基因组洗牌,突变子代谢能力得到提高和改善。ZHAO等[52]利用基因重组技术提高了原菌株解淀粉芽孢杆菌ES–2–4的芬荠素产量。经过两轮基因组重组,获得了1株高产重组FMB72菌株,产量增加了8.30倍;采用基质辅助激光解吸电离飞行时间质谱法,筛选出50个不同表达的斑点(<0.05),根据NCBI数据库,确定了44个蛋白斑点;在44种已鉴定的蛋白质中,信号蛋白ComA 和 Spo0A可能在转录水平上调控芬荠素的合成。可见,通过蛋白组学分析可以揭示解淀粉芽孢杆菌更多的功能及分子机制。
4 展望
结合解淀粉芽孢杆菌多组学分析研究进展,分析归纳其功能分子机制,包括抗性形成、生物膜形成、定殖、促生、诱导植物耐盐性等机制。随着多组学研究深入,其重要功能机理将进一步被阐明。但要看到,其生理功能不仅依赖于自身基因组,也受环境(生境)、相关植物病原菌影响。今后的研究需从两方面入手:①研究其生理功能基因调控网络,明确功能机制复杂性,使用多组学联合阐明其分子机理;②研究“解淀粉芽孢杆菌–植物宿主–植物病原菌”复杂关系,挖掘三者相互作用规律,全面阐明其生理功能的分子机制。
[1] 刘润泽,王世伟,王卿惠,等.解淀粉芽孢杆菌酶系及其应用研究进展[J].高师理科学刊,2019,39(3):70–75. LIU R Z,WANG S W,WANG Q H,et al.Study advances in enzyme systems and their applications of[J].Journal of Science of Teachers' College and University,2019,39(3):70–75.
[2] DING L S,ZHANG S,GUO W B,et al.Exogenous indole regulates lipopeptide biosynthesis in AntarcticPc3[J].Journal of Microbiology and Biotechnology,2018,28(5):784–795.
[3] 闫晓妮,马天有,杜仁佳,等.解淀粉芽胞杆菌胞外抑菌活性物质研究现状[J].中国微生态学杂志,2018,30(2):229–234,249. YAN X N,MA T Y,DU R J,et al.Extracellular antibacterial compounds produced by:research progress[J].Chinese Journal of Microecology,2018,30(2):229–234,249.
[4] SUN J,QIAN S Q,LU J,et al.Knockout ofimproves the bacillomycin D yield based on de novo genome sequencing offmbJ[J].Journal of Agricultural and Food Chemistry,2018,66(17):4422–4430.
[5] YAN Y Z,LIU S Y,WANG D L,et al.Complete genome sequence ofB15 isolated from grape skin,a strain of strong inhibitory activity against fungi[J].Journal of Biotechnology,2016,228:28–29.
[6] GAMEZ R M,RODRÍGUEZ F,BERNAL J F,et al. Genome sequence of the banana plant growth-promoting rhizobacteriumBS006[J]. Genome Announc,2015,3(6):e01391–15.
[7] SUN Z X,HSIANG T,ZHOU Y,et al.Draft genome sequence ofXK-4-1,a plant growth-promoting endophyte with antifungal activity[J]. Genome Announcements,2015,3(6):e01306–15.
[8] JEONG H ,PARK S H ,CHOI S K .Genome sequence of antibiotic-producingstrain KCTC 13012[J].Genome Announcements,2015,3(5):e01121–15.
[9] ZHANG S M,JIANG W,LI J,et al.Whole genome shotgun sequence ofTF28,a biocontrol entophytic bacterium[J].Standards in Genomic Sciences,2016,11:73.
[10] QIN Y X,HAN Y Z,YU Y Q,et al.Complete genome sequence ofL-S60,a plant growth-promoting and antifungal bacterium[J].Journal of Biotechnology,2015,212:67–68.
[11] NASRIN S,HOSSAIN M J,LILES M R.Draft genome sequence ofAP183 with antibacterial activity against methicillin-resistant[J].Genome Announcements,2015,3(2):e00162–15.
[12] ZHAO X,DE JONG A,ZHOU Z J,et al.Complete genome sequence ofstrain BH072,isolated from honey[J].Genome Announcements, 2015,3(2):e00098–15.
[13] MONTOR-ANTONIO J J,SACHMAN-RUIZ B,LOZANO L,et al.Draft genome sequence ofJJC33M,isolated from sugarcane soils in the Papaloapan region,Mexico[J].Genome Announce- ments,2015,3(1):e01519–14.
[14] KIM B Y,LEE S Y,AHN J H,et al.Complete genome sequence ofsubsp.CC178,a phyllosphere bacterium antagonistic to plant pathogenic fungi[J].Genome Announcements,2015,3(1):e01368–14.
[15] QIN Y X,HAN Y Z,SHANG Q M,et al.Complete genome sequence ofL-H15,a plant growth promoting rhizobacteria isolated from cucumber seedling substrate[J].Journal of Biotechnology, 2015,200:59–60.
[16] CHOI S K,JEONG H,KLOEPPER J W,et al.Genome sequence ofGB03,an active ingredient of the first commercial biological control product[J].Genome Announcements,2014,2(5):e01092–14.
[17] KURATA A,HIROSE Y,MISAWA N,et al.Draft genome sequence of the ionic liquid-tolerant bacteriumCMW1[J].Genome Announce- ments,2014,2(5):e01051–14.
[18] LIU X Y,MIN Y,WANG K M,et al.Draft genome sequence ofHB-26[J]. Standards in Genomic Sciences,2014,9(3):775–782.
[19] GUPTA R,VAKHLU J,AGARWAL A,et al.Draft genome sequence of plant growth-promotingstrain W2 associated with(saffron)[J].Genome Announcements,2014,2(5):e00862–14.
[20] NIAZI A,MANZOOR S,BEJAI S,et al.Complete genome sequence of a plant associated bacteriumsubsp.UCMB5033[J]. Standards in Genomic Sciences,2014,9(3):718–725.
[21] MOLINATTO G,PUOPOLO G,SONEGO P,et al. Complete genome sequence ofsubsp.S499,a rhizobacterium that triggers plant defences and inhibits fungal phytopathogens[J]. Journal of Biotechnology,2016,238:56–59.
[22] WANG Y,WANG X,GREENFIELD P,et al.Draft genome sequence ofEBL11,a new strain of plant growth-promoting bacterium isolated from rice rhizosphere[J].Genome Announcements,2014,2(4):e00732–14.
[23] SANTANNA B M M,MARBACH P P A,ROJAS- HERRERA M,et al.High-quality draft genome sequence ofstrain 629,an endophyte from[J].Genome Announcements,2015,3(6):e01325–15.
[24] CAI J,LIU F,LIAO X D,et al.Complete genome sequence ofLFB112 isolated from Chinese herbs,a strain of a broad inhibitory spectrum against domestic animal pathogens[J].Journal of Biotechnology,2014,175:63–64.
[25] WU H,QIAO J,BLOM J,et al.The rhizobacteriumsubsp.NAU-B3 contains a large inversion within the central portion of the genome[J].Genome Announcements,2013,1(6):e00941–13.
[26] SCHOFIELD B J,SKARSHEWSKI A,LACHNER N,et al.Near complete genome sequence of the animal feed probiotic,H57[J].Standards in Genomic Sciences,2016,11(1):1–7.
[27] NIAZI A,MANZOOR S,ASARI S,et al.Genome analysis ofsubsp.UCMB5113:a rhizobacterium that improves plant growth and stress management[J].PLoS One,2014,9(8):e104651.
[28] HOSSAIN M J,RAN C,LIU K,et al.Deciphering the conserved genetic loci implicated in plant disease control through comparative genomics ofsubsp.[J].Frontiers in Plant Science,2015,6:631.
[29] BELBAHRI L,CHENARI BOUKET A,REKIK I,et al. Comparative genomics ofstrains reveals a core genome with traits for habitat adaptation and a secondary metabolites rich accessory genome[J].Frontiers in Microbiology,2017,8:1438.
[30] ZHANG N,YANG D Q,KENDALL J R A,et al. Comparative genomic analysis ofandreveals evolutional traits for adaptation to plant-associated habitats[J].Frontiers in Microbiology,2016,7:2039.
[31] 王世伟,王卿惠,李小鹏,等.镰刀菌分子鉴定与重要应用的研究进展[J].微生物学通报,2018,45(4):907–919. WANG S W,WANG Q H,LI X P,et al.Progress in molecular identification in the genusand its important applications[J].Microbiology China,2018,45(4):907–919.
[32] LIAO Y L,WANG B,YE Y R,et al.Determination and optimization of a strong promoter element fromby using a promoter probe vector[J]. Biotechnology Letters,2018,40(1):119–126.
[33] LIAO Y L,YE Y R,WANG B,et al.Optimization of the purine operon and energy generation infor guanosine production[J]. Biotechnology Letters,2017,39(11):1675–1682.
[34] FAN B,LI Y L,MARIAPPAN A,et al.New SigD-regulated genes identified in the rhizobacteriumFZB42[J].Biology Open,2016,5(12):1776–1783.
[35] ZHAO X,WANG Y,SHANG Q H,et al.Collagen-like proteins (ClpA,ClpB,ClpC,and ClpD) are required for biofilm formation and adhesion to plant roots byFZB42[J].PLoS One,2015,10(2):e0117414.
[36] BUDIHARJO A,CHOWDHURY S P,DIETEL K,et al. Transposon mutagenesis of the plant-associatedssp.FZB42 revealed that theandgenes are involved in plant-microbe-interactions[J].PLoS One,2014,9(5):e98267.
[37] GOODSON J R,KLUPT S,ZHANG C X,et al.LoaP is a broadly conserved antiterminator protein that regulates antibiotic gene clusters in[J]. Nature Microbiology,2017,2(5):17003.
[38] LIU D,LI K Y,HU J L,et al.Biocontrol and action mechanism ofandin soybean phytophthora blight[J].International Journal of Molecular Sciences,2019,20(12):2908.
[39] TANG J Z,DING Y X,NAN J,et al.Transcriptome sequencing and ITRAQ reveal the detoxification mechanism ofGJ1,a potential biocontrol agent for Huanglongbing[J].PLoS One,2018,13(8):e0200427.
[40] ZHAO J,ZHAO P C,QUAN C S,et al.Comparative proteomic analysis of antagonisticQ-426 cultivated under different pH conditions[J]. Biotechnology and Applied Biochemistry,2015,62(4):574–581.
[41] LU J Y,ZHOU K X,HUANG W T,et al.A comprehensive genomic and growth proteomic analysis of antitumor lipopeptide bacillomycin Lb biosynthesis inX030[J].Applied Microbiology and Biotechnology,2019,103(18):7647–7662.
[42] FAN B,LI L,CHAO Y J,et al.dRNA-seq reveals genomewide TSSs and noncoding RNAs of plant beneficial rhizobacteriumFZB42[J]. PLoS One,2015,10(11):e0142002.
[43] KRÖBER M,VERWAAIJEN B,WIBBERG D,et al. Comparative transcriptome analysis of the biocontrol strainFZB42 as response to biofilm formation analyzed by RNA sequencing[J]. Journal of Biotechnology,2016,231:212–223.
[44] IRIZARRY I,WHITE J F.alters gene expression,ROS production and lignin synthesis in cotton seedling roots[J].Journal of Applied Microbiology,2018,124(6):1589–1603.
[45] GAMEZ R M,RODRÍGUEZ F,VIDAL N M,et al. Banana() transcriptome profiling in response to rhizobacteria:Bs006 andPs006[J].BMC Genomics,2019,20(1):1–20.
[46] SHAHZAD R,KHAN A L,WAQAS M,et al.Metabolic and proteomic alteration in phytohormone-producing endophyticRWL-1 during methanol utilization[J].Metabolomics,2019,15(2):1–16.
[47] QIU M H,XU Z H,LI X X,et al.Comparative proteomics analysis ofSQR9 revealed the key proteins involved in in situ root colonization[J].Journal of Proteome Research,2014,13(12):5581–5591.
[48] LIU S F,HAO H T,LU X,et al.Transcriptome profiling of genes involved in induced systemic salt tolerance conferred byFZB42 in[J].Scientific Reports,2017,7(1):10795.
[49] CHAUHAN P S,LATA C,TIWARI S,et al. Transcriptional alterations reveal- rice cooperation under salt stress[J].Scientific Reports,11912(2019).
[50] CHEN L,LIU Y P,WU G W,et al.Beneficial rhizobacteriumSQR9 induces plant salt tolerance through spermidine production[J]. Molecular Plant-Microbe Interactions,2017,30(5):423–432.
[51] ZHAO J F,CAO L,ZHANG C,et al.Differential proteomics analysis ofand its genome-shuffled mutant for improving surfactin production[J]. International Journal of Molecular Sciences,2014,15(11):19847–19869.
[52] ZHAO J F,ZHANG C,LU Z X.Differential proteomics research ofand its genome- shuffled saltant for improving fengycin production[J]. Brazilian Journal of Microbiology,2018,49:166–177.
Advances in application of multi-component analysis on the functional mechanism study of
WANG Shiwei1,2, WANG Qinghui3, ZHAI Liping1,2, LIU Jun1,2, YU Zhidan1,2, XIANG Wensheng3*
(1.College of Life Sciences and Agriculture and Forestry, Qiqihar University, Qiqihar, Heilongjiang 161005, China; 2.Key Laboratory of Resistance Gene Engineering and Preservation of Biodiversity in Cold Areas, Qiqihar, Heilongjiang 161005, China; 3.School of Life Science, Northeast Agricultural University, Harbin, Heilongjiang 150030, China)
have beneficial biological characteristics and can produce many kinds of antimicrobial active substances. They own important physiological and biochemical functions, such as biofilm formation and colonization in plants, promoting plant growth and induction of salt tolerance in plants, etc. In this review, we summarized the molecular mechanisms study using multi-omics analysis progress ofin studying antimicrobial activity, colonization, growth promotion and salt tolerance induction of plants, with the aim of better application of.
; multi-omics analysis; identification; function; molecular mechanisms

S476;Q811.4
A
1007-1032(2020)04-0410-09
10.13331/j.cnki.jhau.2020.04.005
王世伟,王卿惠,翟丽萍,刘军,于志丹,向文胜.多组学分析在解淀粉芽孢杆菌相关功能机制研究中的应用进展[J].湖南农业大学学报(自然科学版),2020,46(4):410–418.
WANG S W,WANG Q H, ZHAI L P, LIU J, YU Z D, XIANG W S. Advances in application of multi-component analysis on the functional mechanism study of[J]. Journal of Hunan Agricultural University (Natural Sciences), 2020, 46(4): 410–418.
http://xb.hunau.edu.cn
2019–10–02
2019–11–28
黑龙江省教育厅基本科研业务费科研项目(No.135309482)
王世伟(1965—),男,山东肥城人,博士,教授,主要从事微生物遗传与发酵工程研究,wsw888535@shou.com;
,向文胜,博士,教授,主要从事酶反应机制和代谢途径的研究,xiagwensheng@yahoo.com.cn
责任编辑:毛友纯
英文编辑:柳正
