印度洋、西北太平洋龟螺属和小龟螺属种类的分类及DNA条形码鉴定
2020-05-23李海涛何静姜重臣陈志云周鹏
李海涛,何静,姜重臣,陈志云,周鹏
(1. 自然资源部南海环境监测中心,广东 广州 510300;2. 中国科学院南海海洋研究所 热带海洋生物资源与生态重点实验室,广东 广州 510301)
1 引言
工业革命以来二氧化碳大量排放导致的全球变暖和海洋酸化,已经对全球海洋生态系统产生了深刻影响[1-3]。无论是钙化生物还是非钙化生物,全球变暖和海洋酸化均会对其造成一系列影响,如影响基因表达、繁殖力、幼体成活率等[4-6]。此外,全球变暖和海洋酸化还会影响物种的分布、生物群落的多样性和结构[7]。
有壳翼足类(Thecosome Pteropods)是海洋浮游动物的重要类群,在海洋食物链中扮演着十分关键的角色,它们不仅是重要的初级消费者,也是其他海洋生物和海鸟的饵料[8-9]。有壳翼足类的外壳主要由文石构成[10],相比方解石,文石更易溶解于海水中[11]。因此,有壳翼足类被广泛用作海洋酸化监测的指示物种[6, 12]。
准确的物种鉴定是开展海洋酸化等环境变化对翼足类影响研究的前提,因为不同的物种会对环境变化产生不一样的响应[13]。然而,我国对翼足类的分类学研究仍远远不足。张福绥[14]在半个多世纪前较为系统地开展了中国海浮游软体动物(包括翼足类、异足类和海蜗牛)的分类学研究,此后国内的分类结论[15]基本沿用了其观点,鲜有其他关于浮游软体动物的分类报道。国外学者[16-17]提出一些新的翼足类分类系统尚未被国内采用,许多分类错误也没得到订正,如张福绥[14]对强卷螺属(Agadina)、厚唇螺(Diacriatrispinosa)和蛆状螺(Cuvierinacolumnella)等种类的鉴定有误[18-20]。DNA条形码技术[21]的出现为物种鉴定提供了新的手段,该技术能有效回避鉴定者对生物个体形态差异的主观认识和判断。近年来,DNA条形码技术已被广泛应用于翼足类的分类学、种类鉴定和地理群体的遗传分化等方面的研究[13,22-23]。
龟螺属(Cavolinia)和小龟螺属(Diacavolinia)是翼足类龟螺科(Cavoliniidae)龟螺亚科(Cavoliniinae)下的2个属。目前,我国只采用了前1个属,将后1个属的种类均归入到前者之中。有壳翼足类的分类主要是根据贝壳的形态特征,但由于其贝壳形态较为简单,因而难以进行准确的物种鉴定[22]。龟螺属和小龟螺属的分类极为混乱,被人为建立了大量的亚种和变形[24-25]。比如,长期以来小龟螺属被认为仅有长吻小龟螺(D. longirostris)1 个种[26-27],后来 van der Spoel等[25]根据细微的形态差异将这1个种分出22个种及4个变形。但通过对比COI序列,分布于东北大西洋的小龟螺属4个形态种被证明为同一物种[28]。因此,龟螺属和小龟螺属的种及种以下分类单元的有效性尚需通过分子手段来加以证实。本研究分析了西北太平洋和印度洋的龟螺属和小龟螺属种类的形态特征,同时结合线粒体COI和16S基因序列对其进行物种鉴定,以期解决两属部分种类的分类混乱和争议,并揭示其地理遗传分化。
2 材料与方法
2.1 样品采集
样品于2016年至2018年采自西北太平洋(含南海)及印度洋海域的12个站位(图1)。使用大型浮游生物网采用垂直拖网和水平拖网相结合的方式采集浮游动物样品,绝大部分样品为夜间用水平拖网的方式采集。海上采集的浮游动物样品经筛绢过滤后用无水酒精固定(酒精使用量约为浮游动物体积的3~4倍),冰冻保存带回实验室。体视镜下对目标种类进行分选和初步的分类鉴定,并编号拍照。共计43个标本用于DNA序列的分析(表1)。
2.2 DNA提取、PCR扩增和测序
取部分或整只动物,采用碱裂解法[29]提取DNA。使用引物LCO-1490(5′-GGT CAA CAA ATC ATA AAG ATA TTG G-3′)和 HCO-2198 (5′-TAA ACT TCA GGG TGA CCA AAA AAT CA-3′)[30]扩增COI基因片段;使用16sar-L(5′-CGC CTG TTT ATC AAA AAC AT-3′)和 16sbr-H(5′-CCG GTC TGA ACT CAG ATC ACG T-3′)[31]扩增 16S rRNA基因片段。
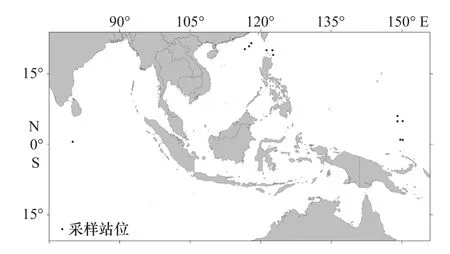
图1 采样站点分布Fig. 1 Distribution of sampling sites
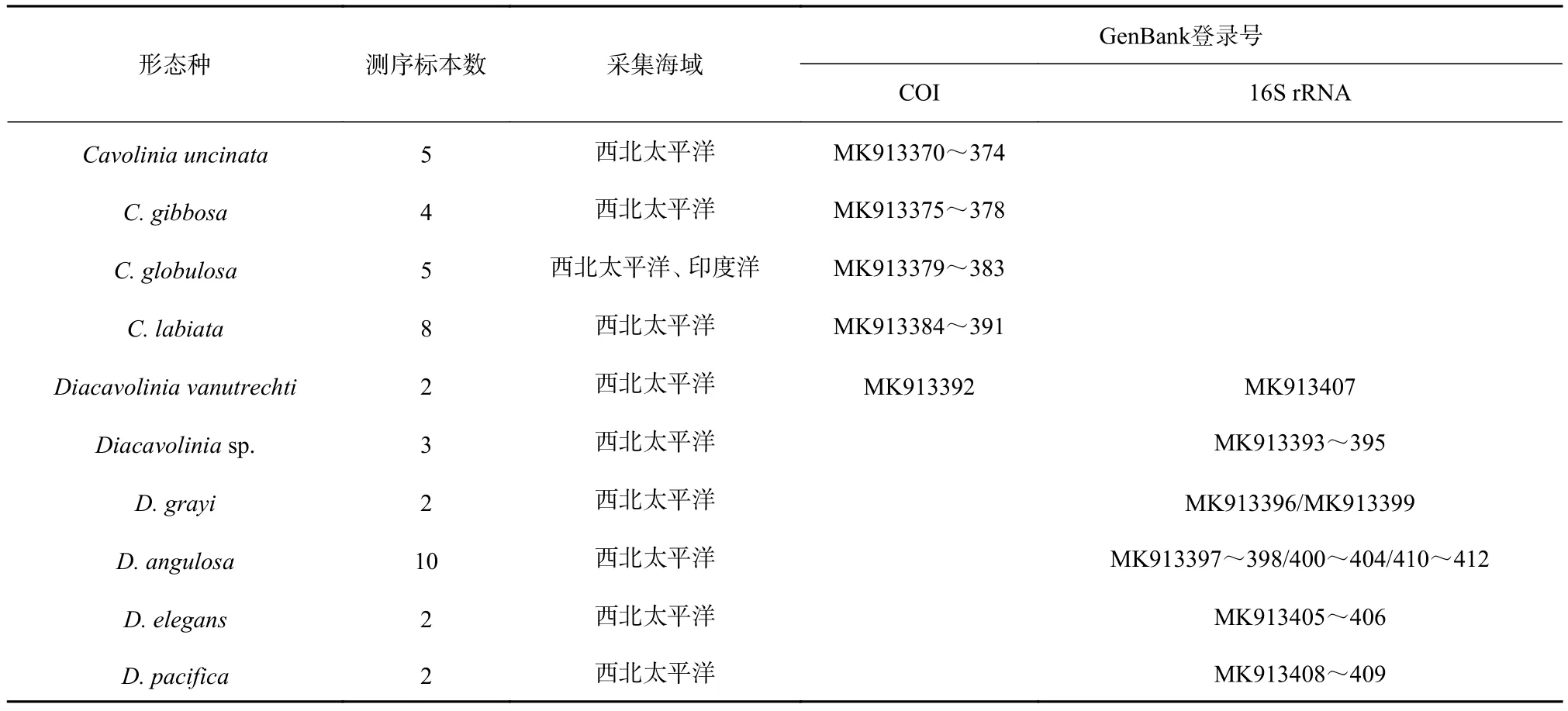
表1 样品采集信息和基因库(GenBank)登录号Table 1 Collection information and GenBank accession numbers for specimens analyzed in this study
PCR反应体系总体积为50 μL,包括:2×扩增缓冲溶液25 μL,脱氧核糖核苷三磷酸(Deoxy-ribonucleoside Triphosphate,dNTP)400 μmol/L,引物各0.3 μmol/L,高保真 PCR 酶(TOYOBO,KOD FX)1.0 U,模板DNA 1 μL,加去离子水补足至50 μL。PCR反应条件为:94℃ 预变性 2 min;98℃ 变性 15 s,53℃ 退火 30 s,68℃延伸60 s,共30个循环;最后68℃延伸10 min。PCR产物送生工生物工程(上海)股份有限公司广州分公司进行回收纯化和测序,COI基因片段进行双向测序,16S rRNA基因片段进行反向引物测序(个别样品为双向测序),测序引物与扩增引物相同。
2.3 系统学分析
拼接、校对后的测序结果,首先利用美国国家生物技术信息中心(NCBI)的BLAST程序(https://blast.ncbi.nlm.nih.gov/Blast.cgi)进行序列同源性检索,并将COI基因序列在综合性序列分析工具软件DAMBE 5[32]中翻译成氨基酸序列,以确保测序获得的序列不是线粒体假基因(Nuclear Mitochondrial Pseudogenes)。本文测序获得的序列连同基因库(GenBank)中下载的其他序列,使用DAMBE 5内置的Clustal W程序进行多重序列比对。使用分子进化遗传分析软件MEGA 7.0[33]计算碱基的组成,基于Kimura 双参数(Kimura 2-Parameter,K2P)模型计算遗传距离,采用邻接法(Neighbor-joining,NJ)构建系统进化树,可靠性经过1 000次自展(Bootstrap)检验。采用进化树分析软件MrBayes 3.2[34]构建贝叶斯树(Bayesian Inference,BI),分子进化模型使用核苷酸替换DNA进化最优模型分析软件jModelTest 2.1[35]根据贝叶斯信息准则(Bayesian Information Criteria)标准计算得出。COI和16S rRNA基因的最佳进化模型分别为HKY+I+G和HKY+G。在马尔可夫链蒙特卡罗(Markov Chain Monte Carlo)参数下运行 300 万代,每100代抽样一次,前2 500代作为老化样本舍去。BI树各分支置信度以后验概率表示。使用进化树作图软件FigTree 1.4.3显示BI树的拓扑结构图。
3 结果
3.1 形态特征
在西北太平洋和印度洋海域共采集到4个龟螺属种类,分别为钩龟螺(C. uncinata)(图2a至图2c)、驼龟螺(C. gibbosa)(图 2d)、球龟螺(C. globulosa)(图 2e)和宽弯龟螺(C. labiata)(图 2f)。其中,钩龟螺的贝壳形态呈连续的变化,小个体的侧棘(侧突起)显得更为发达,尾棘向背方弯曲的程度也更大(图2a),腹壳接近圆形(图2a至图2b);大个体的侧棘不明显,尾棘弯曲程度较小(图2c)。
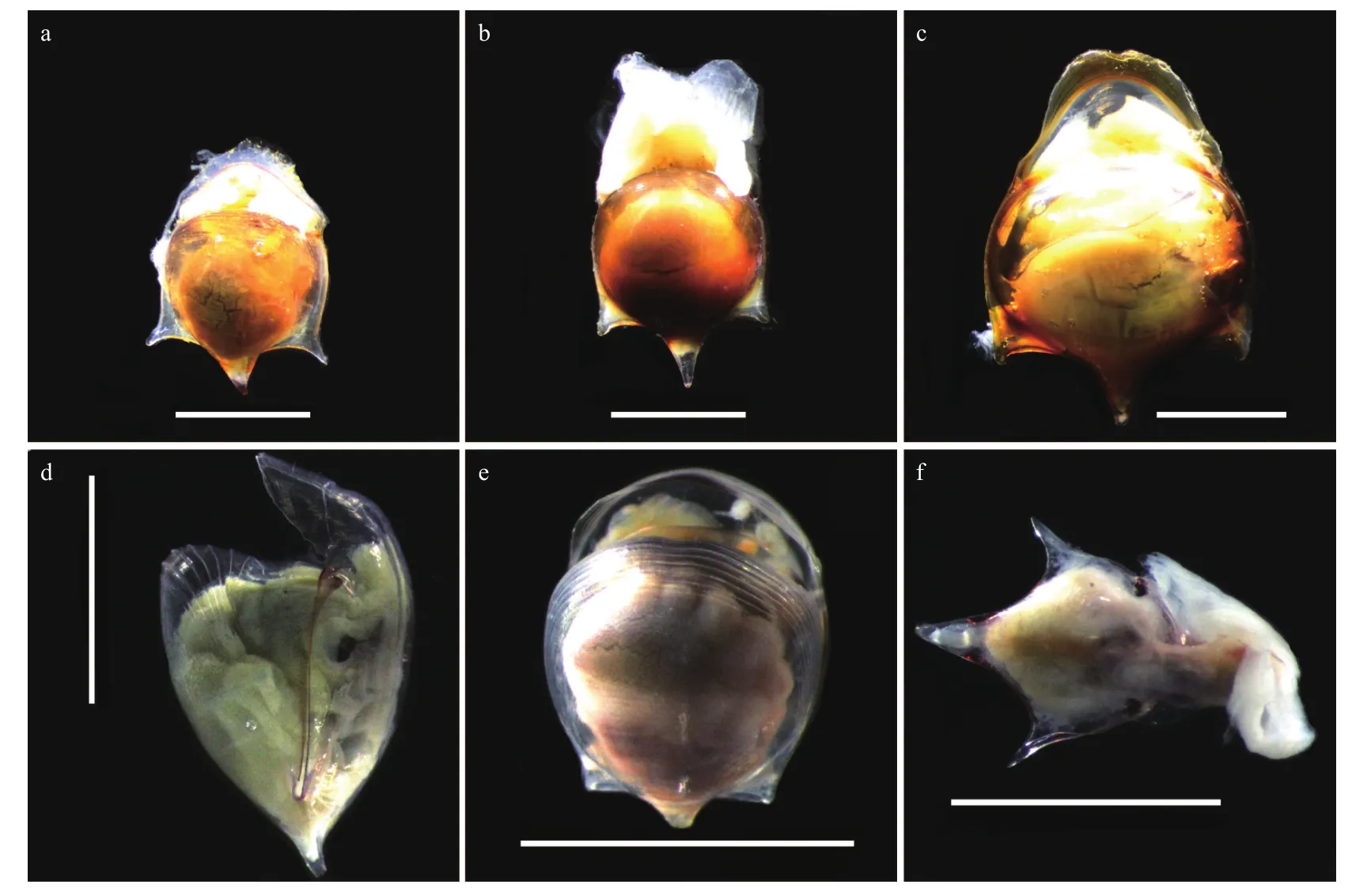
图2 龟螺属种类的贝壳形态(比例尺为5 mm)Fig. 2 Shell morphology of Cavolinia species (scale bars: 5 mm)
小龟螺属具有极其丰富的形态多样性(图3a,图3c,图 3e,图 3g,图 3i至图 3l),其侧突起尖锐(图 3a,图 3c)或极发达(图 3i)或不明显(图 3j至图 3l),背壳前缘部分凹陷(图3e,图3i至图3k)或不凹陷(图3g,图3l)。参照文献[25]进行形态鉴定,部分个体可明确鉴定为D. grayi(图 3a,图 3b)、D. vanutrechti(图 3c,图 3d)、D. pacifica(图 3e,图 3f)、D. elegans(图 3g,图 3h)和D. angulosa(图 3j,图 3k,图 4)等形态种。部分个体难以进行形态种的划分,如图3i所示的标本与D. flexipes(图4)的侧突起一样发达,但前者背壳前缘部分远不及后者宽大。图3l所示的标本,虽然其侧突起不及图3c的发达,但其形态特征与D. vanutrechti的正模标本(图4)十分相似,故将其也定为D. vanutrechti。
3.2 序列分析
测序共获得23条COI序列(GenBank登录号MK913370~MK913392)和20条16S rRNA基因序列(GenBank 登录号 MK913393~MK913412)。本文共计扩增了近30个小龟螺属标本的COI基因,扩增并测序成功的12条序列,经BLAST比对和序列分析后发现,仅有1个标本(图3l)的序列与GenBank中的同种(形态种)具有较高的同源性。其余11条序列与龟螺属和小龟螺属种类的同源性很低,且存在一段3 bp的插入/缺失片段,我们推测这些序列为线粒体假基因(序列未提交GenBank)。这些线粒体假基因均能正确翻译为氨基酸序列,没有产生终止密码子,且核苷酸变异位点大多数(75.5%)为密码子第三位碱基。
真正的线粒体COI序列长658 bp,A、T、G、C平均含量分别为24.3%、40.7%、18.0%和17.0%。线粒体假基因序列长655 bp,A、T、G、C平均含量分别为23.3%、38.3%、20.1%和18.3%。16S rRNA基因序列长 369~372 bp,存在 5个插入/缺失位点,A、T、G、C平均含量分别为30.7%、30.9%、21.5%和17.0%。所有序列的A+T的含量均明显高于G+C的含量。
3.3 系统发育和遗传距离
线粒体COI和16S rRNA基因的BI树和NJ树有大体一致的拓扑结构,本文只显示BI树或NJ树(图4,图5)。在16S rRNA基因系统树中,分布于西北太平洋的小龟螺属D. grayi、D. vanutrechti、D. pacifica、D.elegans、D. angulosa等形态种聚为一个分支,并获得很高的支持率(图4)。因此,这些不同的形态种可能属于同一物种,即长吻小龟螺(D. longirostris)。
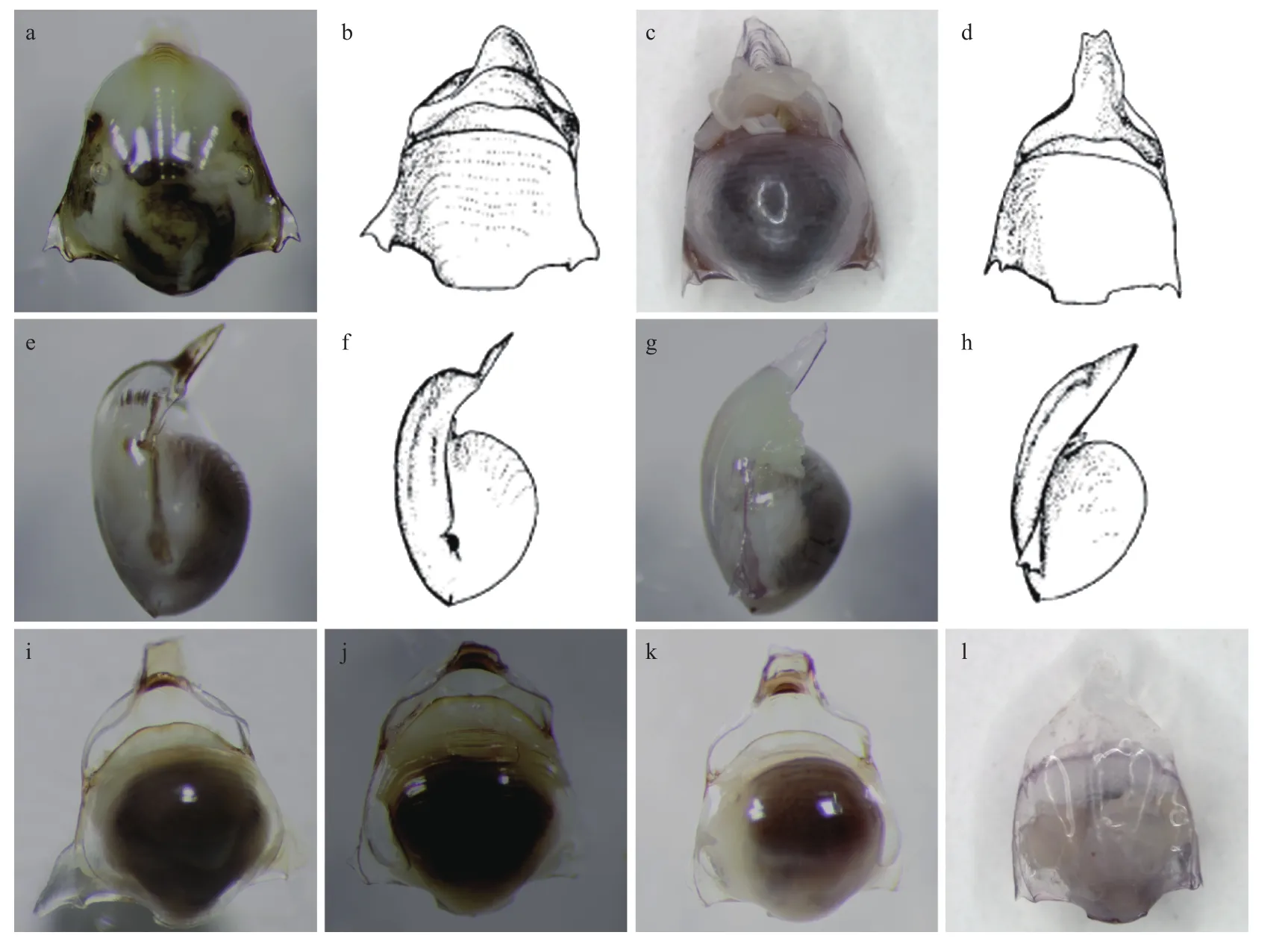
图3 不同形态类型的小龟螺Fig. 3 Different morphotypes of Diacavolinia
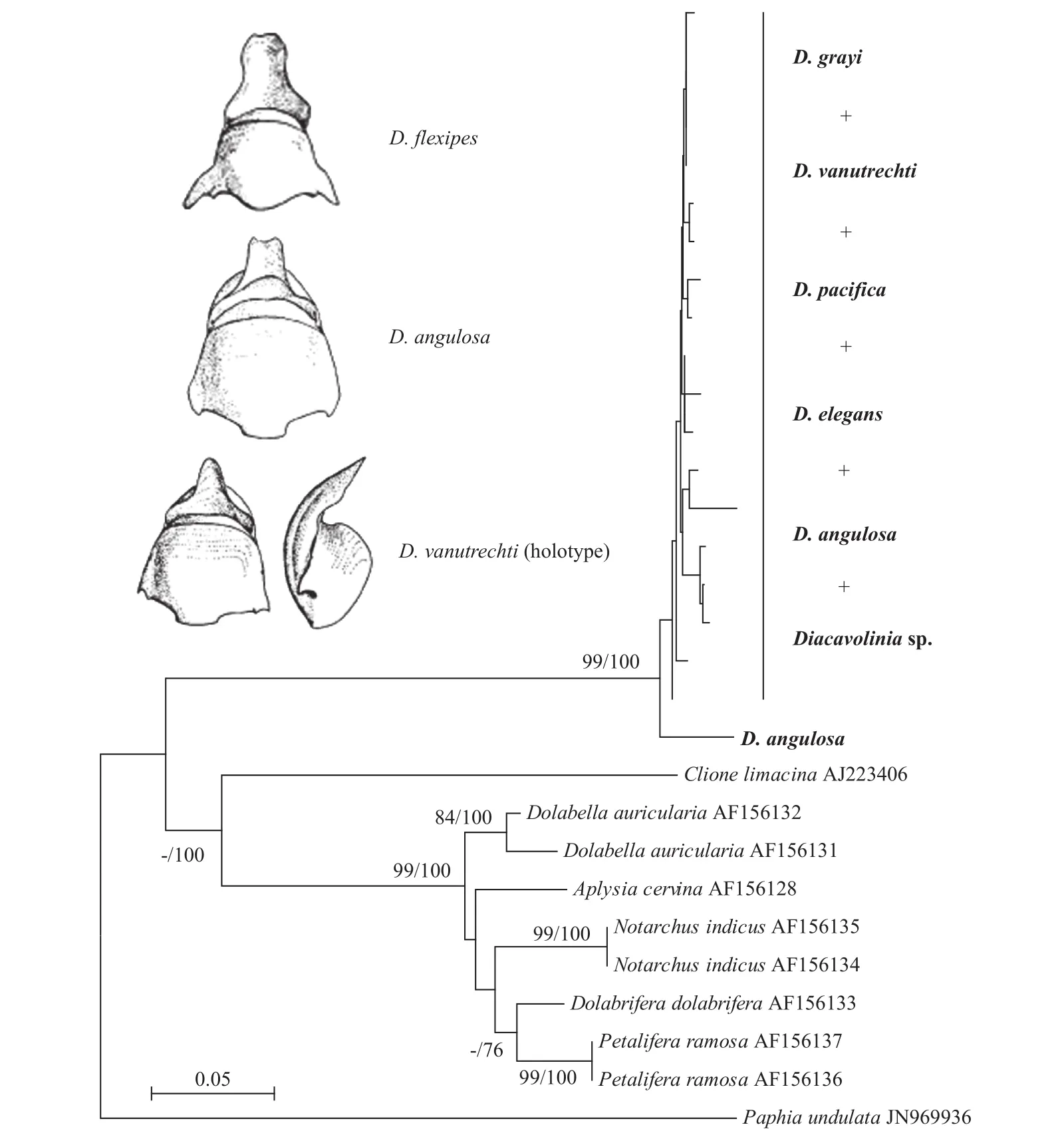
图4 基于16S rRNA基因构建的小龟螺和其他腹足类邻接系统发育树Fig. 4 Neighbor-joining phylogenetic tree of genus Diacavolinia and other gastropods based on mitochondrial 16S rRNA gene sequences
COI基因系统分析结果表明,钩龟螺、球龟螺和长吻小龟螺在系统树中均形成2个地理支系并获得100%的支持率,且3个种类都分别形成大西洋和印度洋-西北太平洋地理支系(图5)。其余种类则在COI系统树中形成单系群。
COI基因的K2P遗传距离分析表明,种内/支系内的遗传距离在0~0.025之间,平均值为0.009;种间/支系间的遗传距离则在0.082~0.393之间,平均值为0.232;钩龟螺、球龟螺和长吻小龟螺3个形态种不同支系间的遗传距离则在0.102~0.246之间,平均值为0.137。16S rRNA基因的K2P遗传距离分析表明,除1条D. angulosa(GenBank登录号 MK913410)与其余序列遗传距离较大外(0.039~0.053,平均值0.044),其余 19 条序列(包括D. grayi、D. vanutrechti、D. pacifica、D. elegans、D. angulosa等形态种)的遗传距离在0~0.033之间,平均值为0.012。小龟螺属与数据库中唯一的1条翼足类16S rRNA基因序列(Clione limacine,AJ223406)的遗传距离在0.400以上。
4 讨论
本文对西北太平洋小龟螺属标本16S rRNA基因序列的分析结果表明,D. grayi、D. vanutrechti、D. pacifica、D. elegans、D. angulosa等形态种可能为同1个种。Maas等[28]的研究结果也表明,东北大西洋小龟螺属的4个形态种实为1种。因此,van der Spoel等[25]基于形态差异对小龟螺属物种的划分无法得到DNA数据的支持,可能过高估计了该属的物种多样性。侧突起的发达程度,背壳前缘的凹陷程度这些连续变异的数量性状显然无法作为种类区分的依据。小龟螺属的种类多样性和分类学问题,尚需通过更大范围的采样并结合多方面的证据来解决。当前,将小龟螺属作为单型属处理更妥,即仅包含长吻小龟螺一个种。
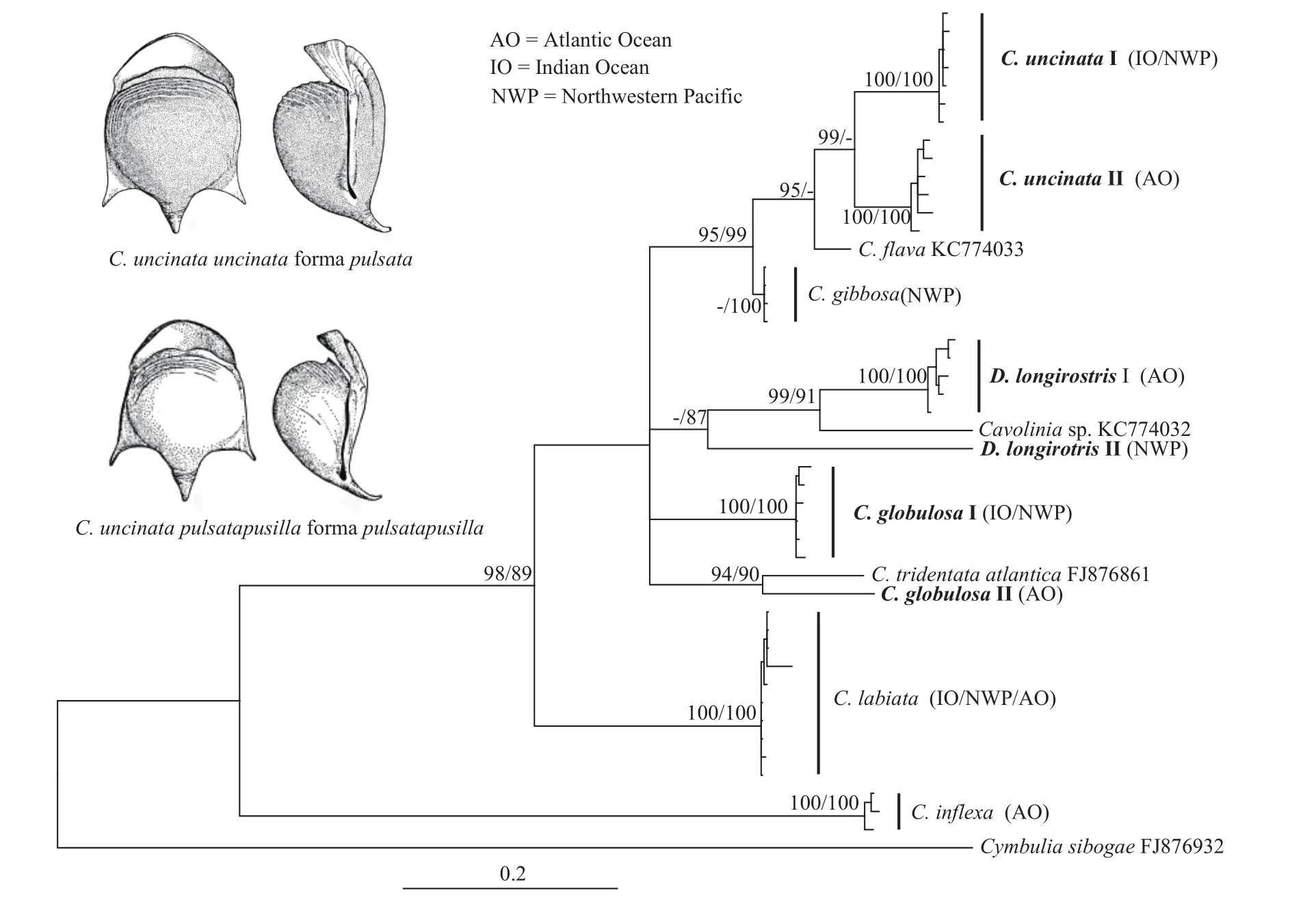
图5 基于COI基因构建的龟螺属和小龟螺属贝叶斯系统发育树Fig. 5 Bayesian inference phylogenetic tree of genera Cavolinia and Diacavolinia based on mitochondrial COI gene sequences
宽弯龟螺在我国被作为C. inflexa的变种来处理[14],前者与后者存在明显的形态差异[36],且系统学分析也支持前者为独立种。钩龟螺被分为2个亚种(图5手绘图),亚种之下又被分为5个变形[24]。这种精细的划分为种类鉴定和其他研究带来极大的混乱。物种具有形态可塑性,栖息环境可能影响其形态特征,不同发育阶段的个体往往也具有一定的差异。我们获得的不同大小的钩龟螺存在一定的形态差异(图2a至图2c),如小个体侧棘的发达程度和尾棘的弯曲程度更大。尤其是我们获得的最大标本(图2c),壳宽可达10 mm,明显大于张福绥[14]和van der Spoel[24]所记录的标本(小于6.5 mm)。
开放的海洋环境没有限制物种分布的地理障碍,终生浮游生物或是其他海洋生物的阶段性浮游幼体也有较强的扩散能力,因而许多物种被认为分布广泛。然而,一些所谓的环球或环极地分布的物种(形态种)实际上是一些局限性分布的隐存种,形态种生物多样性无法反映真实的物种多样性[37-39]。钩龟螺、球龟螺和长吻小龟螺在COI系统树中均形成明显的地理支系,产生了明显的地理遗传分化,不同支系间的K2P遗传距离都大于3%的阈值[21],其内部可能存在隐存种。
线粒体基因在物种鉴定和系统进化等领域得到了广泛应用。然而,线粒体假基因的存在为上述研究造成了一定程度的干扰。线粒体假基因通常极易被通用引物扩增出来,甚至是优先被扩增出来[40]。本文扩增得到真正线粒体COI基因序列的比例极低,这提醒我们在开展DNA条形码研究的时候应防范线粒体假基因的干扰,否则会得出错误的结论。
致谢:中国科学院南海海洋研究所周林滨博士采集了印度洋样品,李开枝研究员在实验过程中给予了帮助和支持,谨致谢忱。
