鳞翅目昆虫基因编辑技术研究进展
2020-03-19程英靳明辉萧玉涛
程英 靳明辉 萧玉涛
(1. 中国农业科学院农业基因组研究所,深圳 518120;2. 中国农业科学院深圳农业基因组研究所,深圳 518120;3. 岭南现代农业科学与技术广东省实验室深圳分中心,深圳 518120)
鳞翅目在分类学上隶属节肢动物门(Arthropoda)六足总纲(Hexapoda)昆虫纲(Insecta),是昆虫纲的第二大目,包括蝶、蛾2类,单个世代经历卵、幼虫、蛹、成虫等4阶段,为全变态昆虫。其种类超过25万种,广泛分布于世界各地。大部分鳞翅目昆虫都是农林、畜牧业的重要害虫,给农牧产业带来巨大的经济损失。基因编辑是指利用核酸内切酶在基因组的特定位置引入双链断裂(Double strand breaks,DSBs),诱导细胞自身的DNA损伤修复机制非同源末端连接修复(Nonhomologous end joining,NHEJ)或同源重组修复(Homology directed repair,HDR)对切口位置进行修复,从而产生基因插入、缺失等多种突变类型。随着基因编辑技术快速发展,鳞翅目昆虫基因编辑研究也取得了一系列重要的成果。本文对3种主要基因编辑技术:锌指核酸酶技术(Zinc Finger Nucleases,ZFNs)、转录激活因子样效应蛋白核酸酶技术(Transcription Activator-Like Effector Nuclease,TALENs)、成簇规律间隔的短回文重复序列及相关核酸酶技术(Clustered Regularly Interspaced Short Palindromic Repeats(CRISPR)/CRISPR-associated system(Cas),CRISPR/Cas)在鳞翅目昆虫基因组编辑中的研究进行简要综述,旨为鳞翅目昆虫功能基因组研究、抗性基因挖掘、建立害虫绿色防控体系提供参考。
1 锌指核酸酶技术及其在鳞翅目昆虫基因编辑的应用
锌指核酸酶(ZFNs)技术是第一代基因编辑技术,1996年Kim等[1]首次成功将其应用于DNA靶向切割,它是一种人工改造的核酸内切酶,每一个ZFNs由两个功能域组成:N末端为锌指蛋白DNA结合结构域,由一系列Cys2His2锌指模块串联组成,每个锌指模块识别并结合一个特异的三联体碱基序列,4-6个锌指模块组合在一起,形成一个锌指蛋白(ZFP),其特异性识别12-18 bp核酸序列。C末端为经过改造的非特异性FokI核酸酶内切酶结构域,FokI以二聚体形式对DNA进行切割。两个锌指蛋白分别识别5'-3'方向和3'-5'方向的DNA双链,双链通常间隔5至7个碱基的靶序列(Spacer),此时FokI核酸酶形成二聚体,切割靶位点,造成DNA双链断裂(图1)。Takasu等[2]通过ZFN mRNA显微注射成功对家蚕尿酸盐代谢通路中尿酸颗粒沉积相关基因BmBLOS2进行敲除,突变后代表现出半透明体色,制备了经典的油蚕表型品系。Merlin等[3]以黑脉金斑蝶的Dpcry2基因为靶标,通过体外合成的ZFNs mRNA显微注射,成功实现了Dpcry2基因敲除,突变后代表现出昼夜节律行为和分子钟机制紊乱。Ma等[4]利用ZFN技术对家蚕丝腺蛋白重链基因BmFibH进行敲除,产生只含有丝胶蛋白的蚕茧,该敲除品系能作为生物反应器实现外源基因稳定高表达。由于设计有效的ZFNs技术难度大,成本较高,实验周期长,ZFNs技术在鳞翅目昆虫研究中的应用报道较少(表1)。
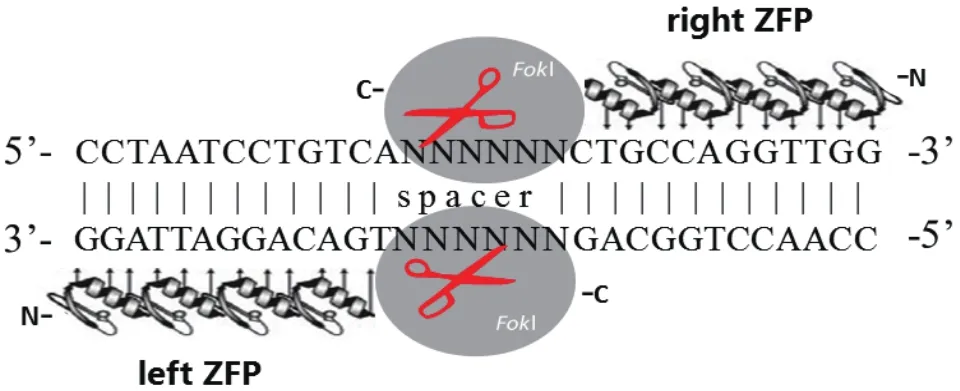
图1 ZFNs结构及工作原理[5-6]

表1 ZFNs、TALENs基因编辑技术在鳞翅目昆虫基因编辑中的应用
2 转录激活因子样效应蛋白核酸酶技术及鳞翅目昆虫基因编辑应用
转录激活因子样效应蛋白核酸酶(TALENs)技术是第二代基因编辑技术,TALENs来源于植物病原菌黄单胞菌属(Xanthomonas),与ZFNs结构相似,每一个TALENs由两个功能域组成:N末端为TALE蛋白结构域,特异性的结合DNA序列,C端为FokⅠ核酸内切酶结构域,非特异的切割靶位点造成双链断裂。每个TALE蛋白由3个部分组成:N-端转运信号;C-端核定位信号NLSs及酸性转录激活结构域AD;中间DNA结合结构域。中间DNA结合结构域由多个串联排列的重复序列单元组成,单个重复序列单元由33-35个高度保守的氨基酸组成,其中第12和13位氨基酸可变,被称为重复可变双残基(Repeat-variable diresidue,RVD),RVD可特异的识别DNA碱基,其中NI识别A;NG识别T;HD识别C;NK识别G;NN识别G或A;NS识别A、C、G、T等,根据不同碱基序列设计相应的TALE模块串,理论上可以靶向任何序列[7-8](图2)。1989年,Bonas首次报道,2009年成功应用于植物细胞基因组编辑[7],迄今已应用于多种鳞翅目昆虫基因编辑研究(表1)。2012年,Ma等[9]首次尝试使用TALENs敲除家蚕BmBLOS2基因,通过显微注射的方法,成功获得BmBLOS2缺失突变个体。通过同时注射两对TALENs,切割靶序列的两个不同位点,实现了家蚕基因组长片段删除。为了提高其编辑效率,Takasu等[10]对TALENs系统进行了进一步的改造,利用改进的TALENs系统pBlueTAL对家蚕BmBLOS2和Bm-re基因进行敲除,编辑效率提高了一个数量级。2014年后,TALENs技术在家蚕基因编辑中的应用快速发展,Xu等[11]利用TALENs对家蚕性别分化相关基因Bmdsx基因雌性特异外显子进行敲除,获得了雌性不育遗传系。日本科学家 Sakurai和 Yang等[12-13]利用 TALENs分别对家蚕和亚洲玉米螟(Ostrinia furnacalis)性信息素相关受体基因进行基因敲除,缺失相关受体的个体出现交配行为异常。为了提高突变的精确性,Ma等[14]将TALEN技术与ssODN联合注射到家蚕胚胎中,实现了家蚕染色体水平的精准靶向编辑,制备了染色体缺失、重复、倒位等多个染色体结构变异模型。将TALENs与微同源介导的末端连接原理结合,Nakade等[15]构建了TAL-PITCh编辑器,成功在家蚕BmBLOS2基因位点插入GFP,该系统极大地提高了基因体内标记示踪效率。迄今,利用上述TAL-PITCh编辑器已成功建立了家蚕W染色体示踪品系及雌性W染色体特异表达Cas9稳定遗传品系[16]。TALENs技术的快速发展及广泛应用极大地推动了鳞翅目昆虫基因编辑研究进展。
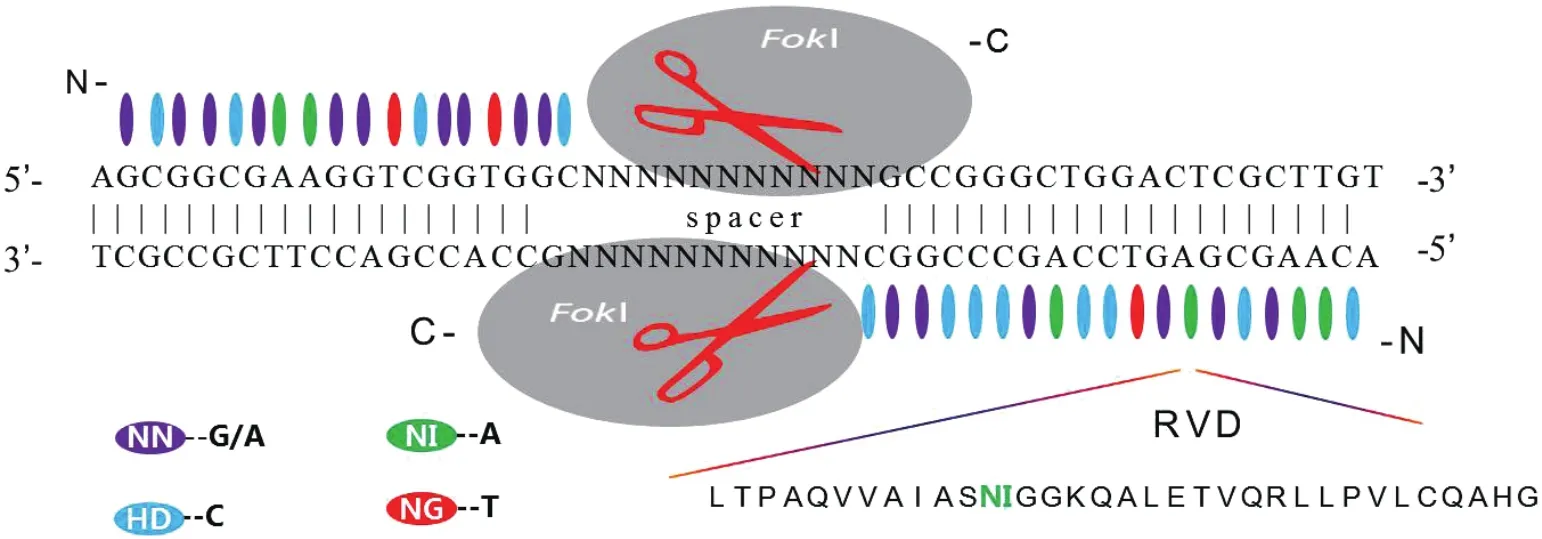
图 2 TALENs结构及工作原理[8,17]
3 CRISPR/Cas技术及鳞翅目昆虫基因编辑应用
CRISPR/Cas基因编辑技术是第三代基因编辑技术,不同于前两代技术,它是一种由RNA引导的核酸内切酶技术,是细菌和古细菌为应对外源核酸物质攻击所演化而来的获得性免疫防御机制,由两部分组成:CRISPR即规律成簇间隔短回文重复序列(Clustered regularly interspaced short palindromic repeats),由前导序列(Leader)、重复序列(Repeat)和间隔序列(Spacer)组成;Cas 即 CRISPR相关蛋白(CRISPR-associated system),其编码核酸切割相关蛋白酶(图3-A)。CRISPR/Cas免疫防御包括3个阶段:接受(Adaptation),pre-crRNA(pre-CRISPR RNA)表达和加工,干扰(图3-B)。接受阶段:外源核酸物质首次入侵细菌基因组时,CRISPR系统会识别外源核酸中的PAM序列(Protospacer adjacent motif,原间隔序列临近基序),并将其上游约 30 nt的序列(原间隔序列Protospacer)剪切后插入自身CRISPR 阵列中的两段重复序列之间(Repeat),形成新的 Spacer,获得免疫记忆。pre-crRNA合成与加工阶段:当外源核酸物质再次入侵细菌基因组时,在前导序列(Leader)的作用下起始转录CRISPR,得到的初级转录产物CRISPR RNA(Pre-crRNA)在相关Cas核酸酶的作用下被加工成成熟的crRNA。干扰阶段:成熟的crRNA 与相关 Cas 蛋白形成复合体,通过碱基互补配对,crRNA识别外源核酸物质中protospacer序列,引导Cas蛋白复合物对外源核酸进行切割,造成双链断裂,从而防止外源核酸入侵[20-21]。95%的古细菌及48%的细菌其基因组中至少存在一个CRISPR基因座,具有极高的物种特异性和多样性,不同菌种识别的PAM序列和Cas蛋白的数量及类型不同。基于Cas蛋白结构及进化关系CRISPR/Cas系统可分为2个大类、6种类型及多种亚型(图3),其中应用较为广泛的有Cas3(Type I)、Cas9(Type II)、Cas10(Type III)、Csf1(Type IV)、Cpf1/Cas12(Type V)、Cas13(Type VI)[20-22]。
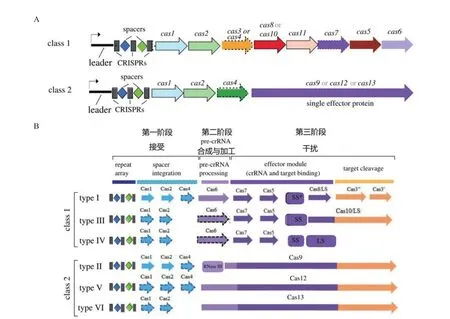
图3 CRISPR/Cas系统结构、分类及功能简介[20-22]
2012年,Jinek等[23]首 次 证 明 改 造 后 的CRISPR/Cas系统可以用于DNA体外靶向切割。Cas9 是Cas蛋白中重要的一员,CRISPR/Cas9 基因编辑系统是细菌II型获得性免疫防御系统经人工改造而成,由Cas9蛋白、pre-crRNA(cr-RNA前体)和 tracrRNA(Trans-activating crRNA)3个 组 分 组成,pre-crRNA与tracrRNA结合,经加工成成熟的发夹结构cr-RNA;cr-RNA通过互补配对的方式引导 Cas9 蛋白对靶序列进行切割,造成DNA双链断裂,启动DNA损伤修复机制,在双链切口位点附近造成删除、插入等多种随机突变。
鳞翅目幼虫除极少数外均取食显花植物,其中大多是农林重要害虫,如棉铃虫、小菜蛾、斜纹夜蛾、甜菜夜蛾、小地老虎、草地贪夜蛾等,对农林业生产造成了巨大经济损失,CRISPR/Cas9基因编辑技术的应用极大的推动了鳞翅目害虫基因功能研究。
2013年,Wang等[24]首次报道CRISPR/Cas9系统可应用于家蚕基因组编辑,通过显微注射体外转录的Cas9 mRNA和sgRNAs,获得了重要标记基因BmBlos2敲除品系,单个sgRNA敲除效率高达94%-95.6%,同时注射两个sgRNA,其大片段敲除效率高达95.5%-100%。Liu等[25]利用CRISPR/Cas技术不仅实现了单位点的小片段敲除、插入,也突破了制备染色体大片段缺失及倒位模型、同时突变6个基因等诸多技术难关。为了进一步解决同源重组效率低下等问题,Zhu等[26]利用CRISPR/Cas9对家蚕NHEJ相关调控因子BmKu70、BmKu80、BmLigIV、BmXLF、BmXRCC4和BmTUDOR-SN进行敲除,敲除细胞中同源重组效率提高了7倍以上。为解决家蚕饲养中病毒感染的问题,Dong等[27]构建了一种病毒诱导的CRISPR/Cas9编辑系统,该系统在病毒感染后能被迅速激活,切割BmNPV病毒中的复制关键因子ie-1,从而有效抑制病毒增殖。CRISPR/Cas9技术应用于鳞翅目昆虫信号通路研究也取得了一定进展。保幼激素(Juvenilehormones,JHs)一直被认为是昆虫幼虫性状维持及向成虫发育的关键因子,Daimon等[4]对JHs合成关键基因JHAMT以及JHs受体Met1和Met2进行了缺失突变,发现敲除后的低龄幼虫并不会存在提前化蛹的情况,对保幼激素在变态发育中的作用有了不同的认识。CRISPR/Cas9也成功应用于miRNA的功能研究,利用CRISPR/Cas9对家蚕miRNA-14进行敲除后,突变后代出现一定程度的发育迟缓现象[28]。截至目前CRISPR/Cas9技术已成功应用于家蚕胚胎发育、体色形成、嗅觉行为、代谢通路等多个领域(表2)。
性信息素具有吸引雄虫进行交配的功能,其代谢相关基因是害虫防治的重要靶标。Koutroumpa等利用CRISPR/Cas9对棉叶虫(Spodoptera littoralis)宿主和性信息素相关受体Orco基因进行敲除,纯合突变个体对宿主植物气味和性信息素嗅觉感受缺陷[29]。斜纹夜蛾嗅觉相关基因SlitPBP3敲除后,突变群体对性信息素的电生理反应敏感性显著降低[30]。棉铃虫性信息素的气味受体16(OR16)敲除后,突变体雄虫不能区分性成熟和未成熟的雌虫,在性信息素主成分Z11-16∶OH作用下与未成熟的雌虫交配,其子代的孵化率和存活率显著降低[31],为上述害虫防治提供了的重要靶标信息。
性别决定机制的解析对害虫遗传发育研究意义重大,其代谢相关通路研究为害虫遗传防治提供了新的思路。鳞翅目性别控制主要由Masculinizer(Masc)、Masc-piRNA、Fem、Fem-piRNA、doublesex(dsx)、Psi、Imp、Siwi等基因组成的信号通路决定[32]。Xu等[33]对家蚕性别分化相关基因Bmsxl,Bmtra2、Bmlmp、BmPSL、BmlmpM、BmPSI和BmMasc进行敲除,进一步解析了家蚕性别决定中上述各基因功能。Bmtra2敲除后性别决定基因Bmdsx非正常转录,雌性胚胎不能正常发育而致死。BmImp或BmImpM基因功能的丧失不影响性别分化。BmPSI和BmMasc基因的突变影响Bmdsx的剪切,导致雌性生殖器官出现在雄性外生殖器中。BmPSI参与调节BmMasc、BmImpM和Bmdsx的表达,为家蚕雄性性别决定中的关键辅助因子。斜纹夜蛾Sldsx基因敲除后雄性睾丸发育异常,外生殖器畸形,无法完成交配[34]。对小地老虎雌性和雄性特异性Aidsx区域进行敲除后,突变后成虫外生殖器畸形或缺失,解剖发现卵巢和睾丸发育异常。触角是区分小地老虎性别的主要标志,Aidsx突变体触角出现多种突变类型,同时性别差异表达相关基因如vitellogenin(Vg),pheromone binding protein(PBP),olfactory receptors(OR)等表达谱均发生相应改变[35]。Wang等[36]研究发现小地老虎性别决定通路中Masc基因敲除后,突变体雄性在外生殖器中表现出严重的结构缺陷,交配行为无法正常进行。
体色相关标记基因的筛选对鳞翅目昆虫遗传学研究有重要意义,Khan等[37]利用CRISPR/Cas9对棉铃虫White、Brown、Scarlet及Ok基因分别进行敲除研究发现,white基因敲除后后代隐性致死,同时影响卵、1龄幼虫、成虫眼的色素沉着。Scarlet基因敲除后1龄幼虫、成虫眼出现黄色素沉着。Ok基因敲除后幼虫呈黑色眼,角质层半透明。Chen等[38]对小地老虎体色相关基因yellow进行了功能验证,鉴定出7个yellow基因,yellow-y基因敲除的小地老虎幼虫呈现出不同于野生型的黄色表型,同时幼虫出现一定程度的脱水现象而导致死亡。
田间害虫防治及新药靶点的发现离不开抗药性机理研究,CRISPR/Cas9技术的出现极大的推动了这一进程。棉铃虫盒式转运蛋白HaABCA2基因敲除后,其对Bt毒素Cry2Aa、Cry2Ab抗性增强,进一步研究发现敲除品系Cry2Aa、Cry2Ab毒素不能与BBMVs结合(Brush border membrane vesicles,中肠刷状缘膜囊泡),而Cry1Ac与BBMVs的结合不受影响,揭示棉铃虫HaABCA2参与Bt毒素Cry2A结合运输[39]。棉铃虫HaABCC2、HaCAD基因敲除后,敲除群体表现出Cry1Ac高抗性,揭示HaABCC2、HaCAD基因参与棉铃虫体内Cry1Ac代谢[40]。棉铃虫CYP6AE基因簇包含9个首尾相连的细胞色素P450氧化酶基因,P450细胞色素氧化酶可以催化上万种生化反应,具备对多种化合物如植物次生物质、杀虫剂、环境化合物等的解毒代谢能力,Wang等[41]采用CRISPR/Cas9基因编辑技术敲除了棉铃虫长达85kb的CYP6AE基因簇,发现该基因簇敲除品系对植物次生性化合物花椒毒素和2-十三烷酮,以及杀虫剂顺式氰戊菊酯和茚虫威的敏感性显著升高。2019年,草地贪夜蛾入侵我国,本课题组率先应用CRISPR/Cas9系统对草地贪夜蛾SfABCC2基因进行敲除,阐述了草地贪夜蛾Bt毒素Cry1F解毒代谢机制[42]。Zuo等[43]通过将ssODN、Cas9蛋白、位点特异sgRNA显微注射入甜菜夜蛾胚胎中,成功制备RyR基因G4946E位置定点突变品系,与小菜蛾、番茄夜蛾中研究相似,RyRG4946E突变后甜菜夜蛾对氯虫苯甲酰胺、氰虫酰胺和氟虫双酰胺的抗性分别增加223倍、336倍和 1 000倍以上。
新型CRISPR/Cas蛋白的发现以及各种基因编辑策略有机结合极大的丰富了鳞翅目基因编辑技术体系。SpCas9来源于酿脓链球菌,仅识别含有“NGG”PAM序列的靶位点,SaCas9来源于金黄色葡萄球菌,比SpCas9蛋白分子量更小,识别以“NNGRRT”序列为PAM的靶位点,AsCpf1识别以“TTTN”序列为PAM的靶位点,而Cas13a能够靶向RNA,使靶位点选择更加灵活。Cas9n(Cas9 nickase)大大提高了 CRISPR/Cas9 系统的特异性,同时显著降低其脱靶效率。dCas9(nuclease-dead Cas9)系统将 Cas9 蛋白切割结构域失活,通过偶联不同的辅助功能蛋白,如FokI、转录激活域、甲基化酶等,可实现更为精确的基因组切割实现靶基因激活、干扰、表观遗传学修饰等。如将dCas9与大鼠胞嘧啶脱氨酶APOBEC1融合,能特异的将胞嘧啶(C)转变为尿嘧啶(U),实现靶位点G·C→A·T定向突变。与APOBEC3A(hA3A)融合可高效介导甲基化胞嘧啶(mC)到胸腺嘧啶(T)的碱基编辑。与海七鳃鳗(Petromyzon marinus)胞嘧啶脱氨酶(PmCDA1)结合构建了新的胞嘧啶编辑器(nCas9-Linker-PmCDA1),在动植物细胞中实现了定点的C→T 和 C→G 突变。PspCas13b酶和ADAR2脱氨酶融合,可高效地修复RNA的单个核苷酸,实现靶位点A·T → G·C定向突变。基于AsCpf1(Cas12a)构建的碱基编辑器可在A/T富集区域进行碱基编辑,极大地拓宽了碱基编辑器的应用范围。
在上述研究基础之上,Ma等[44]研究表明SaCas9和AsCpf1两种核酸酶均可在家蚕BmNs细胞中诱导高效的位点特异性基因编辑,SaCas9敲除效率约为11.8%-11.9%,AsCpf1的敲除效率约 8.7%-16.7%,两者均与SpCas9的敲除效率相当。针对一些特殊的基因,直接敲除会导致转基因后代不能有效存活或传代,Ma等[4]构建了一套组织特异性调控基因编辑策略,通过将丝腺特异性表达Cas9蛋白的品系与组成型表达BmlaminA/C(BmLMN)gRNA品系杂交,实现了BmlaminA/C基因丝腺特异性敲除。Nakade等[15]将CRISPR/Cas9技术与微同源介导的末端连接原理结合,构建了CRIS-PITCh编辑器,大片段敲入效率有了极大提高。张峰和Sam Sternberg团队发现了基于转座子的CRISPR系统,该系统利用转座元件能实现目的基因高效整合到基因组,大片段敲入效率有望进一步提高[45-46]。
4 总结与展望
ZFNs、TALENs 和 CRISPR /Cas 三种基因编辑技术各有优劣。作为基因编辑领域的先行者,ZFNs
技术的应用使基因打靶效率大大提高。但是,由于ZFNs 蛋白序列识别的特点,ZFNs的设计序列依赖性较高,不能满足对全基因组任意基因任意位置进行编辑的需求。同时,其模块化设计及组装难度大,成本高,大部分实验室并不能熟练掌握该技术,其在鳞翅目昆虫中的应用受到了极大限制,仅在少数物种如家蚕中有一定程度的应用。与 ZFNs 技术相比,TALENs其设计及组装较为快捷,所需实验周期也大大缩短,经过较短时间的培训,大部分实验室都能熟练掌握该技术。但是,筛选高效的TALEN模块仍需要大量的实验验证,成本并不低,基于此在鳞翅目昆虫中的应用仅有少量报道。CRISPR/Cas技术的出现颠覆了基因编辑技术领域,不到一周时间实验室就能完全掌握该技术,由于仅需要设计合成一个约20 nt与靶基因互补配对的碱基序列就能识别目的基因,极大的缩小了基因编辑窗口,基本能实现全基因组任意基因位点的编辑。目前,该技术已成功应用于多种鳞翅目昆虫的基因编辑研究。不可忽略的是,CRISPR/Cas系统造成的脱靶率远高于前两种,更高效精确的CRISPR/Cas版本有待开发。

表2 CRISPR/Cas9基因编辑技术在鳞翅目昆虫基因编辑中的应用
相较于模式昆虫,鳞翅目昆虫基因编辑研究还远远滞后,基因导入效率低是主要因素。目前的基因编辑技术对遗传物质导入方式有极高的要求,显微注射通常是最为直接有效的手段,但是对仪器平台以及操作人员熟练程度要求较高。病毒载体具有高效整合、高效表达的特点,在人类疾病研究及基因治疗中发挥着重要的作用,鳞翅目昆虫是许多病毒的天然宿主,通过对这些病毒进行改造,使其能够在活体组织、细胞内完成基因编辑系统的传递,无疑能极大的弥补显微注射技术在鳞翅目昆虫基因编辑应用中的不足。
虽然CRISPR-Cas系统能实现基因组特异位点的高效靶向切割,通过同源重组或非同源重组的方式能够实现目的DNA片段的插入,但是大片段插入以及同源重组效率整体上仍然极低。转座子尤其是DNA转座子结构简单,在转座酶的作用下,能识别特定转座位点,通过剪切、粘贴等一系列过程使基因从一个位置“跳跃”到另一个位置,实现目的基因的高效、位点特异性插入。基于转座子的基因插入对外源基因大小限制较低,可以实现大片段插入,建立基于转座酶的CRISPR系统能很大程度的解决鳞翅目昆虫基因编辑大片段插入效率低及转座随机性问题。类似的,整合酶系统如Flp/FRT、Cre/Lox、φC31/att等具有动植物细胞广泛适用性,无需外源辅助因子实现大片段定点整合及外源基因持续稳定表达,利用CRISPR技术建立基于整合酶的定点整合“停泊位点”(Docking site)品系,亦可应用于鳞翅目基因组大片段整合研究。
大多鳞翅目昆虫均为农林牧重要害虫,科学家们一直致力于建立有害昆虫的遗传防控技术体系,并取得了一定成效。基于基因编辑技术的基因驱动技术,能使有害突变在群体中快速扩散或替代原有基因座,在短时间内达到控制害虫种群的目的。但是,一旦把基因驱动的生物释放至野外,人们便很难对这些生物进行控制,可能会引发一系列的生态安全事件。利用时空表达调控元件给基因驱动装上“开关”,建立可调控的基因驱动技术体系,对鳞翅目昆虫遗传防控技术的建立至关重要。
尽管基因编辑技术有了突飞猛进的发展,但是成功应用于鳞翅目基因组编辑的技术仍屈指可数。随着5 000种昆虫基因组(i5k)计划、昆虫TOP1000计划的推进,众多鳞翅目昆虫全基因组信息有望破译。灵活应用现有的技术开发高效的、鳞翅目昆虫适用的基因编辑的技术体系,利用基因编辑技术对鳞翅目昆虫遗传资源进行深度挖掘,加速昆虫生理生化、遗传代谢、行为机理解析,开发新型经济昆虫,制备昆虫不育系、建立绿色害虫防控体系,大量的工作仍有待开展。
