苦参转录组SSR位点及基因功能注释分析
2019-09-17张宁尹美强谭青青
张宁 尹美强 谭青青
摘要:分析苦参转录组中的简单重复序列(SSR)位点信息,为开发分子标记奠定基础。利用Fastqc软件对苦参转录组测序的原始读长(reads)进行质量评估,再用Trimmomatic软件对reads质量较差的碱基进行过滤,利用Trinity软件对Trimmomatic处理后的reads进行序列组装,之后使用基因组装完整性评估(BUSCO)软件对转录组组装的序列進行质量评估,并分析组装的conting序列的开放阅读框(open reading frame,简称ORF);利用MicroSAtellite(MISA)软件对无冗余独立基因(unigene)进行SSR搜索。利用Trinity软件最终筛选得到23074条ORF信息;使用MISA软件从unigenes序列中发现8 798个SSR位点,分布于7 339条unigene中,总体上unigenes序列中SSR占比为2.16%,SSR位点平均间隔是5.28 bp,其中占比最高的是单核苷重复基序,为50.53%;其次是出现频率分别为22.28%、24.73% 的二、三核苷酸。苦参转录组中SSR类型众多,出现频率高,在后续的苦参遗传性状分析,及次生代谢(苦参碱和黄酮等次生代谢产物)途径等相关基因定位等方面具有很好的应用潜力。
关键词:苦参;转录组;SSR;位点信息;基因功能;分子标记
中图分类号: R285 文献标志码: A 文章编号:1002-1302(2019)07-0041-04
苦参(Sophora flavescens Ait.)是豆科槐属植物,以其干燥根入药,味苦,性寒,具有清热除燥湿、杀虫和利尿等药效。其主要药用成分是生物碱类和黄酮类化合物,已从苦参中分离出生物碱类39个,黄酮类122个成分[1]。苦参主产于山西、陕西、河南、河北等地,在医学临床、农业、畜牧业和日用品等中有广泛的应用[2]。气候的变化和人为过度的采挖造成野生苦参资源数量急剧减少[3]。因此,保护和利用好野生苦参资源是当务之急,势在必行。
分子标记开发可对制定合理有效的种质资源保护策略提供科学依据,但目前还缺乏能够应用于苦参种质鉴定、遗传图谱构建、功能基因定位等研究的简便、高效、稳定且具有种属特异性的分子标记体系。简单重复序列(simple sequence repeat,简称SSR)是由核苷酸构成的重复序列,在真核生物和原核生物基因中都有存在。SSR 位点标记具有在生物中分布广泛、重复类型多样、出现频度高等特点[4],主要应用于分子育种优良基因定位、生物多样性分析、遗传图谱绘制、突变体单核苷酸多态性(single nucleotide polymorphism,简称SNP)位点分析辅助等。传统寻找基因组中SSR标记的方法存在位点开发成本高、步骤较多、操作繁琐等问题[5]。转录组SSR位点开发具有方便快捷、效率高等特点,且成本低廉。SSR开发引物能够直接快速地定位基因信息。随着苦参研究的深入,目前还未发现有关苦参转录组SSR开发的报道。本研究通过分析苦参转录组中的SSR位点信息,为苦参遗传性状分析、次生代谢(苦参碱和黄酮等次生代谢产物)途径、分子标记辅助育种及苦参遗传多样性研究提供依据和参考。
1 材料与方法
1.1 转录组数据来源
从NCBI(美国国家生物技术中心)数据共享平台获得苦参转录组数据,从SRA(Sequence Read Archive)数据库(https://www.ncbi.nlm.nih.gov/sra/)获得苦参叶片RNA-Seq原始测序数据,下载编号是SAMD00029896,使用Illumina HiSeq1000对苦参组织进行建库测序,原始数据reads为 90 bp,采取双端(paired-end sequencing)测序,获得1.3 GB转录组数据,下载网址是ftp://ftp.ncbi.nlm.nih.gov中的DRR031281[6]。
1.2 转录组的从头组装
首先通过Sratoolkit.2.8.2-1将sra格式转录组原始数据转换为fastq格式[7];使用Fastqc软件进行转录组原始数据质量评估,然后,利用Trimmomatic软件对fastq格式的序列进行低质量去除,leading头部去掉质量低于3的碱基,trailing尾部过滤掉质量低于3的碱基,每4个碱基是一个阅读框,4个连续碱基的平均质量低于15的过滤掉,reads中最小长度小于40序列的过滤掉 [8];随后,对高质量reads采用Trinity 软件进行从头(de novo)组装[9],最短contig 长度设置为200 bp(参数为默认参数)。筛选每个基因最长的转录本作为unigene,最后组装得到苦参转录组的全部转录本(包含可变剪切)。
1.3 苦参转录组数据组装完整性评估
选取由Trinity软件组装的序列,使用BUSCO V 2.0.1软件进行苦参叶片转录组数据完整性评价[10]。BUSCO V 2.0.1 软件依据 Ortho DB 数据库,组成了几个大的进化分支单拷贝基因集,将转录本reads拼接结果与该基因集数据进行比较(基因集直接使用 HMMER3与参考数据库比对),依据比对上的比例、完整性评估拼接结果的准确性和完整性。
1.4 ORF预测
使用Trinity软件中的TransDecoder LongOrfs工具对unigene进行开放阅读框(open reading frame,简称ORF)预测,筛选大于100个氨基酸的ORF序列,获得最佳的ORF区域,使用Pfam (http://pfam.xfam.org/)和UniProt(http://www.uniprot.org)数据库对预测结果进行校正,将比对结果保留到Pfam和UniProt数据库的蛋白质序列中[11]。
1.5 SSR位点搜索
使用MISA软件[12]对苦参转录组数据unigene的SSR位点进行定位搜索,查询定位规则是三碱基、四碱基、五碱基和六碱基重复至少5次,二碱基重复不得少于6次,2个SSR位点之间不足100bp则视为复合型SSR。
1.6 含SSR序列的基因功能注释及生物碱基因挖掘
通过diamond blastx和diamond blastp分別将苦参中含SSR的8248条unigene序列与uniprot_sprot、Pfam和eggnog、Kegg、基因本体论(gene ontology,简称GO)等数据库进行比对,比对参数e值<10-5,然后利用WEGO(http://wego.genomics.org.cn/)在线分析工具进行GO功能分类统计,分析含有SSR unigene的功能分布特征;通过与GO库进行比对后,得到的unigene注释结果按照GO数据库的23个类别进行分类统计。通过对WEGO注释结果(3个大类)23个子类更深入分析挖掘苦参碱相关基因,为进一步研究奠定基础。
2 结果与分析
2.1 苦参转录组de novo 组装
从NCBI数据库下载得到的苦参转录组测序(RNA-Seq)数据中共包含14 636 096个双端测序 reads,通过Trimmomatic软件过滤得到14 578 802 个高质量 reads。转录组 de novo组装获得53 179个长度大于200 bp的contigs,拼接获得的长序列(contigs)平均长度为813 bp,最长的 contig为22 546 bp,N50为1 464 bp;筛选每个基因中最长的转录本,共得到54 221条unigenes,平均长度为715.87 bp,最长的unigene 为12 122 bp,N50为1 464 bp(表1)。采用TransDecoder软件中LongOrfs功能进行ORF预测,筛选获得大于100个氨基酸的ORF有29 226个contigs;通过UniProt蛋白质数据库比对获得15 242条蛋白质序列,Pfam数据库比对获得126 429条蛋白质序列;使用TransDecoder最终筛选得到23 074条ORF信息。
contigs 和unigenes的鸟嘌呤(G)和胞嘧啶(C)占比都是44. 8%。从序列长度分布看,序列长度分布在1 000~2 899 bp 的序列大约有19.3%,≥2 900 bp的序列只有0.2%,600~999 bp的序列大约有13.6%,700bp 以下占71.4%(图1)。
2.2 转录组数据完整性评估
对转录组数据进行评估、测序、组装得到的转录序列覆盖所有可能的转录本。评估转录组数据的大小和完整性。依据植物直系同源基因数据集对苦参的转录组数据完整性进行评估,由表2可知,在由苦参转录组序列与植物基因组匹配获得的1440个植物单拷贝直系同源基因中,完全匹配到的直系同源基因( complete)有1000个,占总BUSCO的69.4%,部分片段匹配对应到的单拷贝直系同源基因( fragment)有171个,占总BUSCO的11.9%;没有匹配对应到的植物单拷贝直系同源基因(missing)有269个,占总BUSCO的18.7%,完全匹配到的单拷贝直系同源基因(complete)有973个,占总BUSCO的67.6%,完全匹配到的多拷贝直系同源基因(complete)有27个,占总BUSCO的1.9%。
2.4 转录组中SSR 位点的分布特点
使用 Trinity软件组装得到54 221条unigenes,碱基数为 38 815 308 bp,平均每条unigene长度为715.87 bp;使用 MISA软件搜索得到8 798个SSR位点,存在于7 339条unigenes转录组序列中,包括多个 SSR位点的 unigenes序列有1 173条(包含复合 SSR为551个)占SSR unigenes序列总数的13.33%。总体上unigenes序列中SSR占比为2.16%,SSR位点平均间隔距离是4 411 bp。其中占比最高的是单核苷重复基序,占总SSR 的50.53%;其次是出现频率分别为22.28%、24.73% 的二、三核苷酸。SSR最短平均分布距离是0.99 bp的单核苷酸重复类型,平均分布距离最长的是1.29 bp的五核苷酸重复类型。
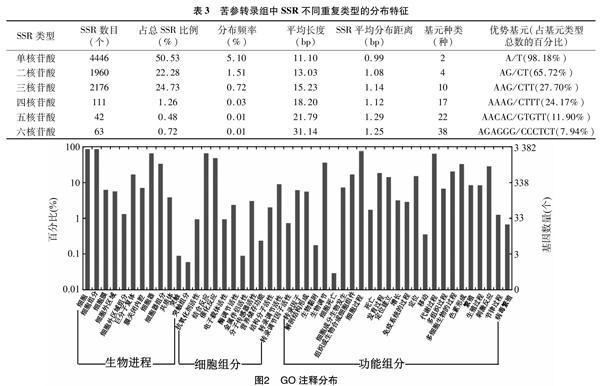
苦参转录组不同重复类型的SSR位点都有多种基元,在考虑碱基互补且包含复合重复基元的情况下,重复类型合计93种,其中六核苷酸38种,五核苷酸22种,四核苷酸类型17种,在筛选的 SSR中单核酸重复优势基元为A/T,占比最高,为总基元类型的98.18%,其次是二核苷酸重复类型优势类型基元AG/CT,为65.72%。三核苷酸重复类型的优势基元是AAG/CTT,占比27.70%;四、五、六核苷酸重复类型的优势基元分别是AAAG/CTTT、AACAC/GTGTT、AGAGGG/CCCTCT,所占的比例分别是24.17%、11.90%、7.94%(表3)。
2.5 转录组SSR 基序重复类型和频率特征
不同重复类型苦参转录组SSR位点分布存在差异(表4)。单核苷酸重复类型设置重复数≥15次作为SSR位点的识别条件,因此在表中未分析单核苷酸类型。除单核苷酸外,各重复类型重复数在5~11次之间,随重复次数的逐渐增加,频率逐步降低。除单核苷酸外,5~7 次是主要集中次数,占SSR类型总数的大多数。
2.6 含SSR序列的基因功能注释及生物碱基因挖掘
为了解含有SSR序列苦参转录组序列的基因功能,本研究通过与公共蛋白数据库进行比对,得到含有SSR序列的分类信息和功能注释。结果发现,uniprot_sprot、Pfam、eggnog、Kegg、GO分别注释到3 094、3 162、3 061、3 138、3 467个基因。
GO注释将基因功能分为生物进程(biological process)、细胞组分(cellular component)、功能组分(molecular function)大类,其下又分了很多子类,从不同角度对基因的功能进行分类注释,各类间互相关联。GO注释可以全面描述苦参中SSR基因和基因产物的属性。将搜索到含有SSR的unigene序列使用blastx比对到蛋白数据库,取比对分值最高的为序列注释信息。细胞组分注释10312条,生物进程注释11 200条,功能组分注释4 376条。将含有SSR序列的3 467條unigene编号后与其对应的GO分类号一起导入到GO分类图形显示在线分析工具WEGO 软件中,得到其基因功能分布(图2)。结果表明,在3 467条unigene序列中注释信息获得23 483个功能注释,平均1条unigene有6.77个GO注释。
苦参主要药用成分是苦参碱和黄酮类物质,通过对含有SSR位点的序列进行GO注释数据挖掘,获得7个生物碱代谢途径相关基因,2个黄酮类生物合成过程相关基因。
3 讨论
苦参转录组 de novo组装获得51 606 个长度大于200 bp的contigs,使用uniprot和Pfam蛋白质数据库进行ORF比对校正,uniprot比对上15 242条蛋白质序列,Pfam数据库校比对上 126 429 条蛋白质序列,TransDecoder最终筛选得到 23 074条ORF信息,unigenes序列长度在700 bp 以下的序列
数大约占总序列数的70%。BUSCO对转录组组装结果:C占比为69.5%,S占比为67.6%,D占比为1.9%,F占比为11.9%,M占比为18.6%,总BUSCOs数目为1 440条。
苦参转录组序列通过MISA搜索到8 798个SSR位点,SSR位点的unigenes序列在苦参转组序列中SSR位点占比为2.16%,平均分布距离4 411 bp出现1个SSR。与其他药用植物比较,高于党参的0.022%[13],低于丹参的0.047%[14],高于西洋参的0.013 3%[15]和人参的0.017 2%[16];与豆科模式植物大豆相比,高于大豆的0.013 5%[17]。表明苦参的SSR位点数量较为丰富。通过对含有SSR位点序列的注释进一步分析获得苦参生物碱相关代谢基因,为后续相关研究提供参考。
本研究结果为苦参转录组数据中的SSR位点分析提供依据。本研究对转录组序列进行了ORF预测,反映了基因组中基因的编码区域,可进一步确定基因位置,省去了SSR引物设计开发过程中的克隆和测序步骤,充分利用了生物信息数据库现有测序数据,降低了开发成本。同时也明确了苦参SSR位点的基本特点,为进一步开发设计新的苦参功能基因SSR 标记奠定了基础。苦参中SSR对于苦参基因功能资源的开发利用、遗传资源评估、丰富的分子标记、种质资源改良和比较基因组学研究都具有重要的价值。
参考文献:
[1]国家药典委员会. 中华人民共和国药典[M]. 北京:化学工业出版社,2015.
[2]张贵君. 精编中草药彩色图谱[M]. 北京:中国医药科技出版社,2016.
[3]张 翅. 苦参茎叶中化学成分的研究[D]. 天津:天津中医药大学,2013.
[4]段永红,渠云芳,王长彪,等. 药用植物苦参SSR-PCR体系的优化与验证[J]. 中国农业大学学报,2014,19(5):95-100.
[5]He J Y,Zhu S,Komatsu K,et al. Genetic polymorphism of medicinally-used Codonopsis species in an internal transcribed spacer sequence of nuclear ribosomal DNA and its application to authenticate Codonopsis Radix[J]. Journal of Natural Medicines,2014,68(1):112-124.
[6]Han R,Takahashi H,Nakamura M,et al. Transcriptome analysis of nine tissues to discover genes involved in the biosynthesis of active ingredients in Sophora flavescens[J]. Biological and Pharmaceutical Bulletin,2015,38(6):876-883.
[7]Staff S. Using the SRA Toolkit to convert .sra files into other formats[EB/OL]. (2015-08-22)[2017-12-06]. http://www.ncbi.nlm.nih.gov/books/NBK158900/.
[8]Bonnal R J P,Ranzani V,Arrigoni A,et al. De novo transcriptome profiling of highly purified human lymphocytes primary cells[J]. Scientific Data,2015,2:150051.
[9]Grabherr M G,Haas B J,Yassour M,et al. Trinity:reconstructing a full-length transcriptome without a genome from RNA-Seq data[J]. Nature Biotechnology,2011,29(7):644-652.
[10]王 林. 白色链霉菌和白背飞虱的基因组学研究[D]. 合肥:中国科学技术大学,2017.`
[11]舒江平,刘 莉,沈 慧,等. 基于系统基因组学分析揭示早期陆生植物的复杂网状进化关系[J]. 生物多样性,2017,25(6):675-682.
[12]王 希,陈 丽,赵春雷. 利用MISA工具对不同类型序列进行SSR标记位点挖掘的探讨[J]. 中国农学通报,2016,32(10):150-156.
[13]王 东,曹玲亚,高建平. 党参转录组中SSR位点信息分析[J]. 中草药,2014,45(16):2390-2394.
[14]邓科君,张 勇,熊丙全,等. 药用植物丹参EST-SSR标记的鉴定[J]. 药学学报,2009,44(10):1165-1172.
[15]杨维泽,金 航,赵振玲,等. 西洋参EST资源的SSR信息分析[J]. 西南农业学报,2011,24(1):275-278.
[16]Li C F,Zhu Y J,Guo X,et al. Transcriptome analysis reveals ginsenosides biosynthetic genes,microRNAs and simple sequence repeats in Panax ginseng C. A. Meyer[J]. BMC Genomics,2013,14:245.
[17]Dreisigacker S,Zhang P,Warburton M L,et al. SSR and pedigree analyses of genetic diversity among CIMMYT wheat lines targeted to different megaenvironments[J]. Crop Science,2004,44(2):381-388. 么大轩,张 彬,刘松涛,等. 基于SNP和SSR对甜玉米种质遗传多样性的评价[J]. 江苏农业科学,2019,47(7):45-50.
