绵羊肺炎支原体RPA检测技术的建立
2019-09-10林裕胜江锦秀张靖鹏游伟胡奇林
林裕胜 江锦秀 张靖鹏 游伟 胡奇林
摘 要:【目的】建立一种快速简便检测绵羊肺炎支原体(Mycoplasma ovipneumoniae, Mo)的方法。【方法】根据Mo膜蛋白P80基因序列,利用Oligo 7软件设计并筛选出特异性扩增引物,通过条件优化建立检测Mo的RPA方法。【结果】该方法可特异性扩增Mo,对其他羊常见病原无特异性扩增,对Mo核酸的检测灵敏度为70 fg·μL-1,与常规PCR敏感性一致。批内和批间结果显示Mo阳性样品均能扩增条带,而阴性对照均无扩增,表明重复性好。对186份临床样品应用建立的RPA方法和常规PCR方法同时进行检测,并对其中40份肺脏样品进行支原体的分离鉴定,结果显示分离鉴定出的13份Mo阳性样品及常规PCR法检测出的95份阳性样品经RPA检测,结果均为阳性。【结论】建立的Mo RPA方法特异性强、重复性好,可作为Mo的快速检测和流行病学调查的一项候选技术。
关键词:绵羊肺炎支原体;重组聚合酶扩增;等温扩增
中图分类号:S 852.62文献标识码:A文章编号:1008-0384(2019)04-416-06
Abstract: 【Objective】 To develop a rapid detecting method for Mycoplasma ovipneumoniae (Mo). 【Method】 Based on the P80 gene sequence, specific primers were designed using Oligo 7 software. Conditions of the recombinase polymerase amplification (RPA) method were optimized for the application. 【Result】 The new assay detected P80 gene in Mo specifically, not any other common pathogens of sheep and goats. The detection sensitivity was 70 fg·μL-1, which was the same as provided by conventional PCR. The inter- and intra-batch tests showed that the modified method could amplify the bands on Mo-positive samples, not on Mo-negative specimens, indicating an acceptable repeatability of the methodology. Furthermore, the RPA assay and conventional PCR methods were simultaneously used on 186 clinic samples, as well as the Mycoplasma isolation and identification on 40 lung samples, to show that the RPA assay positively identified all 13 Mo-positive samples detected by Mycoplasma isolation and identification and 95 positive samples detected by the conventional PCR. 【Conclusion】The newly developed RPA method had desirable specificity and repeatability on Mo detection. It could be applied for rapid detecting and epidemiological studies on Mo.
Key words: Mycoplasma ovipneumoniae; recombinase polymerase amplification; isothermal amplification
0 引言
【研究意義】绵羊肺炎支原体Mycoplasma ovipneumoniae,Mo属于软膜体纲Mollicutes,支原体目Mycoplasmatales,支原体科Mycoplasmaceae,支原体属Mycoplasma,已被确认为引起绵羊和山羊支原体性肺炎(Mycoplasma pneumonia of goats and sheep,MPGS)的主要病原之一[1-4]。Mo的临床症状表现为气喘、高热、咳嗽、渐进性消瘦和肺间质增生性炎症[5-7],该病发病率为20%~30%,在急性发病过程中,病死率可达30%~50% [8-9]。近年来,随着养羊业的发展,该病在国内各省市均有流行发生的报道,给养羊业造成了巨大的经济损失,已成为制约养羊业健康发展的重要疫病之一[10-12]。因此,建立一种快速简便的检测技术对于该病的防控与及时治疗具有重要意义。
【前人研究进展】PCR技术在检测病原体的方法中,被认为优于分离培养法、免疫荧光检测法和ELISA法等,以其能够检测微量的病原DNA而被作为诊断多种疫病的常用方法[13]。然而PCR反应需要特殊的热循环设备,很多基层单位不具备检测条件。重组聚合酶扩增 (Recombinase Polymerase Amplification,RPA)技术 (英国公司TwistDxInc研发)主要依赖于3种酶:能结合单链核酸的重组酶、单链结合蛋白和链置换DNA聚合酶,模拟生物体内DNA复制,在常温反应0.5 h左右即可完成对目的片段的扩增,操作简单,尤其适用于基层使用[14]。目前,RPA技术已经在病毒、支原体、衣原体、细菌、寄生虫等病原检测方面得到了广泛开发与应用,如口蹄疫病毒[15]、非洲猪瘟[16]、山羊支原体山羊亚种[17]、动物衣原体[18]、结核分枝杆菌[19]、疟原虫[20]等。【本研究切入点】迄今为止,尚未见RPA技术应用于Mo的检测。P80基因编码的膜蛋白在免疫特性方面虽未有深入研究,但已应用于遗传进化树分析,且以该基因为靶基因建立的诊断方法已有常规PCR和荧光定量PCR方法[9,21]。【拟解决的关键问题】本研究根据Mo膜蛋白P80基因序列,利用Oligo 7软件设计并筛选出特异性的扩增引物,建立Mo快速简便的检测方法,为基层工作者在Mo临床样品的快速检测和流行病学调查提供一种新的技术支持。
1 材料与方法
1.1 菌株及临床样品
山羊支原体山羊肺炎亚种(Mycoplasma capricolum subsp. capripenumoniae,Mccp)F38株、绵羊肺炎支原体(Mycoplasma ovipenumoniae,Mo)Y98株、山羊支原体山羊亚种(Mycoplasma capricolum subsp.capricolum,Mcc)Califormia Kid株由中国农业科学院兰州兽医研究所储岳峰研究员馈赠。丝状支原体丝状亚种LC型(Mycoplasma mycoides subsp. mycoides LC,Mmm LC)Y-goat株、无乳支原体(Mycoplasma agalactiae,M.agalactiae)PG2株由西南民族大学汤承教授馈赠。牛支原体(Mycoplasma bovis,M.bovis)、大肠杆菌(Escherichia coli,Ec)、精氨酸支原体(Mycoplasma arginine,M.arginini)、溶血性曼氏杆菌(Mannheimia haemolytica,Mh)、巴氏杆菌(Pasteurella multocida,Pm)、金黄色葡萄球菌(Staphylococcus aureus,SA)、莱氏无胆甾原体(Acholeplasma laidlawii,AL)、Mo FJ-CL01株、Mo FJ-ND株、羊口疮病毒 (orf virus,ORFV)及丝状支原体山羊亚种(Mycoplasma mycoides subsp. capri,Mmc)为福建省农业科学院畜牧兽医研究所分离、鉴定和保存。186份临床样品来自于2016-2018年福建省福州市、宁德市、泉州市、南平市、三明市和莆田市,均为临床上疑似羊支原体性肺炎病羊的肺组织和鼻拭子,发病羊表现为咳嗽、呼吸困难、流鼻涕,剖检见胸膜肺炎。
1.2 主要试剂
组织/细菌DNA提取试剂盒购自生工生物工程(上海)有限公司;Nanodrop 2000购自赛默飞 (美国)公司;Twist AmpBasic试剂盒购自TwistDx (英国)公司;胶回收试剂盒购自康宁生命科学 (吴江)有限公司; Ex Taq DNA聚合酶、DL2000 、DL1000 Marker均购自宝生物工程 (大连)有限公司。
1.3 引物设计合成
参照RPA引物设计原则,下载GenBank中登录号为KM435069.1的Mo P80基因序列,利用Oligo 7.9软件设计引物,通过BLAST在线软件比对分析,初步验证其特异性。Mo RPA引物的核苷酸序列为: P1: 5′- CTT AAT GAT GAG CGT AGT AGA TAC CAA ACCAC-3′/P2:5′- GCT GAG GTC GGA TTT GGA CTA ACA ATA TTT G-3′。扩增片段大小为353 bp。常规PCR引物参照林裕胜等[22]的方法,引物均由生工生物工程(上海)股份有限公司合成。
1.4 支原体和细菌核酸抽提
利用组织/细菌DNA提取试剂盒说明书分别提取供试支原体和细菌的DNA,用作RPA和PCR反应的模板。
1.5 RPA的反应条件及优化
参照Twist Amp Basic试剂盒说明书提供的RPA扩增反应体系:取1 μL Mo Y98 DNA作为模板,10 μmol·μL-1上游引物和10 μmol·μL-1下游引物各2.4 μL、Rehydration Buffer 29.5 μL、无核酸酶纯水 12.2 μL和280 mmol·μL-1醋酸镁2.5 μL,总反应体积为50 μL,同時以无菌去离子水作为空白对照,并以试剂盒提供的引物和模板作为阳性对照,用以监控扩增反应是否正常。采用矩阵法,优化反应温度(36、38、40、42℃)和反应时间(20、25、30、35、40 min)。扩增产物纯化后经琼脂糖凝胶电泳检测。扩增目的片段由生工生物工程(上海)股份有限公司进行测序。
1.6 Mo常规PCR及分离鉴定
常规PCR反应体系为:Premix TaqTMVersion 2.0 plus dye 10 μL、上下游引物(10 pmol·μL-1)各1 μL、Mo DNA 2 μL、无菌去离子水补足至20 μL。反应条件:94℃ 2 min;94℃ 30 s、58.5℃ 15 s、72℃ 15 s,30个循环;72℃ 10 min。Mo分离鉴定法参照江锦秀等[23]的方法进行。
1.7 特异性试验
分别以Mo Y98株以及FJ-CL01和FJ-ND株的DNA、Mcc、M.arginini、M.bovis、M.agalactiae、Mh、ORFV、Mccp、Mmc、Mmm LC 、AL、Ec、SA、Pm的核酸作为模板,采用建立的Mo RPA方法进行检测,同时设置空白对照,反应结束后,利用PCR纯化试剂盒回收扩增产物,然后经2%琼脂糖电泳检测。RPA扩增反应的加样顺序和反应条件为:首先将RPA反应体系中除了醋酸镁之外的成分加入到Twist Amp Basic试剂盒提供的冻干酶复合物反应管中,然后将醋酸镁加入到反应管盖子内,盖上盖子瞬时离心,使醋酸镁进入反应体系中,然后将反应管放入38℃恒温水浴锅中孵育30 min。
1.8 敏感性试验
利用Nanodrop 2000测定提取的Mo DNA浓度,将其10倍倍比稀释(101~1010),分别以稀释后的不同浓度的DNA为模板进行RPA反应,以确定该引物的灵敏度,同时进行常规PCR检测,比较两者之间的敏感性。RPA反应体系和条件同特异性试验。
1.9 重复性试验
利用建立的Mo RPA方法对2份Mo阳性样品、Mcc、M.arginini、M.bovis、M.agalactiae、Mh、ORFV、Mccp、Mmc、Mmm LC 、AL、Ec、SA、Pm各1份作阴性对照,反应条件和体系参照特异性试验。进行批内重复试验时,上述样品均设3个重复;进行批间重复试验时,上述样品间隔3 d检测1次,共测定3次。
1.10 临床样品检测
利用建立的RPA方法对186份临床样品进行检测,同时进行PCR检测,并对其中40份肺脏样品进行支原体的分离鉴定,根据检测结果计算检出率及符合率。
2 结果与分析
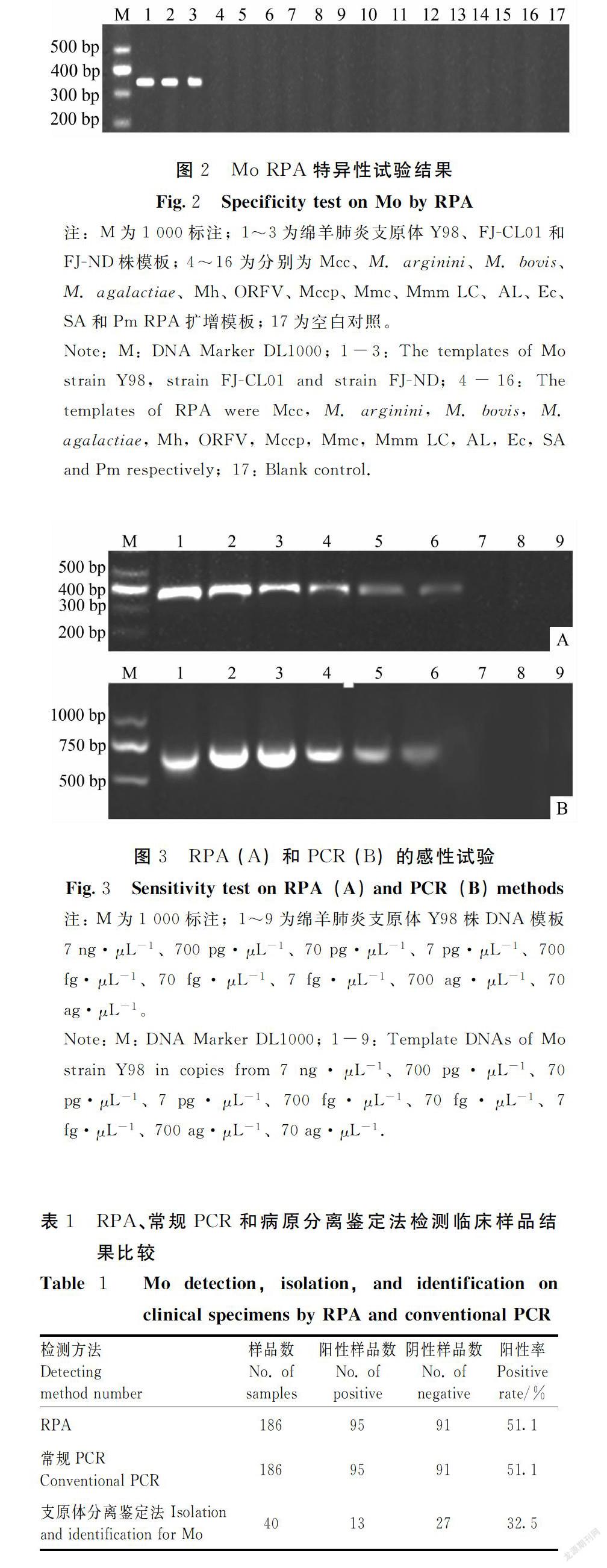
2.1 RPA反应条件的优化及扩增产物的鉴定
经优化后,50 μL反应体系最佳反应时间和温度为38℃反应30 min。应用优化后的体系对Mo Y98株进行扩增并对扩增产物进行测序,结果显示,Mo Y98经琼脂糖凝胶电泳扩增出一条约350 bp的目的片段,与预期大小一致(图1)。测序结果显示, RPA扩增的Mo P80基因序列长度为353 bp,与参考的KM435069.1株序列同源性为99.8%,与预期结果相符。
2.2 特异性扩增
利用建立的RPA检测方法对相应细菌的核酸进行检测,同时设置空白对照。结果显示,仅 MoY98株以及本研究室分离株FJ-CL01和FJ-ND株的DNA扩增出与预期大小相符的目的条带,而Mcc、M.arginini、M.bovis、M.agalactiae、Mh、ORFV、Mccp、Mmc、Mmm LC、AL、Ec、SA、Pm和去离子水样品均为阴性(图2),表明建立的RPA方法具有良好的特异性。
2.3 敏感性试验
经测定, Mo核酸质量浓度为70 ng·μL-1,以10倍系列稀释成7 ng·μL-1、700 pg·μL-1、70 pg·μL-1、7 pg·μL-1、700 fg·μL-1、70 fg·μL-1、7 fg·μL-1、700 ag·μL-1、70 ag·μL-1 进行RPA和常规PCR敏感性试验,结果显示:建立的Mo RPA方法与常规PCR方法的检测限均为70 fg·μL-1,表明RPA与常规PCR方法的敏感性一致(图3)。
2.4 重复性试验
利用建立的Mo RPA方法对2份Mo阳性样品,Mcc、M.arginini、M.bovis、M.agalactiae、Mh、ORFV、Mccp、Mmc、Mmm LC 、AL、Ec、SA、Pm各1份作阴性对照进行批内重复和批间重复试验,结果显示,批内和批间试验结果均一致(图略),即除了2份Mo阳性样品有扩增条带,其余样品均无扩增条带,表明重复性好。
2.5 临床样品检测结果
利用建立的RPA方法对186份临床样品进行检测,同时进行PCR检测,并对其中40份肺脏样品进行支原体的分离鉴定。结果显示(表1),RPA方法Mo阳性率为51.1%(95/186),PCR方法检测Mo阳性率为51.1%(95/186),同时,分离鉴定法的13份Mo阳性样品及常规PCR法检测出的95份阳性样品经RPA检测,结果均为阳性,表明建立的RPA方法与常规PCR方法的符合率为100%,可作为临床样品中Mo快速检测的候选技术。
3 讨论与结论
RPA是近年来新兴起来的一种常温扩增技术,该技术的检测原理是在目的DNA片段扩增过程中,重组酶与引物结合形成的复合物与DNA结合,并沿着DNA链寻找引物的同源序列,一旦引物定位了同源序列,就会发生链交换反应形成并启动DNA合成[24]。同传统的PCR方法相比,该技术具有如下优点:首先是试剂使用方便,方便保存;其次是反应时间短,反应过程一般在40 min内;最后是对设备要求低,仅需一台水浴锅即可完成反应,无需高昂仪器设备。由于该项技术的这些优点,有利于基层单位以及边远山区的推广应用。因此本研究建立了一種以Basic RPA为基础的快速检测Mo的方法。
任何一种的检测技术,都存在优缺点。RPA方法作为一种新型的检测技术,也存在一定的不足,比如引物的设计就没有专门的软件,而且RPA引物相比于常规PCR或荧光定量PCR引物要长10~20个碱基,还不能出现连续重复序列以及发卡结构,且GC含量要控制在30%~70%[25]。由于支原体基因组GC含量偏低,加上RPA引物设计具有以上不足,这就给本研究引物设计造成困难,然而本研究团队在前期建立Mo实时荧光定量PCR、多重PCR的过程中,找到了符合RPA引物设计特性的基因P80,利用Oligo7.9软件针对P80保守性序列设计多对引物,随后通过实验验证以及体系优化筛选到1对效果较好的引物。反应条件优化方面,由于RPA反应体系已经是比较成熟的,因此本研究仅对反应温度和反应时间进行优化,优化后的最佳反应条件为38℃孵育30 min。特异性验证方面,本研究建立的Mo RPA检测方法对Mcc、M.arginini、M.bovis、M.agalactiae、Mh、ORFV、Mccp、Mmc、Mmm LC 、AL、Ec、SA、Pm等均无交叉反应,表明建立的RPA方法特异性强。灵敏度检测结果显示,建立的Mo RPA检测方法的最低检测限为70 fg·μL-1,与常规PCR结果一致,然而本研究建立的方法在灵敏度方面与国内外研究报道还是有一定的差距,如刘占旭等[26]建立的猪嵴病毒RPA诊断方法最低检测限达到了3.36×102 copies·μL-1,比常规PCR灵敏度高100倍;Wang等[27]建立的猪圆环病毒Ⅱ型的qRPA方法最低检测限达到了100 copies·μL-1,比常规PCR灵敏度高10倍;Yang等[28]建立的羊口疮病毒qRPA方法最低检测限为100 copies·μL-1,樊晓旭等[29]建立的塞尼卡病毒RPA-LFD检测方法最低检限为56 copies·μL-1。本研究RAP方法敏感性较低的原因可能是由于Mo基因组GC含量偏低,导致扩增效率低下;此外引物长度过长,也可能是影响敏感性较低的一个原因。重复性验证结果显示,本研究建立的RPA方法可重复性较好。
因此,本研究成功建立了一种具有特异性强、重复性好、操作简便的Mo RPA临床快速检测方法,为Mo的临床快速检测和流行病学调查提供了新的手段。
参考文献:
[1]BESSER T E, FRANCES CASSIRER E, HIGHLAND M A, et al. Bighorn sheep pneumonia: sorting out the cause of a polymicrobial disease [J]. Preventive Veterinary Medicine, 2013, 108(2-3):85-93.
[2]DASSANAYAKE R P, SHANTHALINGAM S, HEMDON C N, et al. Mycoplasma ovipneumoniae can predispose bighorn sheep to fatal Mannheimia haemolytica pneumonia [J]. Veterinary Microbiology, 2010, 145(3-4):354-359.
[3]NIANG M, ROSENBUSCH R F, ANDREWS J J, et al. Demonstration of a capsule on Mycoplasma ovipneumoniae [J]. American Journal of Veterinary Research,1998, 59(5):557-562.
[4]NIANG M, ROSENBUSCH R F, DEBEY M C, et al. Field isolates of Mycoplasma ovipneumoniae exhibit distinct cytopathic effects in ovine tracheal organ cultures [J]. Zentralbl Veterinarmed A,1998, 45(1):29-40.
[5]ETTORRE C, SACCHINI F, SCACCHIA M, et al. Pneumonia of lambs in the Abruzzo region of Italy: anatomopathological and histopathological studies and localisation of Mycoplasma ovipneumoniae [J].Veterinaria Italiana, 2007, 43(1): 149-155.
[6]RIFATBEGOVIC M, MAKSIMOVIC Z, HULAJ B,et al. Mycoplasma ovipneumoniae associated with severe respiratory disease in goats [J]. Veterinary Record, 2011, 168(21): 565.
[7]BESSER T E, CASSIRER E F, POTTER K A, et al. Association of Mycoplasma ovipneumoniae infection with population-limiting respiratory disease in free-ranging Rocky Mountain bighorn sheep (Oviscanadensiscanadensis) [J]. Journal of Clinical Microbiology, 2018, 46(2): 423-430.
[8]YANG F, DAO X, RODRIGUEZ-PALACIOS A, et al. A real-time PCR for detection and quantification of Mycoplasma ovipneumoniae [J]. Journal of Veterinary Medical Science, 2014, 76(12): 1631-1634.
[9]林裕勝, 江锦秀, 江斌, 等.绵羊肺炎支原体、丝状支原体山羊亚种和山羊支原体山羊肺炎亚种多重PCR检测方法的建立[J]. 中国预防兽医学报, 2018, 40(9): 812-817.LIN Y S, JIANG J X, JIANG B, et al. Development of a triplex PCR assay for detection of Mycoplasma ovipneumoniae, M.mycoides subsp. capri and M.capricolum subsp. capripneumoniae [J]. Chinese Journal of Preventive and Veterinary Medicine, 2018, 40(9): 812-817.(in Chinese)
[10]LI M, MA C J, LIU X M, et al. Molecular cloning of HSP70 in Mycoplasma ovipneumoniae and comparison with that of other mycoplasmas [J]. Genetics and Molecular Research, 2011, 10(2): 834-848.
[11]YANG F, TANG C, WANG Y, et al. Genome sequence of Mycoplasma ovipneumoniae strain SC01[J]. Journal of Bacteriology, 2011, 193(18): 5018.
[12]RONG G, ZHAO J M, HOU G Y, et al. Seroprevalence and molecular detection of Mycoplasma ovipneumoniae in goats in tropical China [J]. Tropical Animal Health and Production, 2014, 46(8):1491-1495.
[13]赵军, 王川庆, 吴艳阳, 等. 一种用于检测胞内劳森菌的RPA引物及其检测方法:CN201710652790.5 [P]. 2018-08-24.ZHAO J, WANG C Q, Wu Y Y, et al. A RPA primer for detection of intracellular Lawson bacteria and its detection method:CN201710652790.5 [P]. 2018-08-24.(in Chinese)
[14]EULER M, WANG Y J, OTTO P, et al. Recombinase polymerase amplification assay for rapid detection of Francisella tularensis [J]. Journal of Clinical Microbiology, 2012, 50(7):2234-2238.
[15]ABD EI W A, EI-DEDB A, EI-THOLOTH M, et al. A portable reverse transcription recombinase polymerase amplification assay for rapid detection of foot and mouth disease virus [J]. PLoS One, 2013, 8(8):e71642.
[16]王建昌, 王金鳳, 刘立兵, 等. 非洲猪瘟病毒RPA等温检测方法的建立[J]. 中国动物检疫, 2016, 33(7): 78-81, 94.WANG J C, WANG J F, LIU L B, et al. Rapid and sensitive detection of African swine fever virus by recombinase polymerase amplification [J]. China Animal Health Inspection, 2016, 33(7): 78-81, 94.(in Chinese)
[17]LILJANDER A, YU M, O'BRIEN E, et al. Field-applicable recombinase polymerase amplification assay for rapid detection ofMycoplasma capricolum subsp. capripneumoniae[J]. Journal of Clinical Microbiology,2015, 3(9): 2810-2815.
[18]费媛媛, 李兆才, 娄忠子, 等.动物衣原体RPA检测方法的建立[J]. 中国兽医科学, 2018, 48(2): 142-147.FEI Y Y, LI Z C, LOU Z Z, et al. Detection of Chlamydia in livestock by recombinase polymerase amplification [J]. Chinese Veterinary Science, 2018, 48(2): 142-147.(in Chinese)
[19]BOYLE D S, MCNEMEY R, TENG L H, et al. Rapid detection of Mycobacterium tuberculosis by recombinase polymeraseamplification[J].PLoS One, 2014, 9(8): 103091.[20]KERSTING S, RAUSCH V, BIER F F, et al. Rapid detection of Plasmodium falciparum with isothermal recombinase polymerase amplification and lateral flow analysis [J] .Malar Journal, 2014 (13): 99.
[21]林裕胜, 江锦秀, 张靖鹏, 等. 绵羊肺炎支原体TaqMan 荧光定量PCR检测方法的建立[J]. 中国预防兽医学报, 2018, 40(4): 316-320.LIN Y S, JIANG J X, ZHANG J P, et al. Establishment of a TaqMan real-time fluorescent quantitative PCR assay for detection of Mycoplasma ovipneumoniae [J]. Chinese Journal of Preventive Veterinary Medicine, 2018, 40(4): 316-320.(in Chinese)
[22]林裕胜, 江锦秀, 江斌, 等. ORFV和Mo双重PCR检测方法的建立及应用[J]. 农业生物技术学报, 2017, 25(8): 1374-1380.LIN Y S, JIANG J X, JIANG B, et al. Establishment and application of a duplex PCR assay for detection of Orf virus (ORFV) and Mycoplasma ovipneumoniae (Mo) [J]. Journal of Agricultural Biotechnology, 2017, 25(8): 1374-1380.
[23]江锦秀, 林甦, 林裕胜, 等. 绵羊肺炎支原体FJ01-CL株的分离和鉴定[J]. 福建农业学报, 2015, 30(5): 430-434.JIANG J X, LIN S, LIN Y S, et al. Isolation and identification of Mycoplasma ovipneumoniae FJ01-CL [J]. Fujian Journal of Agricultural Sciences, 2015, 30(5): 430-434.(in Chinese)
[24]吴耀东, 徐民俊, 郑文斌, 等.重组聚合酶扩增技术及其在动物病原快速检测中的应用[J]. 中国兽医学报, 2016, 26(10): 1797-1802.WU Y D, XU M J, ZHENG W B, et al. Development of recombinant polymerase amplification technology and its applications in quick diagnosis of animal pathogen [J]. Chinese Journal of Veterinary Science, 2016, 26(10): 1797-1802.(in Chinese)
[25]秦立得, 南文龙, 陈义平, 等. 重组聚合酶扩增技术及其在动物病毒检测中的应用[J]. 中国动物检疫, 2017,34(5): 81-85.QIN L D, NAN W L, CHEN Y P, et al. Recombinase polymerase amplification and its application in detection of animal diseases [J]. China Animal Health Inspection, 2017, 34(5):81-85.(in Chinese)
[26]刘占旭,刘新生,方玉珍, 等.猪嵴病毒RPA快速诊断方法的建立和应用[J]. 中国兽医科学, 2018, 48(9): 1073-1079.LIU Z X, LIU X S, FANG Y Z, et al. Development and application of a recombinase polymerase amplification assay for rapid diagnosis of porcine kobuvirus [J]. Chinses Veterinary Science, 2108, 48(9): 1073-1079.(in Chinese)
[27]WANG J, WANG J, LIU L, et al. Rapid detection of Porcine circovirus-2 by recombinase polymerase amplification [J]. Journal of Veterinary Diagnostic Investigation, 2016, 28(5):1-4.
[28]YANG Y, QIN X, WANG G, et al.Development of a fluorescent probe-based recombinase polymerase amplification assay for rapid detection of Orf virus [J]. Virology Journal, 2015, 12(1): 206.
[29]樊曉旭, 宋翥远, 赵永刚, 等. 塞尼卡病毒重组聚合酶扩增-侧流层析试纸条检测方法的建立[J]. 中国预防兽医学报, 2018, 40(5): 406-410.
FANG X X, SONG Z Y, ZHAO Y G, et al. Establishment of recombinase polymerase amplification-lateral flow dipstick for the detection of Seneca valley virus [J].Chinese Journal of Preventive Veterinary Medicine, 2018, 40(5): 406-410.(in Chinese)
(责任编辑:张 梅)
