南瓜MSAP 体系的优化及引物筛选
2019-09-09姜立娜周俊国
江 毅 姜立娜周俊国
(河南科技学院园艺园林学院,河南省园艺植物资源利用与种质创新工程研究中心,河南新乡 453003)
DNA 甲基化是引起植物表观遗传学改变的重要因素,通过检测甲基化状态及其改变动态,可以揭示生物体正常状态下基因表达的时空调控以及因甲基化错误造成的发育障碍和疾病。检测DNA 甲基化的方法有重亚硫酸氢盐法、甲基化敏感扩增多态性(methylation sensitive amplification polymorphism,MSAP)分析、甲基化检测芯片技术和甲基化测序等,利用这些技术来确定某个碱基位点的甲基化情况。其中,MSAP 分析因操作简便、重复性好、费用低等优点被广泛应用,是植物DNA 甲基化水平检测的重要技术(Avramidou et al.,2015)。MSAP 技 术 是 在AFLP(amplified fragment length polymorphism)技术的基础上建立起来的,主要是利用可识别5′-CCGG 序列并且对DNA 甲基化敏感性不同的同裂酶HpaⅡ和MspⅠ,与识别GAATTC 序列的EcoRⅠ配对组合对基因组进行酶切,然后再加上相应的限制性内切酶的接头,利用接头序列设计引物,对5′-CCGG 位点甲基化进行特异性扩增。HpaⅡ和MspⅠ对甲基化的敏感程度不同,HpaⅡ不能切割双链内外侧甲基化,即不能切割“CmCGG、mCCGG”和“mCmCGG”,但可切割单链甲基化的序列;而MspⅠ能切割单链或双链 “CmCGG”,不能切割“mCCGG”甲基化序列。MSAP 在全基因组范围检测CCGG 位点的胞嘧啶甲基化变化,从而产生不同酶切产物,最终产生可见的具有不同酶切形式的谱带,以明确DNA甲基化信息。但是,MSAP 技术的局限性是不能完成非CCGG 位点的胞嘧啶甲基化检测。自Xiong等(1999)首次将MSAP 技术用于检测DNA 甲基化以来,这项技术已运用于医学(Shridhar et al.,2015;吴璇 等,2018)、植物(黄韫宇 等,2013;黄豪永,2014)、动物(吴宇,2017;曼则热 · 朱尔丁 等,2018)等多个领域。
南瓜为葫芦科南瓜属(Cucurbita)中的一年生草本植物,起源于美洲大陆,是世界范围内广泛栽培的传统作物。南瓜根系发达,种质资源丰富,有较高的经济价值和观赏价值(林德佩,2000)。随着分子植物育种技术的飞速发展,有研究者运用SSR(朱海生 等,2018)、AFLP(李雪,2014)、SNP(周洋洋 等,2018)等分子标记技术深入分析南瓜果肉、蔓性、果皮颜色等性状的基因遗传信息,而关于南瓜MSAP 技术的应用较少(Aliki et al.,2019)。本试验以南瓜自交系北观为材料,利用正交设计的方法优化南瓜MSAP 技术中的重要影响因素,并最终筛选出36 对多态性引物,以期为南瓜表观遗传分析奠定基础。
1 材料与方法
1.1 试验材料与试剂
试验材料为南瓜高代自交系北观,系印度南瓜种(Cucurbita maxima),由河南科技学院南瓜育种课题组提供。试验时间为2018 年10~12 月,试验地点为河南科技学院分子实验室。
试验所用植物基因组DNA 提取试剂盒、限制性内切酶EcoRⅠ(15 U · μL-1)、HapⅡ(HpaⅡ,MspⅠ)(10 U · μL-1)购自北京百泰克生物技术有限 公 司;T4 DNA ligase(350 U · μL-1)、Taq DNA聚合酶(5 U · μL-1)、dNTP Mixture(2.5 mmol · L-1)购自宝生物工程有限公司;试验所用引物由美国英杰生命技术有限公司合成。
1.2 试验方法
1.2.1 基因组DNA 的提取及检测 当南瓜幼苗生长至两叶一心时,取真叶0.5~1.0 g,分别用SDS 法(刘殿林 等,2002)、CTAB 法(纪元 等,2012)、SDS-CTAB 法(徐彦军和刘洋,2012)和试剂盒法进行基因组DNA 提取。其中,试剂盒法提取DNA 按照试剂盒说明书进行。提取结果用1%琼脂糖凝胶电泳检测,利用核酸蛋白检测仪测定A260/A280和A260/A230波长下的OD 值以及DNA 的浓度。
1.2.2 MSAP 两步式酶切时间与酶量的优化 选用
HapⅡ(HpaⅡ,MspⅠ)与EcoRⅠ对基因组DNA进行两步式酶切。第一步酶切体系为21 μL,包括基 因 组DNA 500 ng,HapⅡ与2.1 μL 10×L Buffer反应液,剩余用ddH2O 补足。其中酶切时间优化设置5 个水平,分别为2、4、6、8 h 和10 h;HapⅡ用量设置3 个水平,即0.5、1.0 μL 和1.5 μL,在PCR 仪中37 ℃酶切。酶切结束后,取酶切产物8 μL,加入2 μL 溴酚蓝混合,用1%琼脂糖凝胶电泳检测,确定最佳酶切时间和酶量。
第二步酶切体系为25 μL,包括21 μL 第一步酶切产物,2.9 μL 10 × H Buffer,EcoRⅠ不同酶量设置3 个水平,即0.4、0.6 μL 和0.8 μL,剩余用ddH2O 补足。将上述体系于PCR 仪中37 ℃酶切,酶切时间优化设置4 个水平,分别为6、8、10 h和12 h,用1%琼脂糖凝胶电泳检测,确定最佳酶切时间和酶量。
1.2.3 MSAP 连接体系的建立 EcoRⅠ接头合成:取EA1 和EA2 引物干粉,离心后用TE 缓冲液稀释至浓度为10 μmol · L-1,分别取稀释液50 μL,在200 μL PCR 管中混匀,在PCR 仪中进行接头处理:94 ℃ 4 min,65 ℃ 10 min,缓慢冷却至室温,4 ℃保存备用。HapⅡ接头合成同EcoRⅠ接头的合成。
取 第 二 步 酶 切 产物21 μL 加入T4 DNA Ligase,EcoRⅠ接头EA1+2、Hap Ⅱ接头MH1+2(5 μmol · L-1)各1.0 μL,2.8 μL 10×T4 DNA Ligase Buffer,于16 ℃进行连接。其中,T4 DNA ligase的用量分别为0.5、1.0 μL,连接时间优化设置6 个水平,分别为2、4、6、8、10、12 h,用1%琼脂糖凝胶电泳检测,确定最佳连接体系。
1.2.4 MSAP 预扩增体系的优化 利用正交设计L16(45)试验(表1),对MSAP 预扩增体系中的连接产物、Taq 酶、dNTP Mixture、Mg2+和引物5个因素进行筛选,确定预扩增体系的最佳水平。预扩增反应PCR 程序为:94 ℃ 2 min;94 ℃ 30 s,56 ℃ 1 min,72 ℃ 1 min,共20 个循环;72 ℃ 5 min;4 ℃保存;预扩增产物用1%琼脂糖电泳凝胶检测,确定最佳扩增体系。
1.2.5 MSAP 选择性扩增体系 在上述试验的基础上,将相应的预扩增产物分别稀释40、80、120、160 倍进行选择性扩增,以确定最佳稀释倍数。
选择性扩增体系为25 μL,其中预扩增产物稀释后的模板4.0 μL,其他与预扩增体系相同。选择性扩增的PCR 程序为94 ℃预变性1 min;94 ℃变性30 s,65 ℃退火30 s,72 ℃延伸1 min,共13个循环(每个循环退火温度降0.7 ℃);94 ℃变性30 s,56 ℃退火30 s,72 ℃延伸1 min,共23 个循环;72 ℃延伸10 min,4 ℃保存。PCR 扩增产物通过1%琼脂糖凝胶电泳进行检测。1.2.6 MSAP 体系验证及引物筛选 利用优化的体系,对9 个EcoRⅠ引物(E1~E9)和9 个Hap Ⅱ(HpaⅡ,MspⅠ)引物(MH1~MH9)组成的81对引物组合进行筛选(表2),通过6%聚丙烯酰胺凝胶电泳检测(郭小菲 等,2014),验证MSAP 体系的可行性。

表1 MSAP 预扩增体系正交设计表 μL

表2 MSAP 分析所用引物
2 结果与分析
2.1 不同方法提取DNA 的比较

图1 不同DNA 提取方法电泳检测结果编号1~3 为CTAB 法;4~6 为SDS-CTAB 法;7~9 为SDS 法;10~12 为试剂盒法。
MSAP 分析对基因组DNA 的质量要求较高,本试验分别使用SDS 法、CTAB 法、SDS-CTAB法和试剂盒法提取基因组DNA,所得DNA 用1%琼脂糖凝胶电泳检测。结果如图1 所示,用CTAB法提取的DNA 没有DNA 条带,只有一片弥散;采用SDS-CTAB 法提取的DNA 可以看到明显的DNA 条带,但浓度较低且操作流程耗时较长;采用SDS 法提取的DNA 拖带明显,无法满足试验所用;试剂盒法提取DNA,条带清晰明亮。结合表3 中测定的基因组DNA 浓度以及A260/A280、A260/A230的数值,最终选择试剂盒法提取的南瓜基因组DNA 进行后续试验。
2.2 酶切体系优化
酶切效果对后期试验结果有较大的影响。用Hap Ⅱ对基因组DNA 进行第一步酶切,由图2 可以看出,Hap Ⅱ用量和酶切时间对基因组DNA 酶切效果没有明显影响,在试验中为节约时间和成本,故选择37 ℃酶切2 h,Hap Ⅱ用量0.5 μL。
用EcoRⅠ内切酶进行第二步酶切,经1%琼脂糖凝胶电泳检测,结果如图3 所示,1~6 泳道弥散较为明显,7~9 泳道弥散隐约可见,10~12 泳道没有明显残留,说明酶切完全;考虑试验时间和经济成本,故确定第二步酶切时间为6 h,EcoRⅠ用量为0.4 μL。
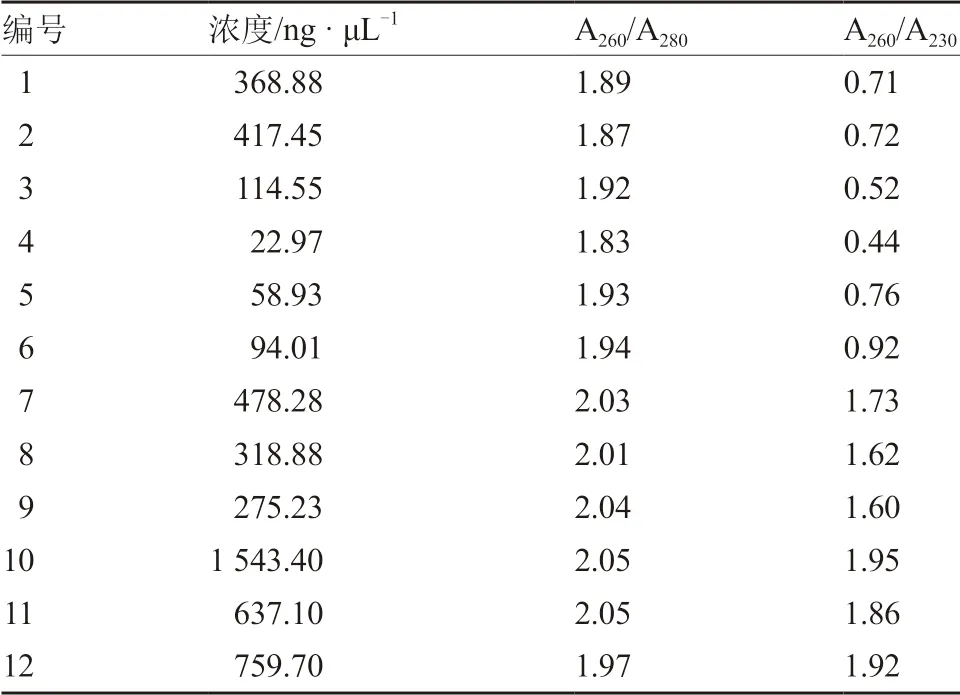
表3 不同DNA 提取方法浓度检测
2.3 连接体系优化
连接时间对南瓜MSAP 的影响较大。由图4可以看出,各泳道均有弥散,而11 泳道弥散的连续性较好,说明连接效果较好,故本试验选择连接时间为12 h,T4 DNA Ligase 用量为0.5 μL。
2.4 预扩增体系正交优化

图2 不同酶切时间与HapⅡ用量电泳结果M 为DL 2000;1、2、3 泳道为酶切10 h;4、5、6 泳道为酶切8 h;7、8、9 泳道为酶切6 h;10、11、12 泳道为酶切4 h;13、14、15泳道为酶切2 h;1、4、7、10、13 泳道 Hap Ⅱ用量为0.5 μL,2、5、8、11、14 泳道Hap Ⅱ用量为1.0 μL;3、6、9、12、15 泳道 Hap Ⅱ用量为1.5 μL。

图3 不同酶切时间与EcoRⅠ用量电泳结果M 为DL 2000;1、2、3 泳道为酶切12 h;4、5、6 泳道为酶切10 h;7、8、9 泳道为酶切8 h;10、11、12 泳道为酶切6 h;其中1、4、7、10 泳道EcoRⅠ用量为0.4 μL;2、5、8、11 泳道EcoRⅠ用量为0.6 μL;3、6、9、12 泳道EcoRⅠ用量为0.8 μL。
对16 个处理的预扩增产物的电泳分析结果表明(图5),PCR 扩增结果具有较大差异。泳道4、6、8、10、11 弥散较亮,说明预扩增产物浓度较高;2、3、5、7、9、12 泳道弥散隐约可见,其他泳道则无弥散。综合来看,表1 中6 号处理可作为预扩增反应最佳体系,即1.5 μL 10×PCR Buffer(Mg2+plus),2.0 μL连接产物,0.6 μL Taq 酶,2.0 μL dNTP Mixture,0.4 μL(10 μmol · L-1)上下游引物。

图4 优化连接体系电泳结果M 为DL 2000;1、2 泳道为连接2 h,T4 DNA Ligase 用量分别为0.5、1.0 μL;3、4 泳道为连接4 h,T4 DNA Ligase 用量分别为0.5、1.0 μL;5、6 泳道为连接6 h,T4 DNA Ligase 用量分别为0.5、1.0 μL;7、8 泳道为连接8 h,T4 DNA Ligase 用量分别为0.5、1.0 μL;9、10 泳道为连接10 h,T4 DNA Ligase 用量分别为0.5、1.0 μL;11、12 泳道为连接12 h,T4 DNA Ligase 用量分别为0.5、1.0 μL。
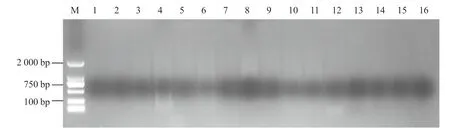
图5 正交优化预扩增电泳结果M 为DL 2000;1~16 为不同处理编号,各体系用量对应表1。
2.5 预扩增产物不同稀释倍数优化
预扩增产物稀释不同倍数后作为选择性扩增模板,扩增结束后用1%琼脂糖凝胶电泳检测。由图6 可以看出,泳道3 较其他泳道明亮,条带清晰,说明扩增产物浓度较高,故将预扩增产物稀释120倍作为选择性扩增模板。

图6 预扩增产物不同稀释倍数电泳结果M 为DL 2000;泳道1~4 分别为预扩增产物稀释40、80、120、160 倍。

图7 MSAP 体系验证及部分引物筛选电泳结果M 为DL 500;其他泳道为不同引物组合。
2.6 利用聚丙烯酰胺凝胶电泳验证MSAP 体系及引物筛选
为证明上述试验的可行性,利用6%聚丙烯酰胺凝胶电泳验证MSAP 体系,并从81 对引物中筛选出36 对适于南瓜MSAP 分析的引物。试验结果显示(图7),引物条带清晰丰富,多态性好。
3 结论与讨论
MSAP 是利用对DNA 甲基化敏感性不同的同裂酶切割DNA,从而产生不同酶切产物,经过添加接头、PCR 扩增、PAGE 电泳、银染,最终产生可见的具有不同酶切形式的谱带,以明确DNA 甲基化信息。MSAP 分析在操作时需要有较高质量的基因组DNA 为前提,若DNA 中含有酚类、RNA酶类等杂质则会影响后续试验进度。在本试验中,利用4 种不同DNA 提取方法进行南瓜基因组DNA的提取,最终确定试剂盒法提取的DNA 条带单一,浓度适中,更能满足试验要求。
MSAP 能够识别多种生物的甲基化比例,具有普遍性,利用基因组DNA 即可进行遗传多样性分析(邱峥艳 等,2012)。在建立南瓜MSAP 体系时,根据预试验结果综合考虑每一步因素的体系范围,利用两歩式酶切,分别优化酶切时间和酶量,因为这两个条件是酶切完全的关键因素,若酶切不完全会造成甲基化分析不准确,酶切过度则会出现非特异性片段。本试验对第一步酶切时间优化设置5 个水平,HapⅡ用量优化设置3 个水平,得出当酶切2 h、酶量为0.5 μL 时,酶切较完全;第二步酶切同样优化酶切时间和EcoR Ⅰ用量,经过电泳检测证明,酶切时间6 h、酶量为0.4 μL 时效果较好。连接体系是在酶切完全的基础上添加接头,因此先要处理接头,使接头由单链变为双链,在本试验中连接时间为12 h、T4 DNA Ligase 用量为0.5 μL 的效果较好,这与吕晓婷等(2012)、周萌萌等(2018)优化MSAP 体系的结果不同,若连接时间过短,会造成连接不充分,影响片段扩增。
MSAP 分析扩增反应关系到DNA 全甲基化、未甲基化、半甲基化概率。在预扩增试验中,由于影响PCR 扩增的因素较多,故利用正交设计优化了5 个主要影响因子,从而为整个试验节约了成本和时间,优化最佳体系为连接产物2.0 μL,10×PCR Buffer(Mg2+plus)1.5 μL,dNTP Mixture(2.5 mmol · L-1)2.0 μL,Taq DNA 聚合酶(5 U · μL-1)0.6 μL,上下游引物(10 μmol · L-1)各0.4 μL;将预扩增产物作为选择性扩增体系的模板,稀释120 倍可以得到清晰的条带。上述所优化的MSAP 体系,经聚丙烯酰胺凝胶电泳检测显示体系稳定,条带清晰,可重复,同时也筛选出36 对适于南瓜MSAP分析的多态性引物,为后续开展南瓜和其他葫芦科作物表观遗传研究提供一定的理论基础。
