基于全基因组SNP标记的抗旱冬小麦品种的遗传多样性分析
2019-03-26左煜昕马靖福刘媛张沛沛栗孟飞程宏波陈思瑾杨德龙
左煜昕,马靖福,刘媛,张沛沛,栗孟飞,程宏波,陈思瑾,杨德龙
(1.甘肃省干旱生境作物学重点实验室,甘肃 兰州 730070;2.甘肃农业大学生命科学技术学院,甘肃 兰州 730070)
小麦(TriticumaestivumL.)是世界上仅次于水稻的主粮作物,为人类提供20%的热量和蛋白质,其产量高低直接关系到世界粮食安全[1-2].在我国,小麦作为四大主粮作物之一,根据小麦的生产特性及地理环境,小麦的生产区域可以划分为北方冬麦区、南方冬小麦区和春麦区,并且北方冬麦区为全国小麦的主要生产区域,其产量超过全国小麦产量的60%[3].随着全球气候变暖,旱灾频发,导致该区域小麦减产严重[1,4-5].因此,深入研究不同地理区域的冬小麦抗旱基因的遗传多样性,挖掘优异抗旱种质资源,对冬小麦抗旱遗传改良具有重要意义.
小麦遗传资源多样性的研究为小麦新品种的选育提供了理论指导[6].吴秀婷等[7]利用分蘖、株高、叶片数、单株总叶面积、抽穗时间、灌浆时间和籽粒产量等形态学指标对小麦遗传多样性分析发现,育种亲本的单一导致育成品种的遗传多样性下降,增加了今后新品种育成的难度.近年来,随着小麦基因组测序的深入,分子标记得到迅速发展,尤其是单核苷酸多态性(single nucleotide polymorphism,SNP)标记由于其具有数量多、分布广、高度稳定、适于高通量检测、易于基因分型等特点[8-9],被广泛应用地应用于作物遗传多样性研究[10].Akhunov等[9]报道了Illumina技术平台开发的90K SNP芯片技术,并将其成功应用于四倍体小麦[9]和六倍体小麦[12-13]的多态性检测.Ren 等[14]利用覆盖小麦全基因组的946个SNP多态性位点对150份硬粒小麦种质进行遗传多样性分析,发现在该材料群体中小麦品种的遗传多样性呈地方品种到早期选育种逐渐降低的趋势,并且硬粒小麦品种的遗传多样性随着生态区域的变化而变化,以欧洲和美洲的品种遗传多样性最高;曹廷杰等[15]采用高密度SNP标记90K芯片对河南省近几年审定小麦品种的遗传基础分析发现,在小麦A、B和D 3个基因组上SNP标记的密度不同,呈现出B>A>D的分布趋势,并发现在小麦的4D染色体上分布的多态性标记最少,检测的种质材料间遗传相似度较高,种质遗传多样性逐渐降低,需要引入新的种质资源来拓宽小麦种质的遗传基础.此外,SNP标记在小麦遗传图谱构建、基因精细定位、农艺性状关联分析、分子标记辅助育种等方面发挥着重要作用[7].本研究利用35K基因芯片对不同来源的124份小麦抗旱种质材料进行基因分型,解析其遗传多样性背景和亲缘关系,为旱地冬小麦育种亲本选配提供理论依据.
1 材料与方法
1.1 供试材料
以前期筛选的来源于不同生态区的124份优异抗旱品种为试验材料(表1).这些材料在多年的田间表型鉴定中,其产量性状和与抗旱相关的生理性状表现出显著的差异[16-17].
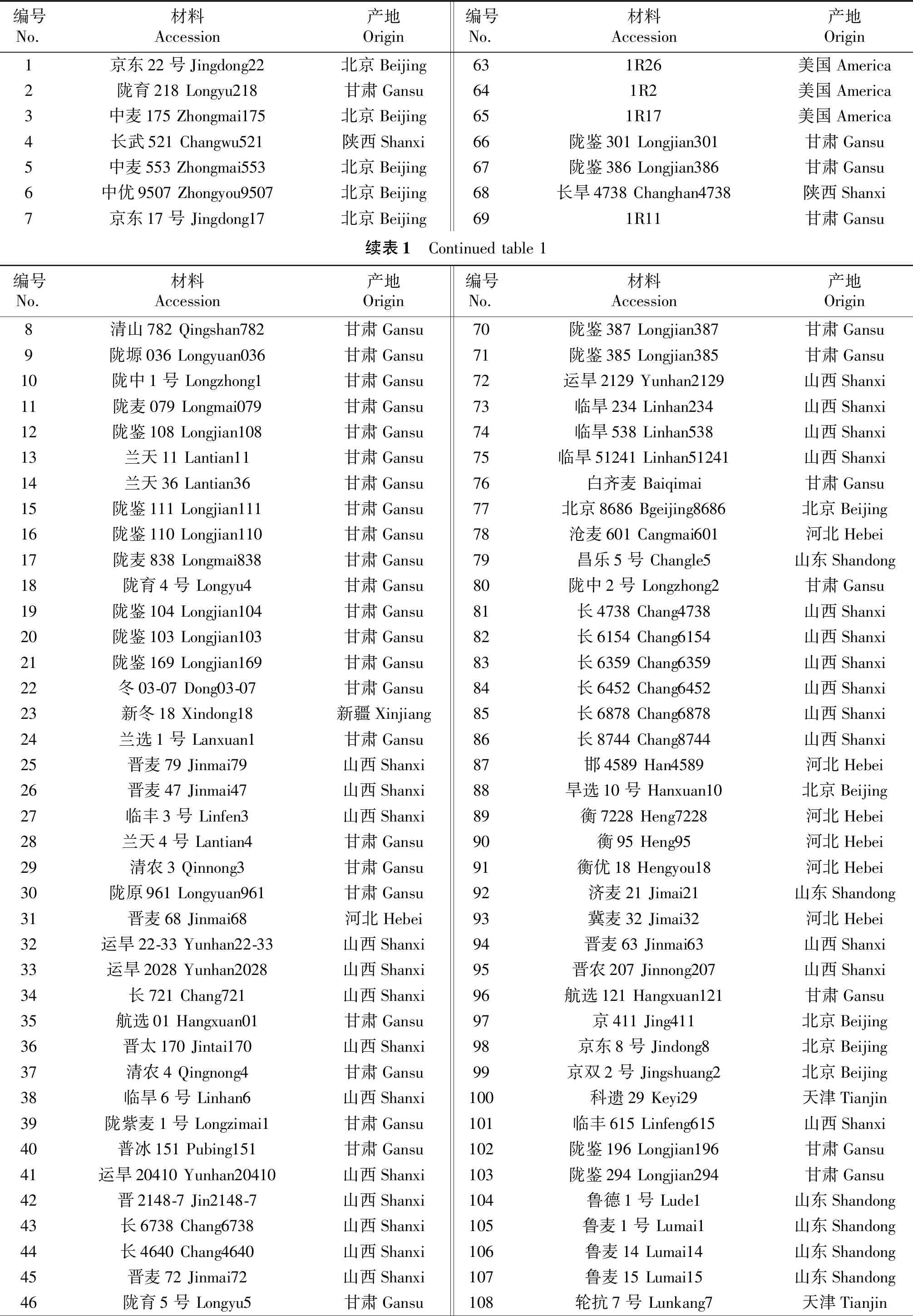
表1 供试小麦材料信息
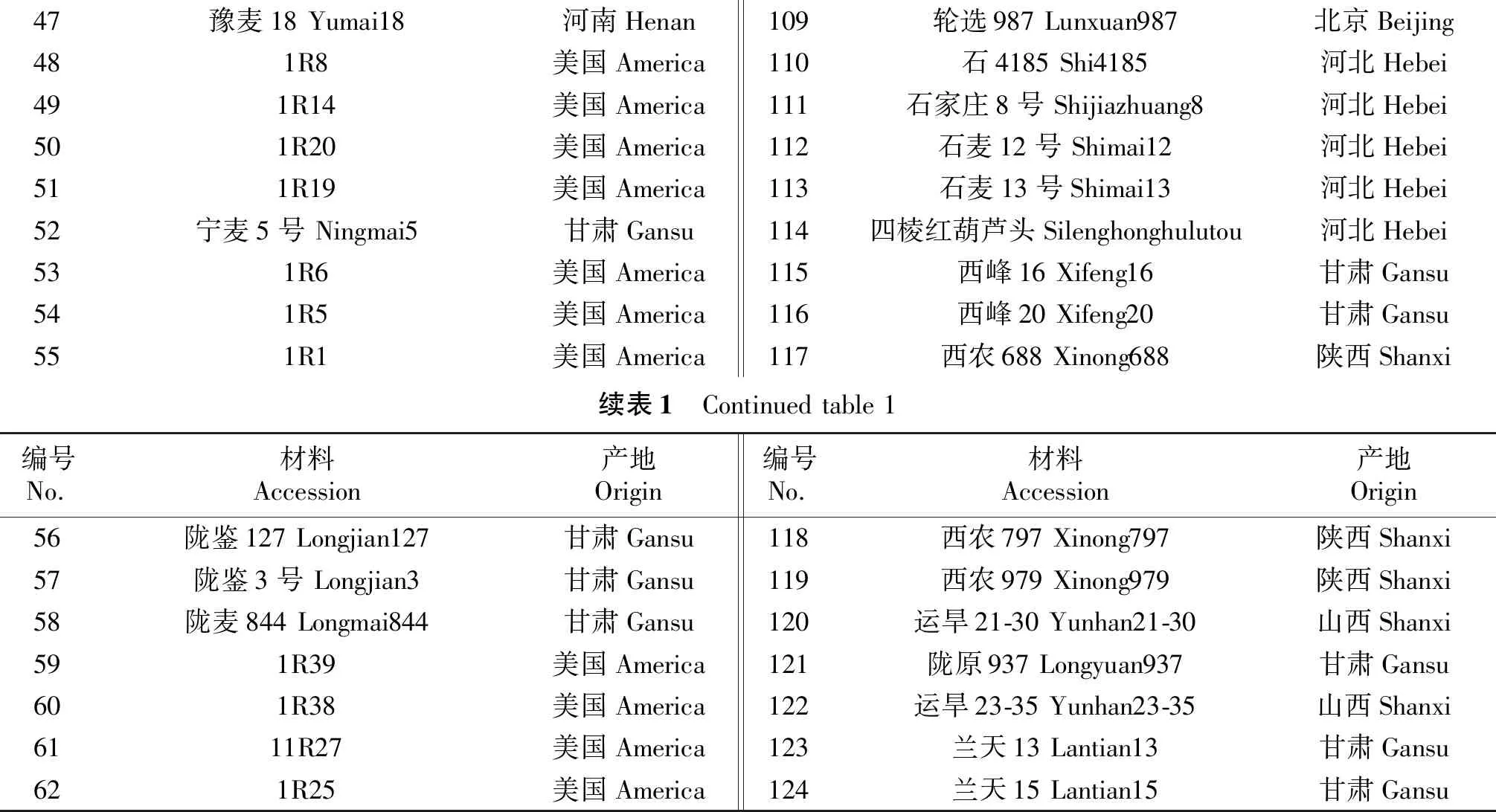
47豫麦18Yumai18河南Henan109轮选987Lunxuan987北京Beijing481R8美国America110石4185Shi4185河北Hebei491R14美国America111石家庄8号Shijiazhuang8河北Hebei501R20美国America112石麦12号Shimai12河北Hebei511R19美国America113石麦13号Shimai13河北Hebei52宁麦5号Ningmai5甘肃Gansu114四棱红葫芦头Silenghonghulutou河北Hebei531R6美国America115西峰16Xifeng16甘肃Gansu541R5美国America116西峰20Xifeng20甘肃Gansu551R1美国America117西农688Xinong688陕西Shanxi续表1 Continuedtable1编号No.材料Accession产地Origin编号No.材料Accession产地Origin56陇鉴127Longjian127甘肃Gansu118西农797Xinong797陕西Shanxi57陇鉴3号Longjian3甘肃Gansu119西农979Xinong979陕西Shanxi58陇麦844Longmai844甘肃Gansu120运旱21-30Yunhan21-30山西Shanxi591R39美国America121陇原937Longyuan937甘肃Gansu601R38美国America122运旱23-35Yunhan23-35山西Shanxi6111R27美国America123兰天13Lantian13甘肃Gansu621R25美国America124兰天15Lantian15甘肃Gansu
1.2 试验方法
1.2.1 小麦基因组DNA提取 采用水培法将小麦幼苗培养至两叶一心,剪取各品系小麦幼嫩叶苗约3 g,置于液氮中冷冻并迅速研磨成粉末.参照CTAB法提取该群体小麦基因组DNA,并用1%的琼脂糖凝胶电泳检测提取的DNA质量,用紫外-可见光光度计测定所提取的DNA在230 nm,260 nm和280 nm处吸光值,检测提取的DNA浓度和相对纯度,D260/D230>2且D260/D280>1.8表示没有RNA污染.
1.2.2 SNP标记分析 小麦基因组DNA送中玉金标记(北京)生物技术股份有限公司进行35K基因芯片扫描分析.基因芯片包括35 143个SNP位点,覆盖小麦全基因组,其中18 689个位点具有品种间的多态性,剔除基因型检出率小于80%以及最低基因频率小于5%的SNP 位点,剩余15 947个SNP位点,用于遗传多样性分析.
1.2.3 SNP遗传多样性统计 利用PowermarkerV2.3.5软件对具有多态性的SNP位点进行一般统计量分析,计算等位位点频率、等位位点数目、遗传多样性指数以及多态性信息量(polymorphism information content,PIC)等参数值.在PowermarkerV2.3.5软件中采用非加权平均法(unweight pair method using arithmetic averages,UPGMA),在遗传相似系数(genetic similarity,GS)的基础上,对该群体材料进行分类,并在Figtree软件中对聚类图进行编辑.
2 结果与分析
2.1 小麦SNP位点遗传多样性分析
利用筛选得到覆盖小麦整个基因组的具有品种间多态性的15 947个SNP标记,对124份小麦抗旱种质材料进行遗传多样性分析,共检测出31 894个等位变异,每个位点均检测到2个等位变异.在PowermarkerV2.3.5软件中计算得到多态性SNP等位变异的频率在0.28~0.97,平均每个SNP等位变异频率为0.75;每个SNP位点遗传多样性指数在0~0.75,平均每个位点遗传多样性指数为0.35;每个SNP位点多态性信息量(PIC)在0~0.70,平均每个位点的多态信息量(PIC)为0.31.
2.2 小麦不同基因组水平上遗传多样性分析
通过对不同基因组遗传多样性分析发现,多态性SNP标记在小麦3个基因组上分布显著不同(图1).其中A、B和D基因组上分别分布了5 010、5 924和5 013个多态性SNP位点,平均每个基因组等位位点频率为0.75、0.74和0.77.分析不同基因组遗传多样性和多态性信息量发现,B基因组遗传多样性较高(0.36),A基因组次之(0.35),D基因组最低(0.33);B基因组的PIC(0.32)较高,A基因组(0.31)次之,D基因组最低(0.29).
2.3 小麦不同同源群水平上的遗传多样性分析
对124份小麦抗旱种质在不同同源群进行遗传多样性比对发现,SNP标记在小麦7个同源群间分布差异显著(图2).其中,第II同源群上分布的标记数目最多(2 945个),第IV同源群最少(1 579个),平均每个同源群上的标记数为2 278个.每个同源群上SNP标记的等位位点频率为0.72(第Ⅰ同源群)~ 0.79(第Ⅶ同源群),平均为0.75.多样性指数在0.31(第Ⅶ同源群)~0.39(第Ⅰ同源群),平均为0.35.PIC值在0.27(第Ⅶ同源群)~ 0.34(第Ⅰ同源群),平均为0.31.由此看出,在小麦同源群间PIC值和遗传多样性指数均表现为第Ⅶ同源群较低,第I同源群最高.
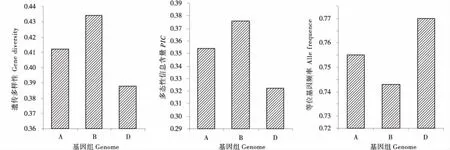
图1 小麦种质材料不同基因组水平上的遗传多样性Figure 1 Genetic diversity of different genomes in wheat accessions

图2 小麦群体不同染色体同源群水平上的遗传多样性Figure 2 Genetic diversity of different homologous groups in wheat population
2.4 小麦不同染色体水平上的遗传多样性
由表2看出,小麦每条染色体分布的多态性SNP标记在441(4D)~1079(5B),平均为759个;每条染色体上的单个SNP标记等位位点频率在0.70(1B)~0.81(4D),平均为0.75;每条染色体上的遗传多样性指数在0.27(4D)~0.41(1B),平均为0.34;PIC值在0.24(4D)~0.36(1B),平均为0.31.由此看出,4D染色体的遗传多样性指数较低,而1B染色体的遗传多样性指数较高.

表2 小麦种质材料不同染色体水平上的遗传多样性
2.5 小麦群体SNP标记的UPGMA聚类分析
通过PowerMarkerV3.2.5计算得出小麦品种间的平均遗传相似系数为0.31,主要分布于0.29~0.36之间,占48.3% (图3).采用UPGMA法在GS为0.38处将124份种质材料分为七类(图4).第Ⅰ类材料共14份,主要包括北京,甘肃,山东和山西品种;第Ⅱ类材料共33份,主要是北京,甘肃和山西品种,还有部分山东和美国材料;第Ⅲ类共有11份材料,主要包括山西,北京和甘肃品种;第Ⅳ类共17份材料,包含甘肃,山西,陕西和美国品种;第Ⅴ类共16份材料,主要有甘肃,山西和河北品种;第Ⅵ类共23份材料,主要是甘肃,山西,河北和美国材料;第Ⅶ类共9份材料,由甘肃,河北和山西品种组成.从分类情况来看,地域来源相同的小麦种质,其遗传基础较为相似,同时也具有较为接近的遗传系数,因此容易被分到同一类群中.而有些相同地域来源的小麦种质并没有聚为同一个类群,表现出该群体小麦种质遗传基础的多样性和复杂性.

图3 小麦品种遗传相似系数的分布Figure 3 Distribution of genetic similarity of wheat accession
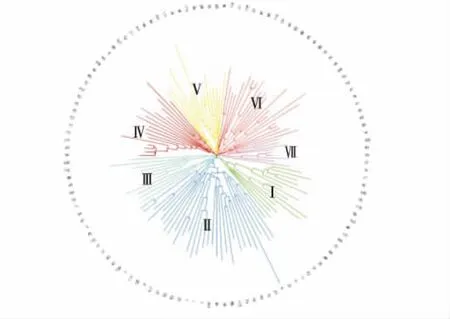
图4 基于SNP标记的小麦种质材料NJ 聚类图Figure 4 Neighbor-joining(NJ) tree for the wheat population based on SNP analysis data
3 讨论
利用分子标记技术分析小麦种质资源的遗传多样性,是阐明小麦遗传基础及改良小麦种质资源的有效途径[18].近年来,SSR等分子标记技术已经在国内外广泛的应用于小麦遗传多样性的研究[19-21],但是利用SNP标记技术分析小麦种质资源遗传多样性的研究还相对较少.本研究通过选用15 947个覆盖小麦全基因组的具有多态性的SNP标记,对124份小麦材料的遗传多样性和亲缘关系进行分析.通过对小麦各基因组、同源群以及染色体的遗传多样性进行研究发现,在小麦的3个基因组中,D基因组的遗传多样性显著低于A基因组和B基因组,在小麦的七个同源群中,第Ⅶ同源群的遗传多样性指数较低,并且在小麦21条染色体中,4D染色体的遗传多样性指数最低.相关研究表明,在B基因组上存在许多的与产量和品质相关的基因,因此B基因组获得了较大的遗传多样性[22-23],而D基因组由于在六倍体化的时候仅有部分Ae.Stragulata品系参与到小麦的多倍体化过程中,且在六倍体化时D基因组与A基因组和B基因组未形成四倍体,从而导致小麦D基因组的遗传多样性较低[23].在前人的研究中发现,山羊草属种的D基因组与小麦D基因组的遗传多样性具有很大的差异,且山羊草属种的D基因组与小麦D基因组完全同源,因此山羊草属种D基因组上的抗病,抗逆的优良基因可以通过简单的同源重组转入到小麦基因组中,丰富小麦的遗传多样性[24].
在聚类分析中发现,地域来源相同的小麦种质,其遗传基础较为相似,同时也具有较为接近的遗传系数,因此容易被分到同一类群中.而有些相同地域来源的小麦种质并没有聚为同一个类群,表现出该群体小麦种质遗传基础的多样性和复杂性.相同地理来源的小麦种质材料也未被完全分到一个类群中,说明地域的差别与聚类的结果没有必然的联系,引起这种地域性差异缩小的原因可能是在小麦长期的选择或育种过程中促进了种质材料间基因的交流[17,19].
遗传基础是小麦育种过程中的一个重要限制因子,种质材料的遗传基础狭窄是导致小麦育种工作难以取得突破性进展的关键因素[21,25-26].因此,对群体内的种质材料进行亲缘关系和遗传多样性分析,在指导小麦育种的工作方面具有重要意义[18,20,27].而现在的小麦育种通常过于关注产量性状而忽视其它优异性状,这导致了小麦遗传多样性的丧失,降低小麦品种对环境的适应性.因此在进行育种工作时通过引进优良的亲本种质材料,提高不同遗传背景种质材料之间的交流频度,拓宽小麦种质材料的遗传基础,进而突破小麦育种的瓶颈,从而培育出对环境适应性更强的小麦品种.
