CRISPR/Cas9技术定点编辑水稻谷蛋白基因GluA3
2019-02-26林冬枝董彦君
周 优,林冬枝,2,董彦君,2,3*
(1上海师范大学生命科学学院,上海 200234;2上海师范大学遗传研究所,上海 200234;3上海市植物分子科学重点实验室,上海 200234)
水稻是世界上最重要的粮食作物之一,为全球半数以上人口的主食,也是人体摄入植物蛋白的主要来源。稻米贮藏蛋白约占其干重的8%—16%,其含量及组分很大程度上决定着稻米的品质和营养价值。因此,提高稻米蛋白含量并改良其组分是水稻品质改良的重要目标。稻米中的蛋白根据其溶解特性可分为三大类,即盐溶蛋白(包括清蛋白和球蛋白)、醇溶蛋白和溶于酸或碱的谷蛋白,其中以谷蛋白为主,约占全蛋白的80%,而醇溶蛋白只占5%—10%[1-5]。与醇溶蛋白相比,谷蛋白具有易消化、富含赖氨酸、精氨酸等特点,是优良的植物蛋白[4]。Tanaka 等[6]从稻米蛋白中分离出两种蛋白体,即蛋白体I(protein body,PB-I)和蛋白体Ⅱ(PB-Ⅱ)。其中,以醇溶蛋白为主的PB-I由10 ku、13 ku、16 ku和57 ku 的多肽构成,以谷蛋白为主的PB-Ⅱ主要含有22 ku、26 ku、37 ku、38 ku和39 ku的多肽组分。科研工作者已鉴定出13 ku醇溶蛋白组分缺失的突变体[7-8]和低谷蛋白的突变体(如Lgc-1)[9]以及谷蛋白57 ku前体的积累缺少突变体[10]。研究表明,水稻谷蛋白基因原始翻译产物是一类氨基酸残基数目在 495—500 aa、大小约为 57 ku 的前体H。这些前体经过穿膜、剪切和酶解等一系列复杂过程最终形成成熟的22 ku和37—39 ku谷蛋白亚基并贮存于蛋白体中。目前,多数水稻谷蛋白基因已被分离克隆,根据序列相似性可将其分为GluA和GluB两个亚家族[11]。GluA亚家族至少包含3个不同编码基因,即GluA1、GluA2和GluA3[12]。近年来,水稻谷蛋白突变体的遗传变异机理和育种等方面的研究取得了明显进展[13]。在遗传机理方面,日本科学家[9]对利用低谷蛋白突变材料NM67育成的Lgc-1相关品种的低谷蛋白特性进行了深入研究,揭示了Lgc-1突变体的低谷蛋白特性是由于GluB亚家族基因GluB4和被剪切了部分片段的GluB5基因以尾对尾反向重复形成的结构造成的。即在GluB4和GluB5之间缺失一段4.5 kb的片段,由于GluB4和GluB5的同源性极高,在 RNA拼接过程中产生了发夹式的RNA双链结构,导致了Lgc-1突变体的低谷蛋白性状。对于某些特定的人群如肾脏病、糖尿病患者来说,大量吸收消化谷蛋白会导致病情恶化。因此,育种家们利用低谷蛋白Lgc-1突变体材料选育出低谷蛋白水稻品种‘春阳’(http://ineweb.narcc.affrc.go.jp/search/ine.cgi?action=inedata_top&ineCode=HOK01830)和‘W3660’[14],又利用Lgc-1低谷蛋白相关品种与球蛋白缺失突变体配组育成了具有更低谷蛋白含量的水稻品种‘LGC-Katsu’和 ‘LGC-Jun’[15]。本研究组也以水稻品种‘春阳’作为亲本育成了能辅助治疗肾病的低谷蛋白品种‘益肾稻1号’(http://www.jyb.cn/high/gdjyxw/201311/t20131104_558 198.html)。因此,水稻谷蛋白突变体不仅是进行真核生物贮藏蛋白生物合成和基因表达调控研究的重要材料,也是培育功能性专用水稻品种不可或缺的优良种质资源[13,16]。众所周知,CRISPR/Cas9是一种快速有效的基因组定点编辑技术,已成为植物分子育种的重要手段。目前,未见利用CRISPR/Cas9技术创制水稻谷蛋白基因突变体的报道。本研究尝试利用CRISPR/Cas9技术对水稻谷蛋白基因GluA3(LOC_Os03g31360)进行定点编辑,以期为改善水稻谷蛋白品质的育种提供中间材料和理论参考。
1 材料与方法
1.1 试验材料
本试验的遗传转化材料为浙江省嘉兴市农业科学院选育的优质高产粳稻品种‘嘉花1号’,在南方粳稻区广泛种植。试验水稻材料种植于人工气候箱以及上海师范大学植物园内的封闭试验基地,常规水肥管理。
1.2 载体构建及转基因研究
本试验使用的CRISPR/Cas9 载体由华南农业大学刘耀光院士[17]提供,U6a载体部分序列如图1A所示,Cas9 载体结构如图1B所示。利用CRISPR引物设计软件 (http://crispr.dbcls.jp/)针对水稻谷蛋白基因GluA3(LOC_Os03g31360)第4外显子设计19 bp靶位点序列5’-TCAGGAAGTGCGCGGAAGA-3’,PAM序列为 TGG(图 1C),设计的靶点接头引物为Target-F/R(表1)。基因靶序列利用NCBI 网站上的BLAST程序进行筛选,未找到脱靶序列。
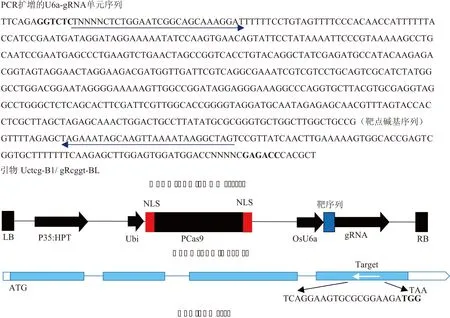
LB:载体左边界;HPT:潮霉素;Ubi:Ubi启动子;pCas9:Cas9蛋白;OsU6a:水稻U6a启动子;gRNA:向导RNA;RB:载体右边界图1 CRISPR/Cas9载体及基因结构图Fig.1 CRISPR/Cas9 vector and gene structure map

引物名称引物序列(5’-3’)Target-FGCCGTCAGGAAGTGCGCG-GAAGATarget-RAAACTCTTCCGCGCACTTCCT-GAGluA3-FAGAACGCACTCCTTTCACCTGluA3-RTGCTTGTTGATGGCATTTCT-TAGluA3-Q-FCCGTCGTTTGCTTGTTCCTCGluA3-Q-RACAGTGCGAATCGGCTCAAAOsActin-FAGGAAGGCTGGAAGAGGACCOsActin-RCGGGAAATTGTGAGGGACATHPT-FGATGTTGGCGACCTCGTATTHPT-RGTGTCACGTTGCAAGACCTG
1.3 PCR鉴定分析及 qRT-PCR 分析
取试验材料单株叶片,采用CTAB法[18]提取全基因组DNA,用于PCR扩增或于-20℃冰箱中保存。PCR体系参照张建辉等[19]方法,PCR扩增产物在1.5%琼脂糖凝胶上进行电泳,电泳结束后EB溶液显色并拍照,进行DNA测序(上海美吉生物公司)。
利用RNA提取试剂盒(北京全式金生物技术有限公司的TransZol Plant)提取水稻根、茎、幼叶、倒二叶、剑叶、穗以及授精15 d后的未成熟胚乳组织中总RNA。以提取的RNA为模板,用反转录试剂盒(北京全式金生物技术有限公司的TransScript One-Step gDNA Removal and cDNA Synthesis SuperMix)合成cDNA,然后进行定量PCR(qRT-PCR)分析,以水稻OsActin基因作为内参基因。qRT-PCR体系为:cDNA 模板1 μL,2×SYBR qPCR Mix (TOYOBO)10 μL,正反向引物(10 μmol/L)各1 μL,ddH2O补足至20 μL。PCR扩增程序为:95℃预变性3 min;95℃ 10 s,60℃ 30 s,72℃ 20 s,40个循环。采用2-ΔCT法分析数据,确定目的基因的相对表达量。
1.4 突变体农艺性状调查
水稻生长成熟后,对‘嘉花1号’及突变体材料的株高、有效穗、结实率、千粒重、每穗粒数和单株产量进行考查。考察6个单株,获得的数据采用Excel 2010进行统计分析。
1.5 种子谷蛋白的提取及SDS-PAGE电泳分析
参照曲乐庆等[4]方法提取‘嘉花1号’、突变体以及Lgc-1材料的成熟种子谷蛋白,略加改动。具体步骤:将单粒种子用研钵磨碎,加入1 mL 0.5 mol/L NaCl和10 mmol/L Tris-HCl(pH 6.8),旋涡震荡混匀,15 000 r/min 离心 10 min,弃上清,将水溶性清蛋白和盐溶性球蛋白去掉;然后向沉淀中加入1 mL 75%乙醇,旋涡震荡混匀,15 000 r/min离心10 min,弃上清;再向沉淀中加入0.5 mL 65℃预热的蛋白抽提液5 mol/L Urea、4%(质量分数,下同) SDS、5% β-ME、20%甘油和0.1 mol/L Tris-HCl(pH 6.8),充分旋涡震荡后室温静置过夜。电泳上样前离心10 min (10 000 r/min),取10 μL上清进行SDS-PAGE (分离胶15%,浓缩胶7.5%),恒压 90 V,垂直电泳270 min左右。电泳后用考马斯亮蓝G-250染色。
2 结果与分析
2.1 GluA3基因特异性表达验证
通过对‘嘉花1号’编码谷蛋白的基因GluA3(LOC_Os03g31360)进行测序分析,发现该基因开放阅读框(ORF)序列与‘日本晴’相同,属于典型的粳稻材料。为了验证GluA3基因是否具有胚乳特异性表达,对其植株根、茎、幼叶、倒二叶、剑叶、穗以及授精15d后的未成熟胚乳组织进行转录表达分析,结果只在授精15d后的未成熟胚乳中检测到该基因mRNA转录本,并有高表达(图2A),这与日本晴基因表达数据库(http://ricexpro.dna.affrc.go.jp/GGEP/graph-view.php?featurenum=14894)结果完全一致(图2B),证实GluA3基因参与水稻种子中谷蛋白的合成。
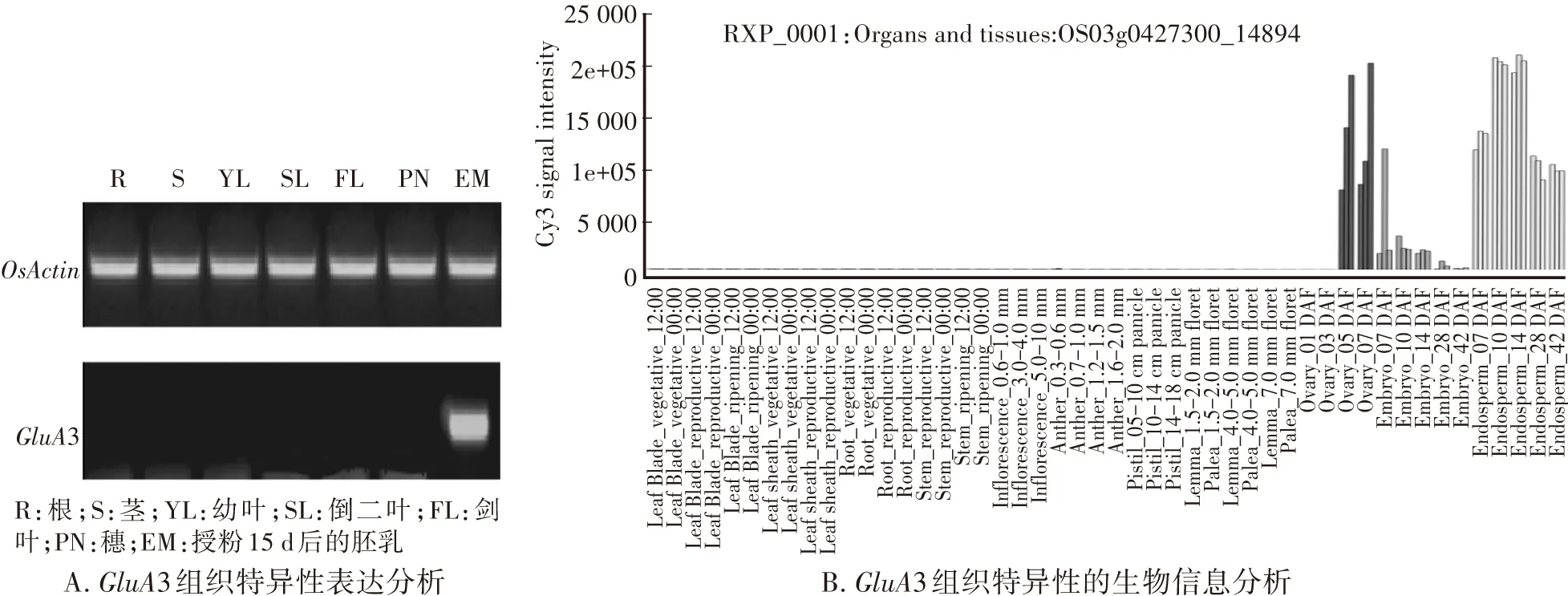
图2 GluA3基因表达的组织特异性Fig.2 The tissue specific expression of GluA3 gene
2.2 CRISPR/Cas9表达载体的构建与转基因植株获得
首先通过网址http://rice.plantbiology.msu.edu/cgi-bin/gbrowse/rice/#search获得水稻谷蛋白基因GluA3(LOC_Os03g31360)全长cDNA 序列,结合生物信息学网站http://crispr.dbcls.jp,通过序列比对分析,在第4外显子上找到一条长度为 19 bp 特异性较好的靶点序列(5’-TCAGGAAGTGCGCGGAAGA-3’)(图1C),将其构建到表达载体中(图1B),将连接了MH载体的终产物转入大肠杆菌,在含有卡那霉素的LB固体平板培养基上37℃过夜培养,挑取单菌落并在试管内摇菌过夜。提取质粒,用引物Uctcg-B1 (5’-TTCAGAGGTCTCTCTCGACTAGTGGAATCGGCAGCAAAGG-3’)和gRcggt-BL (5’-AGCGTGGGTCT-CGACCGACGCGTCCATCCACTCCAAGCT-3’)进行PCR扩增,扩增产物进行测序验证(上海美吉生物公司),将序列结果正确的质粒转化农杆菌EHA 105,用于侵染水稻‘嘉花1号’的愈伤组织,获得组培植株后,利用载体特异性引物HPT-F/R(表1)扩增检测,获得阳性转基因植株23株。
2.3 GluA3基因靶位点突变类型分析
为了检测23株阳性转基因植株是否成功编辑,利用GluA3基因序列特异性引物GluA3-F/R(表1)对上述阳性转基因植株进行扩增测序,发现20个T0代独立转基因系在PAM附近位置发生了编辑。进一步将其中的一个T1代植株移栽到试验田,对GluA3基因测序发现,该基因靶位点突变位点发生分离,M1是在PAM (TGG)第1个碱基(T)至PAM前3个碱基(AGA)共缺失4bp(AGAT)(图3A),M2则是在PAM位置前后共缺失45bp (5’-GAAGATGGAATTCTTTCCTGCCATGTGGCTAACCATGGAGTCAGG-3’),并插入59bp (5’-TTGTGATCATGTTGACATTGAAAACCAACCCTGACTCCATGGTTAGCCACATATGAAGA-3’)(图3A)。两种突变均引起编码的氨基酸发生改变,且翻译提前终止,影响GluA3蛋白的结构和功能。进一步对T1代28个单株进行DNA提取,用基因序列特异性引物进行PCR扩增和测序分析,通过与‘嘉花1号’(WT)序列比对,发现此突变株为嵌合体,分离出两种纯合突变种(图3A);利用特异性引物HPT-F/R进行扩增,发现转基因载体和基因靶位点在后代中发生了分离(图3B);获得T1代无选择标记基因突变株。
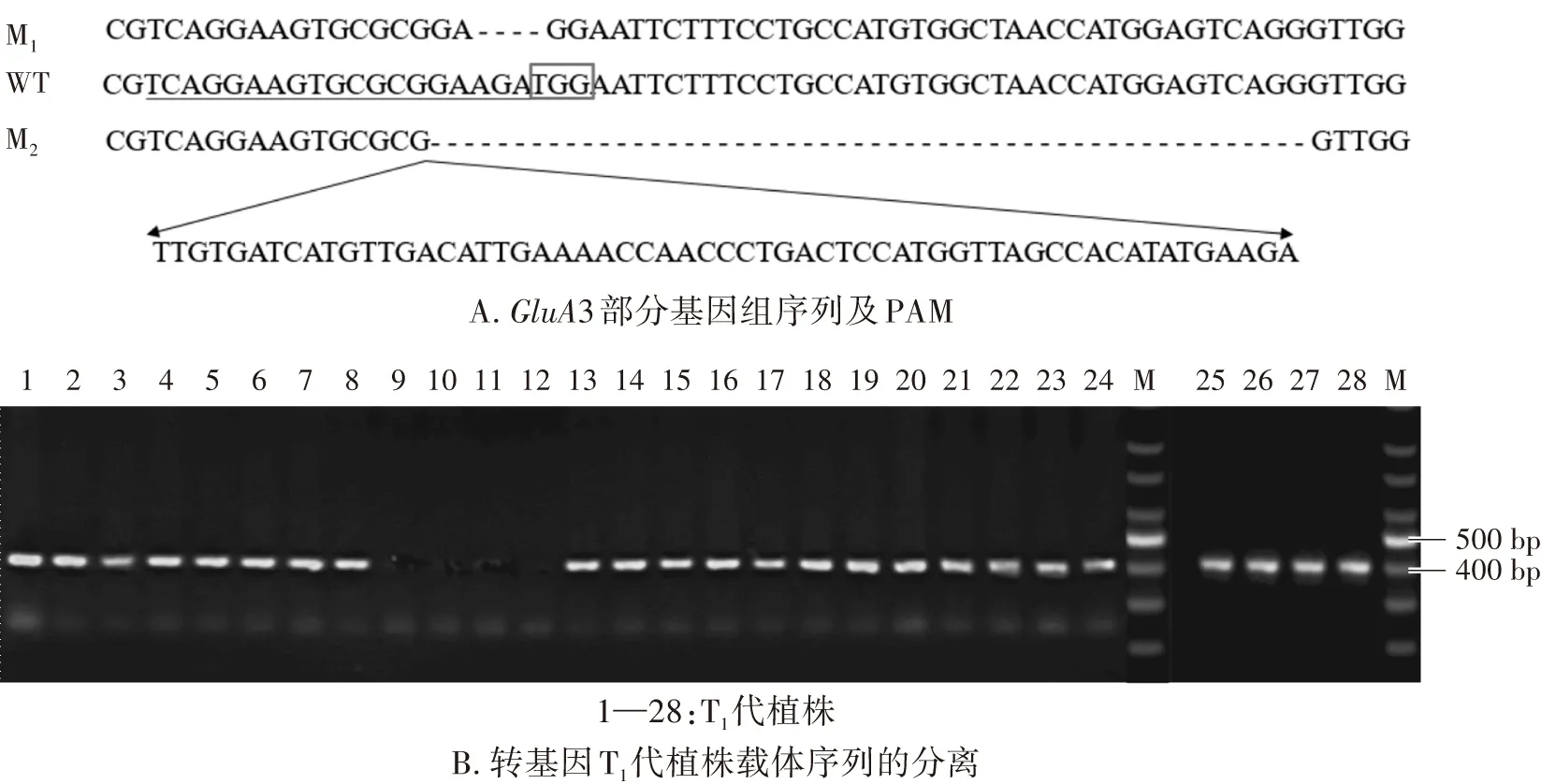
图3 CRISPR/Cas9/sgRNA介导的GluA3编辑Fig.3 CRISPR/Cas9/sgRNA-mediated editing of GluA3
2.4 突变体RNA表达水平以及稻米谷蛋白含量
上述2种类型不包含转基因载体序列的纯合型突变水稻(M1和M2)与‘嘉花1号’表现出相似的植株表型(图4A)。为了明确基因编辑是否影响GluA3基因的转录表达,利用qRT-PCR技术对授精15d后的‘嘉花1号’和突变体未成熟胚乳中GluA3 mRNA表达水平进行比较分析,发现2种突变材料中的GluA3 表达水平均明显下调,大片段缺失/插入的M2更加明显(图4B),表明GluA3基因第 4外显子上4个碱基缺失(M1)和大片段缺失/插入(M2)都影响了该基因的转录水平。同时,为了明确GluA3基因的突变及转录下调是否影响谷蛋白含量以及组分,采用 SDS-PAGE 对‘嘉花1号’、M1和M2水稻糙米种子谷蛋白含量及组分进行了分析。为了提高试验的可靠性,以低谷蛋白突变体Lgc-1作为对照,结果表明:Lgc-1与‘嘉花1号’相比,22—23 ku和37—39 ku的多肽组分明显减少[9],说明本测试方法和结果是可靠的。如图4C所示,M1和M2谷蛋白含量与‘嘉花1号’相比,表现出明显下降趋势,尤其是M2,表明GluA3 突变可引起谷蛋白含量变化。
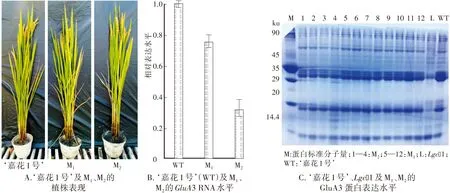
图4 ‘嘉花1号’和GluA3基因编辑后代表型、RNA及其谷蛋白表达的检测Fig.4 Phenotype,expression detection of RNA and gluten of ‘Jiahua 1’,M1 and M2
2.5 GluA3基因编辑后代农艺性状
为了验证GluA3突变是否与水稻产量农艺性状相关,对‘嘉花1号’及其T2代突变体材料的产量相关农艺性状进行了考察。通过生物统计分析,发现除株高、穗长以及千粒重没有明显差异外,穗数、每穗粒数、结实率以及单株产量均存在显著差异(表2),突变体材料分别比‘嘉花1号’下降37%、13%、18%和60%以上。

表2 ‘嘉花1号’和突变体水稻农艺性状的表现
3 讨论
CRISPR/Cas9技术是继RNAi技术、锌指核酸酶和TALEN核酸酶之后最新发展起来的一种可精确定点编辑基因组DNA的新技术,具有设计构建简单、快速等优点。该技术首先在动物中广泛应用,随后科研人员将其应用于植物基因编辑[20]。目前该技术也已成功应用于水稻分子育种[21]。如沈兰等[22]敲除中国东北粳稻品种控制粒型大小的GS3基因和每穗粒数的Gn1a基因,发现这2个基因在不同遗传背景下对产量的影响是多样的;邵高能等[20]对水稻第8染色体上编码甜菜碱醛脱氢酶的基因Badh2进行了编辑,获得香稻材料;周文甲等[23]同时对水稻抽穗期基因Hd2和香味基因Badh2进行编辑,获得早熟香味水稻品种。本研究也成功地应用CRISPR/Cas9 基因编辑技术对水稻种子谷蛋白GluA3基因进行了编辑,获得了2种类型谷蛋白GluA3基因的突变体。
通过CRISPR/Cas9技术定点编辑水稻中谷蛋白基因改变谷蛋白含量及其组分,可以改善水稻蛋白品质。如提高稻米谷蛋白含量能提升其营养价值,低谷蛋白品种如‘阳春’、Lgc-1[9]和‘益肾稻1号’能用于肾病患者的食疗辅助。本研究在水稻谷蛋白GluA3基因第4外显子区域设计序列特异性靶位点进行转基因特异性敲除,成功获得了GluA3基因的2种突变体材料,这2种突变体材料的GluA3 RNA表达水平均降低,蛋白翻译提前终止,M1突变体的GluA3 蛋白分子量由原来56.03 ku 变为51.13 ku,而M2突变体变为51.94 ku,导致突变体中GluA3蛋白功能改变,且稻米中谷蛋白含量表现出下降趋势。目前研究表明,稻米中谷蛋白是由多基因控制,通过对水稻全基因组数据库(http://rice.plantbiology.msu.edu/cgi-bin/gbrowse/rice)搜索发现,至少有16个谷蛋白基因(LOC_Os01g55630等)[24]。由此,今后可以进一步利用CRISPR/Cas9技术创制一系列适应市场需求的不同类型谷蛋白含量/组分的稻米新品种。另外,本研究得到2种GluA3突变体(M1、M2),除了株高、穗长以及千粒重变化不大,其余调查农艺性状(穗数、每穗粒数、结实率以及单株产量)均受到严重负面影响,这可能是GluA3基因一因多效的负作用,也可能是试验脱靶或组织培养过程中其他基因突变所致,还需进一步探究。本研究后续将对突变体材料与野生型材料进行回交,进一步研究GluA3基因与穗数、每穗粒数、结实率以及产量的关系。
