东乡野生稻与日本晴多态性标记的开发
2019-01-17马小定唐江红张佳妮黎毛毛韩龙植
马小定 唐江红 张佳妮 崔 迪 李 慧 黎毛毛,* 韩龙植,*
东乡野生稻与日本晴多态性标记的开发
马小定1唐江红2张佳妮2崔 迪1李 慧3黎毛毛3,*韩龙植1,*
1中国农业科学院作物科学研究所 / 农作物基因资源与基因改良国家重大科学工程, 北京 100081;2重庆师范大学, 重庆 401331;3江西省农业科学院水稻研究所, 江西南昌 330200
东乡野生稻是分布于全球最北端的一种普通野生稻, 与亚洲栽培稻基因组差异较大, 目前缺少覆盖其全基因组的分子标记。本文以东乡野生稻和日本晴为材料, 通过筛选已有的1017个标记, 并利用东乡野生稻基因组重测序信息设计的217个InDel标记, 共检测出203个标记在东乡野生稻与日本晴间呈现多态性。这些标记均匀分布于12条染色体, 平均间隔1.9 Mb, 基本覆盖东乡野生稻全基因组区域。通过对籼粳亚种的检验分析, 发现该套多态性分子标记在东乡野生稻与粳稻杂交后代群体基因型分析上具有较高的应用价值。本研究结果为发掘东乡野生稻的有利基因以及分子标记辅助选择育种提供了有力的工具。
InDel标记; 基因组重测序; 粳稻
东乡野生稻于1978年在我国江西省东乡县东源乡被发现, 当地称之为“野禾”, 后经水稻和植物学相关专家鉴定, 确定为普通野生稻[1-2], 而且是迄今为止发现的分布于全球最北端(28°14¢N)的普通野生稻。东乡野生稻具有丰富的遗传多样性, 而且携带耐寒、耐旱、耐贫瘠、广亲和性、野败育性恢复性、胞质雄性不育、抗病虫和高产等相关基因, 对水稻基础研究和产业发展有着举足轻重的作用, 被誉为“野生植物类的大熊猫”[3]。
分子标记是继形态标记、细胞标记和生化标记后发展起来的一种简单易用的遗传标记。现在水稻研究中被广泛使用的分子标记有简单序列重复(Simple Sequence Repeat, SSR)标记、插入/缺失(Insertion-Deletion, InDel)标记和单核苷酸多态性(Single Nucleotide Polymorphism, SNP)标记。SSR标记是根据广泛存在于基因组序列中的、由1~6个核苷酸为重复单位组成的串联重复序列而设计的分子标记。由于其分布广泛、多态性丰富, 在不同作物功能基因组研究中发挥了重要的作用。InDel标记是指根据相同位点序列由不同数目的核苷酸插入或缺失形成的差异而设计的多态性标记[4]。InDel标记具有基因组差异位点丰富(例如, 日本晴和93-11基因组序列中平均每953 bp就存在1个InDel[5])、染色体位置明确、条带清晰、结果可靠等优点[6-7]。SNP标记是在基因组水平上单个核苷酸的变异引起的DNA序列多态性, 形式包括单碱基的缺失、插入、转换及颠换等。SNP标记遗传稳定性高, 位点丰富且分布广泛, 检测快速并且容易实现自动化分析[8-10]。但是目前由于检测方法及成本的限制, 还未被广泛采用。除了上述分子标记外, 近年来由于测序技术的发展和成本的降低, 一些研究机构开始使用全基因组测序技术进行起源进化和功能基因组学的相关研究。全基因组重测序是对已知基因组序列的物种进行不同个体的基因测序, 并在此基础上对个体或者群体进行差异性分析。通过全基因组重测序分析, 可以找到大量的SNP、InDel、结构变异(Structure Variation, SV)和拷贝数变异(Copy Number Variation, CNV)等位点[11]。
前期, 虽然已有一些研究者利用东乡野生稻为供体构建了染色体片段置换系, 但是使用的主要是SSR标记, 而且基因组覆盖不完全[12]。主要是由于东乡野生稻和亚洲栽培稻基因组存在较大的差异, 多态性分子标记数量和质量不佳。本研究以东乡野生稻和日本晴为材料, 通过筛选现有分子标记, 以及利用东乡野生稻基因组重测序数据设计的InDel标记, 形成一套覆盖全基因组的、条带清晰、结果可靠的分子标记, 为东乡野生稻的基础研究和育种利用提供有力的遗传工具。
1 材料与方法
1.1 研究材料
东乡野生稻取自江西省农业科学院水稻研究所保存的东乡野生稻庵家山居群中的C35单株, 日本晴来源于中国农业科学院作物科学研究所国家种质库。按照常规大田种植方法进行东乡野生稻的种植和栽培稻日本晴育苗移栽。
1.2 基因组重测序
分别从3个东乡野生稻植株上取幼嫩的叶片, 然后立即在液氮中冷冻并储存在–80℃冰箱备用。用DNeasy plant mini kit (QIAGEN)试剂盒提取基因组总DNA, 经NanoDrop、Qubit和琼脂糖凝胶检测DNA提取的质量和浓度。5 μg DNA用于北京诺禾致源科技股份有限公司(北京)基因组重测序(Illumina HiSeq2500)。测序下机的原始数据经过滤, 去除接头序列和低质量测序数据获得有效数据。有效测序数据通过BWA软件[13]比对到参考基因组(ftp://ftp.ensemblgenomes.org/pub/plants/release-28/fasta/ oryza_sativa/dna/), SAMTOOLs软件[14]检测长度小于50 bp的小片段InDel, ANNOVAR软件[15]对检测出的InDel注释。
1.3 InDel标记的设计
根据东乡野生稻基因组重测序结果, 对比日本晴基因组序列信息(IRGSP-1.0.28版), 以每隔1.5~3.0 Mb的距离在各染色体上查找对应的插入缺失位点, 在位点前后各选择120 bp序列, 利用Primer Premier 5.0设计引物。引物长度一般在19~30 bp之间,m值控制在(60±1)℃, 产物设计长度110~180 bp。
1.4 DNA提取、PCR扩增及产物检测
分别从东乡野生稻和日本晴植株上取新生的嫩叶, 采用常规CTAB法提取基因组DNA[16]。
PCR体系10 μL包含模板DNA 0.5 μL、10×PCR buffer 1.0 μL、10 mmol L–1dNTPs 0.3 μL、10 μmol L–1正反引物各0.5 μL、DNA聚合酶0.5 U, 用ddH2O补齐至10 μL。PCR程序为94℃ 预变性5 min; 94℃ 30 s, 59℃ 30 s, 72℃ 30 s, 35个循环; 72℃延伸5 min; 16℃保存。
PCR产物加2 μL 6×Loading buffer, 混匀后取3.5 μL于8%聚丙烯酰胺凝胶电泳槽中, 180 V稳压电压10 min后调电压至250 V, 再电泳大约70 min, 电泳结束后用银染法染胶显色[17]。
2 结果与分析
2.1 多态性分子标记开发
针对东乡野生稻、日本晴和东乡野生稻/日本晴杂合体(将两者DNA等量混合, 模拟杂合体基因组DNA)样本, 利用实验室现有的661对SSR标记引物和356对InDel标记引物进行多态性检测, 获得有多态性的标记81 个, 包括42个SSR标记和39个InDel标记, 多态率为8%。
为了获得更多的多态性标记, 对东乡野生稻进行了基因组重测序, 通过与对照基因组比对, 分别在染色体基因上游、外显子、内含子和基因下游区共检测到373,501个InDel。根据此结果, 设计了217对InDel标记引物, 经过检测, 其中122个标记显示出多态性, 多态率为56% (图1)。综合以前和本次设计的引物, 共获得203个东乡野生稻与日本晴间有多态的分子标记。每条染色体上分子标记数目为9~30个, 平均间隔为1.2~2.6 Mb (表1和附表1)。

图1 多态性分子标记筛选
1: 东乡野生稻; 2: 日本晴; 3: 东乡野生稻/日本晴杂合体; M: 标准分子量。
1: Dongxiang wild rice; 2: Nipponbare; 3: artificial F1hybrid between Dongxiang wild rice and Nipponbare; M: DNA size marker.

表1 各染色体多态性标记分布
为了方便研究者直观地查看标记在染色体上的分布, 利用MapDraw (2.1)软件, 以标记的物理位置为参数制作了203个多态性标记在染色体上的分布图谱(图2)。由图谱可知, 标记在染色体上的分布比较均匀, 基本完全覆盖了12条水稻染色体。
2.2 多态性分子标记的应用
为检验所开发的多态性分子标记在其他亚洲栽培稻中的应用价值,随机选取了59个多态性分子标记, 以5个籼稻和5个粳稻品种为样本, 以东乡野生稻和日本晴为对照检测(图 3)发现, 在粳稻品种中有84.7%的基因型与日本晴一致, 11.6%的基因型与东乡野生稻一致; 在籼稻品种中有73.9%的基因型与东乡野生稻一致, 13.5%的基因型与日本晴一致。而在检测到的异于对照基因型数量中, 籼稻(10.9%)要远远高于粳稻品种(1.7%)(表2和附表2)。因此, 如果利用本套多态性分子标记进行基因型分析, 其在分析东乡野生稻/粳稻杂交后代群体基因型上的应用价值高于分析东乡野生稻/籼稻杂交后代群体。
3 讨论
分子标记是现代遗传学和分子生物学研究最常用的技术之一, 随着技术的发展, 分子标记的种类也在不断发展, 例如SNP、CNV标记等。东乡野生稻做为一种普通野生稻, 与亚洲栽培稻基因组具有较大的差异。直接利用常规的SSR标记以及基于籼稻与粳稻设计的InDel标记来研究东乡野生稻, 或者开展分子标记辅助选择育种具有一定的局限性。例如, 本研究直接筛选常用的SSR和InDel标记, 仅仅只有8%的引物存在多态性。因此, 开发一套覆盖东乡野生稻全基因组的, 并且与亚洲栽培稻间存在多态性的分子标记至关重要。本研究在筛选现有分子标记的基础上, 利用东乡野生稻基因组重测序信息设计的InDel标记, 共获得了203个多态性分子标记, 均匀分布于12条染色体上, 标记平均间隔1.9 Mb, 可以满足基因初定位和标记辅助选择育种的需要。
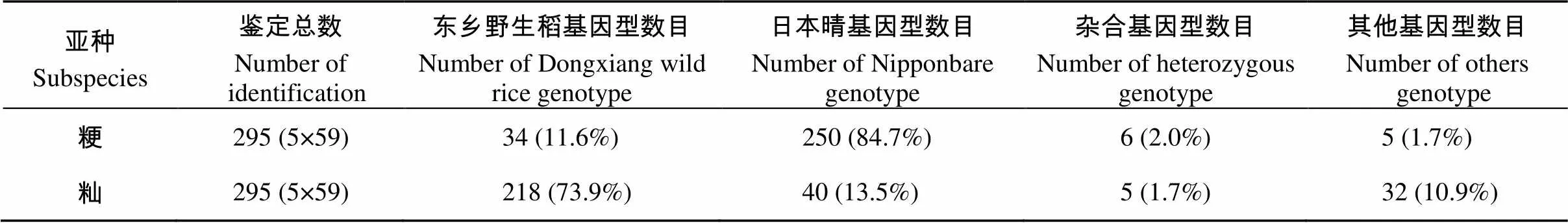
表2 多态性标记籼梗亚种基因型鉴定一致性分析
该分析选取了不同省份的5个粳稻(辽盐241、松粳8、吉粳88、淮稻9号、合系6号)和5个籼稻(黄花占、白玉丝苗、扬稻6号、湘晚籼12号、鄂中5号)品种, 利用59个多态性标记进行检测分析。括号中的数字代表本基因型占总检测基因型数目的比例。
Fivecultivars (Liaoyan 241, Songgeng 8, Jigeng 88, Huaidao 9, and Hexi 6) and fivecultivars (Huanghuazhan, Baiyusimiao, Yangdao 6, Xiangwanxian 12, Ezhong 5) were selected for genotype identification by 59 polymorphic molecular markers. The number in parenthesis represents the proportion of current genotype to total detected genotypes.
1: 东乡野生稻; 2: 日本晴; 3: 辽盐241; 4: 松粳8; 5: 吉粳88; 6: 淮稻9号; 7: 合系6号; 8: 黄花占; 9: 白玉丝苗; 10: 扬稻6号; 11: 湘晚籼12号; 12: 鄂中5号。
1: Dongxiang wild rice; 2: Nipponbare; 3: Liaoyan 241; 4: Songgeng 8; 5: Jigeng 88; 6: Huaidao 9; 7: Hexi 6; 8: Huanghuazhan; 9: Baiyusimiao; 10: Yangdao 6; 11: Xiangwanxian 12; 12: Ezhong 5.
分子标记的开发基本上都是基于2个或者多个特定样本, 因此获得的分子标记的应用范围具有一定的局限性。本研究获得的203个多态性分子标记是基于东乡野生稻和日本晴开发的, 通过对5个籼稻和5个粳稻品种的检验, 发现其在粳稻品种中检测的基因型84.7%与日本晴一致, 而在籼稻品种中的基因型73.9%与东乡野生稻一致。如果利用该套分子标记检测东乡野生稻与籼稻杂交的后代, 可能会有大约74%的分子标记不能表现出多态性; 而在检测东乡野生稻与粳稻杂交后代中, 大约会有85%的标记表现出多态性。因此, 该套分子标记在分析东乡野生稻与粳稻杂交后代群体的基因型上具有较大的应用价值。
附表 请见网络版: 1) 本刊网站http://zwxb.chinacrops. org/; 2) 中国知网http://www.cnki.net/; 3) 万方数据http:// c.wanfangdata.com.cn/Periodical-zuowxb.aspx。
[1] 邬柏梁, 何国成, 白国章, 吴蔼如. 我省东乡县一带发现野生稻. 江西农业科技, 1979, (2): 6–7. Wu B L, He G C, Bai G Z, Wu A R. Wild rice found in Dongxiang County in our province., 1979, (2): 6–7 (in Chinese with English abstract).
[2] 潘熙淦, 饶宪章. 东乡野生稻观察及特征鉴定报告. 江西农业科技, 1982, (7): 5–9. Pan X G, Rao X Z. Report of Dongxiang wild rice observation and characterization., 1982, (7): 5–9 (in Chinese with English abstract).
[3] 黄依南, 黄国勤. 东乡野生稻的发现、价值与保护. 农业资源与环境学报, 2012, 29: 13–15. Huang Y N, Huang G Q. The discovery, value and protection of Dongxiang wild rice., 2012, 29: 13–15 (in Chinese with English abstract).
[4] 刘丹, 孙玉友, 魏才强, 解忠, 李洪亮, 张巍巍, 程杜娟, 孙国宏, 徐德海. InDel分子标记及其在水稻研究中的应用. 种子, 2017, (9): 47–52. Liu D, Sun Y Y, Wei C Q, Xie Z, Li H L, Zhang W W, Cheng D J, Sun G H, Xu D H. InDel molecular marker and its application in rice () research., 2017, (9): 47–52 (in Chinese with English abstract).
[5] Shen Y J, Jiang H, Jin J P, Zhang Z B, Xi B, He Y Y, Wang G, Qian L L, Li X, Yu Q B, Liu H J, Chen D H, Gao J H, Huang H, Shi T L, Yang Z N. Development of genome-wide DNA polymorphism database for map-based cloning of rice genes., 2004, 135: 1198–1205.
[6] 冯芳君, 罗利军, 李荧, 周立国, 徐小艳, 吴金, 陈宏伟, 陈亮, 梅捍卫. 水稻InDel和SSR标记多态性的比较分析. 分子植物育种, 2005, 3: 725–730. Feng F J, Luo L J, Li Y, Zhou L G, Xu X Y, Wu J, Chen H W, Chen L, Mei H W. Comparative analysis of polymorphism of InDel and SSR markers in rice., 2005, 3: 725–730 (in Chinese with English abstract).
[7] 初志战, 郭海滨, 曾栋昌, 刘耀光. 籼粳稻基因组295个InDel 标记的开发. 作物学报, 2016, 42: 932–941. Chu Z Z, Guo H B, Zeng D C, Liu Y G. Development of 295 InDel markers forandrice.2016, 42: 932–941 (in Chinese with English abstract).
[8] Taillon-Miller P, Gu Z J, Li Q, Hillier L, Kwok P Y. Overlapping genomic sequences: a treasure trove of single nucleotide polymorphisms., 1998, 8: 748–754.
[9] 唐立群, 肖层林, 王伟平. SNP分子标记的研究及其应用进展. 中国农学通报, 2012, 28(12): 154–158. Tang L Q, Xiao C L, Wang W P. Research and application progress of SNP markers., 2012, 28(12): 154–158 (in Chinese with English abstract).
[10] Yu H H, Xie W B, Wang J, Xing Y Z, Xu C G, Li X H, Xiao J H, Zhang Q F. Gains in QTL detection using an ultra-high density SNP map based on population sequencing relative to traditional RFLP/SSR markers., 2011, 6: e17595.
[11] Abyzov A, Urban A E, Snyder M, Gerstein M. CNVnator: an approach to discover, genotype, and characterize typical and atypical CNVs from family and population genome sequencing., 2011, 21: 974–984.
[12] 李德军. 江西东乡普通野生稻渗入系的构建及高产QTL定位. 中国农业大学博士学位论文, 北京, 2003. Li D J. Development of Introgression Lines of Common Wild Rice (Griff) from Dongxiang in Jiangxi Province and Mapping QTLs for Improving Yield. PhD Dissertationof China Agricultural University, Beijing, China, 2003 (in Chinese with English abstract).
[13] Li H, Durbin R. Fast and accurate short read alignment with Burrows-Wheeler transform., 2009, 25: 1754–1760.
[14] Li H, Handsaker B, Wysoker A, Fennell T, Ruan J, Homer N, Marth G, Abecasis G, Durbin R. The Sequence Alignment/ Map format and SAMtools.,2009, 25: 2078–2079.
[15] Wang K, Li M, Hakonarson H. ANNOVAR: functional annotation of genetic variants from high-throughput sequencing data., 2010, 38: e164.
[16] Murray M G, Thompson W F. Rapid isolation of high molecular weight plant DNA., 1980, 8: 4321–4325.
[17] Bassam B J, Caetano-Anollés G, Gresshoff P M. Fast and sensitive silver staining of DNA in polyacrylamide gels., 1991, 196: 80–83.
Development of molecular markers polymorphic between Dongxiang wild rice andrice cultivar ‘Nipponbare’
MA Xiao-Ding1, TANG Jiang-Hong2, ZHANG Jia-Ni2, CUI Di1, LI Hui3, LI Mao-Mao3,*, and HAN Long-Zhi1,*
1National Key Facility for Crop Gene Resources and Genetic Improvement / Institute of Crop Sciences, Chinese Academy of Agricultural Sciences, Beijing, 100081, China;2Chongqing Normal University, Chongqing 401331, China;3Rice Research Institute, Jiangxi Academy of Agricultural Sciences, Nanchang 330200, Jiangxi, China
Dongxiang wild rice is a type of common wild rice () that has the northernmost natural distribution of any wild rice species. The genome of Dongxiang wild rice differs from that of modern cultivated rice varieties (). At present, a set of molecular markers that covers the entire genome of Dongxiang wild rice is lacking. In this study, we used Dongxiang wild rice and therice variety “Nipponbare” as research materials. By screening the existing collection of 1017 SSR and InDel markers and the 217 InDel markers which were designed using Dongxiang wild rice genome resequencing information, we obtained a set of 203 markers polymorphic between Dongxiang wild rice and ‘Nipponbare’, which were relatively uniformly distributed on the 12 rice chromosomes, and basically covered the entire genome, with an average inter-locus distance of 1.9 Mb. Through the genotyping of fiveand fivevarieties, we concluded that those 203 polymorphic molecular markers have a high application value in genotype analysis of Dongxiang wild rice andrice offspring population. These results provide a powerful tool for exploring beneficial genes from Dongxiang wild rice as well as marker-assisted breeding and selection.
InDel marker; genome re-sequencing;rice
2018-07-09;
2018-10-08;
2018-11-06.
10.3724/SP.J.1006.2019.82037
韩龙植, E-mail: hanlongzhi@caas.cn; 黎毛毛, E-mail: lmm3056@163.com
E-mail: maxiaoding@caas.cn
本研究由国家重点研发计划项目(2016YFD0100101, 2016YFD0100301), 国家自然科学基金项目(31501287, 31671664), 国家科技支撑计划项目(2015BAD01B01-1), 中国农业科学院科技创新工程项目, 国家农作物种质资源保护项目(2017NWB036-01, 2017NWB036-12-2), 国家农作物种质资源平台(NICGR2017-001)资助。
This study was supported by the National Key Research and Development Program of China (2016YFD0100101, 2016YFD0100301), the National Natural Science Foundation of China (31501287, 31671664), the National Key Technology Support Program of China (2015BAD01B01-1), the Agricultural Science and Technology Innovation Program of CAAS, the National Crop Germplasm Conservation Project (2017NWB036-01, 2017NWB036-12-2), and the National Crop Germplasm Resources Platform Project (NICGR2017-001).
URL:http://kns.cnki.net/kcms/detail/11.1809.S.20181105.1326.016.html
