河南省鳙养殖群体的遗传多样性与选择压力分析
2018-09-18刘慧芬黄建美陈秋凤陈蕃锦朱国超宋东萦聂国兴李学军
刘慧芬,王 静,黄建美,陈秋凤,陈蕃锦,马 晓,朱国超,宋东萦, 聂国兴,李学军
(河南师范大学水产学院,河南省水产动物养殖工程技术研究中心,河南新乡 453007)
鳙(Hypophthalmichthysnobilis)又叫花鲢,是我国著名的四大家鱼之一,在我国淡水养殖业中占有重要地位。鳙的养殖历史悠久,最早可以追溯到一千多年前的唐代,鳙人工繁殖技术的成功,更是推动了养殖业的发展[1]。根据2017年《中国渔业统计年鉴》的统计,我国鳙产量从2007年的213.5万吨增至2016年的348.02万吨,在淡水养殖鱼类年产量中位居第四,产生了巨大的经济效益[2]。
目前,国内已有许多关于鳙的遗传多样性水平检测和种质评估工作。李思发等[3]利用线粒体DNA分析了长江中下游鳙群体的多样性,发现鳙的遗传多样性丰富,但是群体间存在显著的遗传差异;张德春等[4]采用RAPD方法分析了长江中游鳙自然繁殖群体和人工繁殖群体的遗传变异,发现人工繁殖群体的遗传多样性明显低于自然群体;张敏莹等[5]研究了长江下游鳙放流群体和天然捕捞群体的遗传多样性,发现放流群体与天然捕捞群体间的遗传多样性相差不大;耿波等[6]利用17个鳙微卫星标记分析了四川泸州和江西鄱阳湖两个鳙种群的遗传多样性,发现两个地理种群间存在种群内变异;严骏骢等[7]运用AFLP技术分析了中国土著鳙群体和移居群体间的遗传结构差异,发现移居群体的遗传多样性显著低于国内土著群体,出现显著分化。这些研究主要集中在湖北、湖南、广东、四川、江西、黑龙江等地区,但至今尚未有关于河南省鳙遗传背景的研究报道。
鳙作为河南重要经济鱼类之一,市场需求量较大,既有本地自繁苗种,也有从广东、湖北、安徽、天津等地引进苗种。为了厘清河南地区养殖鳙群体的遗传背景,切实做好亲本遗传管理工作,对河南地区鳙养殖群体的遗传多样性进行检测势在必行。
目前关于鳙遗传多样性的分析多采用DNA分子标记,但至今尚未有基于线粒体COI基因(细胞色素C氧化酶亚基Ⅰ)分析鳙遗传背景的研究报道。COI基因作为线粒体DNA上较为保守的序列,被认为是研究群体遗传多样性非常有效的工具。因此,本研究利用线粒体COI基因对河南省的11个鳙养殖群体进行了遗传多样性分析,研究不同群体鳙的遗传结构和系统发育关系,旨在为河南省鳙养殖业的健康发展提供参考资料。
1 材料与方法
1.1 实验材料采集与处理
鳙样本于2016年8-10月间分别取自河南省11个养殖场(样品具体采集地点见表1)。每个群体随机选15尾,剪取部分肌肉组织保存在95%酒精中,用于总DNA提取。

表1 养殖鳙样品信息
1.2 DNA提取
基因组DNA的提取采用酚-氯仿法,具体步骤如下:取100 mg 左右肌肉组织,加入495 μL裂解缓冲液(1mol/L Tris-HCl 25 μL,0.5 mol/L EDTA 50 μL,NaCl 5 mol/L),5 μL 蛋白酶K(浓度为 20 mg/mL),55 ℃恒温水浴锅消化2~3 h。加入等体积的饱和酚,缓慢转动15 min,12 000 r/min 离心10 min。吸取上清液,加入等体积的氯仿-异戊醇混合液(24∶1),缓慢转动10 min后,12 000 r/min 离心10 min。吸取上清液,加入800 μL提前预冷的无水乙醇(-20 ℃)沉淀DNA,70%乙醇洗涤一次,干燥,加入适量ddH2O溶解。用1%琼脂糖凝胶电泳检测DNA质量,Nanodrop 2 000检测DNA浓度,-20 ℃冰箱保存备用。
1.3 PCR扩增和测序
用于扩增mtCOI序列的引物来自Ivanova等[8],具体序列见表2。PCR反应总体系25 μL,其中包括Buffer 2.5 μL,Mg2+2.5 μL,dNTP 1 μL,DNA 1 μL,上下游引物各0.5 μL,Taq酶0.25 μL,用ddH2O补足25 μL。反应程序:94 ℃预变性4 min,然后进行30个循环,每个循环包括94 ℃变性30 s,52 ℃退火35 s,72 ℃延伸1 min,最后72 ℃延伸10 min,4 ℃保存。PCR产物用1%琼脂糖凝胶电泳检测,凝胶成像系统观察拍照后,送至上海生工生物工程有限公司进行测序。
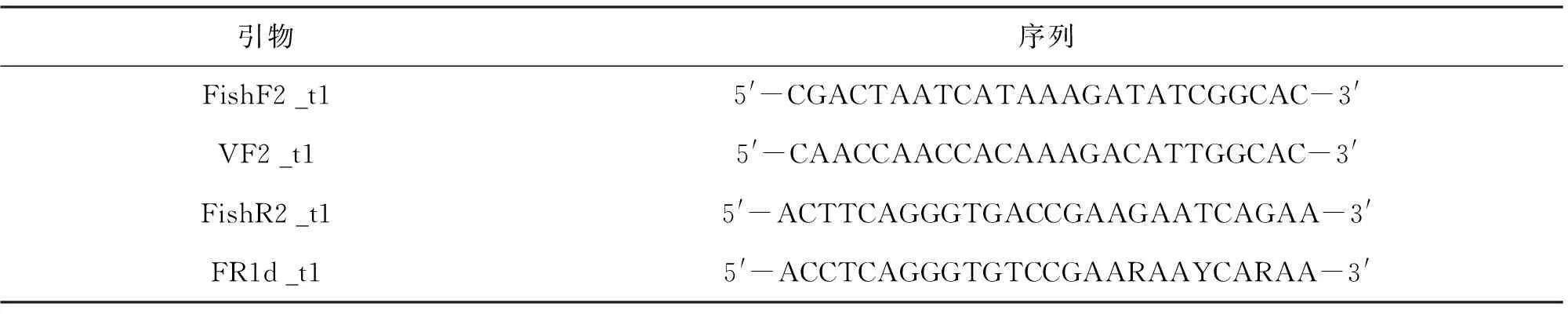
表2 实验中所用引物
1.4 数据分析
测序完成后,DNA序列用CLUSTALX 1.81软件[9]进行比对;用DNAsp 5.0软件[10]统计核苷酸多样性(nucleotide diversity,π)、单倍型多样性(haplotype diversity,Hd)和群体遗传分化系数(FST)。通过PMAL4.3软件[11]和Adaptive Evolution Server在线平台(http://datamonkey.org//dataupload.php)检测鳙养殖群体线粒体COI基因受选择情况,用非同义替代率(dN)与同义替代率(dS)的比值来估算选择压力。用ARLEQUIN 3.1软件[12]进行分子变异分析(AMOVA);用MEGA 4.0软件[13]的Kimura 双参数模型计算单倍型之间的遗传距离,并按照邻接法(neighbor joining method,NJ)构建系统发育树,系统树各节点支持率采用Bootstrap 1 000次重复检验置信度;用TCS 1.2软件[14]构建单倍型网络图。
2 结果
2.1 鳙群体线粒体COI序列和遗传多样性分析
对采自河南地区11个不同养殖场的鳙进行PCR扩增,获得线粒体COI序列,去除两端引物后,得到的目的片段长度为652 bp。经比对分析发现鳙各群体碱基含量差别不大,A、T、C、G平均含量分别为25.3%、29.5%、27.3%、17.9%,A+T含量(54.8%)高于C+G含量(45.2%)。165尾鳙样品共检测到7个基因变异位点,其中简约信息位点5个,单突变位点2个。鳙群体中检测到了14个单倍型(表3),其中群体间共享单倍型10个,特有单倍型4个。各群体单倍型多样性范围在0.714 29~1.000 00之间,其中单倍型多样性最高的是5号群体,最低的是8号群体;核苷酸多样性范围在0.001 27~0.003 24之间,其中核苷酸多样性最高的是5号群体,最低的是2号群体。基因流分析显示11个鳙养殖群体间的平均基因流为5.01。
2.2 鳙群体间的遗传分化与遗传距离
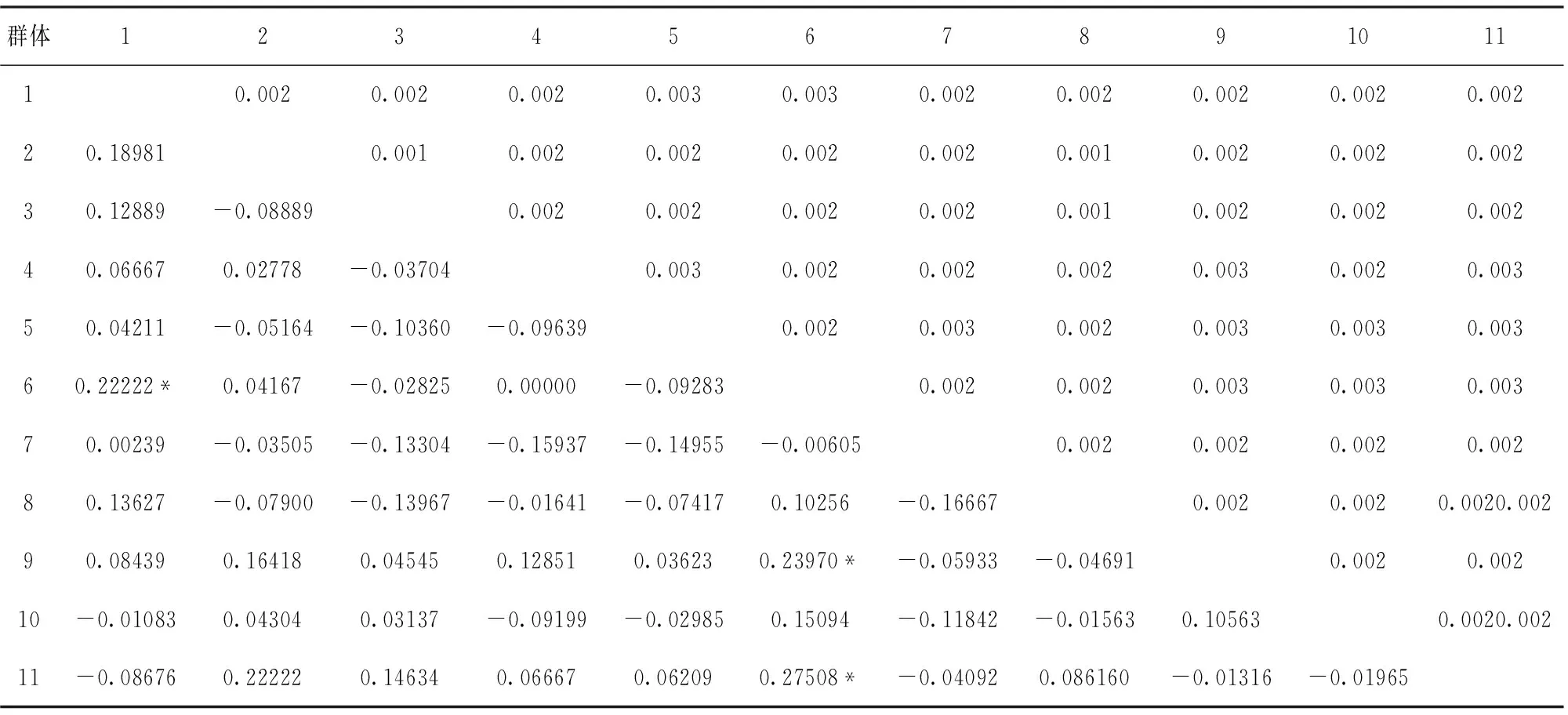
用ARLEQUIN 3.1软件计算不同养殖场鳙群体间的遗传分化(表4),结果显示鳙两两群体间遗传分化系数(FST)值在-0.166 67~0.275 08间,有1组发生极大的遗传分化(FST>0.25),6组发生较大的遗传分化(0.15 对不同养殖场鳙群体进行分子方差分析(AMOVA),结果显示(表5)鳙群体主要的变异来自群体内(97.65%),群体间变异所占比例较低,仅为4.60%,并且3组分子遗传变量间的差异均未达到显著水平。 表3 河南省鳙养殖群体遗传多样性信息 注:Hd为单倍型多样性,π为核苷酸多样性。 表4 养殖鳙群体间遗传分化系数(FST,对角线下方)和遗传距离(D,对角线上方) *表示差异显著(P<0.05)。 表5 养殖鳙群体AMOVA分析 采用NJ法构建系统发育树分析不同养殖场鳙群体间的系统发育关系,结果显示11个养殖场的鳙群体交叉聚在一起,没有形成明显的地理格局分布(图1)。 通过单倍型网络分析,可以看出14个单倍型在各群体中的分布情况以及单倍型之间的演化关系。共享单倍型散布在不同的养殖群体中,没有按照地理区域聚类,这一结果进一步支持了系统发育树的分析结果(图2)。 图1 基于mtCOI基因构建河南地区养殖鳙NJ树 图2 TCS计算mtCOI基因单倍型网络图 为检验是否有选择压力作用在人工养殖鳙群体上,通过PAML软件对11个鳙养殖群体的线粒体COI基因进行选择压力分析和位点检测,结果发现除5号群体外,所有养殖群体的dN/dS比值均为0,表明在这些群体中,所有核苷酸的替换均为同义替换,没有出现氨基酸的改变。当把11个群体混为一组时,检测到83号位点的dN/dS比值为0.17(P>0.05),群体受负向选择影响。综上所述,鳙养殖群体的线粒体COI基因主要受到负向选择影响,没有受到正选择作用。 遗传多样性是评价物种资源状况的重要依据之一,决定了物种的生存力和适应性,是优良性状选育的基础[15,16]。本研究采集的河南省鳙养殖个体有来自湖北、广州、安徽、天津的苗种,也有本地自繁种群,基本上代表了河南省养殖鳙种质资源状况。利用线粒体COI基因对11个鳙养殖群体的遗传多样性进行分析,165尾鳙样品中共检测到14个单倍型,其中群体间共享单倍型10个,特有单倍型4个。这4个特有单倍型中,hap11和hap12为本地自繁苗种所特有,hap2为湖北苗种所特有,hap14为天津苗种所特有,广州和安徽鳙群体没有发现特有单倍型。 单倍型多样性和核苷酸多样性是衡量群体遗传多样性的两个重要参数。在本研究中,11个群体的平均单倍型多样性为0.862 37,平均核苷酸多样性为0.002 23;其中,5号群体(本地自繁)各参数值最高,2号群体(广州)和8号群体(河南驻马店)各参数值较低。本研究11个群体的遗传多样性整体要高于单淇等[17]报道的江西瑞昌(Hd为0.6883,π为0.00511)、湖南长沙(Hd为0.237 9,π为0.001 91)和天津宁河(Hd为0.445 9,π为0.006 67)三个鳙群体。这也可能与所选分子标记不同,线粒体DNA不同区段的进化速率差异有关。 鳙在河南省的养殖模式主要为池塘混养中搭配养殖,但是近年来由于价格提升,才开始增加养殖量。5号群体和8号群体虽然都是人工繁殖群体,但是8号群体遗传多样性明显低于5号群体。养殖群体表现出单倍型多样性贫乏,遗传多样性偏低,反映其育苗亲本数量少。人工养殖条件下依靠少量亲本进行多代的连续繁殖,容易导致群体发生瓶颈效应和近交衰退现象,致使其遗传多样性水平出现下降。 AMOVA分析结果显示,养殖鳙的遗传变异主要来自群体内(97.65%),群体间的遗传差异较低。养殖鳙群体的NJ系统树和单倍型网络图均未检测到明显的地理谱系结构。NJ聚类树发现湖北、广州、安徽、天津以及河南本地鳙群体间存在着较多的交叉和渗透。11个群体单倍型网络聚类关系与NJ聚类的结果类似,各群体单倍型基本交叉在一起。产生这些结果的原因可能是人工繁殖造成的结果。养殖群体在选育过程中是以经济性状为目标,各养殖群体在选育过程中部分亲本的遗传背景可能存在交叉[18,19]。因此,在鳙繁育工作中,需要加强亲本的遗传背景检测,选择遗传变异高的个体作为亲本,才能保证人工繁殖后代遗传多样性和养殖性能。 遗传、变异和选择是生物进化的主要动力[20]。对于大多数养殖鱼类群体,在人工繁殖和养殖过程中,包含着有意或无意的选择。基因所受到的选择压力往往只表现在部分位点上,而不会表现在整条基因序列上[21]。选择压力位点检测结果表明,人工养殖鳙群体线粒体COI基因上留下了选择信号。通常认为非同义突变(dN)与同义突变(dS)的比值大于1,基因受正向选择的影响;dN/dS的比值小于1,基因受纯化或负向选择的影响[22]。本研究中鳙所有养殖群体线粒体COI基因的dN/dS比值均小于1,说明鳙养殖群体主要受到纯化选择影响。从进化的角度看,群体遗传变异是自然环境与人工选择共同作用的结果。在自然环境条件下,没有巨大环境变化时更多受到的是纯化选择[23]。养殖鳙群体的压力分析结果,暗示当前的人工选择对鳙线粒体基因的作用有限。 本研究选用线粒体COI基因检测选择压力。线粒体COI基因编码细胞色素氧化酶,是线粒体氧化呼吸链的重要成员。Liu等[24]分别用线粒体基因和核基因检测了鳅超科鱼类的选择压力,发现线粒体COI基因受负向选择的影响最大。因此,下一步研究我们将选择更多的基因去证实养殖鳙群体受到的选择作用。 综上所述,本研究中河南省11个鳙养殖群体的遗传多样性和遗传分化差异较大,遗传背景存在交叉。鳙作为我国主要的养殖鱼类之一,不同地区间的引进和交流比较频繁。人工养殖鳙需要优良种质,但是不当的引种和人工养殖会使鳙群体出现种质混杂和优良种质退化现象。基于此,建议苗种繁育场在引进苗种时要对其遗传背景进行考察分析,建议加大引种的数量与范围,打破瓶颈效应;另外一方面,人工繁殖时建议进行优良亲本选育,增加繁殖亲本数量,采取家系选育技术提高选育效率,避免近交衰退带来的风险并有利于保持鳙苗种的遗传多样性。

2.3 系统发育关系


2.4 选择压力分析
3 讨论
3.1 河南省鳙养殖群体的遗传多样性
3.2 鳙养殖群体的选择压力
