利用Illumina MiSeq高通量测序技术分析原料乳的菌群分布
2018-08-31于国萍姚宇秀范美婧董良伟
于国萍,陈 媛,姚宇秀,范美婧,刘 鹏,董良伟
原料乳中营养成分比较高,是各种细菌滋生的绝佳环境,原料乳可以为其提供充足的养分和合适的pH值,所以原料乳中有着相对密集的微生物[1],其数量从几百到几千CFU/g不等[2]。其中部分微生物能够改善乳制品的口感等,这些微生物并不都是有益的,有的是有害的,可以引发比较严重的疾病[3]。原料乳中常见的微生物有3 大类,即有益微生物、有害微生物和病原微生物[4]。
原料乳中有益微生物主要是乳酸菌,而乳酸菌主要包括链球菌、明串珠菌和乳杆菌属等。有害微生物常称为腐败菌,其中最主要的就是嗜冷菌。病原微生物通常是致病菌,它即便不改变乳的性质,却也可以造成疾病,引发大规模的感染。原料乳中食源性致病菌主要为大肠杆菌O157:H7、沙门菌、单核细胞性李斯特菌、金黄色葡萄球菌以及志贺菌等[5-8]。
由于乳及乳制品具有较高的营养成分,我国对其的需求越来越大,因此对原料乳的微生物的检测也比较重要[9],原料乳中微生物多样性的检测方法也从原来的纯培养手段转变为分子生物学手段[10-11]。Fricker等[12]采用纯培养和分子方法相结合的手段研究了微生物的多样性,Bonizzi等[13]采用了内转录间隔区序列分析了微生物的构成,Raats等[14]采用变性梯度凝胶电泳(denaturing gradient gel electrophoresis,DGGE)和克隆文库的方法对原料乳及其冷藏过程中菌群的变化进行研究。其中,纯培养的手段比较繁琐,而DGGE等分子生物学技术很难全面地反映微生物的多样性。
随着乳及乳制品的大量生产,我国乳制品的质量问题经常出现[15],这很大程度上是由于乳及乳制品中感染了致病菌,导致食源性疾病的发生。因此,加强原料乳的质量安全检测至关重要,可以防止大规模食源性疾病的发生[16-17]。目前使用的检测方法大多为分子方法[18-22],已有研究分别采用不同的聚合酶链式反应(polymerase chain reaction,PCR)技术检测了乳中致病菌。虽然目前的分子检测方法有很多优势,但是仍旧检测不全面,因此本实验采用高通量测序(high-throughput sequencing,HTS)手段对原料乳的菌种进行检测。
HTS又叫“下一代”测序技术[23-24],主要是能同时对很多样本中提取的DNA进行测序,效率非常高。HTS技术给过去分子水平的生物测序技术带去了革命性的创新,已经普遍利用到关于基因组测序等一系列分子水平的测序方法上[25-27]。本实验主要采用的是Illumina公司的MiSeq测序平台[28],MiSeq个人测序系统采用Illumina的TruSeq边合成边测序技术。第2代测序Illumina MiSeq方法分析有效的避免了通量低、操作复杂和准确率低等缺陷[29-32],具有操作简单、成本较低的优势,并且采用边合成边测序原理,结果可信度高。
本实验主要是利用HTS的方法测定原料乳中菌种的分布情况。采集14 个奶牛场的原料乳,通过HTS的方法(采用Illumina MiSeq系统)测定菌群多样性分布情况,然后根据测序结果,了解原料乳中菌群的分布情况,为构建奶牛养殖风险防控机制,并提出防控奶牛养殖风险的对策提供参考。
1 材料与方法
1.1 材料与试剂
原料乳 黑龙江省内14 个奶牛场提供。
E.Z.N.A.Soil DNA试剂盒 瑞士Omega公司;Qubit 2.0 DNA检测试剂盒 美国Life公司;Taq DNA Polymerase 美国Thermo公司;Agencourt AMPure XP美国Beckman公司。
1.2 仪器与设备
Pico-21型台式离心机 美国Thermo Fisher公司;GL-88B型旋涡混合器 海门市其林贝尔仪器制造有限公司;TND03-H-H型混匀型干式恒温器 深圳拓能达科技有限公司;DYY-6C型电泳仪电源、DYCZ-21型电泳槽北京市六一仪器厂;凝胶成像系统 美国UVP公司;Q32866型Qubit®2.0荧光计 美国Invitrogen公司;T100TMThermal Cyeler型PCR仪 美国Bio-Rad公司。
1.3 方法
1.3.1 样品的采集
在黑龙江省(包括哈尔滨、双城、阿城等地)14 个奶牛场采集原料乳,将样本放在-20 ℃的冰箱里面冻存,并进行后续的实验,按采集的奶牛场的顺序分别标记为1~14。
1.3.2 样品的前处理
将冻存后的样品进行解冻,因为菌株样品和原料乳属于液体,所以其中包括的菌种的数目相对较低,进行后续实验之前一定要将样本混合均匀,样本的菌种比较丰富时,吸取一定量的样本进行离心,倒出液体,留下离心后的菌种。
1.3.3 DNA的提取
DNA的具体提取步骤参照E.Z.N.ATM Soil DNA试剂盒进行,按说明书操作。所提取的DNA于-20 ℃保存备用。
1.3.4 琼脂糖电泳
将提取的DNA进行琼脂糖电泳。将一定量的DNA与一定量的上样缓冲液按比例混合,然后吸取其中的混合液10 μL进行DNA电泳,条件为200 V、45min,在凝胶成像系统中进行观察。
1.3.5 PCR扩增
1.3.5.1 第1轮扩增
PCR采用双引物进行扩增,引物序列是V3-V4通用双引物[33]。主要是先将DNA产物加入试剂盒中的各项物质形成1 个体系,需要加入的物质为PCR缓冲液、核苷酸、DNA样品、Bar-PCR primer F、Primer R、Plantium Taq、双蒸水,使整个反应体系体积为50 μL。
将混合好的物质放入PCR仪中进行扩增,主要步骤是:先进行预变性,然后进行变性过程,需要将其循环多次,最后延伸即可。之后进行第2轮扩增。
1.3.5.2 第2轮扩增
引入Illumina桥式PCR兼容引物,第2次PCR需要加入的物质与第1次类似,只是加入的量不同,同样是使整个反应体系是50 μL。扩增的步骤与方法见1.3.5.1节。
1.3.6 PCR产物的琼脂糖电泳
将PCR后物质进行DNA电泳,步骤同1.3.4节。
1.3.7 DNA纯化回收
将扩增后物质进行DNA的纯化回收,不同微生物的扩增后的物质采用的回收的方法基本一致,不同的是选择的磁珠不同。具体步骤是:用磁珠对扩增后的产物进行吸附,吸附3 次,在吸附的过程中,需要将其放在磁力架上,保证它的吸附力。最后用洗脱液洗脱后保存到小的EP管中,进行后续的实验。
1.3.8 定量混合
利用Qubit 2.0 DNA检测试剂盒对回收的DNA进行定量。最后要将纯化后的DNA与原来的进行混合,使最后进行测序的物质浓度为20 pmol/L。
1.3.9 数据预处理
1.3.9.1 原始序列数据
经过系统整理的数据可以经过一个系统进行转化,成为最初的测序序列,即Raw Data,将其保存到电脑中,它是由所有的序列信息和与其相关的质量数据组成。
1.3.9.2 数据预处理
经过HTS后的数据含有多余的序列,所以在处理数据之前应该先将多余的序列除去,然后才可以进入系统中进行测序。开始先将多余的引物除去,之后成对的read拼接成一条序列,将序列通过系统识别,将不同样本的数据分开,最终对样本中的数据进行检查,去掉不合格的数据,留下最后的有效数据。
1.3.10 去除嵌合体及非特异性扩增序列
在进行PCR扩增的时候,有时候会出现嵌合体,它通常是由于在第1轮扩增过程中的延伸阶段,并没有完全进行,只反应到了一半,使得产物不完整,这些不完整的产物会进行下一次扩增,可能会使其加入一些与之不同的片段,形成一种杂交的DNA,这就是嵌合体。同时也会产生一些非特异性扩增序列。为了确保结果的准确性,需要将这些嵌合体除掉。使用Usearch去非扩增区域序列。先采用uchime验证这个序列是嵌合体,验证成功后将其去除。
1.3.11 分析项目
操作分类单元(operational taxonomic units,OTU)聚类分析:依据每个序列之间的远近程度进行分析,将距离近的归为一个OTU。选择其中最典型的序列进行检测,通常选择序列数目多的作为典型性序列,然后根据OTU的分类情况进行了聚类分析。
Alpha多样性分析:衡量样本物种多样性。计算物种并制作所有样品丰富度指数、Chao、Shannon、Simpson、Coverage等物种多样性指数,箱形图和稀释性曲线。根据各样本的OTU丰度分布情况绘制相对丰度曲线。
物种分类分析:将样本中的序列进行分类后,分类后的序列属于一个菌属,然后根据菌属的数量进行分析。基于物种分类分析,绘制物种分类条形图、物种丰度饼图、物种丰度热图、单样本群落分布丰度柱状图、群落分布分度3D图、样本聚类与柱状图组合分析图、菌群分布条形图等。
1.4 数据分析
利用Mothur 1.30.1软件、FLASH 1.2.3软件、pear 0.9.6软件、Usearch 5.2.236软件、Cytoscape 3.2软件、R 3.2软件等进行数据处理分析。
2 结果与分析
2.1 OTU基本情况
将提取的DNA进行电泳,并将扩增后的产物进行电泳,目的是检测DNA的完整性及其是否能进行后续的实验,扩增后的条带比较亮,说明可以进行后续的实验。OTU指在生物学系统中,为了详细认识样本中菌种的具体分布情况,要将测序的结果按种类开始统计,人为规定一个分组的单位称为一个OTU。一般情况下,认定2 个序列相似度为97%时属于一个菌属,当相似度为99%时,认定其是一个菌种。本实验采用的相似度为97%为一个OTU。
2.2 基于OTU丰度的样本聚类图
样本聚类树图可以直观地反映样本间的相似度,具体方法如下:首先使用描述群落组成关系和结构的算法计算样品间的距离,依据常用的一种算法创造一个或者多个节点的图谱,算法是非加权组平均法,算出一个结构图谱,可以直接观察图谱分析数据,计算样本间距离的方法为布雷柯蒂斯距离。在目前的统计学里,聚类分析全是利用布雷柯蒂斯距离分析的,它能够显示菌落差异,因此本实验使用该参数反映菌落的差异。原料乳的14 个样本的基于OTU的样本聚类树图见图1,树枝的长度代表样本间距离,分支的宽度表示样本间的距离的远近,相似度高的样本距离越小。由图1可以看出,原料乳的14 个样本类聚成2 个主要群体,3、4、5、6、7、8、9、10、11是一类,1、2、12、13、14是另一类。其中12和13是最相似的两个样本。整体看14 个样本,相似度差别略大,可能是由于在黑龙江省不同的区域进行取样,地域的差异性导致的。

图1 基于OTU的样本聚类树图Fig. 1 Clustering dendrogram based on OTU samples
2.3 稀释性曲线
为确定所得到的结果正确性,是否能进行后面的实验,需要进行稀释性曲线分析,它主要是从整个样品的数据里面抽取部分进行分析,分析其所包含的菌种的多少,是通过抽取的个体与物种的数目建立图谱。通过该曲线能够分析样品的菌种数是否丰富,也可以分析结果是否正确。随机选择样本中的一部分序列,将其指代的OTU与序列数绘制稀释曲线,如图2所示,沿着横坐标曲线逐渐稳定时,看出结果分析比较合理,相反,如果曲线不稳定,说明结果不合理,不能进行分析。所以,通过观察该曲线,能看出样本是否检测完全。整个曲线都是依据相似度97%绘制的。由图2可以看出,所有样本的曲线都随着样本中随机抽取序列数增大而逐渐稳定,说明结果比较合理,所含菌种数较多,可以进行后续的测序。
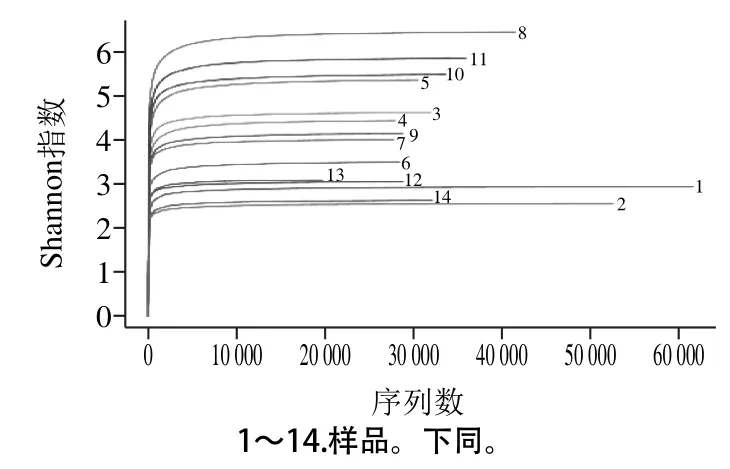
图2 Shannon指数稀释分析图Fig. 2 Rarefaction curves for Shannon index
2.4 Rank-abundance曲线
为了直观地表示每个样本中菌种的数目是否丰富,进一步绘制了Rank-abundance曲线,它与多样性指数一样,可以看出样本的多样性。主要是计算每个样本中的各个OTU包括的序列数目,按照从大到小进行排列,绘制曲线如图3所示。当然,Rank-abundance曲线能够表示样本菌种数目的两大部分,包括样本中所含的菌种的丰度及其是否均匀。曲线的宽度代表了菌种的丰度,曲线越宽,说明所包含的菌种越多;曲线的平稳程度代表了其菌种数目是否均匀,曲线越稳定,说明菌种的构成越均匀。由图3可以看出,所有的样本在横坐标上的宽度的范围分布较广,说明所含菌种的数目较多,组成丰富;同时可以看出曲线比较稳定,说明菌种的构成比较均匀。
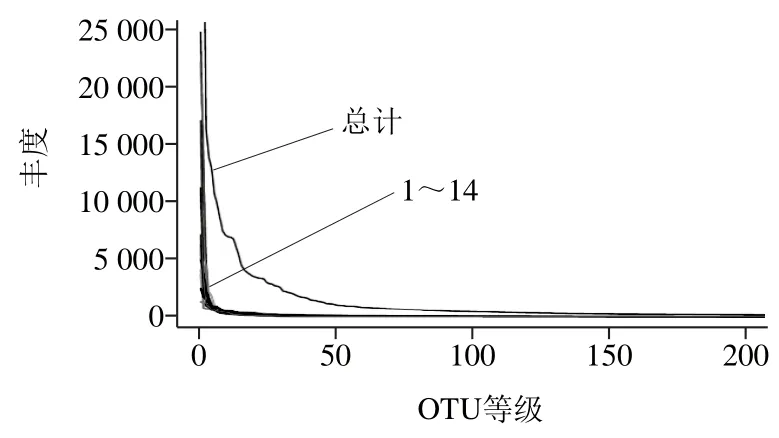
图3 Rank-abundance曲线图Fig. 3 Rank-abundance curves
2.5 物种累积曲线
为了确定样品中的菌种的数目是否会根据样本的数目增加而增加,绘制了物种累积曲线图,由其可看出测序的菌种数是否完全检测出来,也可以看出样本中的菌种数量是否满足需要。根据曲线的走向不但能够知道样本中菌种是否足够,也可以看出样品中物种的丰度。物种累积曲线代表了随着样本的增加是否会有新的OTU出现。在曲线中,随着样本的增加,如果曲线快速增长,说明有更多的OTU检测出来;如果曲线增长缓慢,说明没有更多的OTU检测出来,说明结果合理。因此通过该曲线能够判断样品量是否满足需要,如果曲线快速增长,需要加入足够的样品量满足测序分析,相反的话,就可以直接进行后续的实验。由图4可以看出,沿着横坐标样本的增多,曲线增长比较缓慢,说明没有更多的OTU检测出来,说明样品量足够,可以满足需要。
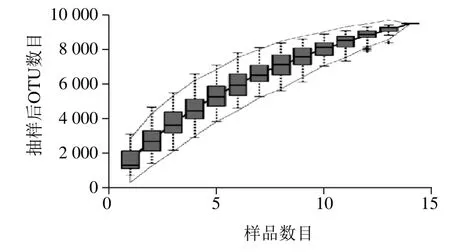
图4 物种累积曲线图Fig. 4 Species accumulation curves
2.6 物种丰度热图
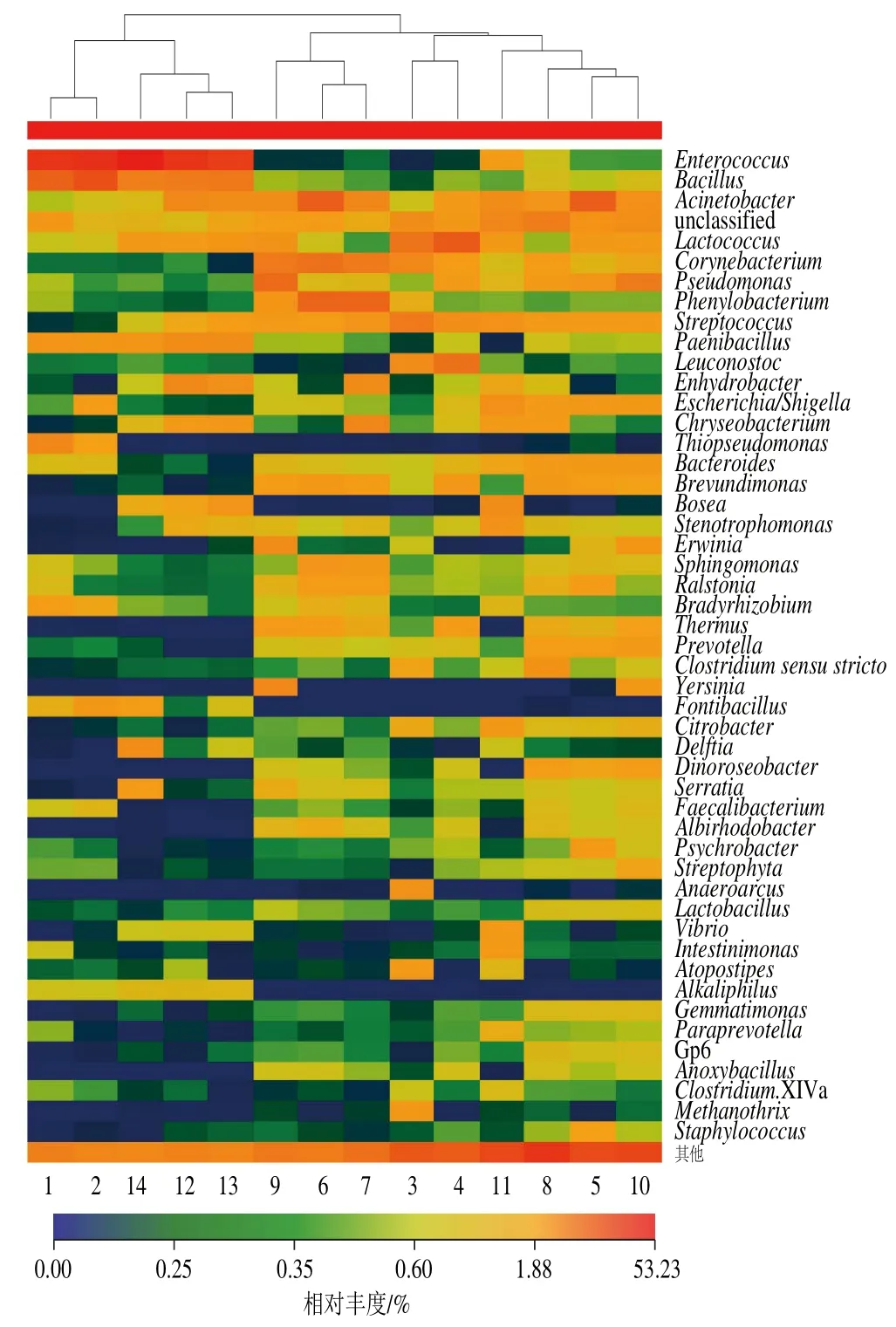
图5 属水平物种丰度热图Fig. 5 Heat map of species abundance at genus level
菌群数量用热图颜色标记,曲线制作过程是将菌落的数量聚类分析,把分析后的结果体现在热图中。原料乳中14 个样本的属水平物种丰度热图,如图5所示,颜色越红说明所含的该菌种的数量越多,颜色越蓝则说明所含的菌种不多。为了展示效果,只显示丰度最高的前50 个物种分类信息,剩余的物种分类合并成其他。由图5可以看出,14 个样本的前5 种菌种的显示颜色比较红,含量比较多,分别是肠球菌属、芽孢杆菌属、不动杆菌属、乳球菌属,其中第4个丰度较高的菌属并没有与之匹配的数据库。其他菌属中有的样本颜色比较红,有的样本颜色比较暗,说明每个样本中菌群的丰度不同,可以看出所有样本的菌群的多样性。
2.7 所有样本菌群分布条形分析

图6 原料乳的菌群分析条形图Fig. 6 Barplot for bacterial genus distribution in raw milk
依据微生物的特性,重点分析菌种的菌群分布,通过菌群分布条形图可以看出菌种的大概分布,所占的比例等,另外还可以看出不同的样本之前的差异。由于菌群的结构图包括单样本群落分布丰度柱状图,群落分布分度3D图,样本聚类与柱状图组合分析图、菌群分布条形图等,本实验用菌群分布条形图来代表。由图6可以看出,原料乳的14 个样品的菌种的分布情况。1、2、12、13、14号奶牛场的原料乳中肠球菌属、芽孢杆菌属较多,3、4号奶牛场中乳球菌属较多,5号奶牛场中不动杆菌属较多,6、7号奶牛场中苯基杆菌较多,8号奶牛场中梭状芽孢杆菌较多,9、10号奶牛场中假单胞菌属较多,11号奶牛场中慢生根瘤菌科中的一个属较多。在结果中关于致病菌的菌种和菌属,可以得知1、2中有金黄色葡萄球菌,2、5、8、10中有志贺菌,其他样本的致病菌暂时没有检测出来。
3 结 论
本实验率先将HTS技术应用在原料乳的菌群的研究中,通过HTS技术了解原料乳中菌群的分布情况。由此可知不同牧场原料乳的菌群存在多样性,位于前列的分别是肠球菌属、芽孢杆菌属、不动杆菌属、乳球菌属,部分原料乳中有金黄色葡萄球菌和志贺菌的存在。相关领域研究者采用不同的方法对原料乳菌群分布进行研究,例如,李博等[34]利用宏观菌落形态学观察的方法检测原料乳中的主要微生物,即在鉴别培养基上培养菌群,观察生长情况,结果表明原料乳中含有乳酸菌、大肠杆菌、嗜冷菌;Pourhanssan等[35]采用革兰氏染色结合镜检的方法对原料乳中的菌群进行了分析鉴定,结果表明原料乳中有大肠杆菌、金黄色葡萄球菌、肠杆菌、克雷白氏杆菌属、绿脓杆菌以及变形杆菌属;相较于HTS,分离培养、生化鉴定无法对难培养或者不可培养的致病菌进行检测,存在特异性差、操作复杂、耗时等缺点,无法实现及时有效检测。刘洋等[36]利用基质辅助激光解吸电离飞行时间质谱鉴定原料乳中细菌菌落分布,鉴定出原料乳中含有乳酸菌、肠杆菌、金黄色葡萄球菌;与这些方法相比,HTS的研究结果更加全面地反映了原料乳菌群分布多样性及丰度信息。此外,夏围围等[37]利用HTS和DGGE分析土壤微生物群落结构的比较研究中,得出HTS能够较为全面和准确地反映土壤微生物群落结构,而DGGE仅能分析有限的优势微生物类群,存在高估物种丰度以及低估微生物群落大小和多样性的可能。因此应该严格控制奶牛生活环境的卫生情况,防止金黄色葡萄球菌和志贺菌污染原料乳,保障原料乳质量安全,为有关部门控制原料乳及储奶设施的卫生条件提供有力的理论和实验依据。
本研究利用HTS方法,对原料乳的菌群分布情况进行了较全面、系统、准确的检测,具有很高的特异性和灵敏度,并为原料乳中致病菌检测提供了技术支持。随着HTS技术的不断推进,相信在不久的将来,它会在食品的检测领域有更广泛和全面的应用前景。
