米槁天然种群遗传多样性的ISSR标记分析
2018-07-25李丽霞刘济明黄小龙柳嘉佳邓明明
李丽霞,刘济明,黄小龙,骆 畅,熊 雪,柳嘉佳,邓明明,李 佳
(贵州大学 林学院,贵州 贵阳 550025)
米槁(CinnamomummigaoH.W.Li)系樟科(Lauraceae)樟属(Cinnamomum)常绿乔木[1],主要分布于云南、贵州和广西等省(自治区),垂直分布海拔300~1 000 m[2]。米槁为贵州省地道中药材,其干燥果实大果木姜子是贵州十大苗药之一[3]。除药用外,米槁还可用作香料和工业原料,是一种多用植物。赵山等[2]、冉光德[4]、郑亚玉等[5]认为,由于近年来对米槁不合理的乱砍滥伐,加上长期只收不种、不保护的恶性循环,以及该物种坐果率低、果实有大小年之分的自然障碍,致使米槁种群天然更新不良,种群数量急剧下降。目前有关米槁的研究主要集中在药用化学成分分离与鉴定、化学成分的生物活性与药理特性等方面[6-11],而对其基础生物特性、生态特性以及针对其种群退化机制原因的探究却鲜有报道。遗传多样性是物种长期进化的结果,是种群生存和发展的前提,对于米槁等生命周期相对较长的林木植物来说,遗传多样性决定了其适应能力,是维持森林生态系统长期稳定的基础[12-13]。分子标记是DNA水平遗传多态性的直接反映,分子标记中简单序列重复区间扩增(Inter-simple sequence repeat,ISSR)结合了RAPD和SSR的优点,目前已广泛应用于植物品种鉴定、遗传作图、基因定位、分类、进化及遗传多样性等方面的研究中,通过分子标记可以有效地指示植物的遗传结构与分化情况,探索植物在物种水平和种群水平的遗传多样性情况[14-15]。在米槁具备良好应用价值的情况下,无论是从保护的角度还是从开发利用的角度,开展米槁种群生存现状和遗传多样性研究,不仅能为米槁遗传资源的遗传改良、有效保护和合理利用提供理论依据,还可为米槁种群退化机制的探讨奠定基础。
1 材料与方法
1.1 材 料
供试材料来源于贵州省罗甸县(LD1、LD2)、册亨县(CH)、贞丰县(ZF)、望谟县(WM1、WM2)、荔波县(LB)、镇宁县(ZN)的8个米槁天然种群(表1),从中采集62株米槁无病虫害的幼嫩叶片,采集时间为2016年7-8月。将采摘的米槁新鲜叶片立即放入装有硅胶的自封袋中,将其带回实验室再置于-70 ℃冰箱中保存备用。
1.2 方 法
1.2.1 DNA的提取与检测 采用新型植物基因组DNA提取试剂盒(天根生化科技有限公司)提取米槁(8个天然种群62株单株)叶片的DNA,放入-20 ℃保存备用。用Eppendorf公司的Biophotometer核酸蛋白分析仪检测所提DNA的含量和纯度,用0.8%琼脂糖凝胶进行电泳,用G-BOX紫外凝胶成像系统观察并拍照记录,检测DNA品质。
1.2.2 ISSR-PCR扩增 参照哥伦比亚大学(UBC)2006年公布的100条ISSR引物序列,由上海英俊生物技术有限公司进行引物合成。从100条ISSR引物中筛选出扩增条带清晰、多态性高、稳定性好的19条引物用于米槁的ISSR分析(引物编号及序列见表2)。反应体系总体积为20 μL,包含MgCl21.8 mmol/L,dNTPs 0.2 mmol/L,引物0.7 μmol/L,模板DNA 90 ng,TaqDNA聚合酶1 U。ISSR-PCR扩增程序为:94 ℃预变性5 min;94 ℃变性30 s,53.0 ℃退火45 s,72 ℃延伸1 min,35个循环;最后72 ℃延伸5 min。扩增产物于4 ℃保存。
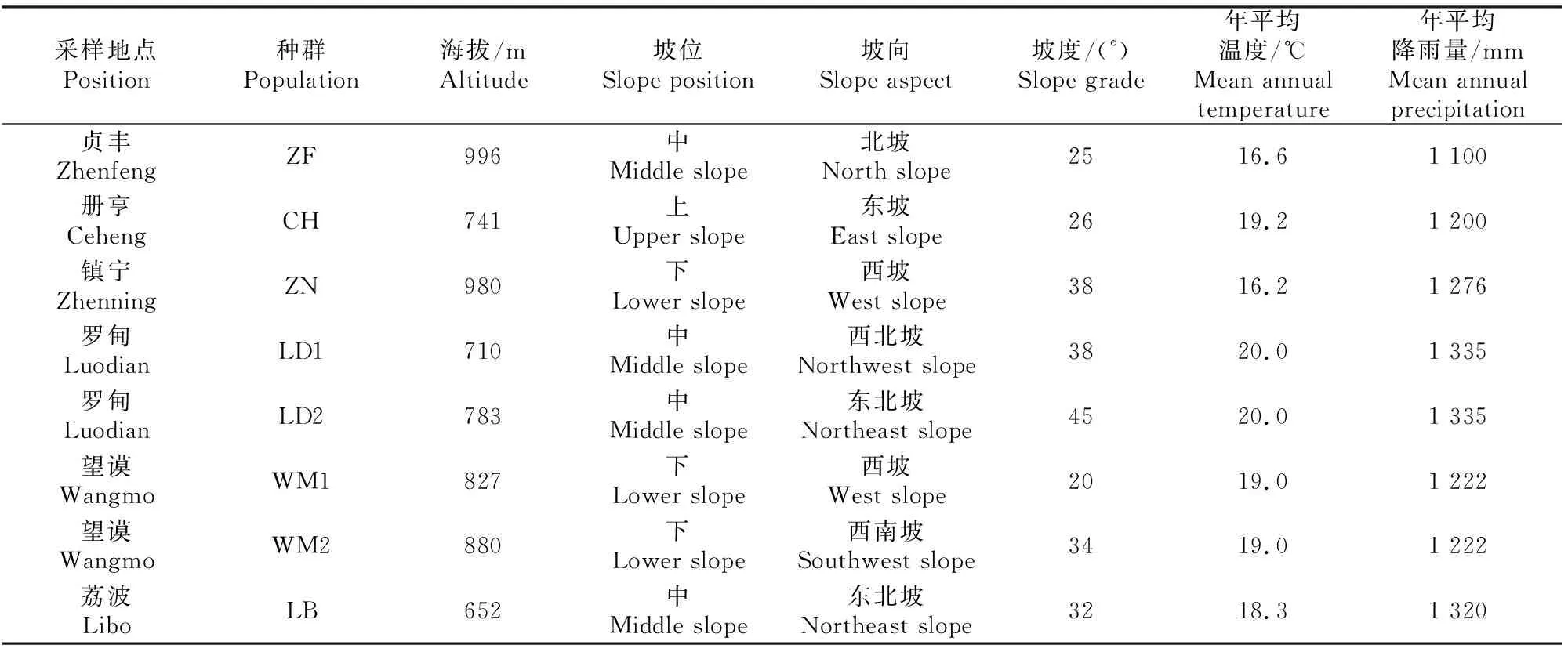
表1 米槁采样地概况Table 1 Description of sampling locations of Cinnamomum migao
1.2.3 ISSR-PCR扩增产物的检测 采用2%琼脂糖凝胶电泳检测PCR扩增产物,缓冲液为1×TAE,电压5 V/cm。在溴酚蓝指示剂距离前沿2~3 cm时停止电泳,采用G-BOX紫外凝胶成像系统观察并拍照记录。
1.2.4 数据分析 采用人工计带法判读电泳图谱中的扩增产物和分子量。相同迁移位置上条带有或无分别计为“1”和“0”,构成“0,1”矩阵。将每个位点视为等位基因M和m,数据“1”代表基因型MM或Mm,数据“0”代表基因型mm,并且假定种群内基因频率处于Hardy-Weinberg平衡状态,用POPGENE32软件计算多态性条带比例(PPL)、观测等位基因数(Na)、有效等位基因数(Ne)、Shannon’s多样性指数(H′)、Nei’s基因多样性指数(H)、基因分化系数(Gst)、基因流(Nm)、遗传相似系数(I)、Nei’s遗传距离(D)等9个相关遗传参数[16]。
采用AMOVA1.55软件对8个米槁种群62株个体进行遗传变异的分子方差分析;用NTSYSpc V2.10e软件对8个米槁种群采用非加权算术平均法(unweighted pair group with arithmetic average,UPGMA)进行聚类分析,建立聚类图;运用GenAlEx软件对62株米槁个体进行主坐标轴分析,建立PCoA 散点聚类图。
2 结果与分析
2.1 米槁物种和种群的遗传多样性
由表2可知,用19条引物对8个米槁种群的62株DNA样品进行PCR扩增,共扩增出186 条重复性高的清晰条带,条带大小为200~3 000 bp,其中多态性条带有167条,多态性条带比例为89.78%。平均每个引物可扩增出9条多态性条带,其中引物UBC846扩增的条带数最多(15条),且扩增条带多态位点比例高达100.00%,引物UBC810扩增条带数最少(6条)。
由表3可知,米槁在物种水平上的多态性条带比例(PPL)为90.86%,在种群水平上的PPL平均为57.26%,其中WM1种群的PPL最高,为70.97%,ZN种群的PPL最低,为30.65%。按PPL排序,各个种群遗传多样性高低的顺序为WM1>LD2=WM2>ZF>LB>CH>LD1>ZN。种群平均观测等位基因数(Na)为1.572 6,Na最高的种群为WM1(1.709 7),最低的种群为ZN(1.306 5),各种群Na大小顺序为WM1>WM2=LD2>ZF>LB>CH>LD1>ZN;物种水平Na为1.908 6。种群有效等位基因数(Ne)平均为1.390 9,最高为 WM1(1.469 6),最低为ZN(1.245 2),各种群Ne大小顺序为WM1>WM2>LB>ZF>LD2>CH>LD1>ZN;物种水平Ne为1.520 9。Nei’s基因多样性指数(H)最高的种群是WM1(0.269 1),最低的是ZN(0.136 2),平均为0.223 3,8个米槁种群H顺序为WM1>WM2>LB>LD2>ZF>CH>LD1>ZN;物种水平的H为0.303 2。Shannon’s多样性指数(H′)种群水平均值为0.328 2,最高的是WM1种群(0.397 4),最低的是ZN种群(0.195 1),各种群排序为WM1>WM2>LD2>LB>ZF>CH>LD1>ZN。H′物种水平(0.453 9)>种群水平(0.328 2),H指数物种水平(0.303 2)>种群水平(0.223 3),Ne物种水平(1.520 9)>种群水平(1.3909 ),由此可见,米槁物种水平遗传多样性较为丰富。
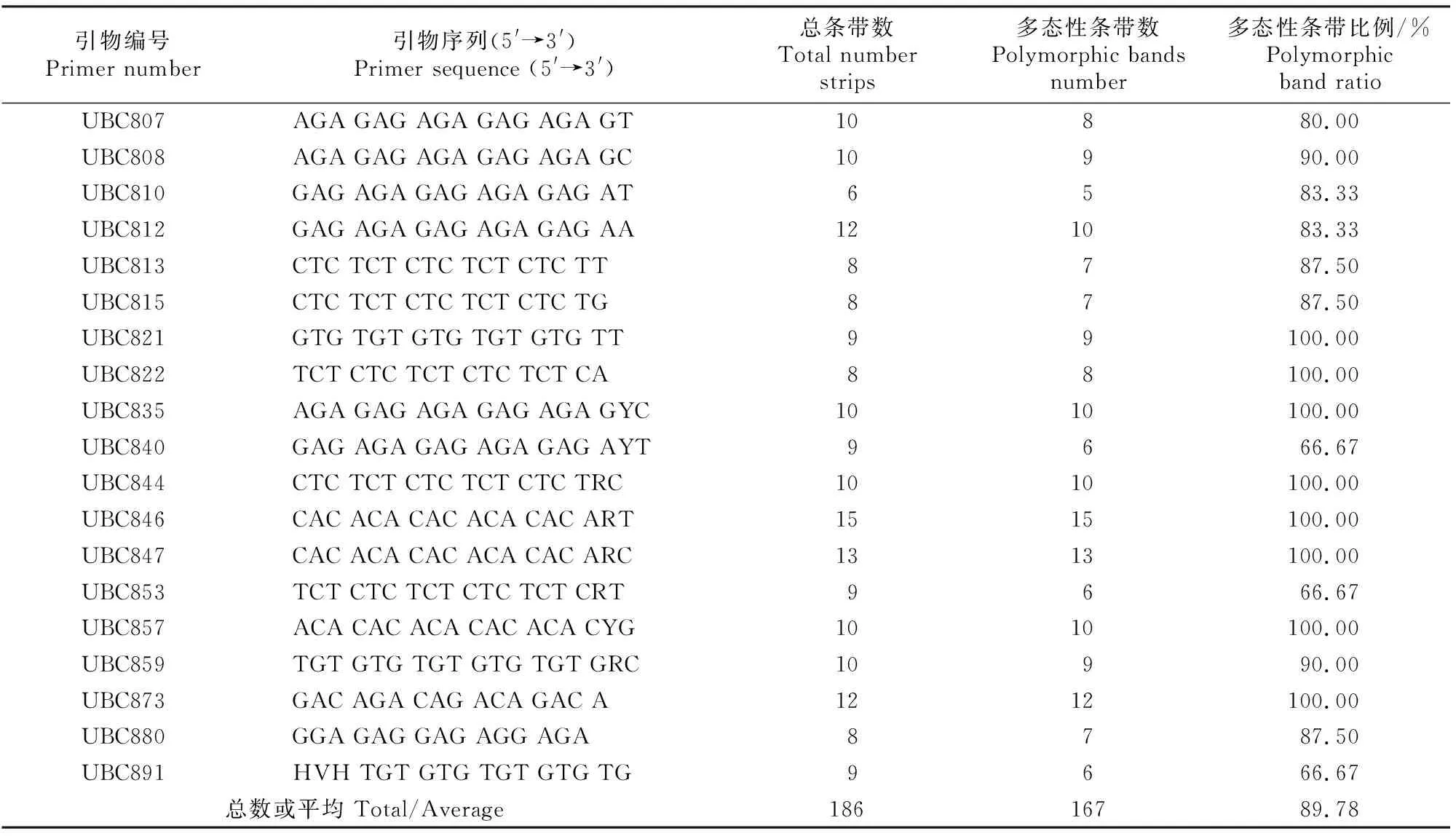
表2 62株米槁单株的ISSR扩增结果Table 2 ISSR amplification of 62 individuals of Cinnamomum migao

表3 8个米槁天然种群的遗传多样性Table 3 Genetic diversity of 8 Cinnamomum migao populations
2.2 米槁种群的遗传结构与分化
利用AMOVA对所有的ISSR标记数据按种群间、种群内两因素阶层进行分子方差分析,结果见表4。由表4可知,米槁总的遗传变异中有16.14%存在于种群间,83.86%存在于种群内(P<0.001),表明种群间和种群内遗传分化均极显著,该分析结果与2.1节分析结果相似。
根据Nei’s基因多样性指数(H)估算的米槁8个天然种群间的基因分化系数(Gst)为0.263 6,即表示种群间变异占总遗传变异的26.36%,种群内变异占比为73.64%。根据Shannon’s多样性指数(H′)估算的种群间遗传多样性所占比例为27.69%,即表示种群间变异占总遗传变异的27.69%,种群内变异占比72.31%。根据H′分析的种群间遗传多样性占比虽略高于根据H估算的Gst值,但两类指数估算的米槁种群遗传分化结果一致,即均以种内遗传变异为主。根据Gst计算出的米槁种群水平上的基因流(Nm)为1.396 8。
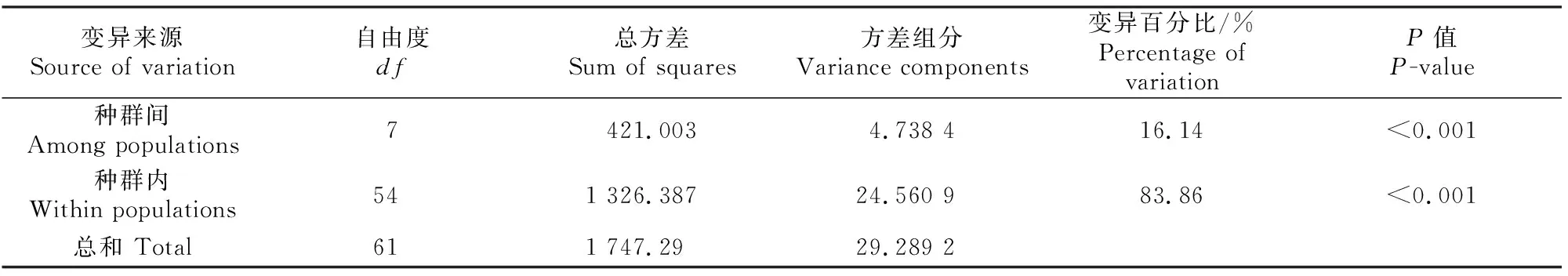
表4 8个米槁天然种群的分子方差分析Table 4 Analysis of molecular variance (AMOVA) for 8 Cinnamomum migao populations
2.3 米槁种群间的遗传距离和聚类结果
基因分化系数(Gst)只能评估种群间的分化程度,不能定量分析种群间的遗传分化,为了进一步分析种群之间的分化程度,利用POPGENE软件计算出种群间的遗传相似系数(I)和Nei’s遗传距离(D),结果见表5。由表5可知,I在0.785 6~0.954 6,这表明种群间的亲缘关系很近。8个米槁种群的D都不高,最大的为LD2和ZN两种群间,但也只有0.241 3,最小的为WM1和WM2两种群间,仅0.046 4。

表5 8个米槁天然种群的Nei’s遗传相似系数和遗传距离Table 5 Nei’s genetic identity and genetic distance among 8 Cinnamomum migao populations
注:表中右上部分为遗传相似系数(I),左下部分为遗传距离(D)。
Note:The data above diagonal are genetic identity (I),while the data below diagonal are the genetic distance (D).
根据种群间的遗传相似系数(I)和Nei’s遗传距离(D),使用NTSYSpcV2.10e软件采用UPGMA法构建了米槁种群的遗传关系聚类图,结果见图1。由图1可知,米槁种群间的遗传距离较小,在遗传距离0.08处,8个种群被划分为2类,即ZN单独为一类,其余7个种群为一类;在遗传距离0.07处,7个种群又被划分为2类,即LD1、LD2、WM1、WM2聚为一类,CH、LB、ZF聚为一类;在遗传距离0.05处,LB单独归为一类,CH、ZF聚为一类;在距离0.045处,LD1、LD2聚为一类,WM1、WM2聚为一类。

图1 基于ISSR标记的8个米槁天然种群的UPGMA聚类图Fig.1 UPGMA dendrogram for 8 natural populations of Cinnamomum migao based on ISSR markers
PCoA散点聚类图体现了个体间的遗传关系。由图2可见,62株米槁个体可明显分为2个谱系,谱系1为LD(LD1、LD2),谱系2包含ZN、ZF、CH、LB,而WM(WM1、WM2)兼具2个谱系的遗传信息,个体之间存在一定差异。

图2 米槁62株个体的PCoA散点图Fig.2 Principal coordinates analysis (PCoA) of 62 samples of Cinnamomum migao
3 讨论与结论
遗传多样性是物种长期进化的产物,也是生存和发展的前提,遗传变异大小决定了一个物种的进化潜力和抗逆境能力,遗传多样性越丰富,其对环境变化的适应能力越强,分布范围也就越广[17]。本试验采用ISSR标记对米槁8个天然种群的遗传多样性及遗传结构进行研究,从条带检测结果可以看出多态性条带比例为89.78%。在米槁种群水平上平均Na为1.572 6,Ne为1.390 9,H为0.223 3,H′为0.328 2, PPL为57.26%,米槁不同天然种群各遗传多样性指标均显示为物种水平>种群水平,说明米槁物种水平遗传多样性较为丰富,进一步证实了陈美兰等[18]对米槁果实大果木姜子遗传多样性的研究结论,即认为米槁在种群间的遗传多样性较低,而在种群内的分化较高,大部分变异存在于种群内,且种群水平的H指数(0.223 3)也与陈美兰等[18]的H指数(0.228 0)大致相当。在物种水平上,与樟科其他植物相比,米槁Shannon’s多样性指数为0.453 9,低于孙林[19]用微卫星SSR标记的天目木姜子(Litseaauriculata)(0.515 0),高于冷欣等[20]应用ISSR标记的红楠(Machilusthunbergii)(0.385)、慈秀芹[21]应用RAPD标记的思茅木姜子(Litseaszemaois)(0.264 2)、江香梅等[22]应用RAPD标记的闽楠(Phoebebournei)(0.368 8)和王中生等[23]标记的舟山新木姜子(Neolitseasericea)(0.282 0) 。米槁种群平均Shannon’s多样性指数H′(0.328 2)高于潘晓华[24]应用RAPD标记的樟树(Cinnamomumcamphora)(0.249 6)。可见,米槁遗传多样性属于中偏高水平,说明米槁的遗传多样性较丰富。
通过对8个米槁种群的ISSR数据进行AMOVA分析,表明米槁总的遗传变异中有16.14%存在于种群间,83.86%存在于种群内,种群间和种内遗传分化均极显著。樟科其他植物的研究结果表明:天目木姜子(Litseaauriculata)有28.36%的遗传变异存在于种群间,71.64%的遗传变异存在于种群内[19];思茅木姜子(Litseaszemaois)表现为27.01%的遗传变异存在于种群间,72.99%的遗传变异存在于种群内[25];舟山新木姜子(Neolitseasericea)的总遗传变异中种群间占36.46%,种群内占63.54%[23]。由此可见,米槁种群间的遗传变异比例较其他樟科植物的低,而种内遗传变异比例较其他樟科植物的高,进一步说明米槁遗传多样性较为丰富。遗传分化系数是判断遗传变异的主要来源,米槁基于Nei’s基因多样性指数的基因分化系数Gst为0.263 6,略低于基于Shannon’s多样性指数估计的分化系数0.276 9,但均说明米槁种群内的遗传多样性较种群间高,同时也说明由于地理隔离的存在,米槁种群间存在一定的遗传分化。
种群间遗传相似系数(I)和Nei’s遗传距离(D)指示8个米槁天然种群间的遗传变异程度较低。基因流(Nm)是影响种群内部和种群间遗传变异程度的重要因素,大的基因流能够阻止种群间的遗传分化,基因流大的物种,种群间的遗传分化小,基因流小的物种,种群间的遗传分化大[26]。当Nm>1时,说明种群间存在一定的基因流动[27]。本试验结果表明,米槁种群水平上的Nm为1.396 8>1,说明米槁种群间存在基因流动,种群间的遗传分化较小,该结果与本试验所得的种群间、种群内的遗传多样性和遗传分化系数的分析结果一致。米槁8个种群62株个体的PCoA散点聚类图显示,62株个体明显分为2个谱系,谱系1为LD的2个种群,谱系2包含ZN、ZF、CH、LB,WM的2个种群兼具2个谱系的遗传信息,这揭示了2个遗传谱系之间可能存在不对称的基因流。谱系2中6个种群个体分布在一个狭窄区域,6个种群个体各自紧密聚集在一起;而谱系1所有个体分布在一个较广泛的区域内,并且同一种群的个体都广泛分布在区域中,表明谱系1种群中的个体之间遗传距离也较大。UPGMA聚类分析结果基本支持上述结果,8个种群划分为3大类,ZN单独为一类,LD 2个种群和WM 2个种群聚为一类,CH、LB、ZF聚为一类;LD1与LD2、WM1与WM2、CH与ZF间遗传距离都较小,遗传相似度较高。聚类图显示地理距离近的种群聚在一起,反映出米槁的遗传变异与生境有一定的关系,从米槁种群的空间分布格局来看,种群遗传变异分布模式基本上与其地理生态格局一致,这与吴雪琴[28]对观光木(Micheliaodora)种群遗传多样性研究的结论一致。
