碱胁迫应答基因GsSnRK1.1与上游调控因子GsGRIK1互作功能分析
2018-07-10朱延明杜建英孙晓丽丁晓东殷奎德王子君朱娉慧
朱延明,杜建英,陈 超,于 洋,孙晓丽,丁晓东,殷奎德,王子君,朱娉慧
(1.东北农业大学植物基因工程实验室,哈尔滨 150030;2.黑龙江八一农垦大学生命科学技术学院,黑龙江 大庆 163319)
植物蔗糖非发酵-1-型相关蛋白激酶-1(Sucrose non-fermenting-1-related protein kinases-1,SnRK1)进化高度保守,是植物碳氮代谢主要调控因子。植物中SnRK1蛋白激酶在序列和功能上与酵母碳源调控蛋白激酶SNF1(Sucrose non-fermenting-1 kinase)和动物AMPK(AMP-activating protein kinase)蛋白激酶同源性较高。Alison等发现黑麦中属于SnRK1亚家族的蛋白激酶可互补SNF1在酵母中利用非葡糖碳源功能[1]。SnRK1是由α、β、γ组成的异源复合体,在植物激素ABA信号传导、能量和代谢调控等过程中起调控中枢作用[2]。研究证明,与SNF1和AMPK相比,植物中SnRK1功能多样性广泛。Ruslana等研究报道,SnRK1蛋白激酶广泛参与调控基因转录,MYBS类转录因子,在植物激素(ABA、细胞激动素、生长素和乙烯)信号途径中扮演关键角色,豌豆VfSnRK1在协调ABA和生长素之间关系中起重要作用,参与细胞激动素信号传导途经[3]。
关于SnRK1在耐盐或耐碱方面调控作用,在植物方面未见报道。酵母研究方面,当酵母中SNF1(SnRK1同源基因),或者其上游激酶(Sak1、Tos3和Elm1)被敲除时,酵母突变株系表现对盐和pH高度敏感[4-5],说明该激酶在酵母耐盐碱非生物胁迫中起关键作用,是本研究探索GsSnRK1.1和GsGRIK1互作行使耐碱功能理论基础;在植物方面,Grahame等研究表明,SnRK1直接调控多种非生物胁迫(盐胁迫、高温胁迫和干旱胁迫)相关基因表达[6]。拟南芥与盐和缺氧(Hypoxia)有关转录因子AtMYC2是SnRK1磷酸化底物,AtMYC2被SnRK1磷酸化后会经蛋白酶体途径降解,说明SnRK1在植物抗盐胁迫途径中起调控作用[7]。
进一步了解GsSnRK1.1上游激活因子,是揭示GsSnRK1.1蛋白激酶新功能关键。Sugden等提出植物SnRK1上游激活激酶存在[8],Wang等报道拟南芥GRIK1和GRIK2(分别称为SNAK2和SNAK1)是与酵母SAK1、TOS3、ELM1和哺乳动物LKB1和CaM⁃KKb有关同源基因[9]。每个GRIK蛋白均可使酵母三重突变体(sak1、tos3和elm1)在非葡萄糖培养基上正常生长[10-11]。结果表明,GRIKs是植物中SnRK1上游激活因子。为验证这一假说,Wei等研究发现拟南芥GRIK1和GRIK2可磷酸化激活SnRK1,而GRIK1(Geminivirus rep-interacting kinases1)是应答细胞外病毒侵害的蛋白激酶,说明植物中SnRK1上游因子具有功能多样性[12]。在非生物胁迫方面,GRIK1磷酸化激活SnRK1家族蛋白激酶响应盐胁迫,且在多重信号通路中起重要作用[13]。但在GRIK1与SnRK1是否参与植物抵御碱胁迫方面,尚无相关研究,本试验针对这一问题开展研究,进一步研究野生大豆GsGRIK1与GsSnRK1互作分子机制。
综上所述,为揭示碱胁迫应答基因GsSnRK1.1与上游调控因子GsGRIK1在野生大豆中互作功能,本研究克隆GsGRIK1和GsSnRK1.1,并通过组织染色和碱处理超量表达目的基因大豆发状根表型分析作功能验证,证明GsGRIK1和GsSnRK1.1具有显著耐碱功能;通过生物信息学软件NCBI和Clust⁃alX 2.0分析,表明GsGRIK1属于类钙调蛋白激酶;利用酵母突变体系统分析证明GsGRIK1和GsSnRK1.1存在互作关系;进一步采用Western blot生化分析证明,GsGRIK1通过钙离子依赖途径磷酸化激活GsSnRK1.1;通过组织染色分析GsGRIK1和GsSnRK1.1在碱胁迫下表达模式,发现其显著响应碱胁迫;通过发根农杆菌遗传转化对大豆毛状根表型分析表明,野生大豆中GRIK1与SnRK1参与植物抵御碱胁迫。为揭示GsSnRK1及其上游调控激酶功能和分子调控机制奠定理论基础。
1 材料与方法
1.1 材料
耐盐碱东北野生大豆(Glycine soja)Gs07256、栽培大豆(Glycine max L.Merr)东农50种子、大肠杆菌(Escherichia coli)DH5α、发根农杆菌(Ag.rhizo⁃genes)K599、酵母表达载体(PYX212和PYX242)以及植物表达载体pCAMBIA1302,均由东北农业大学植物基因工程实验室保存。酵母菌株Msy182(WT)、Msy1193(snf1 Martin)及 Msy923(sak1/tos3/eml1/snf1 Matin,his,ura/TRP/leu)均由哥伦比亚大学提供。胶回收和质粒小提试剂盒购自Transgene公司,引物合成及测序由哈尔滨英俊生物公司及金唯智生物科技有限公司完成。
1.2 方法
1.2.1 GsSnRK1.1与GsGRIK1基因克隆与序列分析
基因克隆:根据野生大豆GsSnRK1.1与Gs⁃GRIK1基因cDNA全长序列,利用Primer 5.0软件设计基因特异PCR引物(见表1)。以野生大豆cD⁃NA为模板,利用PrimeSTAR®HS DNA聚合酶PCR扩增获得目的基因全长CDS区[14-15]。根据PCR产物对照Marker位置回收目的条带。
序列分析:利用NCBI软件对GsSnRK1.1与GsGRIK1在线Blast序列比对,分析其基因结构。对野生大豆、栽培大豆和拟南芥中GsGRIK1同源基因,利用ClustalX 2.0作多重序列比对,参数设定为默认值[16-17]。
1.2.2酵母突变体表型试验
酵母表达载体构建:根据目的基因序列,设计目的基因及其突变体引物(见表1),利用OVER⁃LAP PCR方法扩增目的基因突变体序列,得到GsSnRK1.1-T179A、GsSnRK1.1-T179E、GsSnRK1.1-K49M及GsGRIK1-K146A基因片段。将GsSnRK1.1及其突变体酶切连接整合至酵母表达载体PYX212的Eco RⅠ和SalⅠ位点间,GsGRIK1及其突变体酶切连接至酵母表达载体PYX242的Eco RⅠ和SalⅠ位点间。
互补功能分析:利用TE-LiAC法,将含目的基因GsSnRK1.1及其突变体基因的重组载体、PYX212空载体(负对照)分别转化至酵母突变体Msy1193(snf1)中,并以PYX212-GsSnRK1.1-T179E作为阳性对照,转化菌株均涂布于单缺陷型酵母合成培养基(SD-Ura3)上,30℃恒温箱内倒置培养。挑选单菌落接种到约10 mL YPDA培养基中,于30℃恒温震荡培养24~36 h,收集菌体,将菌液用无菌水稀释至OD600=0.25,梯度(1/10、1/100、1/1 000)稀释。分别取1 μL Msy1193转化子菌液点样至不同碳源(葡萄糖、甘油、乙醇)单缺陷型酵母合成培养基(SD-Leu2)上,30℃恒温箱内倒置培养。每组3个生物学重复,拍照记录。
互作功能验证试验:将含目的基因GsSnRK1.1、GsGRIK1及其突变体基因重组载体共转化至Msy923(sak1/tos3/eml1/snf1)中,并以 PYX212/PYX242空载体为负对照,将两空载体转化至Msy182(WT)作为阳性对照,转化菌株均涂布于二缺陷型酵母合成培养基(SD-Leu2-Ura3)上,30℃恒温箱内倒置培养。挑选单菌落接种到约10 mL YPDA培养基中,于30℃恒温震荡培养24~36 h,收集菌体,将菌液用无菌水稀释至OD600=0.25,再梯度(1/10、1/100、1/1 000)稀释。分别取1 μL Msy923转化子菌液点样至不同碳源(葡萄糖、甘油、乙醇)和不同处理(9 mmol·L-1NaHCO3、1 mol·L-1NaCl、1 mol·L-1山梨醇、1 mol·L-1甘露醇)二缺陷型酵母合成培养基(SD-Leu2-Ura3)上,30℃恒温箱内倒置培养。每组3个生物学重复,拍照记录。
1.2.3目的基因蛋白提取及生化分析
重组酵母细胞接种于50 mL YPD液体培养基中培养,200 r·min-1,30℃震荡培养36 h,达OD600≈3。分别取20 mL菌液低温离心收集菌体。向离心管中加入1 mL裂解缓冲液(含1 μg蛋白酶抑制剂),加入一定量过夜酸洗玻璃珠,混匀后于旋涡振荡仪上振荡2 min,冰浴30 s,重复3次,4℃,12 000 r·min-1离心10 min,收集上清液。将上清液沸水浴5 min,-20℃保存待用。
Western blot生化分析:冰上冻融蛋白提取液,取一定体积样本至新Eppengorf管中,加入8 μL 5×Protein Loading Buffer,沸水浴变性5 min,将样本置于冰上并迅速上样。按常规方法作SDS聚丙烯酰胺凝胶电泳,试验样本每道点样5 μL,80 V电压电泳2 h,然后将凝胶电泳样品转印至ECL膜上,70 V电压转膜2 h。采用脱脂奶粉封闭液封闭蛋白膜1 h,使用GsSnRK1.1特异磷酸化位点Thr179对应的第一抗体Thr172(Phospho-AMPKα)孵育1 h,TBST洗膜3次,10 min·次-1。相对应的第二抗体孵育1 h,TBST清洗3次,10 min·次-1。加DAB显色后用Bandscan显像分析[18]。
1.2.4大豆子叶发状根诱导培养
利用赵晓雯等方法[19],将氯气灭菌大豆东农50种子5 d萌发培养后,重组发根农杆菌K599切割侵染大豆子叶,震荡侵染30 min,将侵染后子叶节转移至共培养基(1/10 B5培养基)中28℃暗培养3~4 d,再转移至含200 mg·L-1阿莫西林-克拉维酸钾的液体B5培养基中冲洗4~5次,除菌,随后转移至含200 mg·L-1阿莫西林-克拉维酸钾的生根B5培养基上,20℃光照培养2周,待子叶叶柄处长出发状根,用于后续试验。
1.2.5GsSnRK1.1与GsGRIK1基因碱胁迫表达模式分析
根据GsSnRK1.1与GsGRIK1基因组序列,设计启动子克隆引物(见表1),以野生大豆基因组DNA为模板,使用TaKaRa的Tks Gflex™DNA Polymerase作PCR扩增,整合至植物表达载体pCAMBIA3301上。将测序正确GsSnRK1.1与GsGRIK1启动子植物表达载体转化至发根农杆菌K599,侵染大豆子叶。对整合目的基因启动子的3周龄大豆发状根碱胁迫处理,碱胁迫处理溶液为含50 mmol·L-1NaH⁃CO3的B5溶液[20],处理时间段为0、6和12 h,利用GUS组织化学染色法检测处理后大豆发状根[21],3次生物学重复,拍照记录。
1.2.6GsSnRK1.1与GsGRIK1基因超量表达大豆发状根碱胁迫表型分析
根据GsSnRK1.1与GsGRIK1基因序列设计PCR扩增引物,GsSnRK1.1-SmaI-FW和GsSnRK1.1-SmaI-RV,GsGRIK1-SmaI-FW和GsGRIK1-SmaIRV(见表1),PCR扩增目的基因,将GsSnRK1.1与GsGRIK1通过酶切分步连接同一植物表达载体pCAMBIA1302。将测序正确的GsSnRK1.1与GsGRIK1基因植物表达载体转化至发根农杆菌K599,使用K599重组菌株侵染大豆子叶。取长势一致不同转基因2周龄大豆发状根,分别置于含30 mmol·L-1NaHCO3的B5培养基上,盐胁迫和碱胁迫处理,继续生根培养15~20 d。每天观察其生长状态,当各样品间表型差异明显时拍照,统计其根长及发状根鲜重并统计分析[22]。试验3次生物学重复。以提取的处理样品DNA为模板,运用基因特异性引物为模板检测处理样品,经检测均为阳性植株。

表1 所用引物Table 1 List of primers
2 结果与分析
2.1 GsSnRK1.1与GsGRIK1基因克隆与序列分析
根据GsSnRK1.1与GsGRIK1基因序列,设计基因特异引物,以野生大豆cDNA为模板,以PCR扩增方法克隆基因。通过测序结果分析,GsSnRK1.1基因长度为1 548 bp,编码516个氨基酸(见图1),受上游激酶调控的特异性磷酸化位点是第179位的苏氨酸(Thr,T),其激活位点为第49位的赖氨酸(Lys,K);GsGRIK1基因长度为1 215 bp,编码415个氨基酸(见图2),其激活位点为第146位的赖氨酸(Lys,K)。 通过NCBI在线Blast分析目的基因,结果表明GsSnRK1.1含有约255个氨基酸AMP⁃KA结合区域,位于第15~270位(见图3A)。AMP⁃KA结构域是广泛存在于植物SnRK1家族蛋白中的一类高度保守结构域。通过ClustalX序列比对发现GsGRIK1内所含CaMKK结构域高度保守(见图4)。尤其是通过序列分析首次证明GsGRIK1属于类钙调蛋白激酶,含有约250个氨基酸CaMKK特异性结构域,位于第130~375位(见图3B),通过钙离子依赖途径调控下游蛋白,本试验结果为国内外首次报道。

图1 GsSnRK1.1基因克隆Fig.1 Cloning of GsSnRK1.1 gene

图2 GsGRIK1基因克隆Fig.2 Cloning of GsGRIK1 gene

图3 GsSnRK 1.1与GsGRIK 1蛋白结构分析Fig.3 Structural analysis of GsSnRK 1.1 and GsGRIK 1 protein

图4 GRIK1家族序列分析Fig.4 GRIK1 family sequence analysis
2.2 GsSnRK1.1与SNF1功能互补分析
为揭示能否利用酵母突变株系统筛选GsSnRK1.1上游调控因子,利用酵母突变体MSY1193(snf1)探究GsSnRK1.1是否可互补酵母中同源基因SNF1功能。因为酵母中同源基因SNF1可调控酵母利用非葡糖碳源,使酵母在低葡萄糖或非葡萄糖条件下正常生长。由此,本研究将GsSnRK1.1及其突变体转入MSY1193(snf1)中,将液体培养基活化的阳性转化子在不同碳源单缺培养基(SD-Ura3)上作表型分析。结果表明,仅含GsSnRK1.1和GsSnRK1.1-T179E(阳性对照)的酵母细胞可在甘油和乙醇非葡糖碳源培养基上正常生长,而含GsSnRK1.1-T179A、GsSnRK1.1-K49M和空载体的阴性对照无法在非葡糖碳源培养基上正常生长(见图5),说明野生大豆GsSnRK1.1可互补酵母中SNF1功能,酵母突变株系统可用于下一步研究。

图5 GsSnRK1.1与SNF1功能互补分析Fig.5 Functional complementation assay of GsSnRK1.1 and SNF1
2.3 酵母中GsSnRK1.1与GsGRIK1互作表型分析
本研究利用酵母四重突变株MSY923(snf1/tos3/eml1/snf1),验证GsGRIK1是否激活GsSnRK1.1,使其行使调控酵母利用非葡糖碳源功能。结果表明,仅含GsSnRK1.1和GsGRIK1酵母细胞以及含GsSnRK1.1-T179E和GsGRIK1-K146A的阳性转化子,可在甘油和乙醇的非葡糖碳源培养基上正常生长,而其他组合酵母细胞无法在非葡糖碳源培养基上正常生长(见图6),说明GsGRIK1可激活GsS⁃nRK1.1,使其行使调控酵母利用非葡糖碳源功能。

图6 酵母中GsSnRK1.1与GsGRIK1互作表型分析Fig.6 Interaction phenotype analysis of GsSnRK1.1 and GsGRIK1 in yeast
为进一步验证GsSnRK1.1与GsGRIK1能否提高酵母耐逆境胁迫能力,将含有共转化质粒的酵母细胞转移到不同胁迫处理培养基上,验证非生物胁迫条件下GsGRIK1是否激活GsSnRK1.1,使其提高酵母耐逆境胁迫能力。发现仅含GsS⁃nRK1.1与GsGRIK1酵母细胞及含有GsSnRK1.1-T179E与GsGRIK1-K146A的阳性转化子可在盐碱胁迫以及模拟干旱胁迫培养基上正常生长,其他组合酵母细胞无法在胁迫处理培养基上正常生长(见图7)。
结果说明,GsGRIK1调控GsSnRK1.1,使其行使提高酵母耐逆境胁迫功能。GsGRIK1与GsS⁃nRK1.1相互作用,能否提高植物耐逆境胁迫能力,需进一步验证。

图7 酵母中GsSnRK1.1与GsGRIK1多种胁迫下表型分析Fig.7 Phenotype analysis of GsSnRK1.1 and GsGRIK1 in yeast under some stresses
2.4 GsGRIK1调控GsSnRK1.1作用机制分析
通过序列分析发现,GsSnRK1.1第179位苏氨酸是GsSnRK1.1激活位点,为探究GsGRIK1调控GsSnRK1.1作用方式,本研究采用检测Thr179磷酸化的抗体Thr172(Phospho-AMPKα)作Western blot分析。结果表明,与未处理组相比,碱胁迫处理后提取蛋白样品GsSnRK1.1磷酸化位点Thr179磷酸化程度明显增加(见图8A),说明GsGRIK1通过磷酸化GsSnRK1.1调控位点Thr179,激活蛋白激酶GsSnRK1.1激酶活性。
分析GsGRIK1序列,发现GsGRIK1可能是一类类钙调蛋白激酶。为进一步了解GsGRIK1调控GsSnRK1.1分子机制,本研究对酵母阳性转化子作碱胁迫(20 mmol·L-1NaHCO3,pH 8.5)处理和 Ca2+参与碱胁迫(20 mmol·L-1NaHCO3+20 mmol L-1CaCl2,pH 8.5)处理,作生化分析。发现有Ca2+参与的碱胁迫处理后GsSnRK1.1的Thr179位点磷酸化程度显著增加(见图8B)。结果表明,GsGRIK1上游调控因子GsGRIK1通过钙离子依赖途径磷酸化GsS⁃nRK1.1调控位点Thr179,调控蛋白激酶GsS⁃nRK1.1激酶活性。
2.5 GsSnRK1.1与GsGRIK1基因碱胁迫表达模式分析
为验证GsSnRK1.1与GsGRIK1是否为碱胁迫应答基因,本研究采用农杆菌介导法用K599侵染大豆子叶,获得分别由GsSnRK1.1与GsGRIK1启动子启动GUS基因表达的大豆发状根,碱胁迫(50 mmol·L-1NaHCO3,pH 8.5)处理后,采用 GUS组织化学染色法作表达模式分析。结果如图9所示,碱胁迫处理的两组样品GUS基因表达量均有表达,未处理染色样品未表达。由此表明,GsSnRK1.1与GsGRIK1可响应碱胁迫。
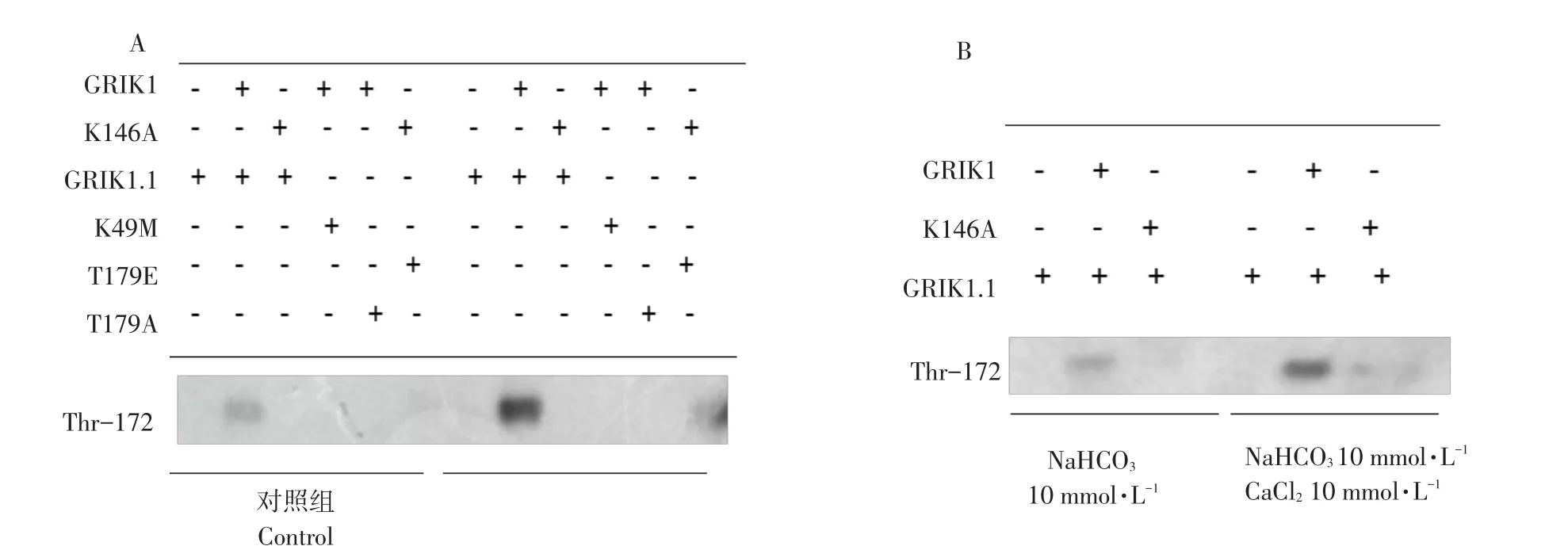
图8 GsSnRK1.1与GsGRIK1酵母中互作生化分析Fig.8 Biochemical analysis of the GsGRIK1 interaction with GsSnRK1.1 in yeast

图9 GsSnRK1.1与GsGRIK1基因碱胁迫下表达模式分析Fig.9 GsSnRK1.1 and GsGRIK1 gene expression pattern analysis under alkali stress
2.6 GsSnRK1.1与GsGRIK1基因超量表达大豆发状根盐碱胁迫表型分析
为进一步阐明野生大豆GsSnRK1.1与GsGRIK1互作是否参与植物碱胁迫通路,通过碱胁迫处理超量表达GsSnRK1.1和GsGRIK1大豆毛状根15 d,作功能表型鉴定及根长根重分析。发现超量表达GsSnRK1.1大豆发状根根长与对照组相比,正常生长条件下根长较短,但在碱胁迫处理条件下根长较长,说明GsSnRK1.1参与植物抵御碱胁迫信号通路;与对照组野生型相比,共表达GsSnRK1.1和GsGRIK1大豆发状根生长状态与野生型一致,但发状根数量较多;在30 mmol·L-1NaHCO3的B5培养基上,共表达GsSnRK1.1和GsGRIK1大豆发状根与其他株系相比生长状态最好,说明GsSnRK1.1和GsGRIK1互作显著提高大豆发状根对碱胁迫耐受性(见图10A);而共表达GsSnRK1.1和GsGRIK1-K146A与过表达GsSnRK1.1大豆发状根生长状态一致,且表型结果与图10B、C根长根重分析结果一致。
以处理样品中提取DNA为模板,运用基因特异性引物为模板检测处理样品,经检测均为阳性植株(见图11)。表明GsSnRK1.1具有提高植物耐碱胁迫能力,受上游激活因子GsGRIK1调控,参与植物抵御碱胁迫信号通路。此结果可填补国内外有关GsSnRK1.1和GsGRIK1互作提高植物对碱胁迫耐受性研究的空白。

图10 GsSnRK1.1与GsGRIK1基因超量表达大豆发状根碱胁迫表型Fig.10 Phenotype of GsSnRK1.1 and GsGRIK1 gene overexpression of soybean hair root under alkaline stress

图11 GsSnRK1.1与GsGRIK1基因超量表达大豆发状根PCR检测Fig.11 PCR identification of GsSnRK1.1 and GsGRIK1 gene overexpression of soybean hair root
3 讨论与结论
3.1 关于SnRK1耐盐或耐碱功能
植物中SnRK1在能源信号传导中起重要作用,且SnRK1可逆蛋白磷酸化是细胞信号响应环境变化重要机制之一。耐盐方面,SnRK1蛋白激酶可参与各种激素信号中枢传递,通过植物细胞信号传导因子及调节机制使植物适应多样环境,如:盐和缺氧(Hypoxia)胁迫[4]。SnRK1蛋白激酶在碱胁迫信号传导中功能和调控机制未见报道。本研究通过表达模式分析、酵母突变株表型分析发现,GsSnRK1.1可积极响应碱胁迫。碱胁迫处理大豆发状根发现,过量表达GsSnRK1.1大豆发状根根长显著高于对照组,说明GsSnRK1.1可正向调控碱胁迫信号通路,提高植物耐碱性,GsSnRK1.1是功能显著耐碱关键调控基因,可用于改良豆科植物耐盐碱基因。正常条件下,过量表达GsSnRK1.1大豆发状根根长较野生型短。Elena等研究发现,植物中SnRK1在胁迫和能源信号传导中起调控中枢作用,但在正常条件下,过量表达AtSnRK1.1拟南芥植株比野生型植株矮小;黑暗和渗透胁迫处理样品中,过量表达AtSnRK1.1拟南芥植株生长状况却明显优于野生型植株。说明过量表达SnRK1可提高植株抵抗盐碱和干旱环境能力[23]。
3.2 关于GsSnRK1.1与上游调控因子GsGRIK1互作功能
当植物受病原菌侵害时,AtGRIK1(Geminivi⁃rus rep-interacting kinases 1)蛋白激酶磷酸化激活AtSnRK1,一方面通过调节植物新陈代谢相关基因,提高植物细胞对病毒抵御能力;另一方面激活被侵染细胞凋亡相关基因,加速细胞凋亡,避免其他组织细胞感染病原菌[24]。但此前关于植物GRIK1调控SnRK1相关功能多限于参与抵御生物胁迫,在非生物胁迫方面少有报道。植物中AtGRIK1磷酸化激活AtSnRK1,可提高拟南芥对盐胁迫抗性,上游调控因子GsGRIK1作用于GsSnRK1.1能否提高植物耐碱性未见报道。本研究中,通过酵母表型分析和生化分析发现,GsGRIK1可激活GsSnRK1.1提高酵母突变株耐盐碱胁迫能力;通过表达模式分析发现,GsGRIK1和GsSnRK1.1参与植物响应碱胁迫;且盐碱胁迫处理转基因大豆发状根,结果表明,在GsGRIK1与GsSnRK1.1共同作用下,可提高大豆发状根对碱胁迫耐受性,说明野生大豆GsGRIK1通过激活调控GsSnRK1.1,参与植物响应碱胁迫,提高植物耐碱性,通过Western blot生化分析,发现GsGRIK1通过钙离子依赖途径磷酸化激活GsS⁃nRK1.1,初步明确GsGRIK1与GsSnRK1.1作用方式与机理。相关研究仅限于其磷酸化作用,Gs⁃GRIK1活性调节未见报道。本研究可为GsGRIK1与GsSnRK1.1参与具体分子通路与作用机制研究奠定理论基础。后续将深入研究GsGRIK1调控GsS⁃nRK1.1参与的具体分子通路,及其在野生大豆中相互作用生理生化机制。
