基于SNP标记的小麦高通量身份鉴定模式
2018-06-13刘丽华庞斌双刘阳娜李宏博赵昌平
刘丽华,庞斌双,刘阳娜,李宏博,王 娜,王 拯,赵昌平
(北京市农林科学院杂交小麦工程技术研究中心,杂交小麦分子遗传北京市重点实验室,北京 100097)
传统的品种鉴定主要依靠田间表型鉴定,该方法易受环境影响,费时费力,难以鉴定亲缘关系,无法保证检测的准确性和时效性。以SSR和SNP标记为代表的DNA指纹检测技术因具备共显性、高多态性、在基因组上均匀分布、高准确性、较好的重复性、较高通量、易实现自动化等优点,可通过构建标准样品的DNA指纹数据库,快速有效地实现品种保护和管理。这两种标记已相继被国际植物新品种保护联盟(International Union for the Protection of New Varieties of Plants,UPOV)[1-2]、国际种子联盟(International Seed Federation,ISF)和国际种子检验协会(International Safe Transit Association,ISTA)推荐作为作物品种鉴定和指纹数据库构建的优选标记。目前,我国已建立了小麦[3]、玉米[4]、水稻[5]等作物的SSR标记品种鉴定标准,为我国农作物品种管理提供了有力的技术支撑。在实际使用过程中,SSR标记具有不易实现数据整合和通量低等缺陷。新发展起来的SNP标记为二等位基因,数据统计简单,同时,国内外大公司相继推出了各种SNP高通量检测平台,能很好地弥补SSR标记的技术缺陷。迄今,SNP标记已被初步用于玉米[6-7]、水稻[8]、大豆[9]、大麦[10-11]、棉花[12]等作物的品种鉴定研究中,应用前景广泛。
现阶段高通量的 SNP 分型方法主要有样本高通量和位点高通量两种平台,不同平台各有优劣。位点高通量平台主要是基于芯片技术的平台,如Illumina公司开发的90K iSelect assay SNP芯片[13]以及中国农科院发布的660K小麦基因组特异SNP芯片,一次实验即可获取几万甚至几十万个SNP位点的数据,适于少量样本大量位点的检测;样本高通量主要有LGC公司推出的KASP(Kompetitive Allele Specific PCR,竞争性等位基因特异性PCR)技术,可在广泛的基因组DNA样品中,对SNPs进行精准的双等位基因判断,具有高通量、高质量、低成本、灵活、快速等优点,适于大量样本少量位点的检测。在实际品种鉴定过程中,既要考虑检测样本量大、高度近似品种少等情况,又要兼顾准确、灵活、经济、快速和通量高等需求,因此,如何利用两种平台研发一种适于小麦高通量身份鉴定的模式显得尤为重要。本研究以380个小麦种质材料为研究对象,利用小麦Illumina 90K SNP芯片进行评价,以期筛选出适合小麦高通量身份(KASP技术)鉴定的最少SNP位点组合,探索小麦品种高通量身份鉴定模式,为小麦品种高通量身份鉴定技术体系的建立和指纹数据库的构建奠定基础。
1 材料与方法
1.1 试验材料
用于候选SNP标记筛选的小麦材料共380个,包括核心种质、引进品种、核心不育系、核心恢复系以及审定品种372个、近等基因系2个、杂交小麦及其双亲3个、DH系样品1个(区分纯合SNP位点的内参)、小麦品种中国春1个(小麦全基因组测序标准参照序列),1个品种的第二次重复(重复率内参)。随机选取95个材料用于核心SNP标记在芯片和KASP平台的验证。
1.2 DNA提取
采用CTAB法[14]提取380个材料的基因组DNA,用TE溶解保存。DNA的质量和浓度用紫外分光光度计和琼脂糖凝胶电泳进行测定,要求DNA样本浓度在50 ng·μL-1以上,OD260/280在1.7~2.1之间,DNA总量大于2 μg。
1.3 基于芯片平台的SNP基因分型
按照Illumina公司提供的基于光纤微珠芯片Infinium技术的操作流程,利用Illumina公司的wheat 90K iSelect Bead Chips全基因组芯片(SNP位点数目为81 587个)对380个样本进行分析。在获得原始数据后将其直接导入Genome Studio软件进行基因分型,以获得每个样本每个位点的SNP基因分型数据。
1.4 基于KASP平台的SNP基因分型
将筛选出的SNP标记侧翼序列信息提交LGC公司(Laboratory of the Government Chemist,Hoddeston,UK)进行KASP引物的设计和合成;PCR体系约为1 μL,包括烘干的DNA(1.5 μL浓度为10 ng·μL-1的DNA ),KASP Mix 0.5 μL(LGC Genomics,Hoddeston,UK),0.014 μL KASP Assay Mix和0.5 μL ddH2O。PCR反应条件为:94 ℃ 15 min;94 ℃ 20 s,61~55 ℃ 60 s(每个循环降低0.6 ℃),10个循环;94 ℃ 20 s,55 ℃ 60 s,26~36个循环。PCR结束后,使用荧光微孔板检测仪进行终点法读数检测结果,Klaster caller软件分析待测样品的SNP位点基因型。
1.5 统计分析
采用杜邦先锋公司提供的Uniqueness软件进行统计分析(基于组合最优化算法),获得小麦品种鉴定最少的SNP位点组合。采用Powermarker V3.25软件[15],基于Nei’s(1983)遗传距离的UPGMA(Unweighted pair-group method with arithmetic means)方法绘制聚类图。
2 结果与分析
2.1 候选SNP位点的筛选结果
利用小麦全基因组SNP芯片(Illumina 90K)对380个小麦材料进行全基因组扫描分析,根据以下原则筛选候选SNP标记:(1)每个SNP位点的得分值(GeneTrain Score)大于0.6;(2)以重复试验样品为材料,以“Edit replicates”为条件进行重复性验证,删除不具备重复性的位点;(3)以杂交种及其双亲为材料,用Edit Parental Relationships进行遗传规律验证,删除亲本与其对应的杂交种基因型不符合遗传规律的位点;(4)以DH系为材料,进行位点可靠性的验证,删除不可靠的位点;(5)删除整体信号弱、数据缺失率大于5%和杂合率较高的位点;(6)根据遗传图谱位置,删除重复或者连锁的位点;(7)筛选单拷贝位点(三种基因型cluster具备明显界限且同时具有AA和BB基因型的位点);(8)筛选MAF值(Minor Allele Frequency,最小等位变异频率)大于0.2的位点。从81 587个SNP标记中最终筛选出高质量、高分辨率、单拷贝、均匀分布于21条染色体的候选SNP标记384个。380个材料在理想SNP位点上的基因分型图(图1)中,195个材料表现为AA基因型,184个材料表现为BB基因型,1个材料(杂交种)表现为AB基因型。剔除杂交种及重复材料后剩余378个常规材料,分析这些材料在384个SNP位点的基因型,结果表明,384个SNP位点能区分其中的376个材料,未区分的两个材料(农大211和农大212)为近等基因系,仅在粒色上有差异。
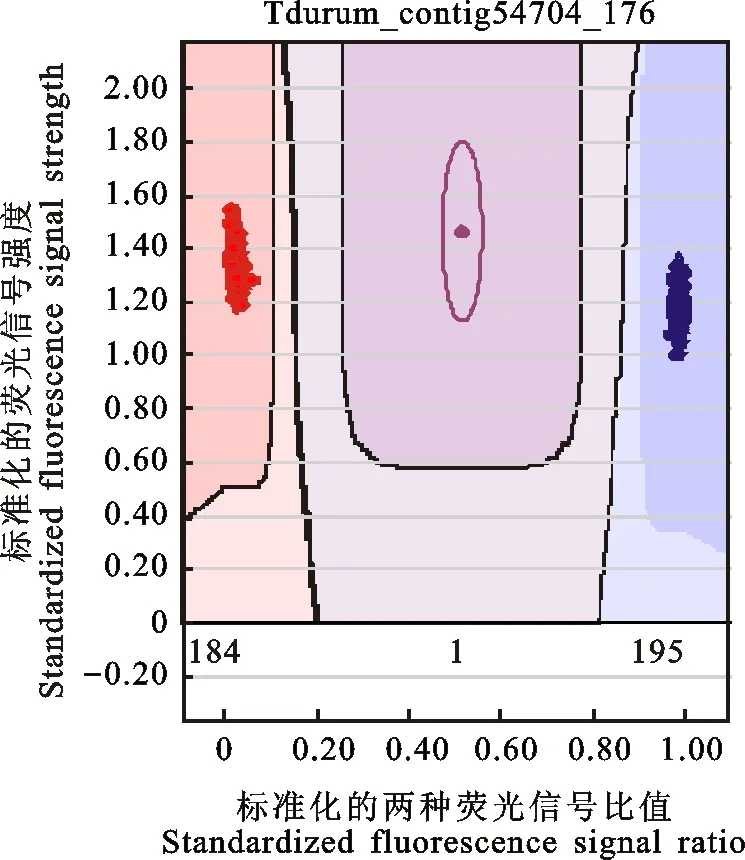
蓝色和红色表示纯合基因型,紫色表示杂合基因型。
Blue and red indicate homozygous genotypes; purple represents heterozygous genotype.
图1380个材料在理想SNP位点上的基因分型图
Fig.1Genotypingof380wheatmaterialsattheperfectgenotypingSNPlocus
2.2 品种鉴定最少SNP位点组合的选择结果
基于378个常规材料384个SNP位点的基因型数据,采用杜邦先锋公司提供的Uniqueness软件的组合最优化算法分析,获得了小麦材料鉴定最少SNP位点组合一套,包括14个SNP位点。图2显示SNP位点的区分能力曲线图,随着SNP位点数的增加,区分材料数不断增加,当位点数达到14个时,区分材料数达到饱和,即14个SNP标记与384个SNP标记的区分能力相同,可区分378个材料中的376个材料,未区分的两个材料仍然是农大211和农大212。基于378个材料14个SNP位点的基因型数据,绘制了378个材料的聚类图(图3),可以很清晰地看见,14个位点仅不能区分农大211和农大212两个材料,其他材料均能区分,区分能力达99.5%。14个SNP标记的组合方式有多种,根据SNP基因分型质量(Genetrain>0.6)、MAF值(MAF>0.2)和染色体分布较均匀等指标,确定了一套小麦材料身份鉴定最佳的14个SNP位点组合(表1)。其中,1B、2A、3B、3D、4A、4B、6A、6B、6D和7B染色体各1个SNP位点,7A和5D染色体各2个SNP位点;最小等位变异频率变化范围为0.37~0.50,平均为0.46;平均基因多样性和多态性信息指数(PIC,Polymorphism Information Content)值分别为0.49和0.37。
2.3 SNP位点在KASP平台上的验证
获取14个SNP位点的侧翼序列,依据KASP原理设计引物,并在KASP平台上进行基因分型。从380个材料中选取95个样品,加上1个阴性对照(H2O),采用设计好的14对SNP引物进行基因分型。结果显示,14个位点均能在KASP平台上获得明确、三型独立的基因分型结果,且与芯片平台结果一致。图4为SNP3位点的芯片和KASP技术分型图,66个样品表现为AA基因型,29个样品表现为BB基因型。
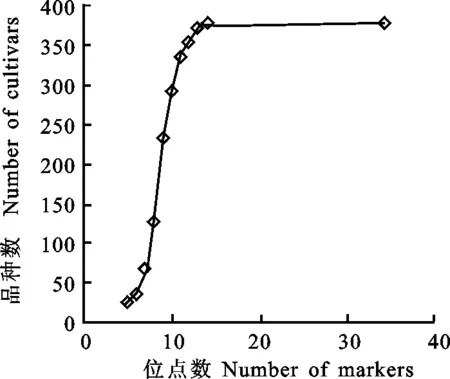
图2 SNP位点组合区分378个品种的能力Fig.2 Ability of SNP loci to distinguish 378 materials

此图旨在直观阐述材料之间的离散程度,至于哪些材料之间有差异或者材料之间差异多少,并不是本文需要表达的观点,加之材料数太多,所以无法也无需在图上标注出材料名称或代号。△△:两个近等基因系。
The purpose of the diagram is to intuitively explain the discrete degree between materials,as for which materials are different or the number of differences between materials is not the point that needs to be expressed in this article. In addition,due to the large number of materials,the name or code of materials can not be marked on the map.△△:Two NILs(near isogenic lines).
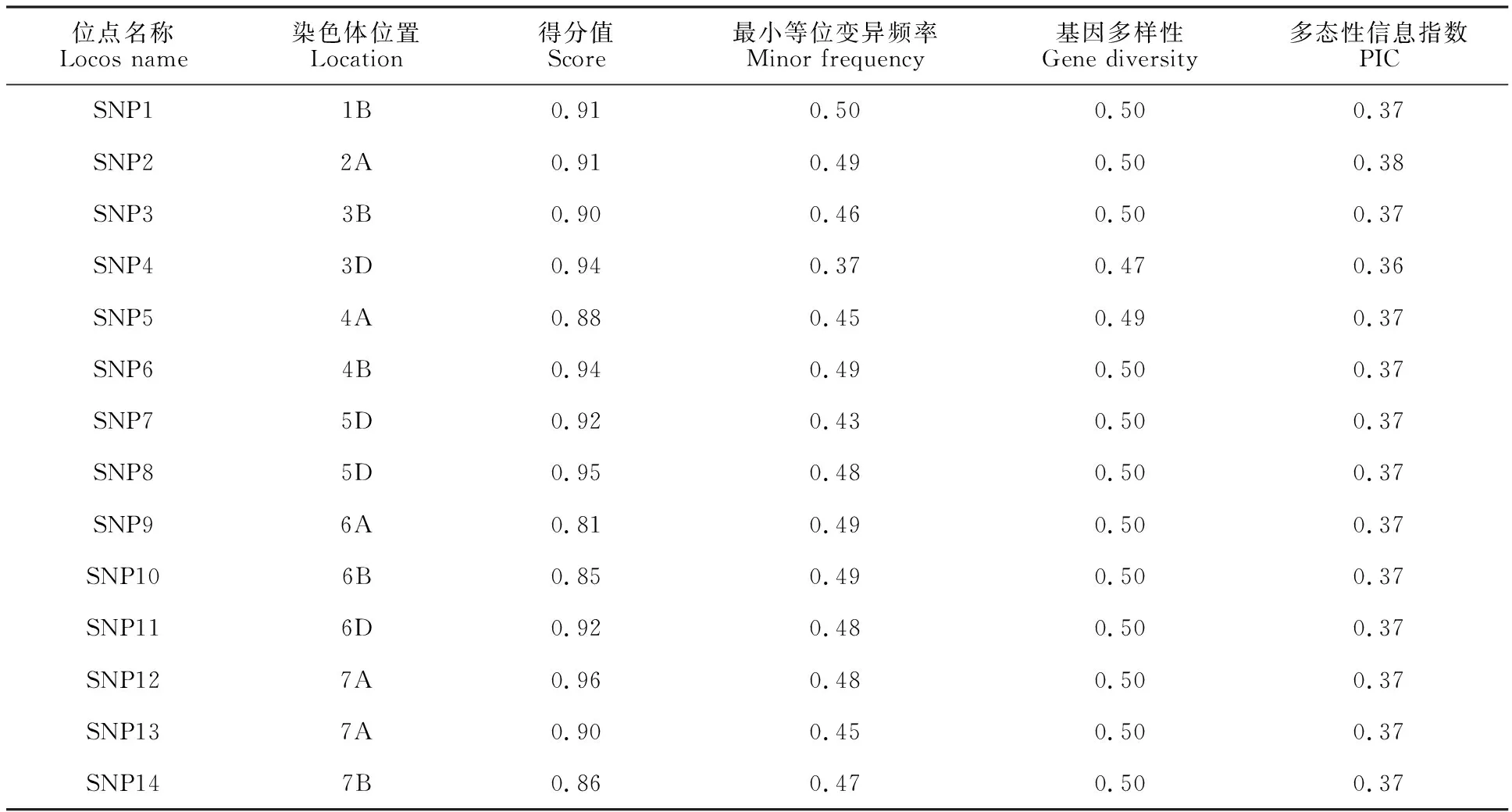
图3 基于UPGMA法的Nei’s遗传距离聚类图
2.4 小麦品种SNP高通量身份鉴定模式
基于以上结果获得的一套小麦品种鉴定最少SNP位点组合(包含14个SNP标记),不仅能将近等基因系以外的材料区分开,而且还兼容芯片和KASP两种平台,说明在实际材料鉴定过程中用少量的SNP位点在KASP平台上快速区分大部分材料是可行的。考虑实际材料鉴定过程中检测样品量大、高度近似材料少等情况,权衡准确、经济、灵活、快速、通量高等需求,材料SNP高通量身份鉴定可采取“核心位点+扩展位点”模式进行。首先启用核心位点,核心位点仅需几十个至上百个位点,在LGC-KASP平台(样本高通量检测平台)上进行,一人一天即可完成上千份样品的基因分型;当少数几对材料用核心位点不能区分时,可启用扩展位点,扩展位点可达几万甚至几十万个,在芯片平台(位点高通量检测平台)上进行,一次实验即可完成几(十)万个位点的基因分型。

蓝色和红色表示纯合基因型,紫色表示杂合基因型。
Blue and red indicate homozygous genotypes; purple represents heterozygous genotype.
图495个样品在芯片平台(A)和KASP平台(B)上的基因分型结果
Fig.4Genotypingresultsof95sampleson90KSNParray(A)andKASPplatform(B)
3 讨 论
单拷贝位点、最小等位变异频率高、无连锁、分布较均匀等是保证品种鉴定准确性的重要因素。本研究通过利用380个材料扫描90K芯片,从81 587个SNP标记中仅筛选出384个高质量、高分辨率、单拷贝、均匀分布于21条染色体的候选SNP标记,选出率只有0.47%,明显偏低,说明90K芯片存在一些不足,如大多数SNP标记为多拷贝位点难以准确分型,多态性较低,连锁或重复标记多,有效标记数量少,D染色体基因组上的标记太少,分布不均匀等。为满足规模化的准确检测与指纹数据库构建的需要,还需采用中国农科院释放的660K芯片进行大量SNP标记的筛选工作,以达到理想的品种鉴定效果。
探索一种准确、经济、灵活、快速、高通量的小麦品种SNP身份鉴定模式是建立高通量鉴定技术体系和指纹数据库构建的前提,本研究提出的材料身份鉴定模式“核心位点+扩展位点”很明显可实现以上目的,但核心位点和扩展位点分别选取多少合适,还需进一步研究与探讨。杜邦先锋公司的研究表明,其选用的16个SNP位点可将公司99%的玉米自交系区分开,不能区分的少量自交系有高度系谱相关性,即使再增加SNPs的数目也只有很小的效果,权衡成本、准确性、高通量等因素,目前仅采取16个SNP位点用于玉米自交系的鉴定。本研究结果显示在已知的378个测试材料中,仅需14个SNP位点即可区分99.5%的材料,与杜邦先锋公司的研究结果比较一致。但从品种鉴定对象来说,杜邦先锋公司的鉴定对象是有限的已知品种,而我国的鉴定对象是无限的未知品种,选择的14个SNP位点作为核心位点是否已经足够,还需进行大量审定标准样品的验证,以确定合适的核心位点数目。
参考文献:
[1] International Union for the Protection of New Varieties of Plants,UPOV.Guidelines for DNA-profiling:Molecular marker selection and database construction “(BMT guidelines”)[M].Geneva,Switzerland:UPOV,2007.
[2] International Union for the Protection of New Varieties of Plants,UPOV.Guidelines for DNA-profiling:Molecular marker selection and database construction “(BMT guidelines”)[M].Geneva,Switzerland:UPOV,2010.
[3] 中华人民共和国农业部.NY/T2859-2015.主要农作物品种真实性SSR分子标记检测—普通小麦[S].北京:中国农业出版社,2015.
Ministry of Agriculture of the People’s Republic of China.NY/T2859-2015.Variety genuineness testing of main crops with SSR markers - Wheat(TriticumaestivumL.) [S].Beijing:China Agriculture Press,2015.
[4] 中华人民共和国农业部NY/T1432-2014.玉米品种鉴定DNA指纹方法[S].北京:中国农业出版社,2014.
Ministry of Agriculture of the People’s Republic of China NY/T1432-2014.Protocol for the identification of maize varieties - SSR marker method[S].Beijing:China Agriculture Press,2014.
[5] 中华人民共和国农业部NY/T1433-2014.水稻品种鉴定DNA指纹方法[S].北京:中国农业出版社,2014.
Ministry of Agriculture of the People’s Republic of China NY/T1433-2014.Protocol for identification of rice varieties-SSR marker method [S].Beijing:China Agriculture Press,2014.
[6] JONES E S,SULLIVAN H,BHATTRAMAKKI D A.Comparison of simple sequence repeat and single nucleotide polymorphism marker technologies for the genotypic analysis of maize(ZeamaysL.) [J].TheoreticalandAppliedGenetics,2007,115:361.
[7]TIAN H L,WANG F G,ZHAO J R,etal.Development of maize SNP3072,a high-throughput compatible SNP array,for DNA fingerprinting identification of Chinese maize varieties[J].MolecularBreeding,2015,35:136.
[8] SHIRASAWA K,SHIOKAI S,YAMAGUCHI M,etal.Dot-blot-SNP analysis for practical plant breeding and cultivar identification in rice [J].TheoreticalandAppliedGenetics,2006,113:147.
[9] YOON M S,SONG Q J,CHOI I Y,etal.BARCSoySNP23:A panel of 23 selected SNPs for soybean cultivar identification[J].TheoreticalandAppliedGenetics,2007,114:885.
[10] JONES H,NORRIS C,SMITH D,etal.Evaluation of the use of high-density SNP genotyping to implement UPOV model 2 for DUS testing in barley [J].TheoreticalandAppliedGenetics,2013,126(4):901.
[11] 张利莎,董国清,扎 桑,等.基于EST-SSR和SNP标记的大麦麦芽纯度检测[J].作物学报,2015,41(8):1147.
ZHANG L S,DONG G Q,ZHA S,etal.EST-SSR and SNP markers based barley malt purity detection [J].ActaAgronomicaSinica,2015,41(8):1147.
[12] KUANG M,WEI S J,WANGY Q,etal.Development of a core set of SNP markers for the identification of upland cotton cultivars in China [J].JournalofIntegrativeAgriculture,2016,15(5):954.
[13] WANG S,WONG D,FORREST K,etal.Characterization of polyploid wheat genomic diversity using a high-density 90 000 single nucleotide polymorphism array [J].PlantBiotechnologyJournal,2014,12:787.
[14] 季 伟,王立新,孙 辉,等.小麦SSR分析体系的简化[J].农业生物技术学报,2007,15(5):907.
JI W,WANG L X,SUN H,etal.Predigestion of wheat SSR analysis protocol [J].JournalofAgriculturalBiotechnology,2007,15(5):907.
[15] LIU K,MUSE S V.Power marker:An integrated analysis environment for genetic marker data [J].Bioinformatics,2005,21(9):2128.
