利用荟萃分析挖掘拟南芥低温胁迫响应基因
2018-04-10段本耀张学杰樊守金张洛艳
段本耀 张学杰 樊守金 张洛艳
(山东师范大学,济南 250014)
低温胁迫是目前威胁全球作物产量的重要非生物胁迫因子之一,严重影响植物的正常生长发育,降低农作物的产量,是农业生产中常见的限制因子。研究表明,每年低温使全球农作物的损失高达数千亿美元[1~2]。在全球环境气候变化的背景下,培育具有较高产量的耐受低温胁迫的作物是解决目前日益严重的粮食危机的有效手段之一。模式植物如拟南芥低温胁迫应答的生理、分子和遗传响应机制对研发低温耐受作物有重要意义,同时还有助于挖掘植物响应非生物胁迫的进化分子基础。
低温胁迫对于植物的生理水平伤害主要体现在水分运输和存贮、矿质营养、光合作用、呼吸作用和总代谢水平的改变[3~5]。植物进化出一套复杂体系来响应低温胁迫,包括信号转导、基因调控、蛋白修饰和代谢调节等生理和生化等途径[3,6~7]。基因及其关联的生物学过程共同构成了上述生理和生化响应机制的分子基础。信号通路和调控途径的参与基因,包括植物内源激素途径关联基因、信号转导基因、转录因子(transcriptional factors,TFs)和蛋白激酶(protein kinases,PKs)等调节基因由于通常位于胁迫响应上游,会首先感受及传导胁迫刺激信号,并调控下游单功能基因的表达,因此被认为发挥更重要的作用。虽然目前的研究已经阐明了一部分植物应答低温胁迫的分子机制,然而由于生物学性状分子基础的复杂性,仍有大量潜在冷胁迫应答关联的单功能基因和调节基因有待挖掘。
微阵列芯片测序是在组学水平挖掘生物学途径的有效手段之一,目前已有数百个非生物胁迫研究采用微阵列芯片测序来挖掘胁迫应答潜在的分子基础[8],目前,十多个拟南芥的低温胁迫应答测序分析已经鉴定出了13 000余个潜在的冷胁迫应答基因。然而,不同实验鉴定的差异表达基因(differently expressed genes,DEGs)却有一定的差异性(heterogeneity)[9~10]。荟萃整合分析是指利用不同的统计学及信息学手段,通过降低不同实验数据间的不一致性挖掘多个实验间的共性的整合分析方法。目前主要报道了4种常用的微阵列芯片表达数据的整合方法:不同实验的表达数据归一化后直接整合(directly merge after normalization),P值整合(Pvalues)、有效样本数目整合(combine effect sizes,ES)和排位整合(combine ranks)等。归一化后直接整合的方法通常受限于要求同一批次的实验或者要求一致的测序平台[11];而P值整合和排位整合则要求测序实验提供包含P值的差异表达基因名单或者基因表达名单[11~13]。
有效样本数目指的是两组样本间在通过数据集(dataset)标准差(s.d)归一化后均值的差异[12]。NetworkAnalyst网络工具是针对微阵列和二代测序所设计的荟萃分析综合工具,其中批量效应矫正分析、数据不一致度的检测及有效样本数目整合分析可以科学地评价和有效地降低不同表达数据间的不一致性,且仅需测序实验提供原始表达数据矩阵(series matrix),因此被广泛的应用到人类疾病关联的基因及microRNA的微阵列测序及二代测序数据分析中[14]。然而基于有效样本数目整合的荟萃分析还未被应用于植物应答非生物胁迫的测序整合分析领域。
在本研究中,我们通过荟萃整合分析拟南芥在低温胁迫条件和标准条件下的微阵列表达谱芯片数据挖掘出了拟南芥中潜在的低温胁迫响应基因。由于所选择表达谱数据间的差异性,我们选择NetworkAnalyst网站的批量效应矫正分析和有效样本数目整合方法来降低实验间的不一致性并整合表达数据。本研究重点关注挖掘拟南芥不同测序实验间共有的响应冷胁迫的分子基础,并基于此来探索植物响应冷胁迫的分子机制,并为抗冷作物的研发提供一定的理论基础。
1 材料与方法
1.1 文献数据库挖掘
本研究采用文献搜索策略在不同数据库中搜索拟南芥的低温胁迫响应表达芯片数据。首先,采用下列关键词在Gene Expression Omnibus数据库(GEO,www.ncbi.nlm.nih.gov/geo/)、PubMed数据库(https://www.ncbi.nlm.nih.gov/pubmed)和ArrayExpress数据库(www.ebi.ac.uk/arrayexpress)中进行低温胁迫关联的文献及表达芯片数据搜索:(((((((((cold damage)OR cold injury) OR cold stress)OR cold harm) OR chilling damage) OR chilling injury) OR chilling stress)) AND ((((sequencing) OR expression) OR profile) OR profiling)) AND ((Arabidopsisthaliana) ORArabidopsis)。数据库搜索的截止时间为2017年4月30日。
1.2 表达芯片数据筛选
将搜索到的文献或者表达芯片数据进行筛选,保留的低温胁迫实验均符合下列标准:实验材料为拟南芥;同时包含低温胁迫处理样本和对照组样本;基因表达测定实验;实验组与对照组间有可比较的实验条件;需提供可下载的表达芯片数据。具有下列任一情况的实验被排除于本次分析:实验材料非拟南芥;没有对照组实验样本;研究材料为非野生型植物材料;采用荧光定量PCR来测定基因的表达量而未进行测序分析;综述类研究;生物信息类研究;荟萃分析类研究。在筛选后的研究中摘录下列信息:GEO关联号(accession number);文献ID(reference ID);测定平台;胁迫处理组及对照组样本数;研究的组织部位;低温胁迫水平(温度);低温胁迫处理时间(表1)。
注:*.相关信息未在原文中列出;UP.上调基因数(TAIR ID);DOWN.下调基因数(TAIR)
Note: *.The information was not specified in the original research; UP. Up-regulated genes’ number(TAIR ID); DOWN. Down-regulated genes’ number(TAIR ID)
1.3 表达芯片数据预处理
在GEO数据库下载筛选出的测序实验的表达数据矩阵,并对下载的数据进行下列预处理:首先,基于不同测序平台提供的ID转换文件,分别将每个实验中的探针ID转换成TAIR基因组ID。删除一个探针ID对应多个TAID基因组ID所对应的表达数据。当多个探针ID对应一个TAIR基因组ID时,采用取均值的方法来处理他们的表达值。随后检查每个表达数据矩阵是否已进行表达值的log2转换。将经过预处理的表达数据上传到NetworkAnalyst网站(http://www.networkanalyst.ca)[12~13]进行个体和批量效应调整分析(individual and batch effect adjustment analysis)。首先对每个表达谱数据进行个体分析,主要包含以下步骤:数据处理(pre-processing)、基因注释(gene annotation)、归一化分析(quantile normalization)和差异表达基因筛选分析。数据处理过程中通过检查基因在样本中的表达量分布删除表达量显著异常的基因。差异表达基因筛选分析中对照组和处理组样本依据原实验中的设计。
1.4 表达芯片数据个体和批量效应矫正分析及荟萃整合分析
利用Limma算法中的t-test分析筛选单个表达谱数据中低温胁迫样本和对照样本间的差异表达基因,界限值(cutoff)为FDR(false discovery rate)矫正后的Pvalue<0.05[12~13]。随后对经过个体分析后的表达数据集进行基于经验贝叶斯法(empirical Bayes approach)批量效应矫正分析,主要目的为降低不同实验间基因的表达率(the expression ratios)差异,使不同实验数据适于后续整合分析。经过批量效应矫正分析的8个表达谱数据被整合成一个数据集,通过数据完整度评价后,利用Cochrans’ Q test分析,评价表达数据集中不同实验间不一致度,并基于此分析结果,选择random effect model模型(REM)进行有效样本数目整合分析,筛选整合后数据集中低温胁迫处理组和对照组中的差异表达基因,界限值为Pvalue<0.05。采用R(version 3.2.4)平台pheatmap软件包进行前25个上调和下调差异表达基因的热图展示。
1.5 功能富集分析
利用R平台topGO软件包[15]对整合后数据集中在低温胁迫样本和对照样本中的差异表达基因进行功能富集分析。在基因功能本体论数据库(Gene ontology,http://amigo.geneontology.org)中下载拟南芥“响应低温胁迫(response to cold)”(GO:0009409)类目中的具有实验证据(experimental evidences,EXP,IDA,IPI,IMP,IGI and IEP)的注释基因,并将上述基因作为已进行功能鉴定的低温胁迫响应基因。不同家族的转录因子、转录调节因子和蛋白激酶下载于iTAK数据库(http://bioinfo.bti.cornell.edu/cgi-bin/itak/index.cgi)。植物激素代谢途径的参与基因下载自基因本体论数据库。
2 结果与分析
2.1 表达芯片数据选择
本研究通过文本筛选利用关键词在GEO数据库、PubMed数据库和ArrayExpress数据库分别初步筛选出1 166篇关联文献、298个测序实验和695个测序实验。通过去除三个数据库间的冗余及非研究性实验,一共获得了693个实验。进一步通过材料与方法中所述的筛选条件进行精细筛选,进一步从59个实验中筛选出8组完全符合条件的含低温胁迫处理样本和对照样本的表达谱测序实验进行荟萃整合分析(GEO关联号:GSE3326,GSE5620,GSE5621,GSE39090,GSE43818,GSE43819,GSE55906,GSE63186)[16~20],其中GSE5621按照实验处理部位将实验划分成幼苗(Shoots)和根(Roots)两组实验文献(表1,图1)。筛选出的8组表达谱测序实验主要基于2个测序平台:AffymetrixArabidopsisATH1 Genome Array(GPL198)和Agilent-021169Arabidopsis4 Oligo Microarray(V4)(GPL9020);两个平台所含的基因数目分别为20 163个和28 401个。8组实验中处理组样本数目范围为3个到12个;对照组的样本数目范围为从2个到18个。8组实验中的植物材料有:整个地上部分、幼苗、嫩芽、叶和根。不同实验中低温胁迫的处理范围为0℃到4℃;低温胁迫的处理时间范围为0.5~504 h。
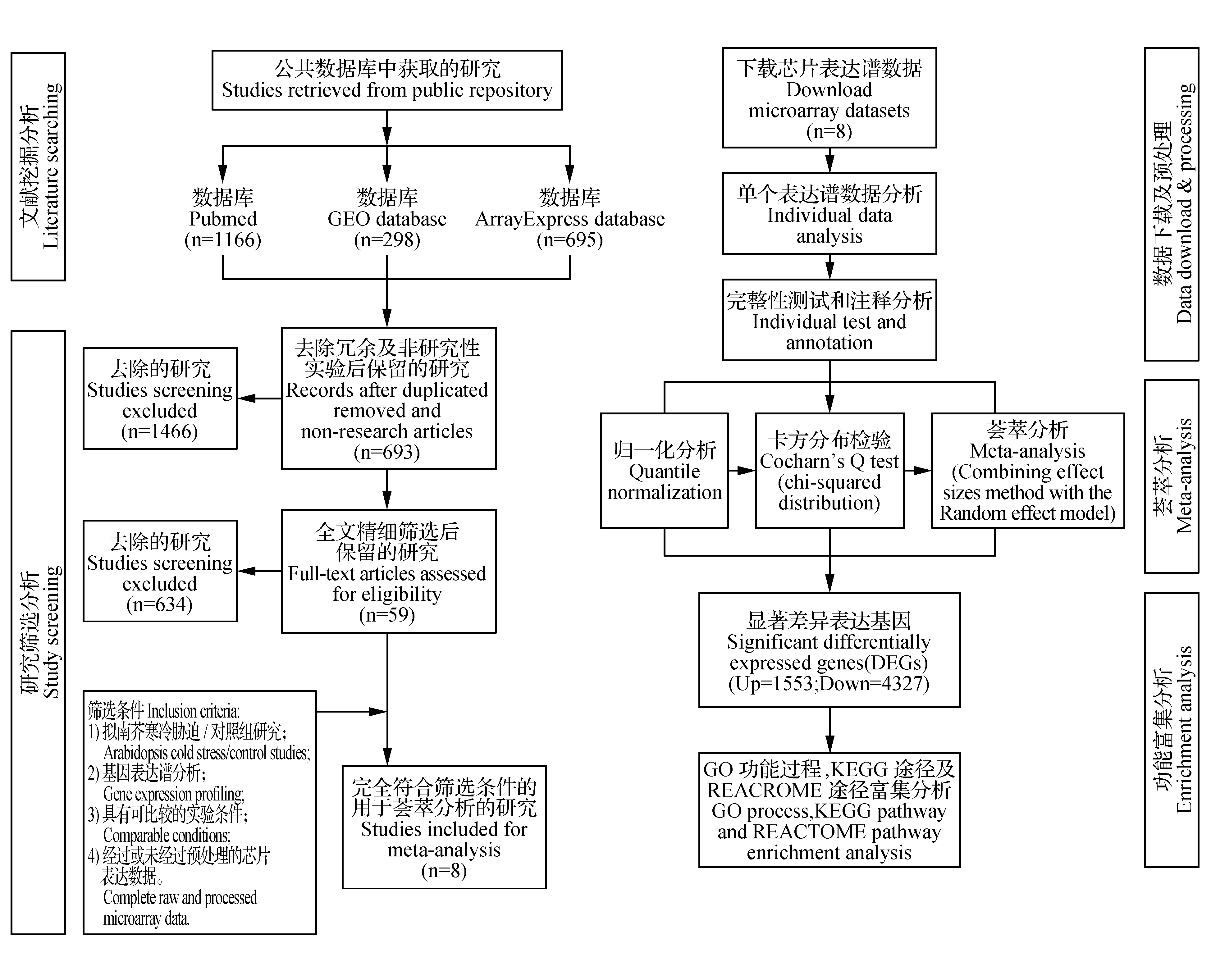
图1 文本挖掘分析、研究筛选/荟萃分析及功能富集分析流程图Fig.1 Flow chart of the literature screening,meta-analysis,enrichment strategy
2.2 个体及批量效应矫正分析结果
8组实验分别进行的个体矫正分析和差异表达基因筛选,一共筛选出12 384个在低温胁迫处理样本和对照样本间差异表达的基因,其中含有7 957个上调表达基因和5 863个下调表达基因(表1)。预处理和归一化的单个表达谱数据上传到NetworkAnalyst网站进行批量效应矫正分析,以减少由于不同批次实验及其他非生物学原因造成的测序数据间的变异,利用主成分分析(Principal Component Analysis,PCA)来展示批量效应矫正分析的效果。批量效应矫正分析前的PCA结果显示,8个实验分布离散,仅来源于同一个实验的样本聚集在一起(图2A);在批量效应矫正分析后的PCA结果中,可以看到8个实验的样本聚集程度显著提高(图2B)。
2.3 荟萃整合分析及差异表达基因筛选
将8个独立的表达谱数据整合后数据集上载到NetworkAnalyst网站进行Cochran’s Q test分析。由于Cochran’s Q test分析显示不同实验间有显著的杂合度(heterogeneities)(图2C),因此采用REM模型进行有效样本数目整合分析来筛选整合后表达数据集中在胁迫组和对照组中差异表达的基因。在Pvalue<0.05界限值下,一共筛选出5 880个差异表达基因,其中包含1 553个上调基因和4 327个下调基因。5 138个荟萃整合分析后筛选出的差异表达基因在单个表达谱数据的差异表达基因筛选分析时也被鉴定为差异表达基因,有742个基因为荟萃整合分析特异的差异表达基因(图2D)。在1 553个荟萃分析所筛选出的上调基因中,有86个基因是已知的低温胁迫功能基因;在4 327个下调基因中有58个是已有功能鉴定的低温胁迫关联基因。表2列出了前20个差异表达基因的名单;图2E展示50个显著上调和下调的基因的表达式样。已报道与响应低温胁迫关联的ERF/AP2转录因子家族中的DREB亚家族成员C-repeat/DRE binding factor 2(CBF2,AT4G25470)和C-repeat/DRE binding factor 3(CBF3,AT4G25480)被鉴定为最显著上调的差异表达基因(表2,图2E)。Octicosapeptide/Phox/Bem1p家族蛋白(AT3G26510)和叶绿素酶1(chlorophyllase 1,AT1G19670)的基因被鉴定为最显著下调的基因(表2,图2E)。
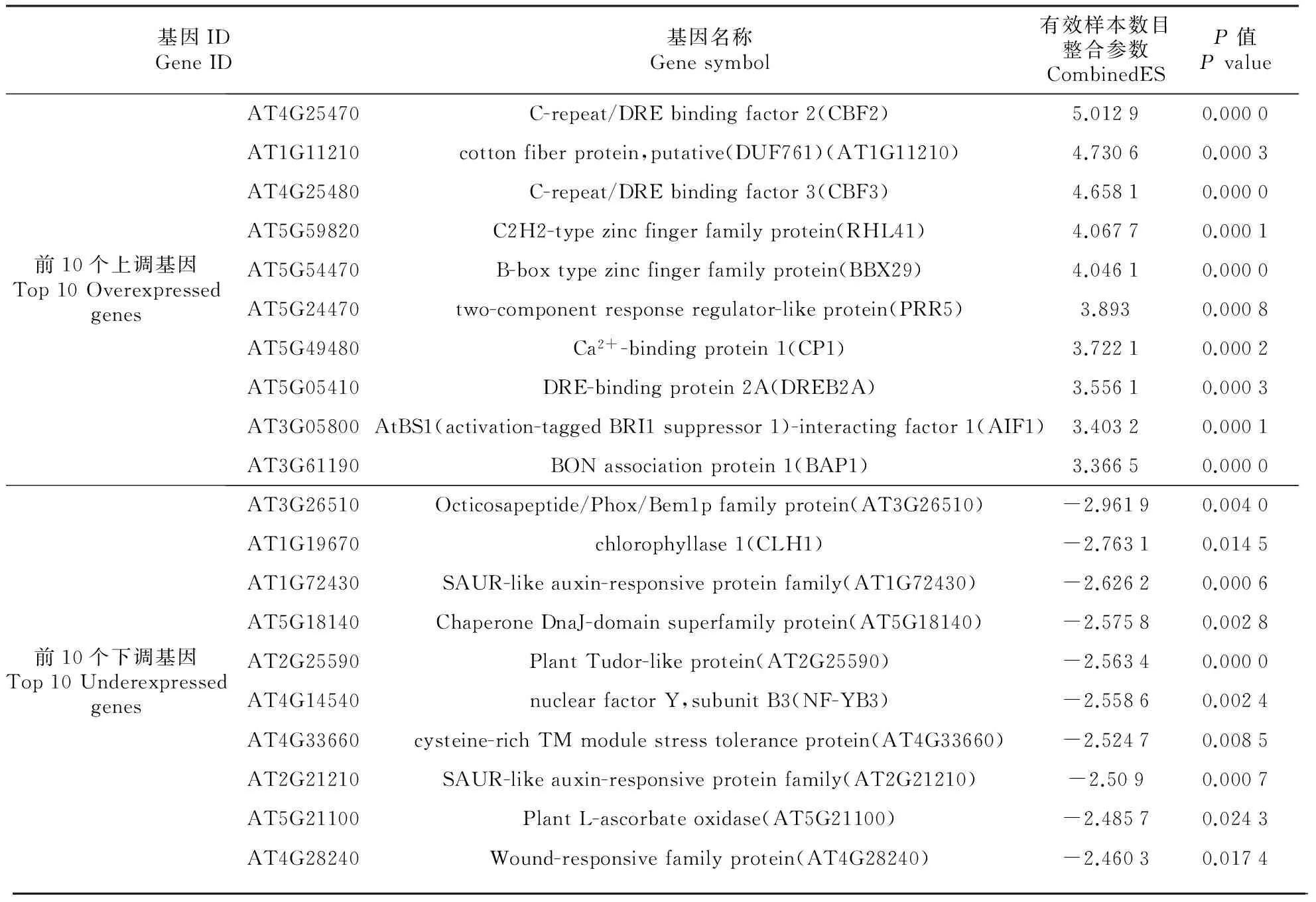
表2 荟萃分析鉴定出的前10个上调基因及前10个下调基因

图2 A.批量效应矫正分析前主成分分析(Principal Component Analysis,PCA)结果;B.批量效应矫正分析后主成分分析结果;C. Cochran’s Q分析的Q-Q plot图;D.荟萃分析筛选出的差异表达基因(Meta-DEGs)和单个分析筛选出的差异表达基因(Individual-DEGs)的venn图;D.前25个上调基因及前25个下调基因在8组实验中的基因表达热图Fig.2 A. The principal component analysis(PCA) for datasets distributions before batch effect adjustment analysis; B. The PCA for datasets distributions after batch effect adjustment analysis; C. The Q-Q plot of Cochran’s Q test; venn diagram of differentially expressed genes identified from the meta-analysis(Meta-DEGs) and those from each individual microarray analysis(Individual-DEGs); D. Gene expression heatmap of the top 25 up-regulated genes and top 25 down-regulated genes from meta-analysis
2.4 差异表达基因的功能富集分析结果
功能富集分析用来挖掘差异表达基因集成员参与的高频(overrepresented)生物学途径和基因功能类目。在Pvalue<0.05的界限值下,1 553个上调基因和4 327个下调基因一共富集到了148个和193个生物学过程类目。表3展示了前15个上调的和前15个下调的生物学过程。其中显著上调的生物学过程有:“响应水缺乏”(GO:0009414,response to water deprivation)、“响应冷胁迫”(GO:0009409,response to cold)、“DNA为模板的转录调控”(GO:0006355,regulation of transcription,DNA-templated)和“响应脱落酸”(GO:0009737,response to abscisic acid)等。其中显著下调的生物学过程有:“氧化还原过程”(GO:0055114,oxidation-reduction process)、“磷酸化作用”(GO:0016310,phosphorylation)、“甾醇生物合成过程”(GO:0016126,sterol biosynthetic process)和“芥子油苷的生物合成过程”(GO:0019761,glucosinolate biosynthetic process)等。

表3 功能富集分析鉴定出的前15个上调及前15个下调生物学过程
2.5 转录因子、激酶及植物激素关联基因的差异表达式样
本研究重点分析了转录因子、激酶及植物激素关联的调控基因在整合后的表达样本中的差异表达式样,以发掘潜在的响应冷胁迫的调控基因。在转录因子的家族成员中,本研究筛选出了26个上调和10个下调的AP2/ERF家族的转录因子(表4),推测其可能与拟南芥响应低温胁迫有关,例如:已报道参与应答渗透胁迫、水胁迫和热胁迫的DREB亚家族A-2成员DRE结合蛋白2A(DREB2A,AT5G05410)和DRE结合蛋白2B(DREB2B,AT3G11020)均在冷胁迫样本中上调。在bZIP家族中,本研究发现有5个上调和10个下调成员可能与响应低温胁迫有关(表4),例如:与盐胁迫和渗透胁迫响应关联的bZIP家族成员AT2G31370和参与调控干旱和盐胁迫过程中钙离子依赖蛋白激酶(Ca2+-dependent protein kinase genes)关联的基因表达的bZIP家族成员BZIP60(AT1G42990)。
表4部分转录因子家族、激酶家族及激素代谢途径中所包含的上调、下调及功能验证基因数目
Table4GenenumbersofDEGsincludedintheTFfamilies,PKsfamiliesandplanthormonepathways
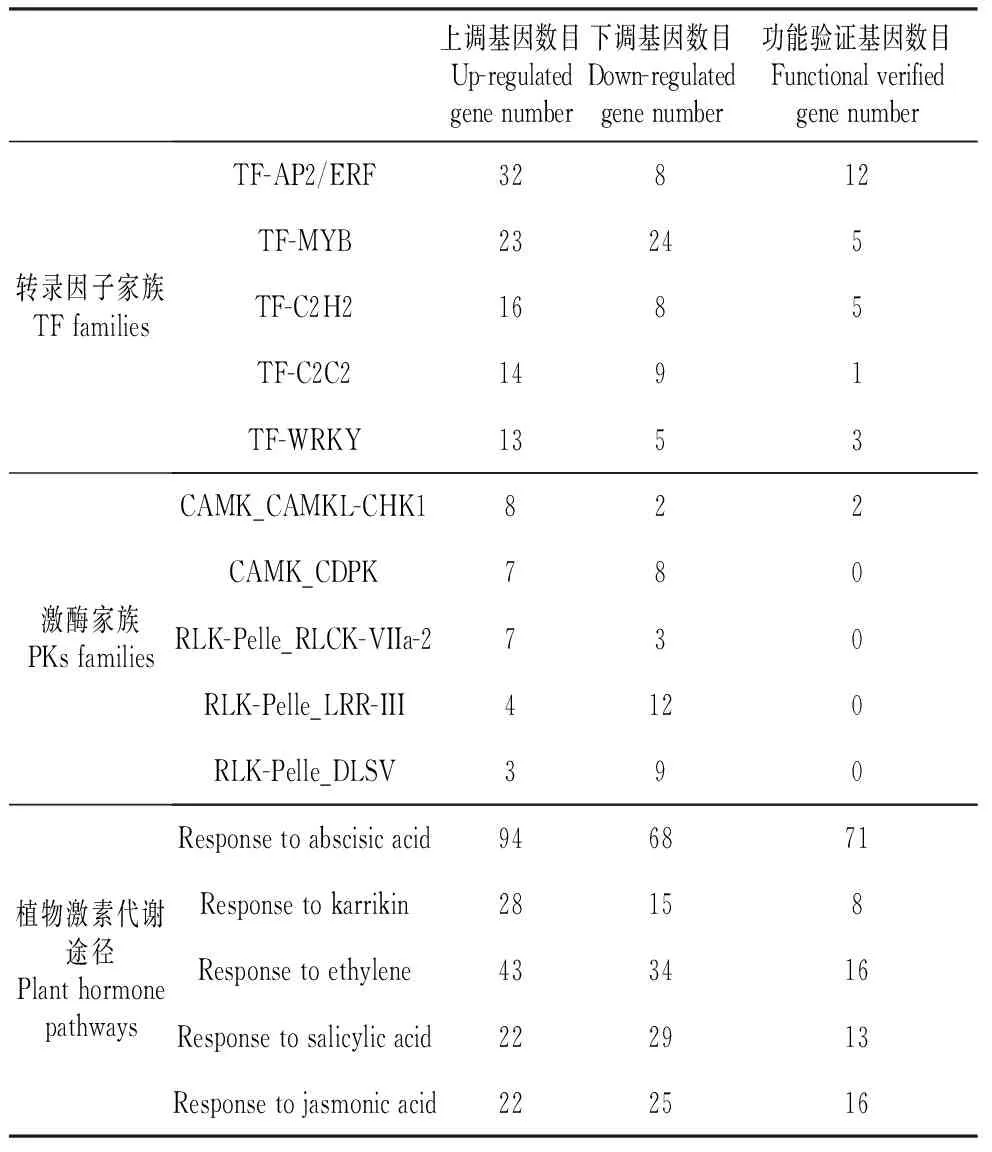
上调基因数目Up-regulatedgenenumber下调基因数目Down-regulatedgenenumber功能验证基因数目Functionalverifiedgenenumber转录因子家族TFfamiliesTF-AP2/ERF32812TF-MYB23245TF-C2H21685TF-C2C21491TF-WRKY1353激酶家族PKsfamiliesCAMK_CAMKL-CHK1822CAMK_CDPK780RLK-Pelle_RLCK-VIIa-2730RLK-Pelle_LRR-III4120RLK-Pelle_DLSV390植物激素代谢途径PlanthormonepathwaysResponsetoabscisicacid946871Responsetokarrikin28158Responsetoethylene433416Responsetosalicylicacid222913Responsetojasmonicacid222516
通过激酶家族成员的表达差异分析,在CAMK_CDPK家族中筛选出7个上调成员和8个下调成员,例如:参与快速防御信号传播和温度波动下的转导途径的钙调蛋白激酶5(CPK5,AT4G35310)和参与干旱和盐胁迫响应的钙调蛋白激酶6(CPK6,AT2G17290)均在冷胁迫处理样本中上调表达。促分裂素原活化蛋白激酶(mitogen-activated protein kinases,MAPK)是植物响应非生物胁迫信号传导通路中的另一个重要成员。本研究中筛选出CMGC_MAPK家族中上调成员3个和下调成员4个,例如MAP激酶11(MAP kinase 11,MPK11,AT1G01560)和MAP激酶5(MAP kinase 5,MPK5,AT4G11330)均在冷胁迫样本中上调表达。
在植物激素关联基因中,本研究鉴定出了与ABA途径关联的94个上调基因和68个下调基因(表4),推测这些基因可能参与拟南芥寒冷胁迫信号响应,如:脱落酸应答元件结合因子1(ABF1,AT1G49720)及plant U-box 9(PUB9,AT3G07360)等。本研究中筛选出38个上调的和33个下调的乙烯途径关联的基因(表4),推测为参与冷胁迫的潜在基因,例如:ERF家族蛋白38(ERF38,AT2G35700)和乙烯应答元件结合因子2(ERF2,AT5G47220)。此外,在与冷胁迫关联的茉莉酸和赤霉素等激素途径中分别有18个上调、25个下调及12个上调、22个下调的基因被推测为冷胁迫潜在参与基因(表4)。
3 讨论
挖掘低温胁迫响应基因或者耐受基因是促进低温胁迫分子机制研究和培育抗低温胁迫作物的有效途径之一。虽然目前已报道了近400个拟南芥低温胁迫应答基因,然而由于植物对低温胁迫的响应是一个非常复杂的体系,涉及到一系列的细胞内信号转导、基因表达调控、蛋白翻译修饰和生理代谢调节等生化和细胞途径[3~5],挖掘植物响应低温胁迫的分子机制仍是亟待解决的关键问题之一。近年来,随着分子生物学、基因工程和基因组学研究方法的快速发展,与植物抗性生理学及生态学结合的植物响应逆境的分子机制研究已经进入了基因组信息时代。伴随着基因芯片测序及高通量测序方法在植物抗逆领域的广泛应用,积累了大量与植物抗逆关联的遗传及表达信息资源,如何科学的整合和利用这些资源成为关键问题。基因芯片荟萃分析可以有效的利用上述资源,对不同实验的表达谱数据进行二次分析,降低不同实验间的不一致性,从而获得更加可靠的结果,挖掘出与植物抗逆性状关联的差异表达基因的深层次信息,并且可以对抗逆性状关联的分子机制作出科学的预测。近年来,整合荟萃分析在基因表达谱分析、基因诊断、药物筛选及序列分析等诸多领域已呈现出广阔的应用前景[6,10~11],但在植物基因响应寒冷胁迫的表达数据整合方面仍有所欠缺。
本研究基于NetworkAnalyst网站的批量效应校正和基于REM模型的有效样本数目整合分析有效地降低了不同实验间的不一致性并进行了科学的荟萃整合分析,并筛选出1 553个上调基因和4 327个下调基因作为不同测序实验响应低温胁迫的共有潜在基因。已有的研究表明,植物激素关联的胁迫感受途径、离子和激酶关联的信号通路及转录因子参与的转录调控过程是拟南芥响应寒冷胁迫的关键途径[3,6,21]。本研究发现,上述三个途径仍有潜在基因可能参与拟南芥寒冷胁迫信号响应。例如,在植物冷胁迫应答过程中,胁迫响应基因表达的通路被分为ABA依赖型和ABA非依赖型[3~5]。在本研究中,鉴定出了94个上调和68个下调基因可能参与拟南芥寒冷胁迫信号响应,如:ABA信号通路及蛋白泛素化途径关联的基因等。此外,乙烯应答因子ERF是植物启动冷胁迫关联基因表达的重要转录因子,主要通过其保守的DNA结合域与乙烯应答元件GCCbox相互作用,以启动胁迫应答基因的表达。本研究通过整合分析预测出的数十个上调及下调的ERF家族转录因子,推测此家族仍有潜在的冷胁迫应答参与基因待通过功能性实验进行验证。
除了上述调控途径,本研究基于功能富集分析发现“细胞内信号传递(intracellular signal transduction)”、“细胞内水缺乏应答(cellular response to water deprivation)”、“昼夜节律(circadian rhythm)”和“防御反应(defense response)”等生物学过程在冷胁迫响应过程中的功能仍有待进一步挖掘。此外,由于脱水、高盐及冷胁迫均会不同程度的影响渗透平衡等内稳态,植物响应不同非生物胁迫转录因子调控机制有一定的相似性[7,22~23],因此本研究挖掘的与其他胁迫关联的功能基因及其参与的生物学过程可能参与低温胁迫应答,例如:“盐胁迫应答的负调控(negative regulation of response to salt stress)”“氧化胁迫应答(response to oxidative stress)”和“干旱恢复(drought recovery)”等。虽然上述潜在的生物学途径关联的基因在植物冷胁迫响应过程中的功能仍需功能实验进行验证,但本研究通过荟萃分析为植物冷胁迫响应的潜在功能基因和潜在分子机制的挖掘提供了更全面和可靠的候选研究对象。
虽然本研究通过荟萃分析挖掘出了参与拟南芥低温胁迫响应的潜在基因及其关联的功能子过程,仍需要指出本研究的优点及局限性。本研究的局限性主要是由所筛选的不同测序实验之间的差异性造成的:我们选择的8个实验包括了5个组织部位,2个低温胁迫条件,8个时间梯度和2个不同的测序平台。首先,不同研究间由于单个实验操作技术、分子遗传因素、环境条件变化等造成的不一致性是很难移除的。其次,由于基因在不同组织部位表达差异造成的不一致性也是很难评价和移除的。上述两个因素构成了本研究的主要局限性。然而,批量效应校正方法被设计用来通过平衡基因的表达波动来降低不同实验间的差异性,再进一步结合基于REM模型的有效样本数目整合分析,可以进一步消减不同表达测序实验间基因表达值的不一致性[12~13]。此外,采用GO功能途径基因功能富集分析可以在差异表达基因的基础上查找与拟南芥冷胁迫响应关联的子过程,潜在的功能基因和子过程共同构成了本研究预测的拟南芥响应低温胁迫的分子基础。植物对低温胁迫响应是一个非常复杂的体系,包括信号转导、基因调控、蛋白修饰和代谢调节等生理和生化途径,基于本研究提供的潜在分子基础,利用遗传学、生物化学、基因组学、转录组学技术相结合,开展拟南芥及其他植物对低温响应的研究,将更深入地阐明植物应答低温胁迫的分子机制,并在此基础上研发抗冷作物。
1.简令成,卢存福,李积宏,等.适宜低温锻炼提高冷敏感植物玉米和番茄的抗冷性及其生理基础[J].作物学报,2005,31(8):971-976.
Jian L C,Lu C F,Li J H,et al.Increment of chilling tolerance and its physiological basis in chilling-sensitive corn sprouts and tomato seedlings after cold-hardening at optimum temperatures[J].Acta Agronomica Sinica,2005,31(8):971-976.
2.Lukatkin A S.Contribution of oxidative stress to the development of cold-induced damage to leaves of chilling-sensitive plants:2.The activity of antioxidant enzymes during plant chilling[J].Russian Journal of Plant Physiology,2002,49(6):782-788.
3.乌凤章,王贺新,徐国辉,等.木本植物低温胁迫生理及分子机制研究进展[J].林业科学,2015,51(7):116-128.
Wu F Z,Wang H X,Xu G H,et al.Research progress on the physiological and molecular mechanisms of woody plants under low temperature stress[J].Scientia Silvae Sinicae,2015,51(7):116-128.
4.Bloom A J,Zwieniecki M A,Passioura J B,et al.Water relations under root chilling in a sensitive and tolerant tomato species[J].Plant,Cell & Environment,2004,27(8):971-979.
5.刘勇,冯美利,曹红星,等.低温胁迫对油棕叶片养分含量变化的影响[J].热带农业科学,2014,34(6):16-19.
Liu Y,Feng M L,Cao H X,et al.Effect of low temperature on the leaf nutrient content of oil palm[J].Chinese Journal of Tropical Agriculture,2014,34(6):16-19.
6.Song B H,Mitchell-Olds T.Evolutionary and ecological genomics of non-model plants[J].Journal of Systematics and Evolution,2011,49(1):17-24.
7.Catalá R,Medina J,Salinas J.Integration of low temperature and light signaling during cold acclimation response inArabidopsis[J].Proceedings of the National Academy of Sciences of the United States of America,2011,108(39):16475-16480.
8.Atkinson N J,Lilley C J,Urwin P E.Identification of genes involved in the response ofArabidopsisto simultaneous biotic and abiotic stresses[J].Plant Physiology,2013,162(4):2028-2041.
9.Yu G W,Zang W D,Yang X Q,et al.Meta-analysis ofArabidopsisthalianaunder abscisic acid and salt stress[J].Journal of Medicinal Plants Research,2011,5(24):5889-5893.
10.Zhang L Y,Zhang X J,Fan S J.Meta-analysis of salt-related gene expression profiles identifies common signatures of salt stress responses inArabidopsis[J].Plant Systematics and Evolution,2017,303(6):757-774.
11.Tseng G C,Ghosh D,Feingold E.Comprehensive literature review and statistical considerations for microarray meta-analysis[J].Nucleic Acids Research,2012,40(9):3785-3799.
12.Xia J G,Benner M J,Hancock R E.NetworkAnalyst-integrative approaches for protein-protein interaction network analysis and visual exploration[J].Nucleic Acids Research,2014,42(W1):W167-W174.
13.Xia J G,Gill E E,Hancock R E.NetworkAnalyst for statistical,visual and network-based meta-analysis of gene expression data[J].Nature Protocols,2015,10(6):823-844.
14.Jha P K,Vijay A,Sahu A,et al.Comprehensive Gene expression meta-analysis and integrated bioinformatic approaches reveal shared signatures between thrombosis and myeloproliferative disorders[J].Scientific Reports,2016,6:37099.
15.Alexa A,Rahnenfuhrer J.topGO:enrichment analysis for gene ontology.[http://www.bioconductor.org/packages/2.11/bioc/html/topGO.html].
16.Lee B H,Henderson D A,Zhu J K.TheArabidopsiscold-responsive transcriptome and its regulation by ICE1[J].The Plant Cell,2005,17(11):3155-3175.
17.Kilian J,Whitehead D,Horak J,et al.The AtGenExpress global stress expression data set:protocols,evaluation and model data analysis of UV-B light,drought and cold stress responses[J].The Plant Journal,2007,50(2):347-363.
18.Guan Q M,Wu J M,Zhang Y Y,et al.A DEAD box RNA helicase is critical for pre-mRNA splicing,cold-responsive gene regulation,and cold tolerance inArabidopsis[J].The Plant Cell,2013,25(1):342-356.
19.Kim Y,Park S,Gilmour S J,et al.Roles of CAMTA transcription factors and salicylic acid in configuring the low-temperature transcriptome and freezing tolerance ofArabidopsis[J].The Plant Journal,2013,75(3):364-376.
20.Chan Z L,Wang Y P,Cao M J,et al.RDM4 modulates cold stress resistance inArabidopsispartially through the CBF-mediated pathway[J].New Phytologist,2016,209(4):1527-1539.
21.Gorsuch P A,Sargeant A W,Penfield S D,et al.Systemic low temperature signaling inArabidopsis[J].Plant & Cell Physiology,2010, 51(9) :1488-1498.
22.Agarwal P K,Shukla P S,Gupta K,et al.Bioengineering for salinity tolerance in plants:state of the art[J].Molecular Biotechnology,2013,54(1):102-123.
23.Munns R.Genes and salt tolerance:bringing them together[J].New Phytologist,2005,167(3):645-663.
