花色素苷合成酶的生物信息学分析
2017-11-09张鹏飞丁楠李萍
张鹏飞,丁楠,李萍*
(西南交通大学生命科学与工程学院,四川成都 610031)
花色素苷合成酶的生物信息学分析
张鹏飞,丁楠,李萍*
(西南交通大学生命科学与工程学院,四川成都 610031)
色素是一类水溶性的类黄酮物质,是植物合成的一类次生代谢产物。花色素合成涉及一系列的代谢反应,花色素苷合成酶(anthocyanin synthesis,ANS)是其合成通路中末端的关键酶,能催化无色花色素转化为有色花色素。目前对ANS的研究表明ANS的表达可调控花色素的积累以及果实和花的颜色。本课题采用生物信息学的方法,对已在 GenBank 上注册的大丁草杂交品种等9种菊科植物ANS的氨基酸序列进行分析。通过此次研究,希望为今后深入研究该类酶的功能和结构特征提供依据。
生物信息学;ANS;分子进化
花色和果色是植物重要的观赏性状,决定着花卉和果实的商品性和价值性[1]。在高等植物中类黄酮(flavonoid)、类胡萝卜素(carotenoid)、和甜菜色素(anthocyanin)是使植物具有无穷魅力的内在原因[2],而类黄酮中的花色素更是对植物的成色至关重要。花色素的合成涉及一系列代谢反应,花色素苷的合成途径中共有20种不同的有机分子参加反应,涉及12种不同的催化酶,这些酶由多个同源的基因编码,共同控制花色素苷合成途径如图1。其中有7种酶在途径中起着较为重要的作用。其中一些酶对合成起到至关重要的作用[3-5],而花色素苷合成酶(ANS)是花色素苷合成途径后期的关键酶。
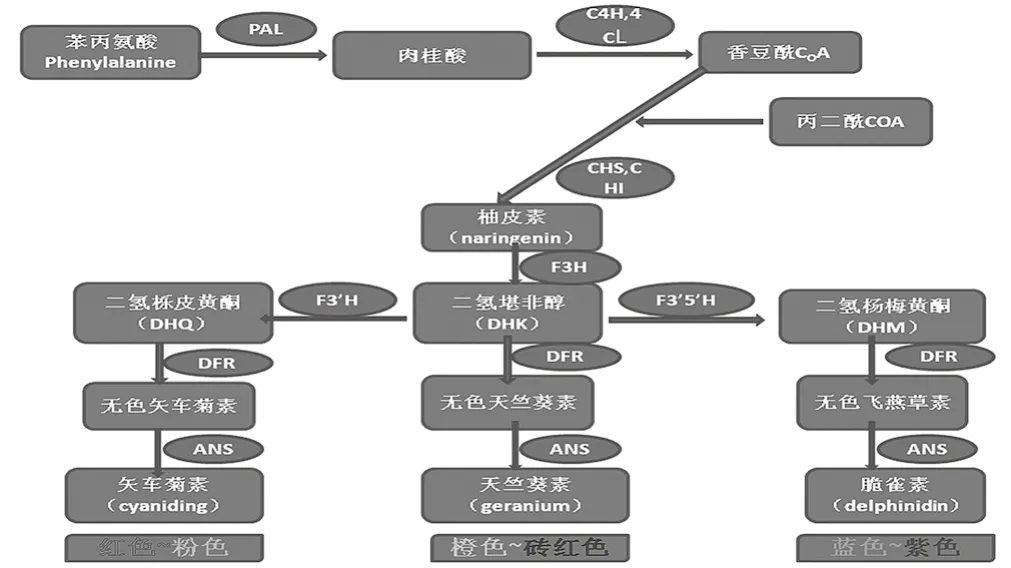
图1 花色素苷生物合成途径
ANS为无色花青素加氧酶,属于2-酮戊二酸铁依赖型双加氧酶,最显著的作用是催化无色花色素转化为有色花色素,在花色素苷合成途径的后期,ANS依赖2-酮戊二酸离子和Fe2+将无色的花色素氧化,产生有颜色的花色素[6]。ANS基因最先从玉米的突变体中通过转座子标签法分离得到,另外在拟南芥、圆叶牵牛、裂叶牵牛、洋桔梗、葡萄和银杏中都有提取[7]。因此本课题在已有ANS研究的基础上,收集9种菊科植物的ANS核苷酸序列和对应的蛋白序列,分析它们的理化性质、结构功能以及进化上的关系,以期为深入探究ANS的性质和在植物生长活动中表现出的更广泛作用提供可靠的理论依据。
1 材料与方法
1.1 试验材料
数据从NCBI 搜索已注册的ANS核苷酸序列及其对应的氨基酸序,筛选出菊科(Asterales)部分序列作为研究材料,包括大丁草杂交品种(Gerbera hybrid cultivar,AAY15743.2)、 紫背菜(Gynura bicolor,BAJ17658.1)、大丽菊(Dahlia pinnata,BAJ21536.1)、瓜叶菊(Pericallis cruenta,ACF75869.2)、莴苣(Lactuca sativa,BAJ10383.1)、水母雪莲花(Saussurea medusa,AAS48200.1)、翠菊(Callistephus chinensis,AAB66560.1)、茼蒿×杭白菊(Chrysanthemum x morifolium,ACF75873.2)、绿毛山柳菊(Hieracium pilosella,ACB56922.1)。
1.2 实验方法
通过ProtParam 在线工具分析ANS基因的核酸序列和氨基酸序列的组成成分和理化性质;通过SignalP[8]来检测ANS基因的信号肽及其剪切位点;使用ProtScale[9]在线工具分析ANS基因的亲疏水性;TMHMM[10]将预测序列中可能含有的跨膜结构,NetNGlyc和NetPhos[11]分别分析序列中的糖基化和磷酸化位点;用MEGA做进化分析,进一步分析其进化关系;使用CDD[12]和SWISS-MODEL[13-15]用来预测ANS的保守结构域、二级结构和三级结构,并与已知的ANS的结构作比较。
2 实验结果
2.1 ANS氨基酸序列理化性质分析
用ProtParam在线工具对以上9中菊科植物ANS序列进行氨基酸理化性质分析,结果如表1。

表1 ANS的氨基酸组成及理化性质分析
由表1可知,九种菊科植物的花色素苷合成酶DNA序列长度、氨基酸数目、分子量、IP都表现出一致性,这是由遗传的同源性所决定的。它们的序列长度在1 000bp-1 500bp之间;氨基酸总数均处在355bp左右;分子量均分布在40KD左右;IP值均低于6;Glu、Leu、Ile、Lys是所选菊科ANS序列共有的主要氨基酸,且9种菊科植物均不含Pyl、Sec;紫背菜、莴苣、水母雪莲花和绿毛山柳菊蛋白为稳定类蛋白,大丁草杂交品种、大丽菊、瓜叶菊、翠菊和茼蒿杭白菊为不稳定性蛋白质。
2.2 ANS核酸序列ORF预测
利用ORF Finder软件查询9种菊科植物ANS核酸序列的开放阅读框(表2)。ORF Finder给出六组参考数据,来自于两条链三联体密码子的不同阅读顺序,而真实结果只可能有一种。由于知道ANS蛋白序列长度,因此所示点开每一条ORF都对应着编码的蛋白质的长度。结果表明9种植物ANS的开放阅读框长度均在1 068bp左右,翠菊ORF偏长为1 276bp。这个结果在一定程度上体现了菊科植物ANS的保守性。
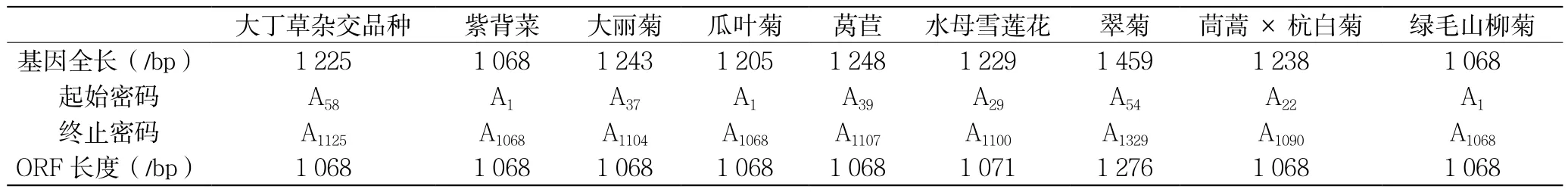
表2 9种菊科植物ANS核酸序列ORF预测和分析
2.3 信号肽的预测和分析
以绿毛山柳菊为研究对象,通过SignalP Server在线工具进行ANS氨基酸序列信号肽的分析。由图2可知九种菊科植物的ANS蛋白序列均无信号肽。图中所示绿色线和蓝色线且值均较低,可以推测以上植物基因在游离核糖体上合成之后,新生成的蛋白质并不进行转运,而仅仅留在细胞质凝胶中催化无色花色素干转化成有色花色素苷从而推动花色素苷合成途径进行。
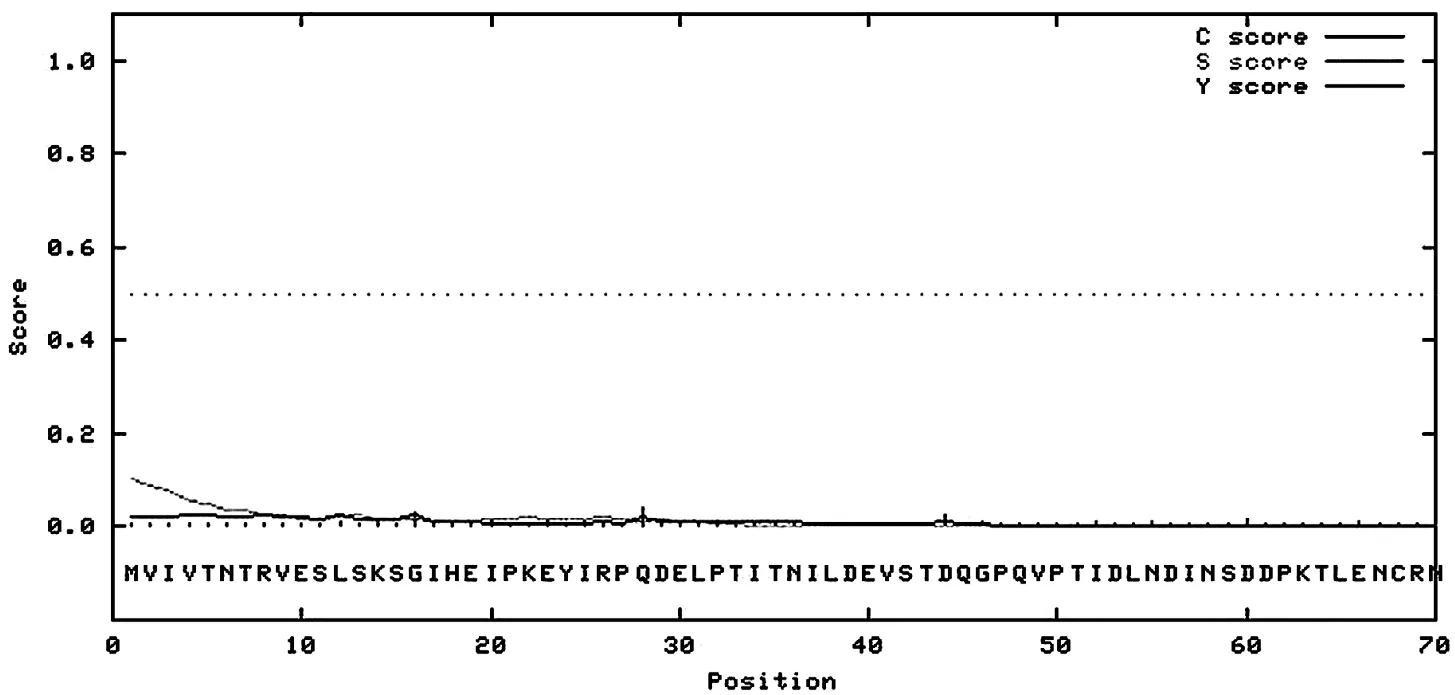
图2 绿毛山柳菊ANS氨基酸序列信号肽预测
2.4 亲水性/疏水性的预测和分析
以绿毛山柳菊为例使用在线工具ProtScale(Hphob./ Kyte &Doolittle)预测结果如图3,绿毛山柳菊ANS多肽链中第114位谷氨酸具有最低值-3.256,亲水性最强;第189位亮氨酸具有最高值2.433,疏水性最强。就整条多肽链而言, 表现为亲水性。通过对上述植物的分析可得到相近的结果。
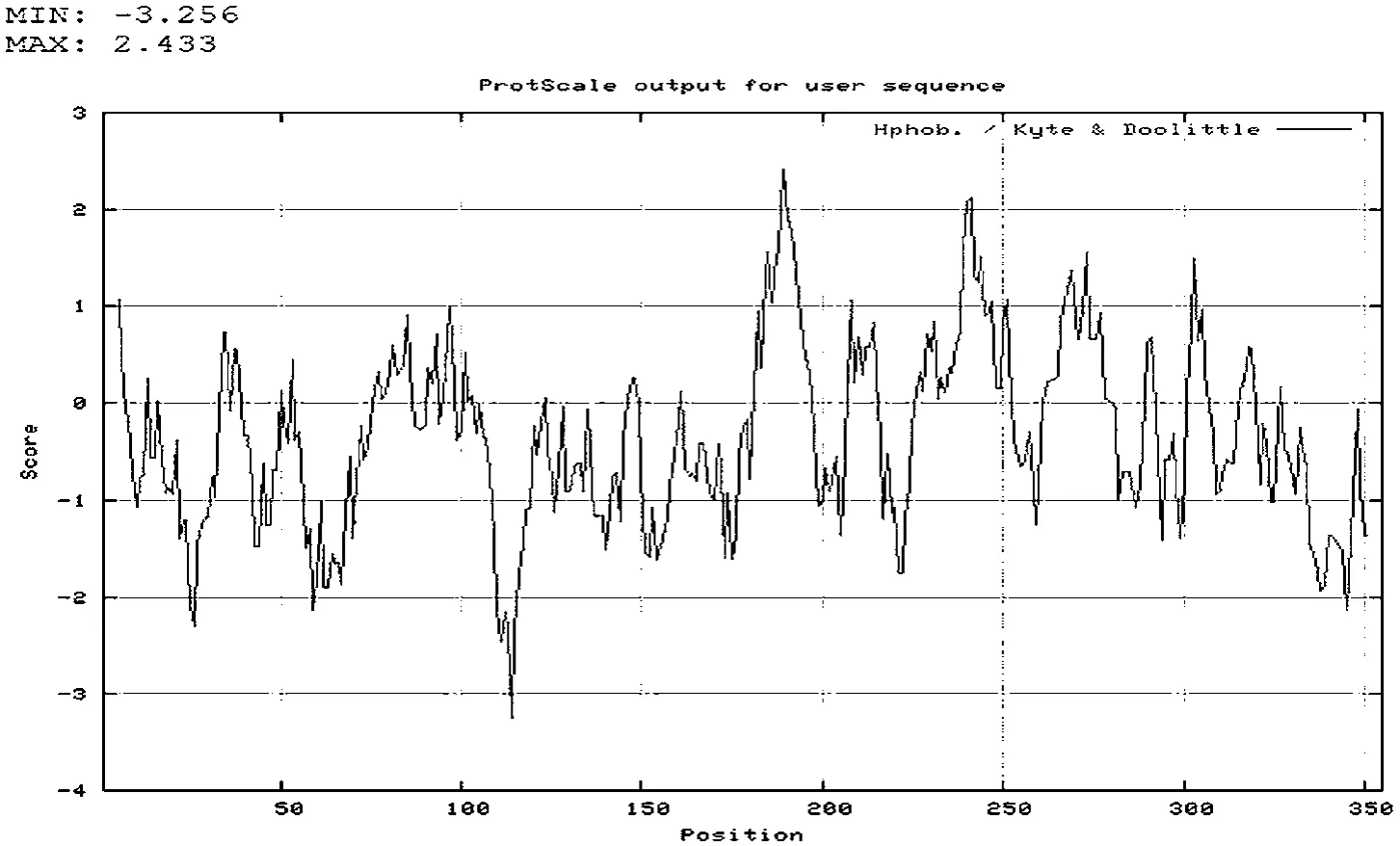
图3 绿毛山柳菊ANS氨基酸序列亲/疏水性预测和分析
2.5 跨膜结构域的预测和分析
以绿毛山柳菊为研究对象,使用在线工具TMHMM Server进行了预测分析。如图4,绿毛山柳菊的ANS氨基酸序列均在膜外,没有穿插在膜之间的跨膜区,其他序列的一侧结果亦是如此。这一结论与亲水性预测结果一致,即所选ANS氨基酸序列均没有跨膜结构。我们可以推测,植物ANS蛋白质在细胞质中合成后并不存在运输,在合成部位或附近产生作用。还可以推测ANS不是分泌型蛋白质。
图4 绿毛山柳菊ANS氨基酸序列的跨膜结构域预测
2.6 糖基化和磷酸化预测和分析
以绿毛山柳菊为例使用在线软件NetNGlyc对ANS氨基酸序列进行糖基化分析。结果如图5,在位置134的天冬酰胺处有蓝色竖线,值为0.6 188,超过设定阀值0.5。便认为此处可能糖基化 。
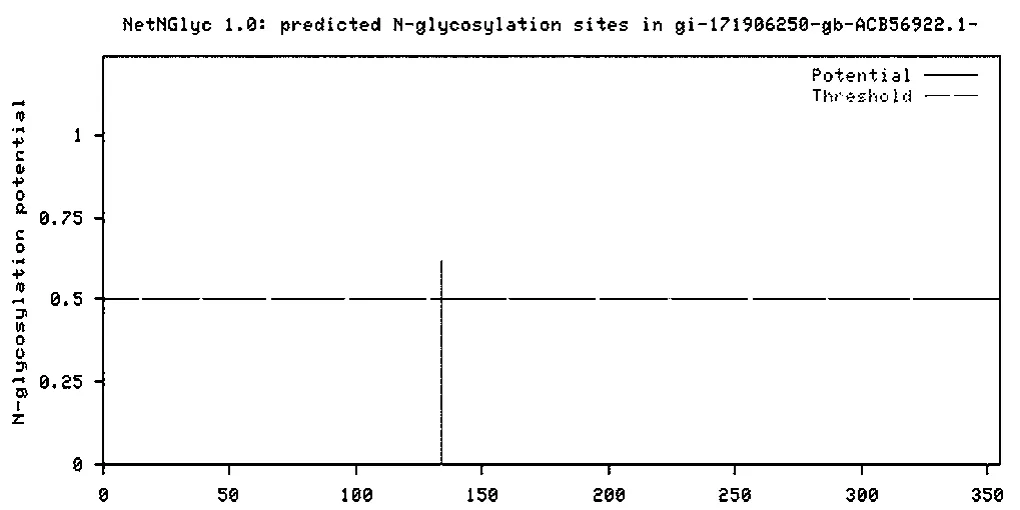
图5 绿毛山柳菊ANS氨基酸序列的糖基化预测
以绿毛山柳菊为研究对象,使用在线软件Netphos分别预测了ANS氨基酸序列中的丝氨酸、苏氨酸、酪氨酸3种不同的磷酸化位点,如图6。结果显示,有许多竖线都超过阈值0.5,这些位点均有较大概率会被磷酸化。其中有8个丝氨酸磷酸化位点(为11、13、15、42、116、121、302和 324位)、5个苏氨酸磷酸化位点(为7、158、163、182和322位)以及3个酪氨酸磷酸化位点(为144、167和285位)。
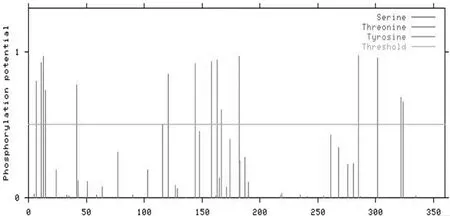
图6 绿毛山柳菊ANS氨基酸序列的磷酸化预测
2.7 序列比对及进化树的构建
用MEGA软件对所选菊科植物和一些不属于菊科、但有同源性的植物如洋桔梗、番薯、裂叶牵牛、圆叶牵牛,这13种植物ANS氨基酸序列一同构建进化树,参数设置为自检举1 000次,模型选择核苷酸p-distance,如图7所示。
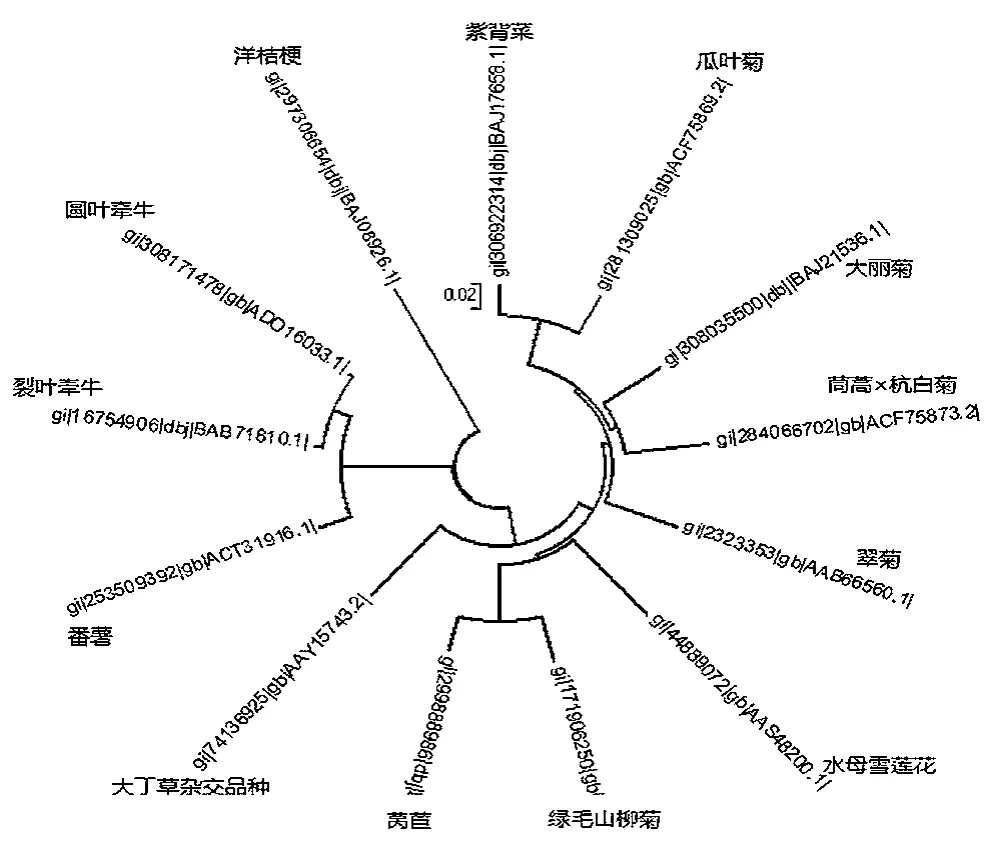
图7 13种植物ANS氨基酸序列构建的进化树
上图所示结果与分类学结论相符合,13条序列明显被分为三大类,属于桔梗科的洋桔梗自成一类,三种旋花科植物番薯、裂叶牵牛和圆叶牵牛被分成一类,9条菊科植物分成一类。同时菊科植物与旋花科植物亲缘关系较近,菊科与桔梗科植物亲缘关系相比之下较远。被分出的旋花科植物中两种牵牛(裂叶牵牛和圆叶牵牛)与另外一种植物番薯的亲缘远近从图中也可以立刻看出。
2.8 二级结构预测
以绿毛山柳菊为例使用在线软件Swiss-Model对ANS氨基酸序列进行预测,结果如图8所示,其二级结构的主要构建是红色的α-螺旋和绿色的无规则卷曲,其次是穿插在α螺旋和无规则卷曲中间的延伸链。9条序列二级结构预测结果均与绿毛山柳菊相似,即他们二级结构的主要构件是α-螺旋和无规则卷曲,其次是延伸链。

图8 绿毛山柳菊ANS氨基酸序列的二级结构预测和分析
2.9 保守结构域的预测和分析
以绿毛山柳菊为例使用在线软件InterproScan分析ANS氨基酸序列,由图9可知,绿毛山柳菊ANS氨基酸序列有一个20G-FeII_Oxy超家族。研究表明,ANS和F3H一样同属于一个氧化戊二酸依赖型加氧酶家族,InterproScan对于保守结构域的预测刚好证明了这一结论。2-酮戊二酸-Fe+-双加氧家族包含2 -酮戊二酸(2OG)和Fe(Ⅱ)依赖氧家族成员。这个家族包括脯4 -羟化酶α亚基C末端。全酶具有活性欧共体,催化反应是:胶原L -脯氨酸+ 2-酮戊二酸+氧气<=>胶原蛋白反式- 4 -羟基- L -脯氨酸+琥珀+二氧化碳。同时还有一个PLN03176 超家族,是cl14733家族的一员。

图9 绿毛山柳菊ANS氨基酸序列保守结构域预测
2.10 三级结构预测
利用Swiss-Model对菊科植物ANS的氨基酸序列进行三级结构的预测,结果如图10所显示菊科植物ANS的空间三级结构均有较大相同点,这与这些菊科核苷酸序列和蛋白质序列本身的相似性有关,同时如前所述菊科植物ANS蛋白中有较为保守的结构域,决定着他们同属于一个家族,三级结构的相似性也证明此结论的准确性。

图10 9种菊科植物ANS氨基酸序列三级结构预测
3 结论
本课题利用生物信息学的知识,利用网络上丰富的软件和信息资源,对9种菊科植物的ANS核苷酸和氨基酸序列进行预测。实验结果表明,菊科植物ANS蛋白质起始密码子为ATG,终止密码子为TGA,相应的蛋白质序列长度分布在355bp左右,相差甚微;ANS蛋白质偏酸性;ANS氨基酸序列所含氨基酸较保守,其中谷氨酸、亮氨酸、异亮氨酸和赖氨酸在所有序列中均占有较高比例,同时全部不含吡咯赖氨酸和硒半胱氨酸;ANS蛋白很可能仅仅停留在细胞质中参与到花色素合成过程中,而不进行转运等;ANS氨基酸序列糖基化分析发现不同植物ANS的糖基化位点不同个数也不同,推测正是由于分子水平糖基化不同,引起了二级结构三级结构折叠和弯曲上的细微差异,但是能进行糖基化的位点只是位于天冬酰胺上,产生N-链接糖蛋白;ANS磷酸化分析看出序列上有很多磷酸化位点,而且出现的位置较统一;9种菊科植物ANS序列相似度极高,在氨基酸序列基础上构建的进化树与形态学特征和物种分类标准基本吻合;菊科ANS氨基酸序列二级结构中含有的主要构件是α-螺旋和无规则卷曲,其次是穿插在整个序列当中的延伸链;20G-FeII_Oxy超家族和PLN03176 超家族是他们共有的保守结构域;三级结构预测通过彩色立体图看出他们在结构上具有极高的相似性。该研究结果可为深入开展ANS酶学特性的分子机理研究提供重要理论依据。
[1]李春雷,崔国新,许志茹,李玉花.植物二氢黄酮醇 4- 还原酶基因的研究进展.生物技术通讯,2009,20(3) :442-445.
[2]刘娟,冯群芳,张杰.二氢黄酮醇 4- 还原酶基因 (DFR) 与花色的修饰.植物生理学通讯,2005,41(6) :715- 719.
[3]Nakatsuka T,Nishihara M,Mishiba K,et al.Two different mutations are involved in the formation of white-flowered gentian plants.Plant Sci,2005,16(9):949-58.
[4]Weiss D,van der Luit AH,Kroon JTM,et al.Petunia homologue of the Antirrhinum majus candi and Zea mays A2 flavonoid genes;homology to flavanone 3-hydroxlase and ethylene-forming enzyme,Plant Mol Biol,1993,22(5):893-897.
[5]高爱红,等.天然食用色素——花青素研究进展[J].保鲜与加工,2001,(3):25-27.
[6]Salisbury FB,Ross CW.Plant Physiology.California:Wads-worth Publishing Company.,1992.323~325.
[7]Xie D Y,Jackson L A,Cooper J D,Ferreira D,Paiva N L.Molecular and biochemical analysis of two cDNA clones encodingDihydrofla2 vonol242Reductase from M edicago truncatula.Plant Physiology,2004,13(4):979 - 994.
[8]Petersen T N,Brunak S,von Heijne G,et al.SignalP 4.0:discriminating signal peptides from transmembrane regions [J].Nature methods,2011,8(10):785-786.
[9]Wilkins MR,Gasteiger E,Bairoch A,et al.Protein identification and analysis tools in the ExPASy server[J].Methods Mol Biol,1999,112:531-552.
[10]A Krogh,B Larsson,G von Heijne,et al.Predicting transmembrane protein topology with a hidden Markov model:Application to complete genomes [J].Journal of Molecular Biology,2001,305 (3):567-580.
[11]Blom N,Gammeltoft S,Brunak S.Sequence and structure-based prediction of eukaryotic protein phosphorylation sites [J].J Mol Biol,1999,294(5):1351-1362.
[12]Marchler-Bauer A,ZhengC,Chitsaz F,et al.CDD:conserved domains and protein three-dimensional structure [J].Nucleic Acids Res,2013,41:348-352.
[13]Biasini M,Bienert S,Waterhouse A,et al.SWISS-MODEL:modelling protein tertiary and quaternary structure using evolutionary information[J].Nucleic acids research,2014.
[14]Kiefer F,Arnold K,Künzli M,et al.The SWISS MODEL Repository and associated resources [J].Nucleic Acids Research,2009,37:387-392.
[15]Guex N,Peitsch M C,Schwede T.Automated comparative protein structure modeling with SWISS-MODEL and Swiss-PdbViewer:A historical perspective[J].Electrophoresis,2009,30(S1):162-173.
Bioinformatics Analysis of Anthocyanin Synthesis in Plants
Zhang Peng-fei,Ding Nan,Li Ping*
(School of Life Science and Engineering,Southwest Jiaotong University,Sichuan Chengdu 610031)
Anthocyanin is a kind of water-soluble flavonoids,which is a class of secondary metabolites produced by plants.The procedure of anthocyanin synthesis involves a series of metabolic reactions,and the key enzyme ANS is just at the end of the pathway of anthocyanin synthesis that catalyzes the conversion of colorless anthocyanin pigment to the colored anthocyanin pigment.Current study of ANS shows that the expression of it can be a regulation factor to the accumulation of anthocyanin,and the controlling of color of fruits and flowers.This research project use the bioinformatics methods,analyze the amino acid sequence of 9 different gerbera hybrids that registered on GenBank .Through this research hope can provide evidence of functional and structural features of this kind of enzyme for further research.
Bioinformatics;ANS;Molecular evolutio
Q946
A
2096-0387(2017)05-0010-06
张鹏飞(1992—),男,甘肃陇西人,硕士,研究方向:生物化学与分子生物学。
李萍(1962—),女,四川成都人,硕士生导师,教授,研究方向:生物化学与分子生物学。
