308例高危妊娠产前诊断CMA技术发现不明确拷贝数变异的结果分析
2017-09-20蒋宇林戚庆炜孟华周希亚郝娜徐钟慧白俊杰欧阳云淑张一休刘俊涛
蒋宇林,戚庆炜*,孟华,周希亚,郝娜,徐钟慧,白俊杰,欧阳云淑,张一休,刘俊涛
(1.中国医学科学院 北京协和医学院 北京协和医院妇产科,北京 100730;2.中国医学科学院 北京协和医学院 北京协和医院超声诊断科,北京 100730;3.北京贝康医学检验所,北京 101111)
308例高危妊娠产前诊断CMA技术发现不明确拷贝数变异的结果分析
蒋宇林1,戚庆炜1*,孟华2,周希亚1,郝娜1,徐钟慧2,白俊杰3,欧阳云淑2,张一休2,刘俊涛1
(1.中国医学科学院 北京协和医学院 北京协和医院妇产科,北京 100730;2.中国医学科学院 北京协和医学院 北京协和医院超声诊断科,北京 100730;3.北京贝康医学检验所,北京 101111)
目的探讨染色体微阵列分析(CMA)在高危妊娠产前诊断中的应用,观察其中临床意义不明的染色体拷贝数变异(VOUS)结果构成及相关特性,分析将该技术广泛应用于产前染色体病诊断临床检测的可行性。方法选择2014年1月1日至2017年6月1日期间于北京协和医院就诊并接受产前诊断,同时进行CMA及核型分析检测的孕妇308例,对其CMA检测的结果进行分类分析并与核型分析相比较,分析其中不明确意义结果的构成以及相关特性。结果所有入组病例中88.0%为各种胎儿结构异常、软指标异常、胎儿生长受限及胎儿水肿。全基因组检测未发现明确染色体拷贝数改变(CNV)或明确多态性改变的病例217例,占70.5%;发现明确致病性染色体数目或片段异常共44例,占14.3%。其中发现染色体非整倍体异常21例,性染色体数目异常6例,染色体致病性CNV结果17例。此外发现不明意义的CNV诊断结果47例,占总病例数的15.3%。其中致病性判读为偏良性的23例,临床意义不明为21例,含符合报告标准的杂合性缺失(LOH)1例,偏致病性的3例。进一步分析各种VOUS病例的片段大小分布及判读依据类型。与核型分析的异常阳性率10.7%比较,全基因染色体芯片诊断明确致病性的染色体异常阳性率为14.2%,诊断阳性率提升3.5%。结论全基因组染色体芯片对于超声发现胎儿结构异常的遗传诊断能力较核型分析有较大的提升,但同时也会有VOUS病例增加遗传咨询的难度。目前国内产前诊断VOUS结果的比例较高,这与本地化数据库不完善,缺乏国内具体的芯片结果判读标准等有关。
产前诊断; 染色体拷贝数变异; 高危妊娠; 染色体微阵列分析; 胎儿超声结构异常
(JReprodMed2017,26(9):863-868)
染色体微阵列分析(CMA)技术是伴随人类基因组计划的完成和近年来分子遗传学发展的新技术。目前已成为人类染色体疾病检测的主要方法。该技术的原理是在微阵列芯片固体表面上集成大量已知序列的基因探针,将被测样品DNA中大量标记的核酸序列与上述探针阵列进行杂交,通过检测相应位置杂交探针的信号,实现特定染色体片段拷贝数(CNV)信息的快速全面检测[1]。与传统细胞遗传学核型分析方法相比,它具有自动化、快速、准确、灵敏度高,以及实现同一芯片上同时大信息量平行检测的优势[2]。正是由于该技术对于染色体病的强大检测能力,2011及2013年美国人类遗传学会(ACMG)发表专家共识将其作为儿童不明原因的智力/体格发育迟缓、自闭症以及超声发现胎儿畸形产前诊断的首选检测方法[3-4],2013年美国妇产科学会(ACOG)也发表指南建议将CMA技术应用于产前诊断的临床检测[5]。2016年ACOG发布的产前遗传病诊断指南中更是将该项技术放在了产前诊断技术体系中重要的地位[6]。尤其在胎儿超声结构畸形的产前诊断方面,研究表明应用CMA进行检测可以较常规核型分析多发现6%的病理意义拷贝数变异[7]。当前在国内产前遗传诊断领域,CMA技术也开始逐步应用于产前临床检测[8]。但于此同时,高精度的遗传检测带来一定比例的临床意义不明的染色体拷贝数变异(VOUS)的结果,胎儿出生后的表型不确定性对于产前遗传咨询带来巨大的挑战[9]。这也成为该技术在临床产前诊断领域广泛开展的重要制约因素。本研究在这种背景下,通过回顾性总结308例连续送检的高危产前诊断标本进行全基因组染色体微阵列芯片检测结果,观察其中不明确意义结果的构成以及相关特性,探讨适宜的染色体变异的致病性判读标准,分析将该技术广泛应用于产前染色体病诊断临床检测的可行性。
资料与方法
一、研究对象
选择2014年1月1日至2017年6月1日期间于北京协和医院就诊,接受羊水产前诊断并进行CMA以及核型分析检测的孕妇共308例,进行羊水穿刺产前诊断时胎儿均存活。每例入组孕妇均按标准操作流程在无菌条件下抽取羊水25~30 ml送实验室检测。
二、实验室检测
所有羊水标本均在北京协和医院产前诊断中心实验室分拣,其中留取15 ml羊水于该实验室进行原位法细胞培养,400条带显带水平进行核型分析,每例计数30个核型,分析10个细胞克隆。其余10 ml左右羊水当日转运到北京贝康医学检验所进行平行的全基因组染色体芯片检测。DNA提取采用北京原平皓生物技术有限公司提供的组织/细胞基因组DNA提取试剂盒,按照相关操作说明提取样本的基因组DNA,全基因组芯片选择美国Affymetrix公司的CytoScan®750K全基因组芯片,检测流程包括消化、连接、PCR扩增、PCR产物纯化及片段化、标记、杂交、洗染及扫描。芯片扫描仪选用Affymetrix公司GeneChip®3000 Scanner with 7G upgrade扫描仪。
三、芯片检测结果的信息分析
芯片检测信息分析和判读软件选用Affymetrix公司提供的Chromosome Analysis Suite (ChAS) 2.1版,同时结合目前国际常用的基因组与表型公共数据库进行检索和判读,包括UCSC Genome Browser(http://genome.ucsc.edu/cgi-bin/hgGateway)、ClinGene(https://www.clinicalgenome.org/)、OMIM(http://omim.org)、DECIPHER(https://decipher.sanger.ac.uk)、DGV(http://dgv.tcag.ca/dgv/app/home)以及PubMed(http://www.ncbi.nlm.nih.gov/pubmed)。
四、全基因组芯片检测结果的判读和报告发放
染色体片段变异的主要报告原则包括:(1)缺失片段大于100 kb,重复片段大于200 kb的染色体CNV;(2)非明确多态性,涉及有OMIM基因的整体缺失重复;(3)片段的重复或缺失涉及OMIM基因部分区域,可能导致基因表达异常;(4)3 Mb以上的连续单亲二倍体(UPD)区域;(5)遗传印记区域的UPD。CNV致病性判读按ACMG相关致病性判读指南[10]进行。对于判读为VOUS的CNV片段,建议孕妇本人及配偶进行亲代检测验证,如果该CNV遗传自表型正常的亲代,判读为VOUS-倾向于良性;若为新生突变,根据指南原则判断为VOUS-不明确意义或VOUS-偏致病性。全基因组芯片检测结果与核型分析结果均独立发放报告。异常诊断结果需结合上述两种方法的检测结果共同判读。
五、病例随访
所有纳入研究的活产新生儿均进行电话或门诊随访,随访妊娠结局83%。引产或流产儿均送病理检查,有条件时进行组织遗传诊断复核。
结 果
一、患者一般情况
符合入组条件的孕妇共308例,平均年龄33.1岁,平均孕次2.6次,产次0.8次,产前诊断检测中位孕周22+3周,最大孕周25+5周。所有产前诊断标本均为羊水标本。主要的产前诊断指证统计见表1,所有入组病例中88.0%为各种胎儿结构异常、软指标异常、胎儿生长受限及胎儿水肿。
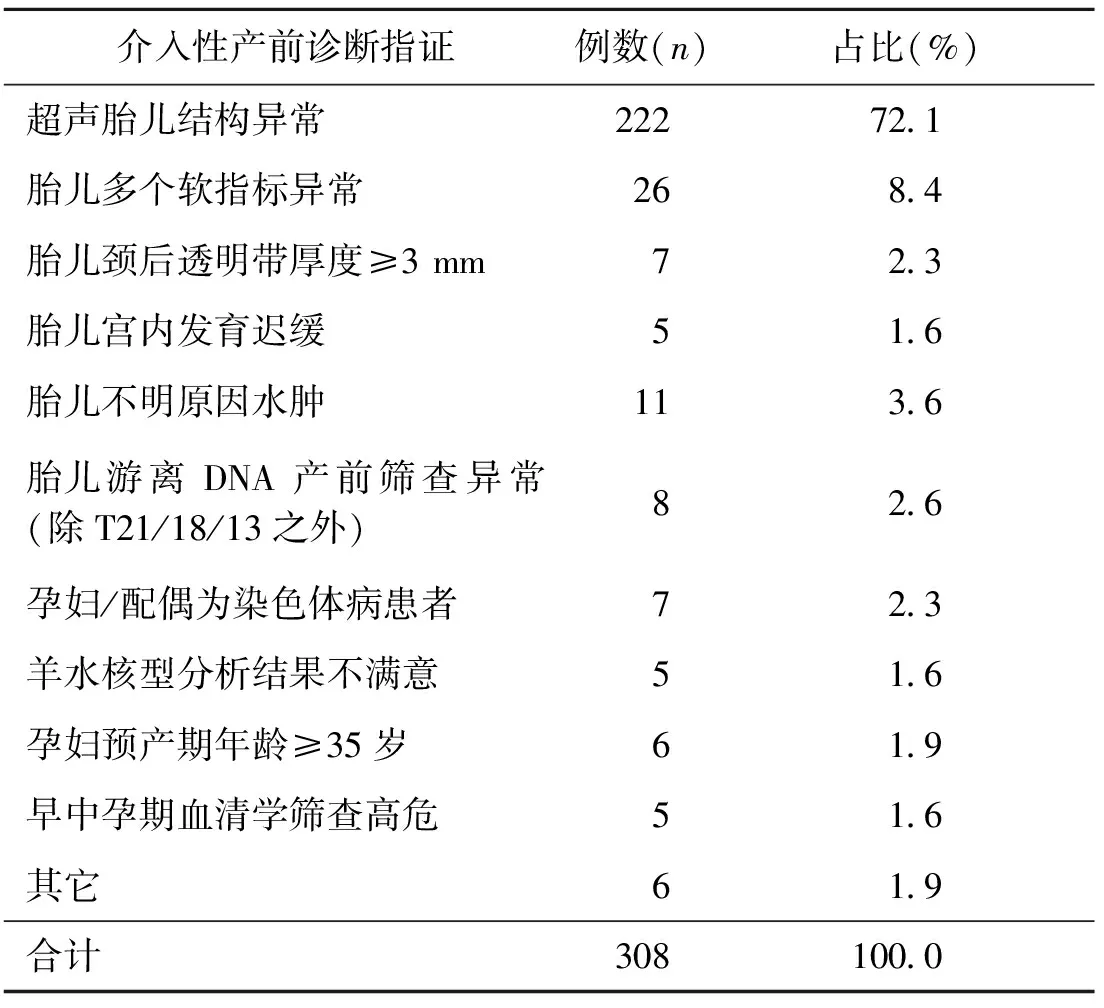
表1 入组患者的产前诊断主要指证
二、检测结果的类型分析
308例患者中,全基因组检测未发现明确拷贝数改变或明确多态性改变的病例217例,占70.5%;发现明确致病性染色体数目或片段异常共44例,占14.3%。其中发现染色体非整倍体异常21例,性染色体数目异常6例,染色体致病性CNV结果17例。此外发现不明意义的CNV诊断结果47例,占总病例数的15.3%。其中根据ACMG指南判读为偏良性的23例,临床意义不明为21例,含符合报告标准的杂合性缺失(LOH)1例,偏致病性的3例。具体类型分布见表2。
三、对于判读为VOUS的结果分析
1.VOUS的片段大小分析:本研究中选用的全基因组染色体芯片为Affymetrix公司的CytoScan®750K,根据芯片技术参数建议的CNV大小报告原则为缺失>200 kb的片段以及重复>400 kb的片段。而本研究实际执行的是更为严格的缺失>100 kb的片段、重复>200 kb的片段以及更小片段但涉及OMIM基因部分外显子区域的CNV片段进行报告的标准,在一些类似国外研究中还有缺失重复≥1 Mb的片段进行报告的标准选择[11]。根据这3种不同的片段大小选择标准,以及是否报告涉及OMIM基因的更小片段改变,将本研究发生的VOUS病例依据不同标准进行分类统计(表3)。其中UPD的报告标准均为连续区域发生UPD≥3 Mb。
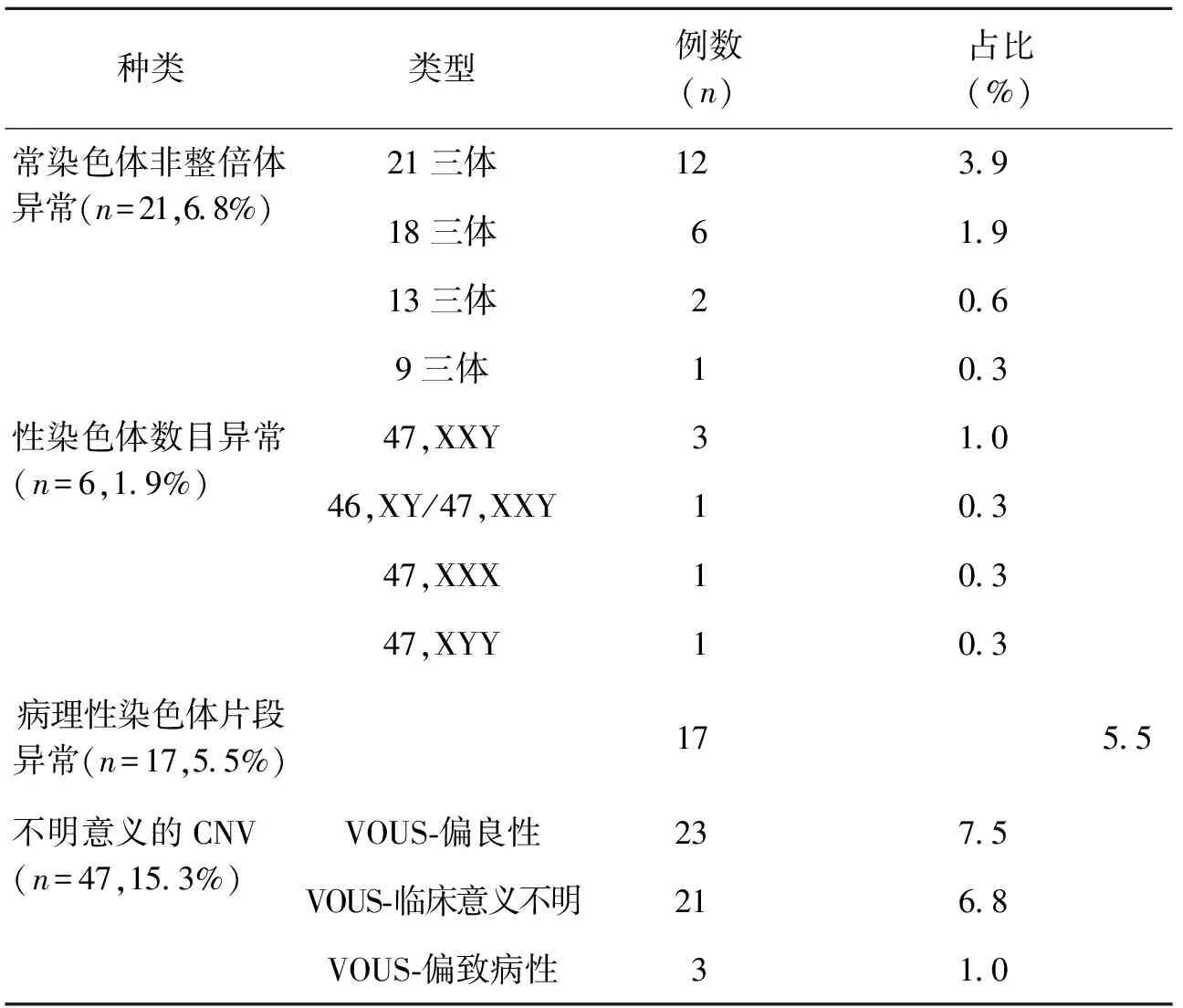
表2 308例患者中诊断的各种染色体及片段异常类型
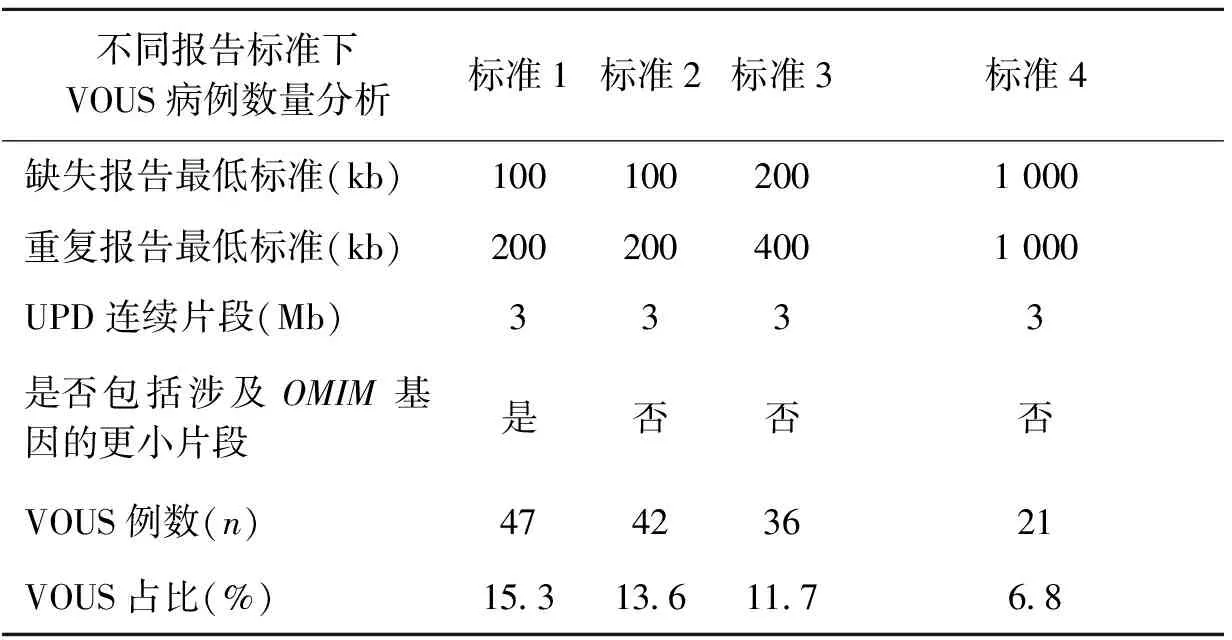
表3 不同报告标准下VOUS片段的检出率
2.VOUS的分类及判断依据类型分析:依据ACMG染色体拷贝数改变致病性的判读指南,以及本实验室的CNV报告原则,本研究的308例检测结果中存在不明确意义的拷贝数改变为56个,分布于47例检测结果中。具体CNV判读分类见表4。其中偏良性、偏致病性及不明意义的VOUS分别占53.6%、3.5%、42.9%。在判读为VOUS-偏良性的CNV中,隐性遗传病携带者以及CNV片段中不含有OMIM基因或基因与疾病无明确相关的情形占83.3%,其余为显性或隐性致病基因发生缺失或重复,且基因剂量效应不明,经过表型正常的亲代验证为遗传性的情形(16.7%)。在判读为VOUS-不明意义的病例中,主要为涉及的CNV内含有显性或隐性基因的重复或缺失,且基因剂量效应不明的情形,以及CNV片段致病性判读矛盾的情形,共占82.6%。仅有的2处连续3 Mb以上的UPD发生于同1例病例中,占所有入组病例的0.3%。2例VOUS-偏致病性的病例中1例为21q21.1q21.2的7.3 Mb缺失,涉及PRSS7 等5个OMIM基因,但该基因的单倍体剂量效应不明,由于缺失片段偏大,综合判读为VOUS-偏致病性;另1例为21q21.1q21.2区域1.1 Mb的缺失,涉及NCAM2这一唐氏综合征候选基因的部分外显子。
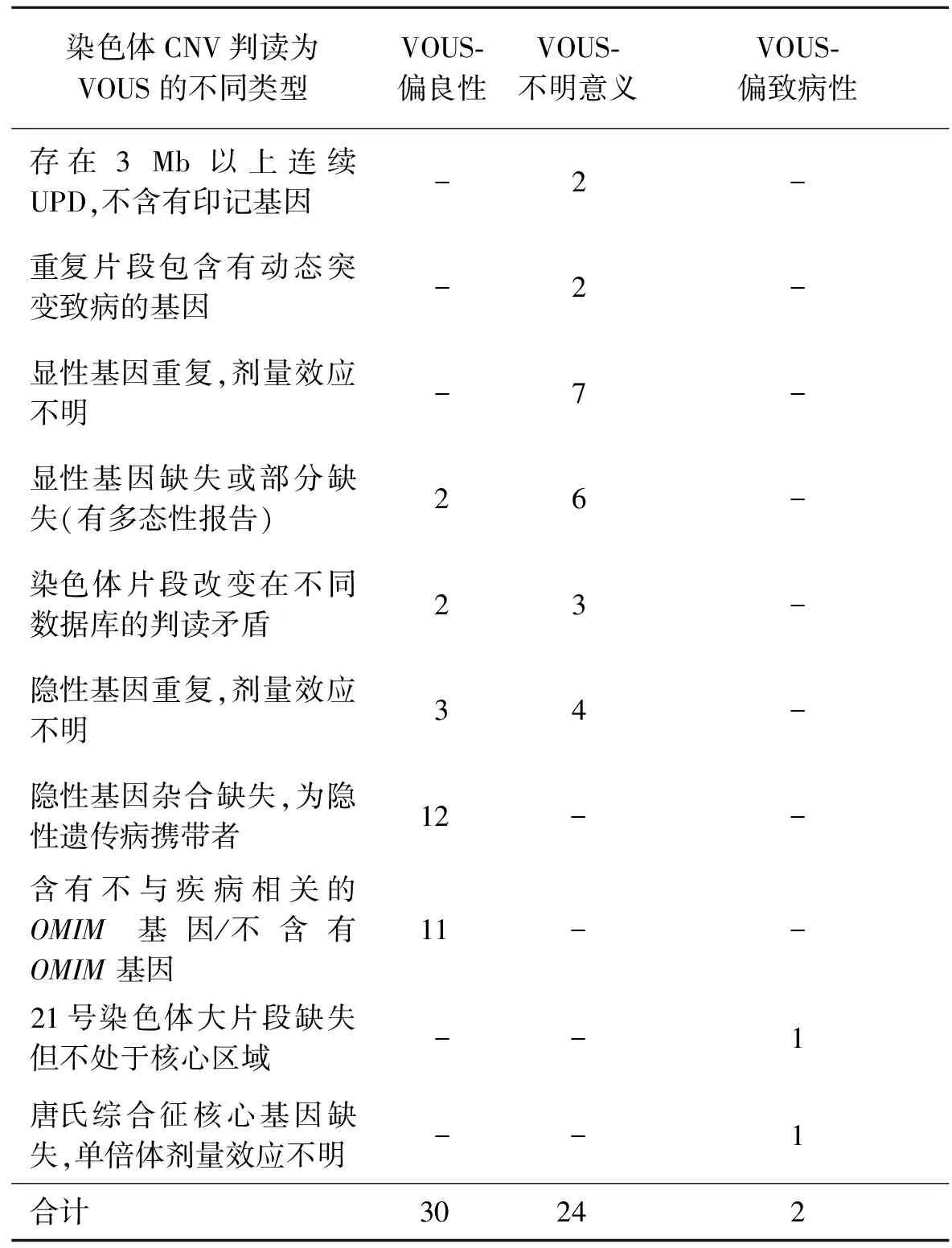
表4 VOUS片段不同分类的判读依据分析
3.入组病例中CMA和核型分析检测能力的比较:308例入组病例同时完成了CMA和核型分析的检测,检测成功率均为100%。两种方法的诊断符合率及全基因组染色体芯片的边际诊断能力见表5。经过比对,未发现核型分析结果异常的病例被CMA检测漏诊的情形,与核型分析异常阳性率10.7%比较,全基因染色体芯片在本研究人群中诊断明确致病性的染色体异常阳性率为14.2%,诊断阳性率提升3.5%。
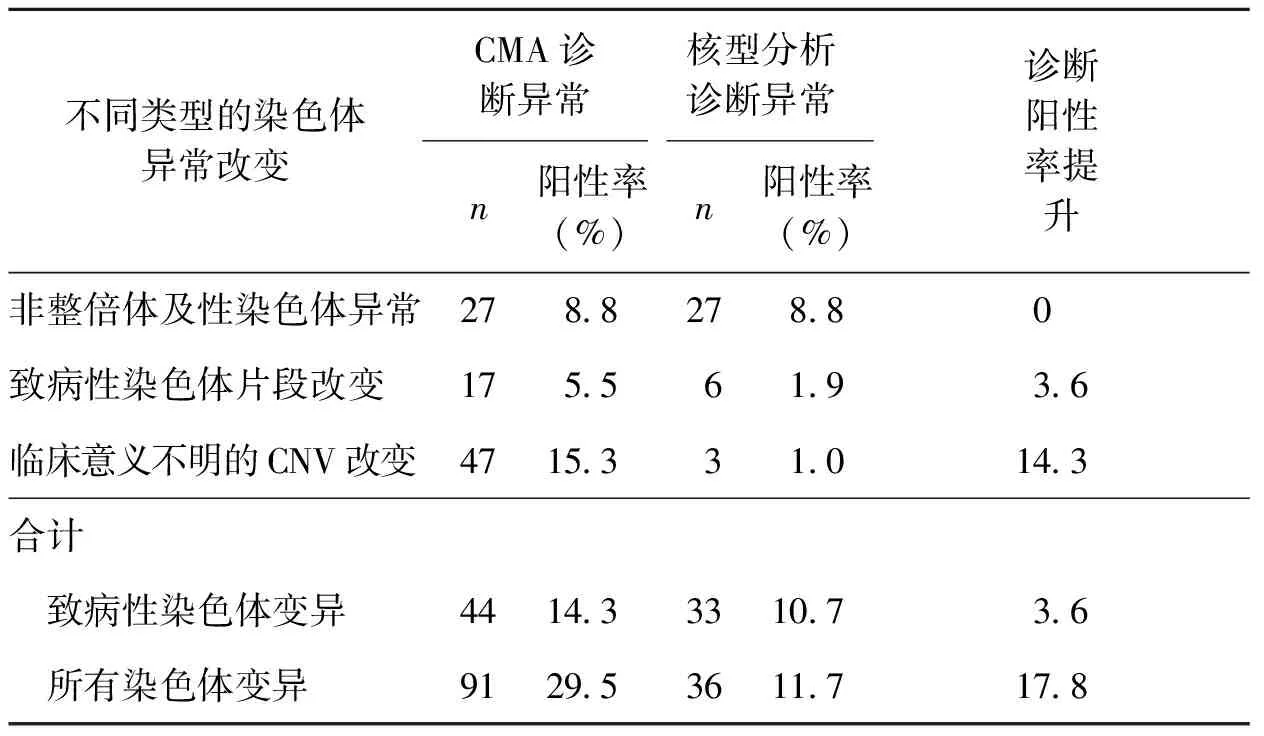
表5 不同染色体异常类型下CMA及核型分析诊断的阳性率分析
讨 论
一、CMA用于产前染色体病诊断的强大作用
对于孕期发现的胎儿多个软指标异常或者胎儿结构异常病例,实现染色体非整倍体异常或微小片段拷贝数异常的精确产前诊断是非常有临床意义的。对于那些确诊有染色体病异常的胎儿,在明确诊断异常来源的前提下可以很好地进行下一胎妊娠再发风险的预判,或者针对发现的亲代染色体异常进行下一胎植入前诊断(PGD)的准备。对于胎儿合并有可生后矫正的结构畸形,除外其染色体病的诊断也可以使孕妇及家人更加放心,坚定他们保留胎儿的决心。本研究的结果显示将CMA技术植入到产前诊断临床检测体系中,对于染色体明确致病的病例诊断可在常规核型分析10.7%的诊断率基础上提升至14.2%,即可多诊断3.5%的染色体异常胎儿。相关的诸多研究也提示类似的结果,说明CMA检测应用在以胎儿超声异常为主的产前诊断人群中能显著提升染色体疾病检出能力[7,12-13]。本研究结果提示,除了产前胎儿超声结构异常外,在产前诊断方面全基因组芯片的临床应用指证还可包括超声软指标异常、不明确的核型分析结果、夫妇之一染色体异常携带者、前次分娩智障等先天缺陷儿、颈后透明带(NT)检测增厚、无创产前筛查提示胎儿存在各种染色体片段改变高风险的产前诊断、不明原因的胎儿宫内发育迟缓等多个方面。
二、不明确意义的检测结果会对CMA在产前诊断中的表现造成困扰及其对策
虽然CMA对于胎儿染色体疾病的诊断能力很强,但该技术在产前病例中的检测常常发现许多与临床表型相关性不明确的拷贝数改变,称之为VOUS[14]。该种拷贝数变异往往是不曾被文献报道过或在公共数据库中未曾收入的罕见变异,拷贝数变异的片段中涉及临床表型功能不明确或剂量效应不明确的基因,或者既存在于正常表型人群中同时也有病理性表型的报导。当产前诊断中出现VOUS往往会对临床遗传咨询带来很大的困难,并给孕妇及家庭带来很多压力,甚至常常导致过度的引产处理。
许多国外相关研究报道VOUS病例往往占全部检测病例的5%以下[15-16]。而本研究中发现的VOUS占所有产前诊断病例的15.3%,按照Affymetrix检测平台通行的拷贝数异常临床报告标准(仅报告≥400 kb的重复片段或≥200 kb的缺失片段)进行修正后,出现VOUS的病例占比降至13.6%。如果将CNV片段长度的报告阈值进一步提升,会显著降低VOUS的报告比率。或者根据美国ACMG的变异报告指南[8]建议,可以将芯片诊断的人群携带率不高的隐性遗传病携带者结果不在报告中体现,则本研究的VOUS比例将下降至11.3%。
造成国内类似不明确意义的检测结果与国外研究相比发生率较高的原因主要有以下几点:(1)国内由于检测费用较高且自费的问题,双亲进行验证性检测的比例不高,造成数据解读困难;(2)国内检测机构的相关工作起步较晚,病例检测数量不大,针对中国人群本地化的检测数据库不完善,更多依赖国外公共数据库进行数据分析;(3)不同的检测平台由于分布探针的密度不同,会造成不同的VOUS发生频率;(4)国内目前尚未形成广泛共识的产前诊断芯片数据判读标准,各家机构的数据判读标准不同,其VOUS的发生频率不一且与国外机构有较大差异。所以呼吁国内相关学术机构尽早开展多中心的机构间检测数据合作,搭建共享的全基因组芯片产前诊断数据平台,并出台统一的结果判读标准,以推动国内产前诊断基因芯片诊断的质量提高。
[1] Brady PD,Vermeesch JR.Genomic microarrays:a technology overview[J].Prenat Diagn,2012,32:336-343.
[2] Lichtenbelt KD,Knoers NV,Schuring-Blom GH.From karyotyping to array-CGH in prenatal diagnosis[J].Cytogenet Genome Res,2011,135:241-250.
[3] Kearney HM,Thorland EC,Brown KK,et al.American College of Medical Genetics standards and guidelines for interpretation and reporting of postnatal constitutional copy number variants[J].Genet Med,2011,13:680-685.
[4] South ST,Lee C,Lamb AN,et al.ACMG Standards and Guidelines for constitutional cytogenomic microarray analysis,including postnatal and prenatal applications:revision 2013[J].Genet Med,2013,15:901-909.
[5] American College of Obstetricians and Gynecologists Committee on Genetics.Committee Opinion No.581:the use of chromosomal microarray analysis in prenatal diagnosis[J].Obstet Gynecol,2013,122:1374-1377.
[6] American College of Obstetricians and Gynecologists’ Committee on Practice Bulletins—Obstetrics;Committee on Genetics;Society for Maternal-Fetal Medicine.Practice Bulletin No.162:Prenatal diagnostic testing for genetic disorders[J].Obstet Gynecol,2016,127:e108-122.
[7] Wapner RJ,Martin CL,Levy B,et al.Chromosomal microarray versus karyotyping for prenatal diagnosis[J].N Engl J Med,2012,6,367:2175-2184.
[8] 染色体微阵列分析技术在产前诊断中的应用协作组.染色体微阵列分析技术在产前诊断中的应用专家共识[J].中华妇产科杂志,2014,49:570-572.
[9] Vanakker O,Vilain C,Janssens K,et al.Implementation of genomic arrays in prenatal diagnosis:the Belgian approach to meet the challenges[J].Eur J Med Genet,2014,57:151-156.
[10] Kearney HM,Thorland EC,Brown KK,et al.American College of Medical Genetics standards and guidelines for interpretation and reporting of postnatal constitutional copy number variation[J].Genetics Med,2011,13:680-685.
[11] Wang H,Chau MHK,Cao Y,et al.Chromosome copy number variants in fetuses with syndromic malformations[J].Birth Defects Res,2017,109:725-733.
[12] Shaffer LG,Rosenfeld JA,Dabell MP,et al.Detection rates of clinically significant genomic alterations by microarray analysis for specific anomalies detected by ultrasound[J].Prenat Diagn,2012,32:986-995.
[13] Pons L,Till M,Alix E,et al.Prenatal microarray comparative genomic hybridization:Experience from the two first years of activity at the Lyon university-hospital[J].J Gynecol Obstet Hum Reprod,2017,46:275-283.
[14] Oneda B,Rauch A.Microarrays in prenatal diagnosis[J].Best Pract Res Clin Obstet Gynaecol,2017,42:53-63.
[15] Schaffer LG,Rosenfeld JA,Dabell MP,et al.Detection rates of clinically significant genomic alterations by microarray analysis for specific anomalies detected by ultrasound[J].Prenat Diagn,2012,32:986-995.
[16] Papoulidis I,Sotiriadis A,Siomou E,et al.Routine use of array comparative genomic hybridization (aCGH) as standard approach for prenatal diagnosis of chromosomal abnormalities.Clinical experience of 1763 prenatal cases[J].Prenat Diagn,2015,35:1269-1277.
[编辑:肖晓辉]
AnalysisofvariationswithclinicallyunambiguoussignificanceinprenatalCMAfor308patientswithhigh-riskpregnancy
JIANG Yu-lin1,QI Qing-wei1*,MENG Hua2,ZHOU Xi-ya1,HAO Na1,XU Zhong-hui2,BAI Jun-jie3,OUYANG Yun-shu2,ZHANG Yi-xiu2,LIU Jun-tao1
1.OB/GYNDepartment,PekingUnionMedicalCollegeHospital,ChineseAcademyofMedicalScience,Beijing100730 2.DepartmentofUltrasound,PekingUnionMedicalCollegeHospital,ChineseAcademyofMedicalScience,Beijing100730 3.BecreativeLab(Beijing)Co.Ltd,Beijing101111
Objective: To explore the application of chromosomal microarray analysis (CMA) in the patients with high-risk pregnancy.
Methods: A total of 308 patients with high-risk pregnant who were admitted to Peking Union Medical College Hospital for prenatal diagnosis,and detected by CMA and karyotype analysis from Jan.1 2014,to Jun.1 2017 were recruited in this study.The CMA results were analyzed and compared with the karyotyping,and the composition and the characteristics of the various clinical unambiguous significances (VOUS) were also analyzed.
Results: A large proportion (88.0%) of the patients was abnormal in fetal structure or soft marker,and had fetal growth restriction or fetal edema.Whole genome detection did not reveal a clear chromosome copy number change (CNV) or clear polymorphic changes in 217 patients (70.5%),and 44 patients (14.3%) were identified with pathological CNV.Among them,21 patients had chromosomal aneuploidy abnormalities,6 patients abnormal sex chromosome number,17 patients chromosome pathogenic CNV.In addition,47 patients had various clinical unambiguous significances of CNV results (15.3%).Among them,23 patients were likely benign,and 21 patients were truly clinical unambiguous significance.There was one patient with loss of heterozygous (LOH) and three patients with likely pathogenic.This study also further analyzed the distribution of fragment size and the type of interpretation based on VOUS in the patients.The positive rate of chromosome abnormality by CMA was 14.2%,compared with 10.7% of the positive rate of karyotyping,and the positive rate increased by 3.5%.
Conclusions: The diagnostic efficiency of genome-wide chromosome microarray chip for fetal structural abnormality is higher than that of karyotype analysis.However,there was also difficulty in the proper genetic counseling for VOUS.At present,the proportion of domestic prenatal diagnosis of clinical unambiguous significance is high,which is related to the low level of local database,lack of domestic specific microarray results interpretation criteria.
Prenatal diagnosis; Copy number variation; High-risk pregnancy; Chromosome microarray analysis; Fetal ultrasound abnormality
10.3969/j.issn.1004-3845.2017.09.003
2017-07-02;
2017-07-19
“十二五”国家科技支撑计划项目(2014BAI06B05)
蒋宇林,男,浙江人,副主任医师,母胎医学专业.(*
)
