动物育种中的统计方法
2017-09-14梅步俊王志华
梅步俊,王志华
(1.内蒙古河套学院农学系,内蒙古 巴彦淖尔 015000;2.内蒙古河套学院土木工程系,内蒙古 巴彦淖尔 015000;3.美国爱荷华州立大学动物科学系,美国爱荷华州 埃姆斯 50010)
动物育种中的统计方法
梅步俊1,3,王志华2
(1.内蒙古河套学院农学系,内蒙古 巴彦淖尔 015000;2.内蒙古河套学院土木工程系,内蒙古 巴彦淖尔 015000;3.美国爱荷华州立大学动物科学系,美国爱荷华州 埃姆斯 50010)
现代动物育种中涉及大量统计问题。由于该领域研究对统计基础依赖性强,系统回顾并梳理动物育种中的统计方法有助于研究者把握这些方法的发展脉络,汲取前人的经验、智慧和教训。本文介绍了现代动物育种中常见统计方法的主要内容,所面临的问题和发展趋势,希望能对广大育种工作者开展进一步研究奠定基础。
动物育种;统计计算;数量遗传;复杂性状
10.16863 /j.cnki.1003-6377.2017.05.003
统计方法使家畜育种完成了从艺术到科学的变革,在这一过程中,许多科学家做出了杰出贡献。大多数家畜育种问题涉及到一系列的定量分析方法和纷繁的数学、统计学计算。例如,选种选配过程可以看做是一个决策问题,可以用线性规划求解;在海量的基因表达数据中挖掘出有生物学意义的基因表达模式实际上是模式识别问题,可以使用聚类分析;预测家畜未来的生产性能或育种值是典型的统计推断问题,育种学家通常使用Henderson的理论解决此类问题。目前,家畜育种中的统计方法依然是许多学术会议的重要议题之一。
1 发展初期成果
将统计方法应用到家畜遗传育种的历史最早可以追溯到Galton(1822-1911)和Pearson(1857-1936)的研究,这些工作实际上早于孟德尔定律被重新发现。1889年,Galton在研究亲本与后代身高之间的关系时发现:后代的身高往往倾向与父母亲身高的中间值,这一趋势被称为“回归现象”。Galton的这项研究为“遗传力”和“预期选择反应”等概念奠定了基础。两个极端亲本群体性状平均值的差异类似于选择差,其子代群体平均值的差异等于选择反应。后代和亲本之间的统计回归是遗传力,Falconer(1913–2004)将选择响应(即遗传获得量GS)对选择差的比值称为现实遗传力[1]。同时,Galton的工作也促进了线性模型在动物育种中的应用,即便到21世纪,动物育种中使用的主要还是线性模型。但是使用非线性模型重新分析Galton的数据发现:父-女、父-子、母-女和母-子身高的回归在67~68英寸处有弯曲。这也说明在不知道明确原因的情况下,依然可以使用统计遗传模型准确地估计遗传参数。Pearson一生写了大量关于性状进化的论文,Henderson在此基础上发表了预测选择偏差的著名论文。Pearson关于选择如何影响群体方差-协方差结构的论文深刻的影响了Henderson,Henderson发展了在正态分布假设和特定选择强度条件下,如何计算方差减小的公式。选择对遗传方差的影响被称为“Bulmer”效应[2]。但是Pearson的公式只是近似值,只适合于候选个体没有亲缘关系和理想分布的情况。但家畜育种中,候选家畜间往往有亲缘关系,信息量也不相等。如参加后裔测定的公畜可能有几千个有记录的后代,而青年公畜往往没有任何后裔生产记录。因此Pearson只提供了比较理想选择方案时的近似公式。
历史上,遗传学面临的一个重要问题是如何统一连续变异的性状和孟德尔性状。Toyama Kametaro(1867–1918)在研究家蚕时发现了第一个动物中的孟德尔性状[3];Yule(1871–1951)第一次统一了连续变异和孟德尔性状,虽然他的观点Pearson并不认同。Fisher和Wright无疑是现代家畜数量遗传学的重要奠基人,他们也是数量遗传学历史上著名的牵扯个人恩怨,充斥恶意人身攻击的Fisher–Wright学术论战的当事人[4]。Fisher(1930)提出了数量遗传学中广泛使用的无穷小模型(infinitesimal model)和方差分析。Wright(1921)使用通径分析和相关分析,提出了近交系数(F);他还推导出孟德尔群体的特性,还包括存在突变的情况下,有限群体随机交配时等位基因频率的分布[5]。Wright还将物理学中描述扩散现象的Fokker-Planck方程(也称为Kolmogorov向前方程)引入群体遗传学[6]。
Fisher的无穷小模型在动物育种中居于重要地位。假设有K个位点,个体的位点k(k=1,2,…,K)贡献A等位基因效应aK(固定值)到基因型值u(加性值):

此处,W是随机指示变量,0、1、2对应该位点的aa、Aa和AA。如果群体处于哈代温伯格(HW)平衡,三种基因型的频率分别为(1-Pk)2、2(1-Pk)Pk和Pk2,这里Pk为位点k随机抽取A等位基因的概率[7]。u的边缘分布依赖于K个位点的联合基因型概率分布。由于u是随机变量的线性组合,如果W是相互独立的(基因型间连锁平衡),随着K的增加,u的分布收敛于正态分布,但是连锁不平衡(LD)会降低收敛率。因为u的均值和方差是有限的,K→∝时单个位点的效应和频率一定变得无限小,取极限时u~N(m,σu2),此处的典型值为0,σu2是加性遗传方差(多基因)[8]。Wright使用相关分析,Malécot使用概率计算分别建立了“配子相似”概念。在此基础上,20世纪60年代Henderson提出奶牛的动物模型,这个模型实际是Fisher模型的向量扩展形式,加性效应u变为育种值向量u,加性遗传方差σu2变为Aσu2,此处是个体间没有近交情况下的加性关系矩阵。A矩阵也可以反映亲缘关系,其元素是两个个体随机抽取一个位点,其等位基因是血缘同源(Identity By Descent,IBD)概率的2倍[9]。
育种值概念的提出也得益于Fisher的另一项贡献,即位点平均基因替代效应。Lush在其家畜育种学课上讲授了这一概念,后来Falconer也在其《数量遗传学导论》一书中介绍了它。和上面相同,假设K个位点处在哈代温伯格(HW)平衡状态,显性效应dk,1-pk=qk,u的平均值为点平均基因替代效应为ak=ak+dk(qk-pk),其AA、和Aa的aa育种值分别为2qkak、(qk-pk)ak、-2pkak。个体育种值u为所有位点育种值之和。育种值依赖于HW假设,其计算公式中的频率和显性偏差是不独立的。因此一般情况下,只有加性效应可以遗传给后代,育种学家最感兴趣的也是ak,狭义的u是只包含加性效应的随机变量(无穷小育种值),可以被定义为所有ak之和。在基因组学出现以前,由于观察不到基因和等位基因效应,推断育种值是传统育种学的核心问题。直到今天,将数量遗传学应用到家畜育种实践时也很少考虑基因,统计方法在家畜育种学中依然起着重要作用,在广泛应用的Henderson方法中,也只有A矩阵考虑遗传(基因)因素。即使在基因组时代,由于使用标记检测QTL需要投入大量经费,企业没有利润可言,因此目前对单个基因对复杂性状的影响依然知之甚少[11]。
2 动物育种中主要问题
在缺乏性状的遗传背景知识时,数量遗传学可以作为获得家畜遗传价值概括性评价的基础。随着人类对生物体代谢途径、基因网络和基因组结构等知识的不断增加,传统数量遗传学方法就略显简单。由于性状之间遗传和环境因素的关联性,我们要使用统计方法合理的分析影响选择的多种效应,就必须使用复杂的多元分析方法[12]。Ronald Fisher(1890–1972)奠定了自然选择的基本理论。动物育种学认为选择进展和加性方差-协方差成正比,在这一观点的启发下,Alan Robertson(1920–1989)进一步发展了自然选择理论,Crow、Kimura和Edwards在文章中给出了该理论较为容易理解的描述。统计方法也是这些自然选择理论的基础,模型的参数估计强依赖于加性遗传假设前提。如果存在非加性遗传变异,为了在模型中考虑未知基因间复杂的交互作用,许多理论的假设都是不切实际的。由于小群体和选择导致的LD使部分遗传方差组分变得很困难。如果基因网络正好处在LD中,推断特定基因对遗传方差的贡献也会变得很麻烦[13]。变异可能产生于直接的代谢途径,也可能间接来源于由LD引起的基因间的相关性。群体遗传学创始人之一的Sewall Wright(1889–1988)引入通径分析来区分直接效应和间接效应,但是这种方法实际上需要考虑基因之间相互关系的背景知识。
现在,我们使用生产性能记录、系谱记录和分子标记信息研究性状的遗传基础,推断家畜遗传价值,寻找基因组区域和表型之间的关联性(即基因组选择)。动物育种中常见的生产性能数据包括:肉用家畜的生长率、采食量;绵羊和山羊的剪毛量和品质;乳用家畜的产奶量、乳成分、繁殖性能和长寿性;多胎品种(如鸡和猪)的产蛋量和产仔数[14]。家畜患病记录(如奶牛乳房炎)往往很难获得,常使用替代变量进行研究,如牛奶的体细胞数(SCC)、体表的寄生虫数量。其它性状,如生存或长寿性状可用删失数据统计方法来处理,即只知道家畜在时刻存活,时刻以后的状态未知;再比如计数性状(如产仔数)或分类性状(如产犊难易性,疾病发展阶段)。因此,家畜育种中的统计模型除使用正态分布外,也使用其它分布,如使用双指数或分布可以使分析更具鲁棒性。
3 动物育种中主要方法
现代育种学之父Lush(1896–1982)认为:可能所有的基因都影响复杂性状。即使在基因组学飞速发展的今天,我们依然不太清楚大多数复杂性状的基因数量,基因的作用机制、等位基因频率及效应等。统计方法将基因组对某个表型的全部效应概括为“基因型值”。表型可由一些数学模型来表示,其中最重要的就是模型中的加性遗传值部分,也被称为育种值。但是,遗传值或模型的其它组分不能被直接观察到,必须由家畜个体及其亲属数据来推断。因为线性模型易于使用,较非线性模型计算强度小,结果便于解释、应用,所以家畜育种中的统计推断过程往往使用线性模型。如果使用大量的基因组标记,理论上可以由此计算家畜的分子相似性,而不再需要详细的系谱记录。但是标记的基因组相似性并不能完全代表致因变异的遗传相似性,除非标记和QTL间有强的LD。QTL也是表示基因组区域和表型有统计显著性关系的抽象概念。动物育种中的标记辅助推断可能最早是Neiman-Sorensen和Robertson在分析牛群体变异时提出的[15]。
虽然许多性状是多基因遗传模式,但是标准的全基因组关联分析(GWAS)却基于表型和单个标记间的回归分析。GWAS结果往往不会出现大量的统计显著性变异,只能解释部分性状变异。不能拒绝GWAS中的零假设往往被认为是多基因模型的佐证,但是从因果论证的角度看是不充分。动物育种数据集可能非常大,如奶牛泌乳记录,且是多元变量(同一模型同时考虑多个性状),多数变量是正态分布(牛奶中的体细胞数浓度和乳房炎指标对数变换后近似为正态分布),但是少数为非正态分布(如离散性状)。数据结构为横断面或纵向数据(肉鸡生长曲线),而且极度不平衡,存在不随机缺失数据。例如,由于选择、生殖障碍或疾病,有第一泌乳期数据的奶牛不一定有第二泌乳期数据[16]。由于一些优秀公牛有更多的后代,数据不完全是随机的,遗传效应的真值不能从环境效应中完全区分出来。家畜育种中的另一个难题是限性性状。
Lush首先将数学模型用在动物育种中,他使用通径分析处理模型中的隐变量。动物育种中的模型往往包括固定效应和随机效应。随机效应包括无穷小模型的,或加性遗传模型的显性和上位效应,群效应、重复测量数据的永久环境效应、窝效应。随机效应是表型之间相关和重复测量数据之间相关性的原因。随机效应的分布由遗传和环境因素的分布参数(方差和协方差)决定。可以将公畜作为固定效应也可以作为随机效应,除非公畜完全近交,公畜的育种值是固定值,但形成配子时不同的等位基因是随机抽样的,会导致遗传上不同的后代。将公畜作为随机效应可以估计育种值,估计的均方误差更稳定,减少预测的过拟合,甚至可以估计没有记录个体的育种值。动物模型中需要估计育种值的个体超过样本数,在基因组时代情况依然一样。但基因组分析模型与数量遗传基本假设有冲突,基因组分析模型使用固定的基因型数据和随机标记效应。大多数动物育种模型认为数据是正态的,有大量的加性基因和微小的替代效应。但是如果认为有无限多的位点或等位基因,发现显著效应的概率就应该是0,但是这明显与分子生物学结果不符,所以MAS(辅助标记选择)将QTL概念引入到动物育种中[17]。
理论上有两种非加性基因效应,显性和上位效应。Comstock和Robinson提出北卡罗林那设计Ⅰ、Ⅱ、Ⅲ估计基因平均显性效应。实际育种中,显性效应主要应用在交配方案问题。但是当显性效应作为随机效应时,因为难以收集携带两个家系等位基因的亲属数据,如全同胞或堂(表)兄妹数据,所以很难获得精确的方差估计。在非近交情况下,加性方差可由A阵构建的显性关系矩阵估计,在近交情况下计算较为复杂。杂交品种往往使用固定效应模型,也可以使用SNP标记估计显性基因组方差,但是由于标记不等于QTL,标记显性方差和遗传方差是有区别的。假设两个等位基因之间无显性,且处于哈代温伯格平衡和LE状态,表型和两个位点等位基因数的线性回归模型为:

此处X1和X2表示给定位点A等位基因的数量,E(.|.)是条件期望[18]。如果回归系数β12为0,则模型变为加性模型。位点1的等位基因替代效应为:

上式表示其决定于位点2的拷贝数。整个群体该性状的平均值为:

因此

和育种值类似,上位效应也依赖于等位基因频率。除非β12非常大,当一个等位基因为稀有基因时,基因频率的改变对平均值的影响主要依赖于加性效应项。即使上位效应对性状有影响,大部分遗传方差也是加性的。因为复杂性状实际上是不同基因编码的酶协同代谢反应的结果,Michaelis-Menten动力学表明底物浓度和反应速率之间是非线性关系,并以非线性方式影响基因产物。近来的文献报道了使用基因组数据发现数量性状中大量基因上位作用的证据。研究中轻易忽略高阶上位作用是不正确的,Taylor和Ehrenreich报道酵母中五个基因之间的交互作用。但是Hill等指出大量上位作用的的上位方差非常小,可能的原因是:如果上位作用具有重要的生物学意义,但是上位效应方差却小于加性效应方差的原因可能是方差组分解释遗传结构的能力是有限度的[19]。Lush指出因为基因间的重组,所以针对上位效应的选择是无效的[20]。因此,育种学家也主要关注育种值对遗传进展的影响,而忽略上位作用在育种中的作用。虽然,Fisher早已提出上位作用的概念,但直到Cockerham和Kempthorne才将这种交互作用剖分为上位组分。Cockerham使用正交多项式,Kempthorne使用IBD概率,他们假设在大的随机群体,且不存在连锁的情况下研究上位作用。上位方差依据影响性状的位点数,可以被剖分为若干正交组分。例如两个位点时,上为方差是加性×加性、加性×显性、显性×加性、显性×显性效应之和。Henderson使用以上结论推断显性和上位遗传效应,并且用BLUP预测总的遗传值。
20世纪60年代,许多家畜或家禽的母体遗传效应逐渐引起育种学家的兴趣。20世纪80年代,动物育种学的主要研究内容是不同环境的方差异质性。表观遗传学一直没有引起统计家畜育种学家的注意,但是Neugebauer建立了以系谱为基础的模型,考虑了父系和母系印记加性效应及其协方差,发现基因组印记可以解释高达25%的加性方差。
4 目前的成就
Lush使用通径系数,建立了评估奶牛公畜遗传值的公式,该模型假设遗传和环境方差是已知的。Robertson研究表明Lush的统计量是群体信息和数据的加权平均值,实际上体现了贝叶斯统计思想。假设公畜的传递力(TA)为s~N(m,vs),如果公畜有n个后代其平均生产性能减去群体平均值为估计为加群平均数:
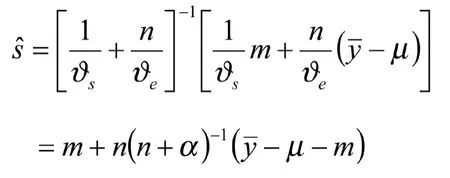
频率学派和以自然函数为基础的统计方法在二十世纪的动物育种领域中居于主要地位。MCMC方法的出现解决了计算高维积分的难题,贝叶斯方法的灵活性和有效性也因此完美的体现在动物育种中。应用最广泛的MCMC方法是Gibbs方法,但是Gibbs方法只适用于分布已知的某些特定的情况[22]。Sorensen首次用Gibbs模拟选择过程中加性遗传方差的变化。随后,贝叶斯统计方法被广泛的用在遗传学的许多领域,如QTL检测、基因定位、系统发育分析、序列比对、群体分化和动植物基因组选择等问题[23]。一些非线性(nolinear)方法也被引入动物育种中用来分析分类或计数性状数据、生存数据和纵向数据,虽然非线性方法在理论上准确性更高,但是在实际应用中,由于计算机计算过程中的舍入误差和非线性往往只存在与数据集的两端等原因,非线性模型并不比线性模型更有优势。混合模型(mixture Model)和稳健分布(robust distribution)也在二十世纪出现在动物育种研究中。除非在实验室研究中,生产实践中动物育种过程也极少出现完全随机交配的理想情况,畜禽群体在历史上的选择过程也不完全清楚。选择和选配如何影响遗传参数估计和预测育种值的准确性等问题依然是育种学家面临的重要问题。
随着全基因组测序技术的发展,大量的二等位基因标记,如SNP标记数据出现动物育种中,动物育种学也因此进入基因组选择时代[24]。Meuwissen首先提出了基因组选择的Bayes A和BayesB方法(Bayes A和BayesB方法的思想或原理之前已经用于解决动物育种问题),其主要过程是通过将数据集拆分为训练集(拟合模型)和测试集(预测),由训练集建立模型估计标记效应或育种值,预测测试集的表型值等[25]。Meuwissen的工作为其后的贝叶斯基因组预测奠定了基础,其后又出现了许多贝叶斯线性回归模型,如 Bayesian Lasso、Bayes C和 Bayes R,这些回归模型基本相同,只是标记先验分布的假设不同。Meuwissen的另一项贡献是在动物育种中引入了交叉验证;为了整合非测序家畜数据和测序家畜数据,提出单步BLUP(SS-BLUP)方法,但是这些方法并没有考虑非加性遗传方差,而检测模型的交互作用需要密集的计算。由于上位效应的回归系数接近于0,问题或模型缩减(shrink),但是基因组分析无疑比传统的基于表型数据的数量遗传学分析存在更多的交互作用检测问题。此外,近几年来,再生核希尔伯特空间回归(RKHS)和神经网络方法被用来检测非加性效应。实际上,广义上讲,BLUP和G-BLUP也是RKHS的特例。
[1]Hill,W.G.,Estimation of realised heritabilities from selection experiments.II.Selection in one direction [J].Biometrics,1972,28(3):p.767-80.
[2]Bulmer,M.G.,The effect of selection on genetic variability:a simulation study[J].Genet Res,1976,28 (2):p.101-17.
[3]Onaga,L.,Toyama Kametaro and Vernon Kellogg[J].silkworm inheritance experiments in Japan,Siam, and the United States,1900-1912.J Hist Biol,2010,43(2):p.215-64.
[4]Dekkers,J.C.,Application of genomics tools to animal breeding[J].Curr Genomics,2012,13(3):p.207-12.
[5]Wright,H.B.,E.J.Pollak,and R.L.Quaas,Estimation of variance and covariance components to determine heritabilities and repeatability of weaning weight in American Simmental cattle[J].J Anim Sci, 1987,65(4):p.975-81.
[6]Gianola,D.and G.J.Rosa,One hundred years of statistical developments in animal breeding[J].Annu Rev Anim Biosci,2015,(3):p.19-56.
[7]Hill,W.G.,Applications of population genetics to animal breeding,from wright,fisher and lush to genomic prediction[J].Genetics,2014,196(1):p.1-16.
[8]Hartley,H.O.and J.N.Rao,Maximum-likelihood estimation for the mixed analysis of variance model[J]. Biometrika,1967,54(1):p.93-108.
[9]Haley,C.S.and S.A.Knott,A simple regression method for mapping quantitative trait loci in line crosses using flanking markers[J].Heredity(Edinb),1992,69(4):p.315-24.
[10]Qanbari,S.,et al.,Classic selective sweeps revealed by massive sequencing in cattle[J].PLoS Genet, 2014,10(2):p.e1004148.
[11]Nishio,M.and M.Satoh,Including dominance effects in the genomic BLUP method for genomic evaluation[J].PLoS One,2014,9(1):p.e85792.
[12]McAdam,A.G.and S.Boutin,Maternal effects and the response to selection in red squirrels[J].Proc Biol Sci,2004,271(1534):p.75-9.
[13]Bijma,P.,Estimating indirect genetic effects:precision of estimates and optimum designs[J].Genetics, 2010,186(3):p.1013-28.
[14]Wang,C.S.,et al.,Response to selection for litter size in Danish Landrace pigs:a Bayesian analysis[J]. Theor Appl Genet,1994,88(2):p.220-30.
[15]Lee,Y.and J.A.Nelder,Analysis of ulcer data using hierarchical generalized linear models[J].Stat Med, 2002,21(2):p.191-202.
[16]Meyer,K.and M.Kirkpatrick,Better estimates of genetic covariance matrices by"bending"using penalized maximum likelihood[J].Genetics,2010,185(3):p.1097-110.
[17].Lander,E.S.and D.Botstein,Mapping mendelian factors underlying quantitative traits using RFLP linkage maps[J].Genetics,1989,121(1):p.185-99.
[18]Hill,W.G.,M.E.Goddard,and P.M.Visscher,Data and theory point to mainly additive genetic variance for complex traits[J].PLoS Genet,2008,4(2):p.e1000008.
[19]Huang,W.,et al.,Epistasis dominates the genetic architecture of Drosophila quantitative traits[J].Proc Natl Acad Sci U S A,2012,109(39):p.15553-9.
[20]Taylor,M.B.and I.M.Ehrenreich,Genetic interactions involving five or more genes contribute to a complex trait in yeast[J].PLoS Genet,2014,10(5):p.e1004324.
[21]Zhang,Q.,et al.,[Comparison of MIVQUE and REML with Monte Carlo simulation[J].Yi Chuan Xue Bao,1995,22(6):p.424-30.
[22]Gjoen,H.M.,H.Simianer,and B.Gjerde,Efficiency of estimation of variance and covariance components from full-sib group means for continuous or binary records[J].J Anim Breed Genet,1997,114(1-6):p. 349-62.
[23]Blasco,A.,The Bayesian controversy in animal breeding[J].J Anim Sci,2001,79(8):p.2023-46.
[24.]Brown,P.O.and D.Botstein,Exploring the new world of the genome with DNA microarrays[J].Nat Genet,1999,21(1 Suppl):p.33-7.
[25].Hayes,B.J.,et al.,Invited review:Genomic selection in dairy cattle:progress and challenges[J].J Dairy Sci,2009,92(2):p.433-43.
Statistical Methods in Animal Breeding
MEI Bu-jun1,3,WANG Zhi-hua2
(1.Agricultural Department,Hetao College,Bayannur 015000,China;2.Department of Civil Engineering,Hetao College,Bayannur 015000,China;3.Department of Animal Science,Iowa State University,Iowa 50010,USA)
Modern animal breeding involves a large number of statistical problems.Because researchers in this field require the foundation of statistical knowledge,a systematic review of the development of statistical methods in animal breeding will allow researchers to take advantage of previous experience and lessons.This article discusses the basic principles of common statistical methods,problems and trends in animal breeding, hoping to provide a foundation for further study of breeders.
animal breeding;statistical calculation;quantitative genetic;complex trait
S813
A
1003-6377(2017)05-0014-07
国家自然科学基金项目(31460594);河套学院教学研究项目(HTXYJZ14005);国家留学基金委项目(201308155140)
梅步俊(1978-),男,副教授,研究方向:统计基因组学。E-mail:meibujun@163.com
2017-06-12,
2017-07-18
