茅山地区南苍术的特异性分子标记研究
2017-08-31王明强汤诗杰薛佳宇
王明强, 汤诗杰, 方 彦, 薛佳宇
〔1. 江苏省中国科学院植物研究所(南京中山植物园) 植物多样性及系统演化研究中心, 江苏 南京 210014;2. 南京森林警察学院, 江苏 南京 210023〕
茅山地区南苍术的特异性分子标记研究
王明强1,①, 汤诗杰1,①, 方 彦2, 薛佳宇1,②
〔1. 江苏省中国科学院植物研究所(南京中山植物园) 植物多样性及系统演化研究中心, 江苏 南京 210014;2. 南京森林警察学院, 江苏 南京 210023〕
以中国野生南苍术〔Atractylodeslancea(Thunb.) DC.〕全部分布区9个居群(包括江苏句容的茅山居群、射乌山居群和小九华山居群,安徽金寨的燕子河居群和天堂寨居群,河南嵩县的五马寺居群和纸坊居群,湖北的保康后坪居群和红安七里坪居群)为研究对象,进行rbcL、matK、psbA-trnH、ITS和nadF片段序列比对及SRAP指纹图谱比较来筛选江苏句容茅山居群南苍术的特异性分子标记。结果表明:通过5个DNA片段的序列比对,江苏句容茅山居群南苍术在nadF片段的第581位点处碱基为T,其余8个居群为A,可以将江苏句容茅山居群与其余8个居群区分开。利用SRAP随机引物筛选出1条江苏句容茅山居群南苍术特有的长度为323 bp的条带,根据该条带序列设计1对SCAR引物,正向引物序列为5′-ATCTTTGCTATTAACTATTAAGCTATTCTTCGCTATTCA-3′,反向引物序列为5′-CGATGTTGAAATTGCTGTCTCTGCTTTCTATTC-3′,该引物可在江苏句容茅山居群所有南苍术单株中扩增得到长度为268 bp的特异性条带,而在其他居群中均不能扩增出该条带。本研究筛选出的2种特异性标记可以为鉴定南苍术的道地性提供可靠的技术支持。
南苍术; 道地性; 茅山地区;nadF片段; SRAP-SCAR
南苍术〔Atractylodeslancea(Thunb.) DC.〕,又名茅苍术,为菊科(Asteraceae)苍术属(AtractylodesDC.)植物,主要分布于江苏、安徽、湖北和河南等狭小区域,生于野生山坡草地、林下、灌丛及岩石缝隙中[1]。苍术是中国传统中药材[2],江苏茅山地区的南苍术质量尤佳,美誉自古有之。《本草经集注》[3]中记录苍术“今处处有之,以蒋山、白山、茅山者为胜”,即今江苏的南京、句容等地,亦可知茅山南苍术早已是道地药材。
彭华胜等[4]观察到,南苍术叶及根状茎的形态随海拔升高呈现连续的变化,且在不同生长期和不同生境,同一居群不同个体间也有较大差异,不同居群的个体从形态上很难区分。在挥发油总含量上,茅山地区南苍术较其他地区的南苍术低[5],且挥发油中的苍术素和苍术酮含量高,不含或含较少的茅术醇和β-桉叶醇[6-7],但挥发油含量的测定周期较长、检测成本较高,且个体间差异大[8],因此,化学成分含量无法成为一种有效而稳定的鉴别依据。
与形态和化学成分相比,植物的基因组DNA是稳定的遗传物质,利用DNA分子标记鉴别道地药材更为快速、准确。已有研究结果显示,利用DNA序列差异可以鉴别道地药材。余永邦等[9]对14个地区太子参〔Pseudostellariaheterophylla(Miq.) Pax〕的ITS序列进行了扩增和测序,结果表明ITS序列可以鉴别江苏产区及其他非道地产区的太子参;张宏意等[10]也用ITS序列鉴别四川、广东、湖北等地的何首乌〔Fallopiamultiflora(Thunb.) Harald.〕;孔德政等[11]用psbA-trnH序列分析牛膝(AchyranthesbidentataBlume)道地产区河南8个区域的野生居群,可以将新乡九莲山、南阳老界岭、信阳灵山和南阳淮源的牛膝鉴别开。南苍术也有类似的研究,如利用trnL-F和ITS[12-13]等序列可以将南苍术与苍术属其他植物区分开,但尚无利用DNA片段序列差异鉴别道地产区与非道地产区南苍术的相关研究报道。
此外,利用随机引物扩增图谱差异也可以鉴定道地药材,如利用RAPD、SSR和ISSR图谱等[14-17]可以区分不同来源的球花石斛(DendrobiumthyrsiflorumRchb. f.)、当归〔Angelicasinensis(Oliv.) Diels〕、铁皮石斛(DendrobiumofficinaleKimura et Migo)和贝母(Fritillariaspp.)等药材,将随机引物扩增出的特有条带测序后转化成特异性SCAR标记,能显著提高随机引物标记的稳定性及可重复性,在川牛膝(CyathulaofficinalisK. C. Kuan)和宁夏枸杞(LyciumbarbarumLinn.)等[18-19]药材的鉴别研究中均有报道。在针对南苍术的研究中,任冰如等[20]利用扩增出的RAPD指纹图谱差异,将南苍术分为南苍术类群和大别山类群;郭兰萍等[21]利用RAPD探讨不同产地南苍术遗传多样性时,认为茅山南苍术单独为一类。但目前尚无利用随机引物的指纹图谱来开发用于南苍术道地性鉴别的特异性SCAR分子标记。
本文利用南苍术不同居群基因组序列差异及特异性分子标记,建立南苍术道地性鉴别的分子鉴别体系,以期为道地药材的种质保护提供简便而稳定的检测方法。
1 材料和方法
1.1 材料
在中国野生南苍术分布区江苏、安徽、河南及湖北采集9个居群100个单株(表1),每株取2~3枚幼嫩且无病虫害叶片,清洗后保存于-80 ℃,用于总DNA的提取。
实验材料经江苏省中国科学院植物研究所杭悦宇研究员鉴定,凭证标本存放于江苏省中国科学院植物研究所标本馆(NAS)。
1.2 方法
1.2.1 样品总DNA提取 每个样品取0.1 g叶片,用EasyPure Plant Genomic DNA Kit试剂盒(北京全式金生物技术有限公司)提取总DNA,溶于200 μL灭菌超纯水中,-20 ℃保存、备用。
表1 南苍术9个居群供试材料基本采集信息
Table 1 Basic collection information of materials tested from nine populations ofAtractylodeslancea(Thunb.) DC.

编号No.采集地Locality株数Numberofindividuals采集号No.ofcollectionAL1江苏句容茅山MaoshaninJurongofJiangsu20201510021-201510040AL2江苏句容射乌山ShewushaninJurongofJiangsu10201510011-201510020AL3江苏句容小九华山XiaojiuhuashaninJurongofJiangsu10200606001-200606010AL4安徽金寨燕子河YanziheinJinzhaiofAnhui10200606021-200606030AL5安徽金寨天堂寨TiantangzhaiinJinzhaiofAnhui10201508011-201508020AL6河南嵩县五马寺WumasiinSongxianofHe’nan10201509001-201509010AL7河南嵩县纸坊ZhifanginSongxianofHe’nan10201509011-201509020AL8湖北保康后坪HoupinginBaokangofHubei10200311001-200311010AL9湖北红安七里坪QilipinginHonganofHubei10201510001-201510010
1.2.2 5个DNA片段序列测定
1.2.2.1 引物信息 对rbcL、matK、psbA-trnH、ITS和nadF5个DNA片段进行扩增。其中,前4个DNA片段为国际通用的条形码片段,为区分物种或居群较常用的DNA片段;nadF片段为作者自行筛选的DNA片段,变异率较高,适合进行种下水平的区分。rbcL、matK、psbA-trnH和ITS片段的扩增引物选择通用引物,根据GenBank数据库中发布的菊科cpDNA序列,利用Oligo 6软件自行设计nadF片段的引物,各引物信息见表2。
表2 南苍术9个居群的PCR扩增片段引物信息
Table 2 Primer information of PCR amplified fragment from nine populations ofAtractylodeslancea(Thunb.) DC.

片段Fragment引物Primer引物序列(5′→3′)Primersequence(5′→3′)来源SourcerbcL1FATGTCACCACAAACAGAAACFa,etal.[22]724RTCGCATGTACCTGCAGTAGCmatKtrnK-3914FGCTCCAGAAGATGTTGATCGBayer,etal.[23]matK-1541RGGGGTTGCTAACTCAACGGpsbA-trnHpsbA-FGTTATGCATGAACGTAATGCTCSang,etal.[24]psbA-trnH-RCGCGCATGGTGGATTCACAATCCTate,etal.[25]ITSny47-FAACAAGGTTTCCGTAGGTGAMark,etal.[26]AST1TGAGGACGCTTCTCCAGACnadFrpl132-FCCGATTCACCAACTCTTATCTATTTCTArpl132-RCATTGGATGTGAAAGACATGTATTGTTC
1.2.2.2 序列扩增、测序及数据处理 每个居群选取5个单株用于DNA片段扩增和序列测定。扩增反应在PerkinElmer GeneAmp PCR System 9600(美国PerkinElmer公司)上进行,试剂购自广州东盛生物科技有限公司。PCR反应体系总体积为50.0 μL,包含模板DNA 1.0 μL,2×Reaction Mix(含20 mmol·L-1Tris-HCl、100 mmol·L-1KCl、3 mmol·L-1MgCl2、400 μmol·L-1dNTPs和0.02 g·mL-1溴酚蓝)25.0 μL,10 μmol·L-1引物各2.0 μL,2.5 U·μL-1TaqDNA聚合酶1.0 μL,最后用双蒸水补至50.0 μL。matK、rbcL、psbA-trnH、ITS和nadF片段的扩增程序均为:95 ℃预变性5 min;95 ℃变性45 s,56 ℃退火45 s,70 ℃延伸90 s,35个循环;最后72 ℃延伸10 min。PCR产物经质量体积分数0.8%~1.0%琼脂糖凝胶电泳后,溴化乙锭(EB)染色,用Gel Doc图像分析仪(江苏省捷达科技发展有限公司)观察。
上述PCR扩增产物交由上海华大基因科技有限公司进行双向测序。测序结果利用Sequencher 4.5软件进行拼接,去除低质量的序列;通过MEGA 5.1对序列进行分析,用NJ(Neighbor-Joining)法构建系统进化树评估南苍术各居群间的亲缘关系。
1.2.3 SRAP分析方法
表3 用于江苏句容茅山居群南苍术特异性条带筛选的SRAP引物
Table 3 SRAP primers used for screening specific bands ofAtractylodeslancea(Thunb.) DC. from Maoshan population in Jurong of Jiangsu

引物Primer引物序列(5′→3′)Primersequence(5′→3′)引物Primer引物序列(5′→3′)Primersequence(5′→3′)SRAP-F1TGAGTCCAAACCGGATASRAP-R5GACTGCGTACGAATTAACSRAP-F2TGAGTCCAAACCGGAGCSRAP-R6GACTGCGTACGAATTGCASRAP-F3TGAGTCCAAACCGGAATSRAP-R7GACTGCGTACGAATTCCASRAP-F4TGAGTCCAAACCGGACCSRAP-R8GACTGCGTACCAATTCGAGSRAP-F5TGAGTCCAAACCGGAAGSRAP-R9GACTGCGTACCAATTCGCCSRAP-F6TGAGTCCAAACCGGACGSRAP-R10GACTGCGTACCAATTCTCCSRAP-F7TGAGTCCAAACCGGACTSRAP-R11GACTGCGTACGAATTTCGSRAP-F8TGAGTCCAAACCGGAGGSRAP-R12GACTGCGTACGAATTGGTSRAP-R1GACTGCGTACGAATTAATSRAP-R13GGCTTGAACGAGTGACTGASRAP-R2GACTGCGTACGAATTTGCSRAP-R14GACTGCGTACGAATTCAASRAP-R3GACTGCGTACGAATTGACSRAP-R15GACTGCGTACGAATTCTGSRAP-R4GACTGCGTACGAATTTGASRAP-R16GACTGCGTACGAATTGTC
1.2.3.1 SRAP引物筛选 参照文献[27-28],用于茅山地区南苍术特异性条带筛选的SRAP引物(表3)由上海华大基因科技有限公司合成,其中正向引物8条,反向引物16条,两两组合共有128种组合方式。分别取每居群所有单株的20.0 μL总DNA混匀,作为SRAP引物筛选的模板。在Biometra TAdvanced型PCR仪(德国耶拿分析仪器股份公司)进行PCR反应,反应体系总体积为20.0 μL,包含DNA模板1.0 μL,2×Reaction Mix(含20 mmol·L-1Tris-HCl、100 mmol·L-1KCl、3 mmol·L-1MgCl2、400 μmol·L-1dNTPs和0.02 g·mL-1溴酚蓝)10.0 μL,10 mmol·L-1正向引物和反向引物各0.7 μL,2.5 U·μL-1TaqDNA聚合酶0.4 μL,用双蒸水补齐至20.0 μL。SRAP分子标记的扩增程序为:94 ℃预变性5 min;94 ℃变性1 min,35 ℃退火45 s,72 ℃延伸1 min,5个循环;94 ℃变性1 min,52 ℃退火45 s,72 ℃延伸1 min,35个循环;72 ℃延伸10 min。扩增产物经质量体积分数2.0%琼脂糖凝胶电泳,EB染色,电压60 V,电泳1.5 h后凝胶成像系统观察、拍照,用2 000 bp DNA Ladder作为分子质量标记。
1.2.3.2 SRAP-SCAR特异性条带的克隆及测序选择江苏句容茅山居群南苍术扩增出的特异性条带,对特异性条带进行切胶回收。将回收的目的DNA片段连接到pMD19-T载体〔宝生物工程(大连)有限公司〕,并将连接产物转入大肠杆菌DH5α感受态细胞〔宝生物工程(大连)有限公司〕。在含有氨苄的细菌培养基上于37 ℃培养12 h。挑取单菌落进行PCR检验,将含有目的条带的克隆交由上海华大基因科技有限公司进行测序。根据DNA序列分析结果,设计1对特异性引物用于后续实验。
1.2.3.3 SRAP-SCAR标记的验证 用合成的SCAR引物对南苍术的9个居群100个单株进行验证。在Biometra TAdvanced型PCR仪进行PCR反应,反应体系总体积为20.0 μL,包含DNA模板1.0 μL,2×Reaction Mix(含20 mmol·L-1Tris-HCl、100 mmol·L-1KCl、3 mmol·L-1MgCl2、400 μmol·L-1dNTPs和0.02 g·mL-1溴酚蓝)10.0 μL,10 mmol·L-1正向引物和反向引物各0.7 μL,2.5 U·μL-1TaqDNA聚合酶0.4 μL,最后用双蒸水补齐至20.0 μL。扩增程序为:95 ℃预变性5 min;95 ℃变性45 s,56 ℃退火45 s,72 ℃延伸30 s,30个循环;72 ℃延伸10 min。PCR扩增产物经质量体积分数1.0%琼脂糖凝胶电泳,EB染色,电压100 V,电泳0.5 h后凝胶成像系统观察、拍照,采用2 000 bp DNA Ladder作为分子质量标记。
2 结果和分析
2.1 5个DNA片段的分析结果
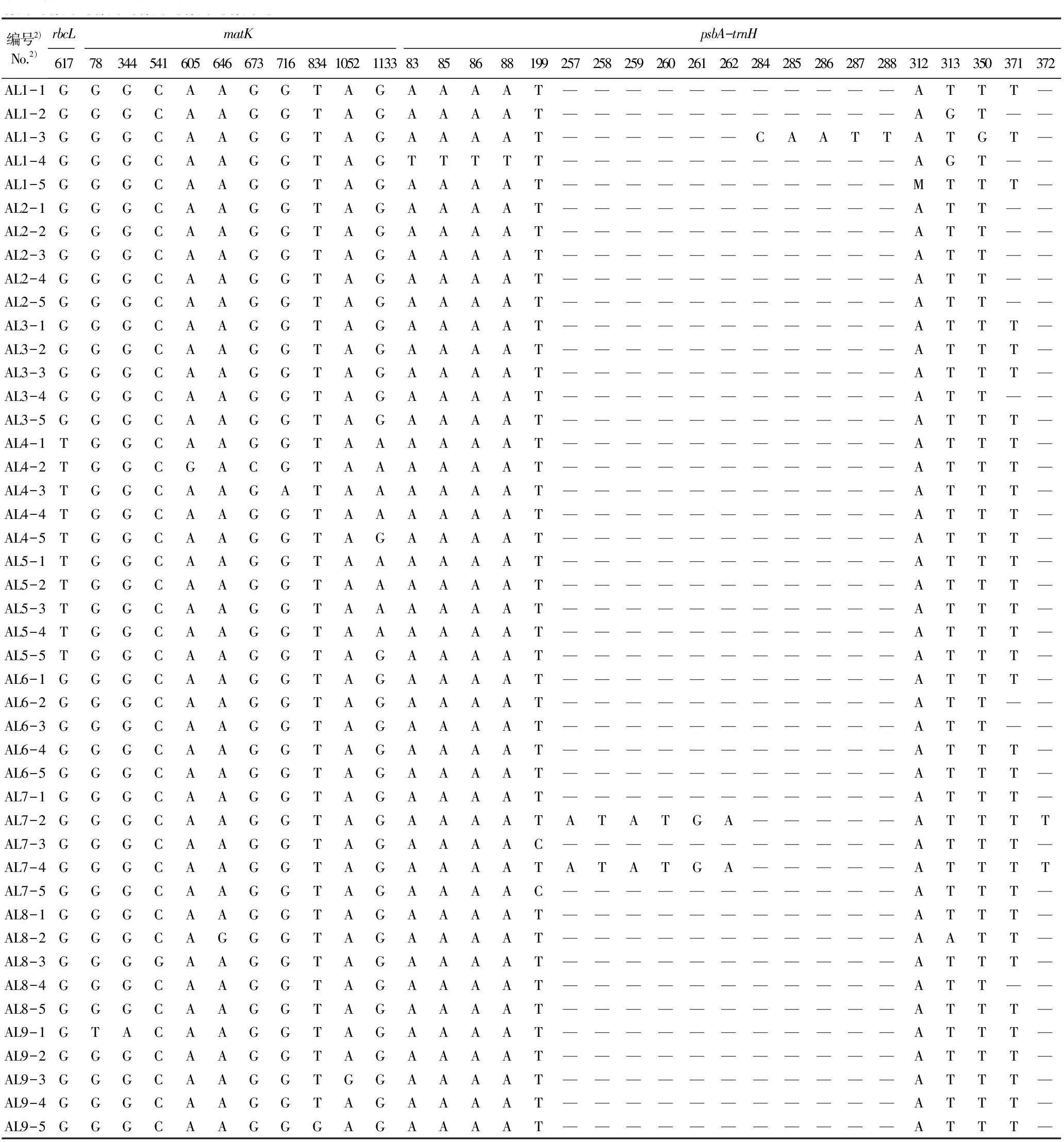
2.1.1 5个DNA片段的特征分析 南苍术9个居群rbcL、matK、psbA-trnH、ITS和nadF片段的变异位点见表4。
由表4可以看出:南苍术9个居群rbcL片段的变异位点较少,安徽2个居群南苍术具有独特的序列变异位点,在第617位点处安徽2个居群为T,而其余7个居群均为G。psbA-trnH片段在南苍术居群间及居群内虽然有变异位点,但只是个别单株的变异,无法代表所在地区的南苍术。ITS片段的变异位点最多,但具有区分度的位点仅有1个,在第66位点处河南2个居群为A,而其余7个居群均为G,能将河南2个居群南苍术鉴别出来;江苏3个居群、安徽2个居群及湖北红安七里坪居群在第9、第42、第55、第87、第115、第160、第197、第217、第403、第510和第597位点处分别为G、C、G、C、A、A、G、T、A、A、G,河南2个居群为A、T、A、T、G、C、C、C、T、T、A,可以将上述居群区分开。但是,matK、rbcL、psbA-trnH和ITS 4个片段均不能将江苏句容茅山居群南苍术与其余居群南苍术区分开。
由表4还可以看出:江苏句容茅山居群南苍术在nadF片段的第581位点处为T,其余8个居群为A,可以将江苏句容茅山居群南苍术与其余8个居群的南苍术区分开。另外,在nadF片段的第76和第343位点处,江苏3个居群为A,其余8个居群为T。在nadF片段的第161位点处,江苏3个居群和安徽金寨天堂寨居群为C,安徽金寨燕子河居群、河南2个居群和湖北2个居群为T。
表4 南苍术9个居群5个DNA片段变异位点的比较1)
Table 4 Comparison on variable sites in five DNA fragments from nine populations ofAtractylodeslancea(Thunb.) DC.1)
编号2)No.2)rbcLmatKpsbA-trnH617783445416056466737168341052113383858688199257258259260261262284285286287288312313350371372AL1-1GGGCAAGGTAGAAAAT———————————ATTT—AL1-2GGGCAAGGTAGAAAAT———————————AGT——AL1-3GGGCAAGGTAGAAAAT——————CAATTATGT—AL1-4GGGCAAGGTAGTTTTT———————————AGT——AL1-5GGGCAAGGTAGAAAAT———————————MTTT—AL2-1GGGCAAGGTAGAAAAT———————————ATT——AL2-2GGGCAAGGTAGAAAAT———————————ATT——AL2-3GGGCAAGGTAGAAAAT———————————ATT——AL2-4GGGCAAGGTAGAAAAT———————————ATT——AL2-5GGGCAAGGTAGAAAAT———————————ATT——AL3-1GGGCAAGGTAGAAAAT———————————ATTT—AL3-2GGGCAAGGTAGAAAAT———————————ATTT—AL3-3GGGCAAGGTAGAAAAT———————————ATTT—AL3-4GGGCAAGGTAGAAAAT———————————ATT——AL3-5GGGCAAGGTAGAAAAT———————————ATTT—AL4-1TGGCAAGGTAAAAAAT———————————ATTT—AL4-2TGGCGACGTAAAAAAT———————————ATTT—AL4-3TGGCAAGATAAAAAAT———————————ATTT—AL4-4TGGCAAGGTAAAAAAT———————————ATTT—AL4-5TGGCAAGGTAGAAAAT———————————ATTT—AL5-1TGGCAAGGTAAAAAAT———————————ATTT—AL5-2TGGCAAGGTAAAAAAT———————————ATTT—AL5-3TGGCAAGGTAAAAAAT———————————ATTT—AL5-4TGGCAAGGTAAAAAAT———————————ATTT—AL5-5TGGCAAGGTAGAAAAT———————————ATTT—AL6-1GGGCAAGGTAGAAAAT———————————ATTT—AL6-2GGGCAAGGTAGAAAAT———————————ATT——AL6-3GGGCAAGGTAGAAAAT———————————ATT——AL6-4GGGCAAGGTAGAAAAT———————————ATTT—AL6-5GGGCAAGGTAGAAAAT———————————ATTT—AL7-1GGGCAAGGTAGAAAAT———————————ATTT—AL7-2GGGCAAGGTAGAAAATATATGA—————ATTTTAL7-3GGGCAAGGTAGAAAAC———————————ATTT—AL7-4GGGCAAGGTAGAAAATATATGA—————ATTTTAL7-5GGGCAAGGTAGAAAAC———————————ATTT—AL8-1GGGCAAGGTAGAAAAT———————————ATTT—AL8-2GGGCAGGGTAGAAAAT———————————AATT—AL8-3GGGGAAGGTAGAAAAT———————————ATTT—AL8-4GGGCAAGGTAGAAAAT———————————ATT——AL8-5GGGCAAGGTAGAAAAT———————————ATTT—AL9-1GTACAAGGTAGAAAAT———————————ATTT—AL9-2GGGCAAGGTAGAAAAT———————————ATTT—AL9-3GGGCAAGGTGGAAAAT———————————ATTT—AL9-4GGGCAAGGTAGAAAAT———————————ATTT—AL9-5GGGCAAGGGAGAAAAT———————————ATTT—
续表4 Table 4 (Continued)

编号2)No.2)ITSnadF942556683871061151601941972174034644675105977576154161343395411468473581628704705AL1-1GCGGCCCAAGGTACTAGTACCATTACTTTTAL1-2GCGGCCCAAGGTACTAGTACCATTACTTTTAL1-3GCGGYCCAAGGTAYTAGTACCAAT—CTTT—AL1-4GCGGCCCAAGGTAYTAGTACCAAT—CTAT—AL1-5GCGGYCCAAGGTACTAGTACCAAT—CTAT—AL2-1GCGGCCCAAGGTACTAGTACCATT—CAAT—AL2-2GCGGCCCAAGGTACTAGTACCAATACAATTAL2-3GCGGCCCAAGGTACTAGTACCAATACAATTAL2-4GCGGCCCAAGGTACTAGTACCAATACAATTAL2-5GCGGCCCAAGGTACTAGTACCAAT—CAAT—AL3-1GCGGCCCAAGGTACTAGTACCAAT—TAAT—AL3-2GCGGCCCAAGGTACTAGTACCAATATAAT—AL3-3GCGGCCCAAGGTACTAGTACCAATACATT—AL3-4GCGGCCCAAGGTACTAGTACCAAT—CAATTAL3-5GCGGCCCAAGGTACTAGTACCAAT—CATTTAL4-1GCGGCCCAAGGTACTAGTTCTTAT—CATT—AL4-2GCGGCCCAAGGTACTAGTTCTTAT—CATT—AL4-3GCGGCCCAAGGTACTAGTTCTTAT—CATT—AL4-4GCGGCCCAAGGTACTAGTTCTTAT—CAT——AL4-5GCGGCCCAAGGTACTAGTTCTTAT—CATT—AL5-1GCGGCCCAAGGTACTAGTTCCTAT—CATT—AL5-2GCGGCCCAAGGTACTAGTTCCTAT—CAT——AL5-3GCGGCCCAAGGTACTAGTTCCTAT—CATT—AL5-4GCGGCCCAAGGTACTAGTTCCTAT—CATT—AL5-5GCGGCCCAAGGTACTAGTTCCTAT—CATT—AL6-1ATAACTCGCGCCTCCTATTGTTAT—CAT——AL6-2ATAACTCGCTCCTCTTATTCTTAT—CATTTAL6-3ATAACTCGCGCCTCCTATTCTTAT—CAT——AL6-4ATAACTCGCGCCTCCTAGTCTTAT—CATT—AL6-5ATAACTCGCGCCTCCTAGTCTTAT—CATT—AL7-1ATAACTCGCGCCTCCTAGTCTTAT—CAT——AL7-2ATAACTCGCGCCTCCTATTGTTAT—CATT—AL7-3ATAACTCGCGCCTCCTATTCTTAT—CATTTAL7-4ATAACTCGCGCCTCCTATTCTTAT—CAT——AL7-5ATAACTCGCGCCTCCTATTCTTAG—CAT——AL8-1GCGGCCCAAGGTACTAGTTCTTAT—CATT—AL8-2RYRGCYCRMGSYWCYWRTTCTTAT—CATT—AL8-3RYRGCYCRMGSYWCYWRTTCTTAT—CAT——AL8-4RYRGCYCRMGSYWCYWRTTCTTAT—CAA——AL8-5ATAGCTCGCGCCTCCTATTCTTAT—CAA——AL9-1GCGGCCCAAGGTACTAGTTCTTAT—CAAT—AL9-2GCGGCCCAAGGTACTAGTTCTTAT—CAAT—AL9-3GCGGCCCAAGGTACTAGTTCTTAT—CAT——AL9-4GCGGCCCAAGGTACTAGTTCTTAT—CAT——AL9-5GCGGCCCAAGGTACTAGTTCTTAT—CATT—
1)rbcL:rbcL片段变异位点的碱基Bases at variable sites inrbcLfragment;matK:matK片段变异位点的碱基Bases at variable sites inmatKfragment;psbA-trnH:psbA-trnH片段变异位点的碱基Bases at variable sites inpsbA-trnHfragment; ITS: ITS片段变异位点的碱基Bases at variable sites in ITS fragment;nadF:nadF片段变异位点的碱基Bases at variable sites innadFfragment. —: 碱基缺失Base missing.
2)AL1-1,AL1-2,AL1-3,AL1-4,AL1-5: 江苏句容茅山居群5个单株Five individuals from Maoshan population in Jurong of Jiangsu; AL2-1,AL2-2,AL2-3,AL2-4,AL2-5: 江苏句容射乌山居群5个单株Five individuals from Shewushan population in Jurong of Jiangsu; AL3-1,AL3-2,AL3-3,AL3-4,AL3-5: 江苏句容小九华山居群5个单株Five individuals from Xiaojiuhuashan population in Jurong of Jiangsu; AL4-1,AL4-2,AL4-3,AL4-4,AL4-5: 安徽金寨燕子河居群5个单株Five individuals from Yanzihe population in Jinzhai of Anhui; AL5-1,AL5-2,AL5-3,AL5-4,AL5-5: 安徽金寨天堂寨居群5个单株Five individuals from Tiantangzhai population in Jinzhai of Anhui; AL6-1,AL6-2,AL6-3,AL6-4,AL6-5: 河南嵩县五马寺居群5个单株Five individuals from Wumasi population in Songxian of He’nan; AL7-1,AL7-2,AL7-3,AL7-4,AL7-5: 河南嵩县纸坊居群5个单株Five individuals from Zhifang population in Songxian of He’nan; AL8-1,AL8-2,AL8-3,AL8-4,AL8-5: 湖北保康后坪居群5个单株Five individuals from Houping population in Baokang of Hubei; AL9-1,AL9-2,AL9-3,AL9-4,AL9-5: 湖北红安七里坪居群5个单株Five individuals from Qiliping population in Hongan of Hubei.
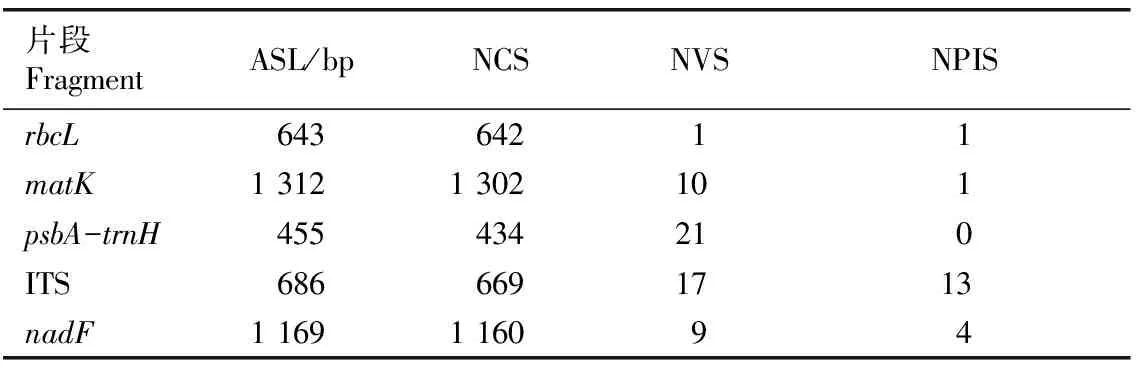
南苍术9个居群rbcL、matK、psbA-trnH、ITS和nadF5个DNA片段的分析结果见表5。由表5可以看出:这5个片段的可匹配序列长度分别为643、1 312、455、686和1 169 bp。当空缺(gap)始终作缺失(missing)处理时,rbcL片段有642个保守位点,保守性最高;psbA-trnH片段有434个保守位点,保守性最低;psbA-trnH片段的变异位点最多,有21个,变异性最大;ITS片段的信息位点最多,有13个。
表5 南苍术9个居群5个DNA片段信息1)
Table 5 Information of five DNA fragments from nine populations ofAtractylodeslancea(Thunb.) DC.1)
片段FragmentASL/bpNCSNVSNPISrbcL64364211matK13121302101psbA-trnH455434210ITS6866691713nadF1169116094
1)ASL: 可匹配序列长度Alignment sequence length; NCS: 保守位点数Number of conserved sites; NVS: 变异位点数Number of variable sites; NPIS: 信息位点数Number of parsim-informative sites.

AL1: 江苏句容茅山居群Maoshan population in Jurong of Jiangsu; AL2: 江苏句容射乌山居群Shewushan population in Jurong of Jiangsu; AL3: 江苏句容小九华山居群Xiaojiuhuashan population in Jurong of Jiangsu; AL4: 安徽金寨燕子河居群Yanzihe population in Jinzhai of Anhui; AL5: 安徽金寨天堂寨居群Tiantangzhai population in Jinzhai of Anhui; AL6: 河南嵩县五马寺居群Wumasi population in Songxian of He’nan; AL7: 河南嵩县纸坊居群Zhifang population in Songxian of He’nan; AL8: 湖北保康后坪居群Houping population in Baokang of Hubei; AL9: 湖北红安七里坪居群Qiliping population in Hongan of Hubei. 分支上数据为支持率Datums above the branches are support rate.图1 基于5个DNA片段联合构建的南苍术9个居群的NJ系统进化树Fig. 1 NJ phylogenetic tree of nine populations of Atractylodes lancea (Thunb.) DC. based on combining five DNA fragments
2.1.2 基于5个DNA片段联合的南苍术居群间聚类分析 基于5个DNA片段联合构建的南苍术9个居群间的NJ系统进化树(图1)显示:与江苏句容茅山居群南苍术亲缘关系较近的是江苏句容的小九华山居群和射乌山居群,其次分别是安徽2个居群、湖北2个居群、河南2个居群,推测江苏句容茅山居群南苍术与其余居群的亲缘关系与南苍术分布的地理距离成正相关。
2.2 SRAP-SCAR分析结果
2.2.1 与茅山地区南苍术特异性相关的SRAP分子标记筛选 应用128对SRAP引物分别对供试南苍术9个居群的总DNA混合样品进行PCR扩增,根据指纹图谱进行筛选,结果显示:在引物对SRAP-F8/R14的PCR扩增图谱中,江苏句容茅山居群在约300 bp处有1条特有条带,而其余8个居群均无此条带(图2)。
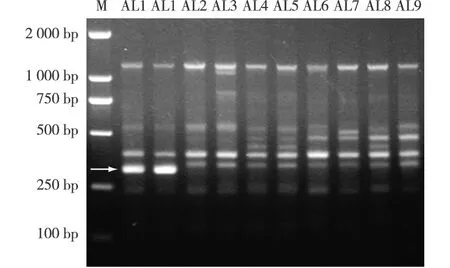
M: DNA marker; AL1: 江苏句容茅山居群Maoshan population in Jurong of Jiangsu; AL2: 江苏句容射乌山居群Shewushan population in Jurong of Jiangsu; AL3: 江苏句容小九华山居群Xiaojiuhuashan population in Jurong of Jiangsu; AL4: 安徽金寨燕子河居群Yanzihe population in Jinzhai of Anhui; AL5: 安徽金寨天堂寨居群Tiantangzhai population in Jinzhai of Anhui; AL6: 河南嵩县五马寺居群Wumasi population in Songxian of He’nan; AL7: 河南嵩县纸坊居群Zhifang population in Songxian of He’nan; AL8: 湖北保康后坪居群Houping population in Baokang of Hubei; AL9: 湖北红安七里坪居群Qiliping population in Hongan of Hubei. 箭头示江苏句容茅山居群特有条带The arrow indicates specific bands of population at Maoshan in Jurong of Jiangsu.图2 引物对SRAP-F8R14的南苍术9个居群总DNA的PCR扩增图谱Fig. 2 PCR amplification pattern of total DNA from nine populations of Atractylodes lancea (Thunb.) DC. by primer pair SRAP-F8R14
2.2.2 茅山地区南苍术特有条带的序列测定及与茅山地区南苍术相关的SCAR引物设计 江苏句容茅山居群南苍术特有条带所包含的序列是该居群的特有序列,根据该序列设计的引物仅在江苏句容茅山居群南苍术单株中扩增得到条带,而在其余8个居群南苍术单株中则无法得到。
对引物对SRAP-F8R14扩增片段的测序结果显示该片段长度为323 bp,其核苷酸序列见图3。
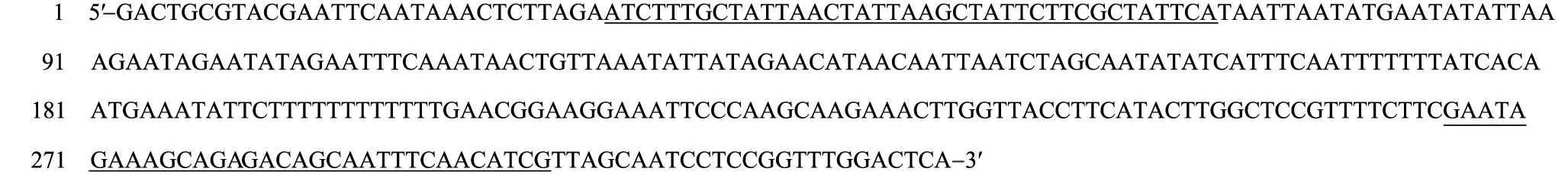
下划线部分为设计的SCAR引物The underline parts are the designed SCAR primers.图3 引物对SRAP-F8R14在江苏句容茅山居群南苍术中扩增出特有片段的DNA序列Fig. 3 DNA sequence of specific fragment amplified by primer pair SRAP-F8R14 from Atractylodes lancea (Thunb.) DC. from Maoshan population in Jurong of Jiangsu
根据引物设计的一般原则,设计1对SCAR引物,正向引物序列为5′-ATCTTTGCTATTAACTATTAAGCTATTCTTCGCTATTCA-3′,反向引物序列为5′-CGATGTTGAAATTGCTGTCTCTGCTTTCTATTC-3′,此对引物扩增出的序列长度为268 bp。
2.2.3 SRAP-SCAR标记在南苍术各居群单株中的验证 用设计的SCAR引物对南苍术9个居群100个单株进行验证,结果见图4。除江苏句容茅山居群南苍术的20个单株在268 bp处扩增出条带外,其余8个居群的所有单株均未在该处扩增出条带。

AL1: 江苏句容茅山居群Maoshan population in Jurong of Jiangsu; AL2: 江苏句容射乌山居群Shewushan population in Jurong of Jiangsu; AL3: 江苏句容小九华山居群Xiaojiuhuashan population in Jurong of Jiangsu; AL4: 安徽金寨燕子河居群Yanzihe population in Jinzhai of Anhui; AL5: 安徽金寨天堂寨居群Tiantangzhai population in Jinzhai of Anhui; AL6: 河南嵩县五马寺居群Wumasi population in Songxian of He’nan; AL7: 河南嵩县纸坊居群Zhifang population in Songxian of He’nan; AL8: 湖北保康后坪居群Houping population in Baokang of Hubei; AL9: 湖北红安七里坪居群Qiliping population in Hongan of Hubei.图4 SRAP-SCAR标记在南苍术9个居群单株中的验证Fig. 4 Identification of individuals from nine populations of Atractylodes lancea (Thunb.) DC. by SRAP-SCAR marker
3 讨 论
3.1 DNA片段鉴别道地产区药材的可行性及片段筛选
药材道地性形成的根本原因是长期地理隔离导致的与其他区域居群产生的遗传差异,实质上是药材基因组DNA序列出现了变异,因此,不同区域同一植物反映在DNA片段上的稳定差异实际上是各区域该植物的特质,就此产生了鉴别的依据。但是,不同DNA片段演化速率的快慢存在差异,作为种下水平的鉴别,必须筛选到演化速率快的片段。尽管不同片段在不同类群植物中演化速率也有差异,但总体上看,rbcL和matK片段的演化速率较慢,往往适用于种或者种以上水平的鉴别,适用于种下水平的往往序列较小,鉴定效果不佳;相对而言,ITS和psbA-trnH等片段更适合遗传差异小的种下水平的鉴定,但是也并非一定有效[29-31]。
郑丽[32]利用rbcL、matK、psbA-trnH和ITS共4个DNA片段作为标记,对苍术属5种植物进行了分子鉴别,认为仅ITS片段能将该属供试的5个种区分开,而其他3个片段则不行,说明该属遗传差异较小、序列较保守,常规片段很有可能无法进行种下水平的区分,还需要寻找更具鉴别度的快速演化片段。本研究选用的5个片段中,matK和rbcL片段的变异位点较少,rbcL片段仅第617位点能鉴别安徽2个居群南苍术,但安徽2个居群间则无法区分。虽然psbA-trnH片段的变异位点较多,如河南嵩县五马寺居群和江苏句容茅山居群均有少数单株存在小片段插入,但只是居群内个别单株间的变异,无法代表所在地区的南苍术。ITS片段的变异位点最多,但具有区分度的仅有1个,能将河南2个居群南苍术鉴别出来,但河南2个居群间也无法区分。nadF片段的变异位点较多,鉴别效果最佳,不仅能将道地产区江苏句容茅山居群的南苍术区分开,而且还可以将江苏句容的射乌山居群和小九华山居群与其他居群分开,但不能区分射乌山居群和小九华山居群。nadF片段由本研究自行筛选得到,位于叶绿体基因组的基因间区,变异率较高,适合进行种下水平的区分。此前的分子鉴别、遗传演化等研究中极少使用nadF片段,该片段为单拷贝片段,长度稳定(1 000 bp左右),两端序列保守(分别为蛋白编码基因nadF和rpl32)[33],引物设计容易,有望被开发为进行种下研究的热点片段。nadF片段在江苏句容茅山居群南苍术的鉴定效率达到100%。
3.2 茅山地区南苍术鉴别的SCAR分子标记的开发意义
利用随机引物的指纹图谱进一步开发的特异性分子标记是专一性、特异性的分子标记,不仅稳定,而且操作十分简单,已经成为检验药材道地性的重要手段。与DNA序列的差异相比,由随机引物进一步开发的SCAR分子标记更适用于遗传背景相近的对象的鉴定。由于随机引物扩增产生的多态性条带能给予更大的筛选范围,更容易发现基因组的差异,因此,种下水平的分子标记筛选常用随机引物的多态性条带来开发,甚至在同一居群不同个体间也能用随机引物来发现差异,进而开发成SCAR分子标记。如韦阳连等[34]采用RAPD引物找到了茅山南苍术不同个体间与苍术素含量相关的分子标记。
本研究开发的江苏句容茅山居群南苍术SRAP-SCAR标记源自不同居群南苍术通过SRAP随机引物扩增后的指纹图谱的比较筛选,但这种标记不用进行序列比对分析,仅用PCR产物进行凝胶电泳检测便可以达到鉴别的目的,操作更加简便,判断也更加直观,对操作者的专业知识要求更低,因此,在应用推广上更具优势。
总体而言,本研究基于nadF片段的序列差异和SRAP的指纹图谱开发的茅山地区南苍术特有的分子标记均可以有效地将茅山地区南苍术与其余居群南苍术区分开。茅山地区南苍术作为南苍术的道地药材,其专一性、特异性鉴别的分子标记的开发对其他道地药材的保护性鉴别也提供了理论和科学依据。
[1] 中国科学院中国植物志编辑委员会. 中国植物志: 第七十八卷第一分册[M]. 北京: 科学出版社, 1987: 23.
[2] 国家药典委员会. 中国人民共和国药典: 2015年版(一部)[M]. 北京: 中国医药科技出版社, 2015: 161.
[3] 陶弘景. 本草经集注[M]. 辑校本. 北京: 人民卫生出版社, 1994: 196-197.
[4] 彭华胜, 王德群. 安徽省术属药用植物过渡类群的居群生物学研究[J]. 中国中药杂志, 2007, 32(9): 793-797.
[5] 朱晓琴, 贺善安. 不同产地苍术药材化学成分的比较[J]. 植物资源与环境, 1994, 3(4): 18-22.
[6] 刘晓宁, 侯芳洁, 谷 巍, 等. 不同产地苍术挥发油特征性成分分析[J]. 南京中医药大学学报, 2009, 25(1): 51-53.
[7] 沈晓婧, 杨文国, 石 莹, 等. 茅苍术与北苍术挥发油的统计分析[J]. 科技创新导报, 2013(34): 67-68.
[8] 吴宝成, 韦阳连, 高 兴, 等. 茅山苍术挥发油组分特征及与根茎鲜质量相关性研究[J]. 林产化学与工业, 2008, 28(5): 84-88.
[9] 余永邦, 秦民坚, 梁之桃, 等. 不同产区太子参的rDNA ITS区序列的比较[J]. 植物资源与环境学报, 2003, 12(4): 1-5.
[10] 张宏意, 石祥刚. 不同产地何首乌的ITS序列研究[J]. 中草药, 2007, 38(6): 911-914.
[11] 孔德政, 张 伟, 李 永. 道地产区不同牛膝种群的遗传多样性分析[J]. 西北农林科技大学学报(自然科学版), 2014, 42(11): 151-155.
[12] 葛燕芬, 杭悦宇, 夏 冰, 等. 5种苍术属药用植物的trnL-F序列测定及种间遗传关系分析[J]. 植物资源与环境学报, 2007, 16(2): 12-16.
[13] PENG H S, YUAN Q J, LI Q Q, et al. Molecular systematics of genusAtractylodes(Compositae, Cardueae): evidence from internal transcribed spacer (ITS) andtrnL-Fsequences[J]. International Journal of Molecular Sciences, 2012, 13: 14623-14633.
[14] 袁英惠. 利用微卫星标记分析球花石斛的遗传多样性并鉴定其药材的地理来源[D]. 南京: 南京师范大学生命科学学院, 2011: 37-42.
[15] 高文远, 秦恩强, 肖小河, 等. 当归药材道地性的RAPD分析[J]. 中草药, 2001, 32(10): 926-929.
[16] 沈 洁, 丁小余, 丁 鸽, 等. 铁皮石斛居群差异的研究Ⅱ: ISSR指纹标记方法的建立与优化[J]. 中国中药杂志, 2006, 31(4): 291-294.
[17] 施云燕, 黄海飞, 朱振洪. 贝母类药材随机引物扩增多态性分析研究[J]. 现代中药研究与实践, 2014, 28(4): 10-13.
[18] 田孟良, 官 宇, 刘 帆, 等. 基于RAPD标记的SCAR分子标记技术鉴定川牛膝及其混淆品[J]. 中国中药杂志, 2010, 35(8): 953-956.
[19] SZE S C W, SONG J X, WONG R N S, et al. Application of SCAR (sequence characterized amplified region) analysis to authenticateLyciumbarbarum(wolfberry) and its adulterants[J]. Biotechnology and Applied Biochemistry, 2008, 51: 15-21.
[20] 任冰如, 贺善安, 於 虹, 等. 用RAPD技术评估苍术居群间的亲缘关系[J]. 中草药, 2000, 31(6): 458-461.
[21] 郭兰萍, 黄璐琦, 王 敏, 等. 南北苍术的RAPD分析及其划分的初步探讨[J]. 中国中药杂志, 2001, 26(3): 156-158.
[22] FAY M F, SWENSEN S M, CHASE M W. Taxonomic affinities ofMedusagyneoppositifolia(Medusagynaceae)[J]. Kew Bulletin, 1997, 52: 111-120.
[23] BAYER R J, GREBER D G, BAGNALL N H. Phylogeny of Australian Gnaphalieae (Asteraceae) based on chloroplast and nuclear sequences, thetrnLintro,trnL/trnFintergenic spacer,matK, and ETS[J]. Systematic Botany, 2002, 27: 801-814.
[24] SANG T. Utility of low-copy nuclear gene sequences in plant phylogenetics[J]. Critical Reviews in Biochemistry and Molecular Biology, 2002, 37: 121-147.
[25] TATE J A, SIMPSON B B. Paraphyly ofTarasa(Malvaceae) and diverse origins of the polyploid species[J]. Systematic Botany, 2003, 28: 723-737.
[26] CHASE M W, COWAN R S, HOLLINGSWORTH P M, et al. A proposal for a standardised protocol to barcode all land plants[J]. Taxon, 2007, 56: 295-299.
[27] GENG J F, ZHU C S, ZHANG X W, et al. A genetic linkage map of non-heading Chinese cabbage[J]. Journal of the American Society for Horticultural Science, 2007, 132: 816-823.
[28] LI G, QUIROS C F. Sequence-related amplified polymorphism (SRAP), a new marker system based on a simple PCR reaction: its application to mapping and gene tagging inBrassica[J]. Theoretical and Applied Genetics, 2001, 103: 455-461.
[29] CBOL Plant Working Group, JANZEN D H. A DNA barcode for land plants[J]. Proceedings of the National Academy of Sciences of the United States of America, 2009, 106: 12794-12797.
[30] KRESS W J, ERICKSON D L. DNA barcodes: genes, genomics, and bioinformatics[J]. Proceedings of the National Academy of Sciences of the United States of America, 2008, 105: 2761-2762.
[31] HOLLINGSWORTH P M. DNA barcoding plants in biodiversity hot spots: progress and outstanding questions[J]. Heredity, 2008, 101: 1-2.
[32] 郑 丽. 苍术属系统发育及药用植物白术的群体遗传和栽培起源研究[D]. 杭州: 浙江大学生命科学学院, 2013: 124-125.
[33] DANIELL H, LIN C S, YU M, et al. Chloroplast genomes: diversity, evolution, and applications in genetic engineering[J]. Genome Biology, 2016, 17: 134.
[34] 韦阳连, 吴宝成, 周义锋. 茅山苍术与苍术素相关的RAPD标记研究[J]. 安徽农业科学, 2008, 36(24): 10368-10370.
(责任编辑: 张明霞)
Study on specific molecular marker ofAtractylodeslanceain Maoshan area
WANG Mingqiang1,①, TANG Shijie1,①, FANG Yan2, XUE Jiayu1,②
(1. Center for Plant Diversity and System Evolution, Institute of Botany, Jiangsu Province and Chinese Academy of Sciences, Nanjing 210014, China; 2. Nanjing Forest Police College, Nanjing 210023, China),J.PlantResour. &Environ., 2017, 26(2): 17-26
The study focuses on nine populations from all distribution areas of wildAtractylodeslancea(Thunb.) DC. in China, including Maoshan population, Shewushan population and Xiaojiuhuashan population in Jurong of Jiangsu, Yanzihe population and Tiantangzhai population in Jinzhai of Anhui, Wumasi population and Zhifang population in Songxian of He’nan, and Houping population in Baokang and Qiliping population in Hongan of Hubei. Taking these nine populations as objects, specific molecular marker ofA.lanceafrom Maoshan population in Jurong of Jiangsu is screened by using sequence alignment of fragments ofrbcL,matK,psbA-trnH, ITS andnadFand comparison on SRAP fingerprints. The results show that through sequence alignment of five DNA fragments, the base at the 581th locus innadFfragment ofA.lanceafrom Maoshan population in Jurong of Jiangsu is A, and that from other eight populations is T, which can distinguish Maoshan population in Jurong of Jiangsu from other eight populations. A specific band with length of 323 bp inA.lanceafrom Maoshan population in Jurong of Jiangsu is screened by SRAP random primers, and a pair of SCAR primers is designed according to the sequence of this band.The forward primer sequence is 5′-ATCTTTGCTATTAACTATTAAGCTATTCTTCGCTATTCA-3′,and the reverse primer sequence is 5′-CGATGTTGAAATTGCTGTCTCTGCTTTCTATTC-3′, which can amplify specific band with length of 268 bp from all individuals ofA.lanceafrom Maoshan population in Jurong of Jiangsu, and cannot amplify any band from other populations. The two specific markers screened in this study can provide reliable technical support for identifying the genuineness ofA.lancea.
Atractylodeslancea(Thunb.) DC.; genuineness; Maoshan area;nadFfragment; SRAP-SCAR
2016-12-08
江苏省林业三新工程项目(LYSX[2015]22)
王明强(1992—),男,安徽阜阳人,硕士研究生,主要从事植物系统与演化方面的研究。 汤诗杰(1969—),男,安徽东至人,博士,副研究员,主要从事园林植物与植物多样性保护方面的研究。
Q946-33; S567.21+1
A
1674-7895(2017)02-0017-10
10.3969/j.issn.1674-7895.2017.02.03
①共同第一作者
②通信作者E-mail: xuejy@cnbg.net
