8种石首鱼类线粒体基因组特征及分子系统进化分析
2017-07-07孙利元杨天燕杨宝清
孙利元, 杨天燕, 孟 玮, 杨宝清, 张 涛
8种石首鱼类线粒体基因组特征及分子系统进化分析
孙利元1, 杨天燕3, 孟 玮4, 杨宝清1, 张 涛2
(1. 山东省水生生物资源养护管理中心, 山东烟台264003; 2. 中国科学院海洋研究所, 山东青岛 266007; 3.中国海洋大学水产学院, 山东青岛 266003; 4. 新疆大学生命科学与技术学院, 新疆乌鲁木齐 830046)
为探讨石首鱼科(Sciaenidae)鱼类分子系统进化关系, 采用生物信息学方法分析了黑鳃梅童鱼()、棘头梅童鱼()、大黄鱼()、小黄鱼()、鮸鱼()、白姑鱼()、黄姑鱼()和皮氏叫姑鱼()共8种石首鱼类线粒体基因组全序列的基本特征。结果显示, 除皮氏叫姑鱼外, 其余7种石首鱼类编码的37个基因排列顺序与脊椎动物线粒体基因组相同。基因组碱基分布存在不均衡现象, A+T含量高于G+C含量。线粒体基因组的基因变异位点分析结果表明, ND4和ND5基因可作为基因的辅助分子标记, 应用于石首鱼类群体遗传学的研究中。黄鱼亚科5种鱼类13个蛋白质编码基因的比值远低于1, 显示出较强的纯化选择。皮氏叫姑鱼与其他石首鱼间的遗传距离均较大且亲缘关系较远, 暗示叫姑鱼属或为石首鱼类中较为原始的类群。基于线粒体基因组全序列构建NJ系统树支持黄鱼亚科和白姑鱼亚科亲缘关系较近的形态学结论。而基于去除控制区后序列和13个蛋白质编码基因序列构建的系统树则表明两亚科鱼类间的差别在非编码区更为明显。
石首鱼类; 线粒体基因组序列; 系统进化
石首鱼类隶属鲈形目(Perciformes)、鲈亚目(Percoidei)、石首鱼科(Sciaenidae), 是中国海洋经济鱼类中产量最大的群类。中国沿海是石首鱼类的重要分布区之一, 有17属30种[1], 其中山东近海有6属8种[2]。20世纪60~90年代, 老一辈鱼类学家在对石首鱼类外部形态特征研究的基础上, 结合鳔和耳石的形态和式型的比较分析, 初步弄清了石首鱼类的亲缘隶属关系, 也为中国鱼类分类研究开辟了一条新途径[3-5]。近年来, 随着分子生物学理论和PCR技术的发展, 采用DNA分析的方法已逐渐成为研究和解决鱼类系统发育问题的主要手段。如蒙子宁等[6]、张永等[7]和陈泉梅[8]基于线粒体16S rRNA序列分析了中国近海部分石首鱼类分子系统进化关系; 田兰香等[9]通过比较长度为381 bp的Cyt基因序列, 探讨了7种石首鱼类的系统发育情况; 柳淑芳等[10]系统分析了19属30种石首鱼科鱼类线粒体基因, 剖析了该基因在石首鱼科鱼类系统进化研究中的应用潜力; 马春艳等[11]测定了核内重组激活RAG-1基因序列, 并分析了中国沿海9属13个种的石首鱼间的差异。迄今尚未见运用线粒体基因组全序列对山东近海石首鱼类系统发育关系进行分析研究。本研究选取山东近海分布的8种石首鱼科鱼类, 分析其线粒体基因组基本特征、蛋白质编码基因及系统发育关系等, 以期为山东近海石首鱼类分子系统发育及种质资源的研究提供遗传信息, 同时也为寻找该类群合适的分子标记提供参考资料。
1 材料和方法
1.1 数据获取
从Genbank数据库查询并下载了《山东鱼类志》[2]所记载的8种石首鱼类线粒体DNA全序列, 这8种鱼分别为梅童鱼属()的黑鳃梅童鱼()和棘头梅童鱼(); 黄鱼属()的大黄鱼()和小黄鱼(); 鮸鱼属()的鮸鱼(); 白姑鱼属()的白姑鱼(); 黄姑鱼属()的黄姑鱼(); 叫姑鱼属()的皮氏叫姑鱼()。
1.2 数据处理
采用DNAstar 7.10和muscle3.8.31软件对全部序列进行多重比对编辑; 通过Dnasp 5.10.1软件[12]计算序列碱基组成和变异位点; PAMLX 1.3.1软件[13]计算13个蛋白编码基因的同义替换率(synonymous substitution rate,)和非同义替换率(non-synonymous substitution rate,); Mega 6.0软件[14]基于蛋白质编码基因串联后序列计算不同种间遗传距离; PAUP 4.0软件[15]基于Kimura双因子参数模型, 分别对线粒体基因组全序列、去除控制区后的序列和13种蛋白质编码基因构建邻接系统进化树, 1 000次重抽样检验系统树中节点的自引导值。
2 结果
2.1 线粒体基因组序列特征及差异位点分析
8种石首鱼类线粒体DNA全序列长度在16 442 bp~ 19 154 bp, 其中棘头梅童鱼线粒体基因组长度最短, 皮氏叫姑鱼最长。除皮氏叫姑鱼外, 其余7种石首鱼类线粒体基因组均包括了13个蛋白质编码基因、22个转运RNA基因和两个核糖体RNA基因, 其排列顺序与脊椎动物线粒体基因组相同。分别计算8种石首鱼类线粒体全基因组A、T、G、C碱基含量如表1所示, 除皮氏叫姑鱼C碱基含量最低、T碱基含量最高以外, 其余7种鱼类均表现为G碱基含量最低、C碱基含量最高。所有序列A+T含量均略高于G+C含量, 这与脊椎动物偏好A、T碱基相一致。
分析比较了5种黄鱼亚科(Lamichthyinae)鱼类线粒体基因组13个蛋白质编码基因和2个核糖体RNA基因位点变异情况(表2), 可以看出15个基因变异程度在10.25%~30.12%。从基因的保守程度来看, 排在前3位的是12S rRNA、16S rRNA和COIII基因, 其变异位点比例分别为10.25%、11.46%和17.71%。从变异位点数来看, ND5的变异位点数最多, 为427个, COI次之, 为399个。

表1 8种石首鱼类线粒体基因组序列特征
2.2 蛋白质编码基因特征分析
8种石首鱼类的13个蛋白质编码基因中, 除ND6由轻链编码, 其余均由重链编码。采用Mega软件分析其密码子组成情况(表3), 除、、、和基因编码氨基酸的数量在不同石首鱼类中存在差异以外, 其余8个蛋白质编码基因所编码氨基酸的数目完全相同, 分别为ND2(348)、ND3(116)、ND4L(99)、ND4(460)、COII(230)、COIII(261)、ATP6(228)、ATP8(56)。8种石首鱼类的13个蛋白编码基因中, 除黄姑鱼ND4基因和白姑鱼ND5基因以GTG为起始密码子, 其余均以ATG作为起始密码子。、、、、和基因出现了不完全终止密码子, 其中ND2基因除黄姑鱼和皮氏叫姑鱼以T碱基结束, 其他均为TA结束。
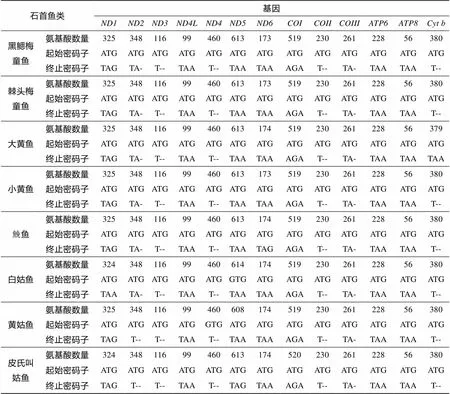
表3 蛋白质编码基因的氨基酸数量和起始、终止密码子
2.3 选择压力分析
进化速率通常受到稳定性选择、突变和定向选择控制。一般而言, 同义突变不受自然选择作用, 而非同义突变则相反。为了检验石首鱼类线粒体基因组选择压力, 选取了黄鱼亚科的黑鳃梅童鱼、棘头梅童鱼、大黄鱼、小黄鱼和鮸鱼为代表, 估算了线粒体基因组中13个蛋白质编码基因的非同义替换率和同义替换率, 并构建二者比值的柱状图如图1所示。13种蛋白编码基因的值在0.0234~0.2338之间且均小于1, 显示出较强的负(纯化)选择。
2.4 遗传距离及系统发育关系分析
将13个蛋白编码基因序列首尾串联后, 利用Mega软件计算Kimura 2-Parameter遗传距离, 从表4可以看出, 8种石首鱼类两两间平均遗传距离为0.239, 其中皮氏叫姑鱼和白姑鱼遗传距离最大, 为0.409, 小黄鱼和黑鳃梅童鱼遗传距离最小, 为0.006。计算3个亚科间的遗传距离显示, 黄鱼亚科和白姑鱼亚科遗传距离较小, 为0.230; 叫姑鱼亚科和白姑鱼亚科、黄鱼亚科遗传距离分别为0.399和0.402。黄鱼亚科鱼类平均遗传距离为0.130(0.006~0.177); 白姑鱼亚科为0.243。
选取鲈形目、攀鲈科(Anabantidae)的攀鲈()为外群, 分别基于整个线粒体基因组全序列、去除控制区后的序列和13种蛋白质编码基因构建NJ系统发育树如图2所示。前两种聚类树中, 黄鱼亚科的5种鱼类均以相同的顺序层层聚到一起, 且皮氏叫姑鱼和外群聚类并出现在聚类树的根部, 但基于整个线粒体基因组序列构建的系统树中白姑鱼亚科的白姑鱼和黄姑鱼聚为一支后, 再与黄鱼亚科鱼类聚类; 而基于去除控制区后的序列构建的系统树中白姑鱼和黄姑鱼分别与黄鱼亚科鱼类聚类。去除控制区后的序列和13种蛋白质编码基因构建的系统树几乎完全一致, 仅部分节点的支持率存在差别。
3 讨论
由于DNA双链之间存在选择压力及自然突变的差异, 基因组上碱基的分布往往存在着不均衡的现象[16]。本研究中8种石首鱼类线粒体基因组A+T含量均略高于G+C含量, 呈现出明显的碱基偏好性。Brown等[17]通过研究发现, 线粒体DNA 2/3复制周期时间内都在H-链上, 形成没有保护的单链状态, 更加容易产生氧化和水解。H链和L链的突变压力不同直接影响了碱基组成的不对称。此外, 皮氏叫姑鱼基因组序列较其他7种鱼类长2 655 bp~2 712 bp不等, 通过同源序列的比对发现, 其线粒体基因组全序列包含5个非编码区, 且tRNAVal、12S rRNA、16S rRNA和tRNAPhe这4个基因的排列顺序与其他石首鱼类有变化[16]。
基因的非同义替换率与同义替换率的比较是分子进化研究的重要内容[18]。根据中性进化理论[19-20], 由于同义替换并不影响编码氨基酸的变化, 考察非同义替换率与同义替换率的比率, 可用来判断是否有选择压力作用于这个蛋白质编码基因。当比值大于1时, 认为有正选择效应; 当该值等于1时, 认为存在中性选择; 当该值小于1时, 认为有净化选择[21]。本研究中5种黄鱼亚科鱼类的13个蛋白质编码基因,比值均远小于1, 表明这些基因受到净化选择压力, 其非同义替换速率比同义替换速率低很多。基因的均值最低, 仅为0.0234, 说明其承受的选择压力和功能束缚最为强烈。、和3个基因的均值最高, 分别为0.2338、0.1328和0.125, 其承受的选择压力较弱。
a. 基于线粒体基因组全序列; b. 基于去除控制区后序列; c. 基于线粒体基因组13个蛋白质编码基因序列
a. Based on the complete mitochondrial genome; b. Based on the sequences with the control region deleted; c. Based on 13 protein-coding genes
线粒体基因序列通常作为DNA条形码, 广泛应用于物种分类和鉴定[22-25]。从本研究结果看出在15个基因中,基因变异位点数为399个, 仅次于(427)、略高于(314)。选择合适的分子标记对于种群遗传的研究至关重要, 综合上述研究结果, 推测和基因或可作为基因的辅助备选分子标记, 应用于石首鱼类群体遗传学的研究中。
通过对遗传距离统计发现, 皮氏叫姑鱼与其他石首鱼间的遗传距离均较大, 表明叫姑鱼亚科与其他石首鱼类的亲缘关系较远。蒙子宁等[6]和田兰香等[9]分别采用16SrRNA和基因对石首鱼类系统发育的研究中也得出了类似结论。从系统发育关系来看, 3种NJ树都显示皮氏叫姑鱼单独聚类于系统树的基部, 该结果暗示叫姑鱼属或为石首鱼类中较为原始的类群, 与朱元鼎等[3]将叫姑鱼亚科作为石首鱼类中最先分化的一支的分类相一致。
朱元鼎等[3]和成庆泰等[4]将黄鱼属、梅童鱼属、鮸鱼属归属于黄鱼亚科, 认为其耳石形态与白姑鱼亚科相近, 似与白姑鱼属的关系更为密切。本研究基于线粒体基因组全序列构建系统树中, 黄鱼亚科的5种鱼类聚到一起后又与白姑鱼亚科的2种鱼类相聚, 该结果支持这两个亚科亲缘关系较近的形态学结论。而基于去除控制区后序列和13个蛋白质编码基因序列构建的系统树也得到了类似的结果, 两个亚科的7种鱼类聚成了一支, 略有不同的是两亚科间的支持率不高, 表明两亚科鱼类间的差别在非编码区更为明显。
[1] 刘效舜. 黄渤海区渔业资源调查与区划[M]. 北京: 海洋出版社, 1990: 191-200. Liu Xiaoshun. Fishery resources investigation and regionalization of Bohai and Yellow Sea[M]. Beijing: Ocean Press, 1990: 191-200.
[2] 成庆泰, 周才武. 山东鱼类志[M]. 济南: 山东科学技术出版社, 1997: 282-293. Cheng Qingtai, Zhou Caiwu. Fish fauna of Shandong[M]. Jinan: Shandong Science and Technology Press, 1997: 282-293.
[3] 朱元鼎, 罗云林, 伍汉霖. 中国石首鱼类分类系统的研究和新属新种的叙述[M]. 上海: 上海科学技术出版社, 1963. Zhu Yuanding, Luo Yunlin, Wu Hanlin. Sciaenid fishes classification system research and new species description in China[M]. Shanghai: Shanghai Science and Technology Press, 1963.
[4] 成庆泰, 郑葆珊. 中国鱼类系统检索(上册)[M]. 北京: 科学出版社, 1987: 317-324. Cheng Qingtai, Zheng Baoshan. Chinese fish retrieval system (Vol.1.) [M]. Beijing: Science Press, 1987: 317- 324.
[5] 孟庆闻, 陈立行. 鱼类分类学[M]. 北京: 中国农业出版社, 1995: 713-728. Meng Qingwen, Chen Lixing. Fish Taxonomy[M]. Beijing: China Agriculture Press, 1995: 713-728.
[6] 蒙子宁, 庄志猛, 丁少雄, 等. 中国近海8种石首鱼类的线粒体16S rRNA基因序列变异及其分子系统进化[J].自然科学进展, 2004, 14(5): 514-521. Meng Zining, Zhuang Zhimeng, Ding Shaoxiong, et al. The analysis of mitochondrial 16S rRNA gene sequence variation and molecular system evolution of eight sciaenid fishes in Chinese offshore[J]. Progress in Natural Science, 2004, 14(5): 514-521.
[7] 张永, 马春艳, 马凌波, 等. 基于16S rRNA部分序列探讨中国近海十三种石首鱼类的分子系统进化关系[J]. 海洋渔业, 2010, 32(3): 276-282. Zhang Yong, Ma Chunyan, Ma Lingbo, et al. Molecular phylogenetic relationships of 13 Sciaenidae species in China Sea areas based on 16SrRNA fragement sequences[J]. Marine Fisheries, 2010, 32(3): 276-282.
[8] 陈泉梅. 中国石首鱼科鱼类分子系统学研究[D]. 广州: 暨南大学, 2007. Chen Quanmei. Molecular phylogeny of the Sciaenidae in China[D]. Guangzhou: Jinan University, 2007.
[9] 田兰香, 梁冰, 张树义, 等. 细胞色素b基因序列与7种石首鱼类的系统进化[J]. 台湾海峡, 2004, 23(4): 436-443. Tian Lanxiang, Liang Bing, Zhang Shuyi, et al. Phylogenetic relationships of 7 Sciaenidae species based on cytochrome b gene sequences[J]. Journal of Oceanography in Taiwan Strait, 2004, 23(4): 436-443.
[10] 柳淑芳, 陈亮亮, 戴芳群, 等. 基于线粒体基因的DNA条形码在石首鱼科(Sciaenidae)鱼类系统分类中的应用[J]. 海洋与湖沼, 2010, 41(2): 223-232. Liu Shufang, Chen Liangliang, Dai Fangqun, et al. Application of DNA barcoding genefor classifying family Sciaenidae[J].Oceanologia et Limnologia Sinica, 2010, 41(2): 223-232.
[11] 马春艳, 马凌波, 倪勇, 等. 基于RAG1基因的中国近海13种石首鱼科鱼类系统进化关系[J]. 水产学报, 2012, 36(1): 9-16. Ma Chunyan, Ma Lingbo, Ni Yong, et al. Molecular phyologenetic relationships of 13 Sciaenidae species in the China Sea based on RAG1 gene sequences[J]. Journal of Fisheries of China, 2012, 36(1): 9-16.
[12] Rozas J. DNA sequence polymorphism analysis using DnaSP. bioinformatics for DNA sequence analysis, methods in molecular biology[M]. New Jersey: Humana Press, 2009: 337-350.
[13] Xu B, Yang Z. PAMLX: a graphical user interface for PAML[J]. Molecular Biology and Evolution, 2013, 30(12): 2723-2724.
[14] Tamura K, Stecher G, Peterson D, et al. MEGA6: molecular evolutionary genetics analysis version 6.0[J]. Molecular Biology and Evolution, 2013, 30(12): 2725- 2729.
[15] Swofford D L. Paup 4.0 for Unix/Vms: phylogenetic analysis using parsimony[M]. Sunderland: Sinauer Associates Inc, 1999.
[16] 钟东, 赵贵军, 张振书, 等. 基因组内碱基分布整体均衡与局部不均衡的研究进展[J]. 遗传, 2002, 24(3): 351-355. Zhong Dong, Zhao Guijun, Zhang Zhenshu, et al. Advance in the entir balance and local unbalance of base distribution in genome[J]. Hereditas, 2002, 24(3): 351-355.
[17] Brown W M, Prager E M, Wang A, et al. Mitochondrial DNA sequences of primates: tenpo and mode of evohxtion[J]. Journal of Molecular Evolution, 1982, 18: 225-239.
[18] Xu T J, Tang D, Jin X X. A surprising arrangement pattern and phylogenetic consideration: the complete mitochondrial genome of Belanger’s croaker Johnius belangerii (Percoidei: Sciaenidae)[J]. Mitochondrial DNA, 2015, 26(4): 655-657.
[19] Nei M, Kumar S. Molecular evolution and phylogenetics[M]. New York: Oxford University Press, 2000: 252-265.
[20] Kimura M. The neutral theory of molecular evolution[M]. Cambridge: Cambridge University Press, 1983.
[21] Ohta T. The nearly neutral theory of molecular evolution[J]. Annual Review of Ecology and Systematics, 1992, 23: 263-286.
[22] Yang Z, Bielawski J P. Statistical methods for detecting molecular adaptation[J]. Trends in Ecology and Evolution, 2000, 15(12): 496-503.
[23] 裴男才, 陈步峰. 生物DNA条形码: 十年发展历程、研究尺度和功能[J]. 生物多样性, 2013, 21(5): 616- 627. Pei Nancai, Chen Bufeng. DNA barcoding of life: a classification of uses according to function and scale after ten years of development[J]. Biodiversity Science, 2013, 21(5): 616-627.
[24] 莫帮辉, 屈莉, 韩松, 等. DNA条形码识别I. DNA条形码研究进展及应用前景[J]. 四川动物, 2008, 27(2): 303-306. Mo Banghui, Qu Li, Han Song, et al. DNA barcoding identification I. research progress and applied perspective of DNA barcoding[J]. Sichuan Journal of Zoology, 2008, 27(2): 303-306.
[25] 丁兰平, 马元元, 黄冰心. DNA条形码技术在大型海藻学研究中的应用及前景[J]. 海洋科学, 2012, 36(11): 103-110. Ding Lanping, Ma Yuanyuan, Huang Bingxin. The application and perspective of DNA barcoding technology on the macroalgae[J]. Marine Sciences, 2012, 36 (11): 103-110.
[26] 张辉, 线薇薇. DNA条形码在长江口鱼类浮游生物生态学研究中的意义[J]. 海洋科学, 2015, 39(4): 135- 137. Zhang Hui, Xian Weiwei. The importance of DNA barcoding on the ichthyoplankton ecological study in the Yangtze estuary[J]. Marine Sciences, 2015, 39(4): 135-137.
(本文编辑: 谭雪静)
Analysis of the mitochondrial genome characteristics and phylogenetic relationships of eight sciaenid fishes
SUN Li-yuan1, YANG Tian-yan3, MENG Wei4, YANG Bao-qing1, ZHANG Tao2
(1. Shandong Hydrobios Resources Conservation and Management Center, Yantai 264003, China; 2. Institute of Oceanology, Chinese Academy of Sciences, Qingdao 266007, China; 3. Fisheries College, Ocean University of China, Qingdao 266003, China; 4. College of Life Science and Technology, Xinjiang University, Urumqi 830046, China)
To explore the phylogenetic relationships of sciaenid fishes, the basic characteristics of mitochondrial genomes in eight sciaenid fishes (,,,,,,and) were revealed by comprehensive bioinformatic analysis. The results showed that mitochondrial genomes contained 37 standard vertebrate genes, and the order was identical except for inThe base distribution was not balanced, and the content of A+T was higher than that of G+C. Genetic variation analysis of 13 protein-coding genes and 2 ribosomal RNA genes among sciaenid fishes showed that the ND4 and ND5 genes could be used as supplementary molecular markers to the COI gene and applied to research on population genetic diversity. Theratios of 13 mitochondrial protein-coding genes in five Pseudosciaeninae fishes were much lower than 1, indicating strong purifying selection. The genetic distances betweenand other sciaenid fishes were high, and relationships were distant, which suggested thatfishes might be the relatively primitive group. A neighbor-joining phylogenetic tree based on the complete mitochondrial genome supported the morphological conclusion that fishes of Pseudosciaeninae and Argyrosominae were closely related. Furthermore, phylogenetic trees based on sequences deleting the control region and 13 protein-coding genes showed that the differences between these two subfamilies were much more evident.
Sciaenid fishes; mitochondrial genome sequence; phylogenetic
Apr. 22, 2016
[Department of Ocean and Fisheries of Shandong Province Program, No. 270006-FZLX-2015-00-1]
S917.4
A
1000-3096(2017)03-0048-07
10.11759//hykx20160422005
2016-04-22;
2016-08-03
山东省海洋与渔业厅资助项目(No. 270006-FZLX-2015-00-1)
孙利元(1980-), 男, 山东招远人, 工程师, 主要从事海洋渔业资源学研究, E-mail: heroland80@163.com
