利用表达谱芯片数据筛选不同表达丰度的内参基因
2017-03-13胡世贤陆佳涛徐福意晁天柱周梁良李凯周宇荀肖君华
胡世贤,陆佳涛,徐福意,晁天柱,周梁良,李凯,周宇荀,肖君华
(东华大学生物科学与技术研究所,上海 201620)
利用表达谱芯片数据筛选不同表达丰度的内参基因
胡世贤,陆佳涛,徐福意,晁天柱,周梁良,李凯,周宇荀,肖君华*
(东华大学生物科学与技术研究所,上海 201620)
目的 基于表达谱芯片的检测结果,筛选多个内参基因,用于小家鼠肝脏组织中具有不同表达丰度基因的定量检测。方法 应用表达谱芯片技术,完成小鼠肝脏组织的表达谱检测;依据基因表达丰度将基因分为3组,并进一步通过变异系数(coefficient of variation, CV)组内筛选候选内参基因;采用实时荧光定量PCR技术(real-time quantitative polymerase chain reaction, qPCR)和geNorm软件确定内参基因。结果 表达谱芯片成功采集超过60000个小鼠肝脏组织中转录本的表达量数据,并将之分为低、中、高3个组合。最终筛选了低表达Casp2和Lrrc14、中表达Nrd1和Trpc4ap、高表达Atp5a1和Clu,共6个内参基因。结论 基于表达谱芯片数据筛选的6个内参基因,可适用于qPCR技术准确定量小家鼠肝组织转录组中不同表达丰度基因的表达量。
内参基因;表达谱芯片;实时荧光定量PCR;geNorm软件
血脂水平异常是动脉粥样硬化、心脏病和脂肪肝密切相关的重要危险因素[1]。随着系统遗传学的蓬勃发展,研究者们利用高血脂小鼠模型,结合基因组学、转录组学、表型组学等知识,在血脂相关遗传学研究中取得了丰硕的成果[2]。其中,基因表达分析在转录组水平上,是研究血脂等复杂性状不可或缺的方法[3]。常用的技术如基因表达谱芯片,操作方便,通量高,可准确快速得到完整的全基因组表达丰度数据,但是昂贵的价格限制了其应用。与之相比,具有高灵敏性,高重复性,高特异性的实时荧光定量PCR技术是核酸分子定量的“金标准”[4]。
在qPCR相对定量实验中,目前被广泛采用的是2-△△CT法。△Ct表示目标基因和内参基因Ct值的差异,-△△Ct表示两样本间△Ct之差。当目标基因和内参基因扩增效率相差不大时,两样本间检测基因相对表达量差异即为2-△△Ct[5]。因此,定量的准确性与内参基因的选择密切相关。理想的内参属于维持细胞基本生物学特征的基因,应具备两个重要特点:1)表达稳定,不受实验环境的影响[6];2)与待测基因表达水平相近[7]。系统遗传学研究中,内参基因常被用于qPCR在多样本中定量数十甚至上百个目标基因,从而挖掘基因共表达网络[8-10]。1999年Suzuki 等[11]提出的Gapdh、B2m等常用内参基因不具有通用性,一方面是由于其表达水平受到不同实验条件的影响,另一方面传统内参基因的表达丰度较高,难以定量表达丰度较低基因[12]。因此,Radoni等[13]采用qPCR技术,针对13种已知候选内参基因获取ΔΔCT最稳定的2个基因作为内参;Almeida 等[14]采用qPCR array,在51个候选内参基因中筛选内参基因,其表达水平差异达到300倍。但是,从少量已知候选内参中选择内参基因的方法,很难兼顾到表达稳定性和表达丰度。
本研究以不同1号染色体替换系品系[15]的高血脂小鼠模型为样本,采用表达谱芯片技术获取小鼠肝脏组织的RNA表达数据,并进一步利用qPCR和geNorm软件筛选内参基因,以期适用于qPCR定量肝组织转录本中不同丰度基因的表达量。
1 材料与方法
1.1 动物实验与RNA提取
8周龄SPF级C57BL6/J(B6)小鼠(雄性14只),体重22~25 g,购自上海斯莱克实验动物有限公司[ SCXK (沪) 2012- 0002]。动物实验遵守1988年动物管理条例。实验在东华大学生物科学与技术研究所屏障动物实验设施进行[ SYXK (沪) 2014- 0022]。取野生小家鼠来源1号染色体替换系品系B6-Chr1KM、B6-Chr1HZ、B6-Chr1SJ和近交系B6的8周龄雄鼠,每个品系各14只,品系内分为2组(每组7只)分别喂食40%高脂饲料和10%对照饲料(上海诺宝生物科技有限公司)至20周龄。颈椎脱颈处死20周龄雄鼠后,即刻摘取肝脏组织,采用RNAiso Plus试剂(上海麦约尔生物技术有限公司)进行RNA抽提。以Nanodrop 2000c超微量分光光度计(Thermo Fisher Scientific,美国)和1% 甲醛变性琼脂糖凝胶电泳鉴定RNA的质量和完整性,-80℃保存备用。
1.2 表达谱芯片检测和筛选候选内参基因
采用Mouse Transcriptome Assay 1.0(Affymetrix)芯片,对样本进行表达谱检测(上海其明信息技术有限公司)。用Expression Console和Transcriptome Analysis Console采集并分析数据,根据芯片信号值完成基因分组,每组筛选CV最低的基因(共21个)作为候选内参基因。用Oligo7跨越内含子设计引物,由上海生工生物工程有限公司合成,引物序列信息见表1。
1.3 qPCR验证
采用RevertAid First Strand cDNA Synthesis Kit试剂盒(上海麦约尔生物技术有限公司)逆转录cDNA,操作步骤依据说明书进行。qPCR采用2 × SYBR Green/Rox qPCR Master Mix(北京百泰克生物科技有限公司)试剂盒,ABI 7900 real-time PCR system和7900 System Software-SDS 2.2 (Applied Biosystems)分析样本,反应体系和反应条件依据说明书进行。
1.4 筛选内参基因
采用geNorm v3.5 软件( https://www.biogazelle.com )根据平均变异度(M < 0.5)筛选稳定表达的基因,每组选取M值最低的2个基因为内参基因。
2 结果
2.1 芯片检测分析
小鼠肝组织表达谱芯片检测结果如图1所示:转录本的平均信号值在3~20之间,将信号值在3~7的转录本定义为低表达组,包括54180个转录本;信号值在7~13为中表达组,包括9163个转录本;信号值在13~20为高表达组,包括2192个转录本。
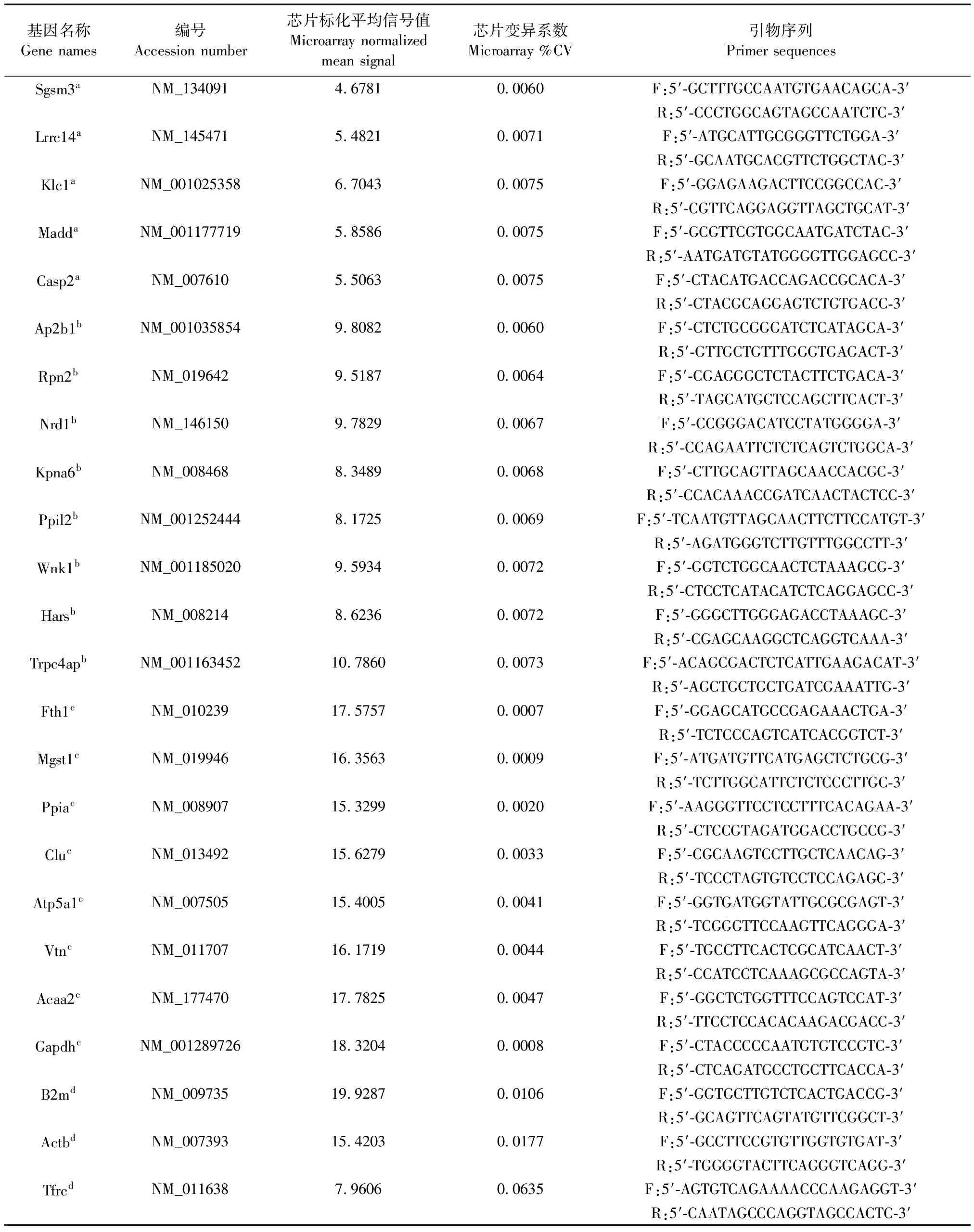
表1 基因芯片表达分析所筛候选内参
注:a. 低表达水平基因;b. 中表达水平基因;c. 高表达水平基因;d. 传统内参基因。
Note. a indicates low expression genes; b indicates moderate expression genes; c indicates high expression genes and d indicates traditional reference genes.
2.2 候选内参基因
根据CV越小基因表达水平越稳定,在组合内选择CV最小的转录本为候选内参基因。如表1所示,每个组合内初步筛选的候选内参,包括Sgsm3、Lrrc14、Klc1、Madd、Casp2、Ap2b1、Rpn2、Nrd1、Kpna6、Ppil2、Wnk1、Hars、Trpc4ap、Fth1、Mgst1、Ppia、Clu、Atp5a1、Vtn、Acaa2和Gapdh,所有候选内参基因表达量CV均低于1%。
2.3 qPCR验证
qPCR的检测结果如图2所示,21个候选内参基因相对表达量差异达到15 个Ct值(不考虑个别离群值),约105,并可以明显的分为3个组合,低表达组合为Sgsm3、Lrrc14、Klc1、Madd和Casp2,Ct值范围25到28;中表达组合为Ap2b1、Rpn2、Nrd1、Kpna6、Ppil2、Wnk1、Hars和Trpc4ap,Ct值范围21到25;高表达组合为Fth1、Mgst1、Ppia、Clu、Atp5a1、Vtn、Acaa2和Gapdh,Ct值范围15到20。
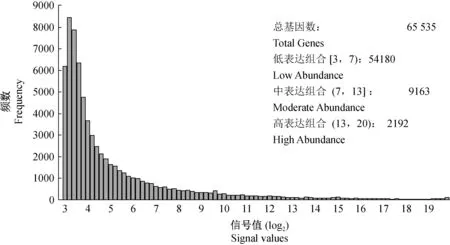
注:Y轴表示芯片信号值(log2)。X轴表示区间内转录本频数。图1 小鼠肝脏基因表达量分布Note. The Y-axis indicates values (log2) of microarray. The X-axis indicates frequency of transcripts in intervals.Fig.1 Distribution of the mouse liver gene expression abundance

注:X轴表示候选内参基因,Y轴表示Ct值。蓝色为低表达内参,橙色为中表达内参,绿色为高表达内参,灰色为对照内参基因。图2 qPCR检测小鼠肝脏候选内参表达量。Note. X-axis indicates candidate reference genes and Y-axis implies Ct value. Blue, orange, green and gray colors represent for genes of low abundance, moderate abundance, high abundance and reference, respectively.Fig.2 Candidate reference genes of the mouse liver tested by qPCR.
2.4 内参基因
geNorm软件分析结果如图3所示,21个候选内参基因除Ppia和Fth1 M值均大于0.5,其他候选内参M值都在0.5以下,其中Casp2和Lrrc14基因M值最小。在每个表达组合内,我们各选取了2个M值最小的基因作为理想的内参(表2),低表达组合为Casp2和Lrrc14,中表达组合为Nrd1和Trpc4ap,高表达组合为Atp5a1和Clu。

注:Y 轴表示候选内参基因的平均表达稳定度 M 值,X 轴表示看家基因表达稳定性由左向右逐步提高。图3 候选内参表达稳定性分析Note: Y-axis indicates the average expression stability values ( M) , and X-axis indicates the candidate reference genes according to their increasing expression stability from left to right.Fig.3 Stability analysis of candidate reference genes
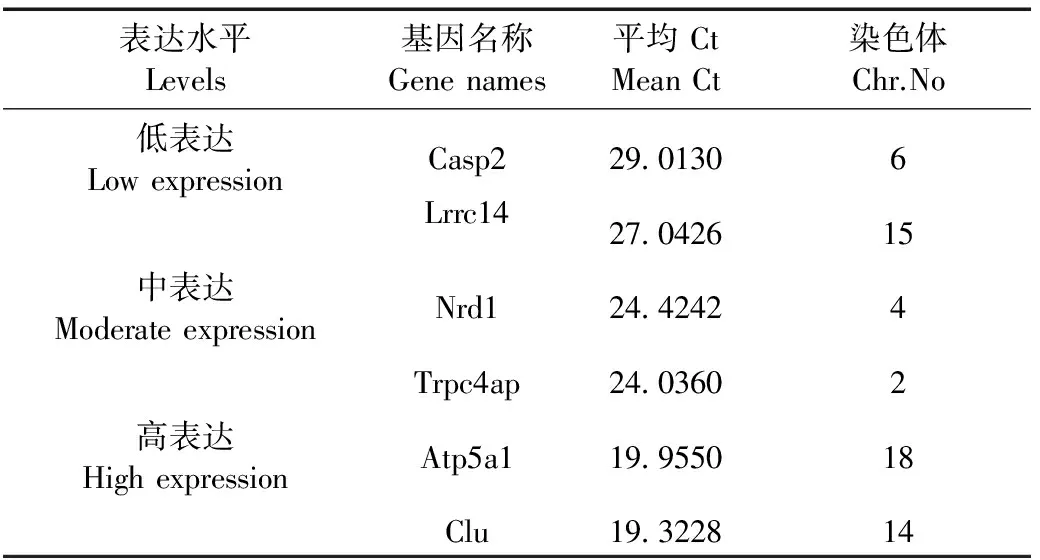
表2 优选内参基因
3 讨论
模式动物小鼠是血脂相关复杂疾病研究的重要模式动物,且发现不同的近交系小鼠对动脉粥样硬化、脂肪肝等疾病的易感性不同。准确定量小鼠肝组织转录组中血脂代谢相关基因表达量情况,可构建基因共表达网络,发现血脂代谢调控通路中的关键基因,从而促进血脂异常相关疾病的遗传学研究。qPCR是定量技检测基因表达量的最佳选择,但qPCR在相对定量过程中需要用内参基因来标化检测基因的表达量,再用2-△△CT法得到不同样本间相对表达水平差异,而选择合适的内参基因是影响qPCR相对定量准确度的重要因素[16]。因此针对小鼠肝组织转录组的qPCR定量检测,需要筛选变异系数小、稳定表达的看家基因。
本研究结果揭示,在表达谱芯片检测结果中,65 536个检测转录本的丰度差异超过105。根据变异系数筛选的21个候选内参基因中,包含传统常用内参基因Ppia和Gapdh[17],说明其在小鼠肝脏组织中具仍能稳定表达。针对常用于小鼠肝脏转录组[12]的内参基因B2m,Actb,Tfrc的表达谱数据分析结果发现,B2m、Actb和Tfrc表达量的CV分别为1.06%,1.77%,6.35%,均高于21个候选内参基因,说明其表达稳定性较差,并不适用于本研究。23个基因qPCR检测结果与表达谱芯片数据一致,除Actb表达水平在2种检测方法中呈现了较大波动,同样说明常用内参基因Actb不适用于本研究。geNorm软件是基于不同条件下2个内参基因相对表达水平比值为定值的原理分析表达稳定性,表达水平比值变异度M的增加意味着内参稳定性的下降。因此内参基因的M值越小,就表示该基因越稳定。此外,单个内参基因标化数据导致定量结果偏差数倍[18],多个内参基因能提高定量准确度[19,20],但内参基因数量过多也有操作不便,经济成本高的缺点。因此,本研究在每个表达水平组合里,各选取了2个M值最低的内参基因。
总之,基于表达谱芯片数据挖掘内参基因的方法,能快速、精确的优选转录组中表达水平最稳定、表达丰度差异超过1000倍的内参基因。筛选的内参基因适用于qPCR相对定量小鼠肝组织中基因的表达量,对系统遗传学中qPCR转录组表达分析具有重要的意义。
[1] 朱竟赫, 赵金明, 秦文艳,等. 高脂血症实验动物模型研究概述[J]. 实验动物科学, 2012, 29(2):48-52.
[2] Leduc MS, Lyons M, Darvishi K, et al. The mouse QTL map helps interpret human genome-wide association studies for HDL cholesterol[J]. J Lipid Res, 2011, 52(6):1139-1149.
[3] Civelek M, Lusis AJ. Systems genetics approaches to understand complex traits[J]. Nature Rev Genet, 2014, 15(1):34-48.
[4] Yuan J S, Reed A, Feng C, et al. Statistical analysis of real-time PCR data[J]. BMC Bioinformatics, 2006, 7(4):563-569.
[5] Livak KJ, Schmittgen TD. Analysis of relative gene expression data using real-time quantitative PCR and the 2(-Delta DeltaC(T)) method[J]. Methods, 2001, 25(4):402-408.
[6] Lercher M J, Urrutia A O, Hurst L D. Clustering of housekeeping genes provides a unified model of gene order in the human genome[J]. Nature Genet, 2002, 31(2):180-3.
[7] Silver N, Best S, Jiang J, et al. Selection of housekeeping genes for gene expression studies in human reticulocytes using real-time PCR[J]. BMC Mol Biol, 2006, 7(1):1-9.
[8] Punt S, Houwing-Duistermaat JJ, Schulkens IA, et al. Correlations between immune response and vascularization qRT-PCR gene expression clusters in squamous cervical cancer[J]. Mol Cancer, 2015, 14(1):1-12.
[9] Spurgeon SL, Jones RC, Ramakrishnan R. High throughput gene expression measurement with real time PCR in a microfluidic dynamic array[J]. PloS One, 2007, 3(2):1662.
[10] Horst OV, Horst JA, Samudrala R, et al. Caries induced cytokine network in the odontoblast layer of human teeth[J]. BMC Immunol, 2011, 12(1):1-13.
[11] Suzuki T, Higgins PJ, Crawford DR. Control selection for RNA quantitation[J]. Biotechniques, 2000, 29(2):332-337.
[12] Hellemans J, Mortier G, Paepe AD, et al. qBase relative quantification framework and software for management and automated analysis of real-time quantitative PCR data[J]. Genome Biol, 2007, 8(2): R19.
[13] Radoni A, Thulke S, Mackay I M, et al. Guideline to reference gene selection for quantitative real-time PCR[J]. Biochem Biophys Res Comm, 2004, 313(4):856-862.
[14] Almeida TA, Quispe-Ricalde A, Oca FMD, et al. A high-throughput open-array qPCR gene panel to identify housekeeping genes suitable for myometrium and leiomyoma expression analysis[J]. Gynecol Oncol, 2014, 134(1):138-143.
[15] Xiao J, Liang Y, Li K, et al. A novel strategy for genetic dissection of complex traits: the population of specific chromosome substitution strains from laboratory and wild mice[J]. Mamm Genome, 2010, 21(7-8):370-376.
[16] Sellars MJ, Vuocolo T, Leeton LA, et al. Real-time RT-PCR quantification of Kuruma shrimp transcripts: A comparison of relative and absolute quantification procedures[J]. J Biotechnol, 2007, 129(3):391-399.
[17] Kouadjo KE, Nishida Y, Cadringirard JF, et al. Housekeeping and tissue-specific genes in mouse tissues[J]. BMC Genomics, 2007, 8(1): 127.
[18] Olsvik PA, Kai KL, Jordal AEO, et al. Evaluation of potential reference genes in real-time RT-PCR studies of Atlantic salmon[J]. BMC Mol Biol, 2005, 6(22):1-9.
[19] Jain M, Nijhawan A, Tyagi AK, et al. Validation of housekeeping genes as internal control for studying gene expression in rice by quantitative real-time PCR[J]. Biochem Biophys Res Comm, 2006, 345(2):646-651.
[20] Hoerndli F J, Toigo M, Schild A, et al. Reference genes identified in SH-SY5Y cells using custom-made gene arrays with validation by quantitative polymerase chain reaction[J]. Analyt Biochem, 2004, 335(1):30-41.
Screening of reference genes of different abundance using gene expression microarrays
HU Shi-xian, LU Jia-tao, XU Fu-yi, CHAO Tian-zhu, ZHOU Liang-liang, LI Kai, ZHOU Yu-xun,XIAO Jun-hua*
(Institute of Biological Sciences and Biotechnology, Donghua University, Shanghai 201620, China)
Objective Based on the data detected using gene expression microarray, to select multiple reference genes and use them to quantify transcriptome genes of different abundance in the mouse liver tissue. Methods To detect global transcriptome genes in the mouse liver tissues using gene expression microarray. All the genes were sorted into different groups according to their expression level, followed by coefficient of variation (CV) analysis. Real-time quantitative polymerase chain reaction (qPCR) and geNorm software were used to further identify the reference genes. Results The expression levels of over 60,000 genes were obtained from microarray screening, and divided them into low, moderate and high expression groups. Finally six reference genes were screened, i.e. Casp2 and Lrrc14 as low abundance, Nrd1 and Trpc4ap as moderate abundance, and Atp5a1 and Clu as high abundance genes. Conclusions The six reference genes derived from microarray data can be used to accurately quantify the global transcriptome genes with various expression levels.
Reference genes; Gene expression microarrays; Quantitative real-time PCR; geNorm software; Mouse; Liver
XIAO Jun-hua,E-mail: xiaojunhua@dhu.edu.cn
国家自然科学基金面上项目(编号:31371257);上海市科委关键项目(编号:12140900404,13140900300,15140900500)。
胡世贤(1991-),男,硕士研究生,研究方向:医学分子遗传学。E-mail: dhu.sxhu@hotmail.com
肖君华(1968-),男,教授,研究方向:医学分子遗传学。E-mail: xiaojunhua@dhu.edu.cn
Q95-33
A
1005-4847(2017) 01-0008-06
10.3969/j.issn.1005-4847.2017.01.002
2016-06-28