CIT2基因对突变酿酒酵母木糖利用影响的研究
2016-12-22李维维陈朝儒曲娟娟顿宝庆李桂英
李维维, 张 骥, 陈朝儒, 王 智, 曲娟娟, 顿宝庆*, 李桂英, 路 明
1.中国农业科学院作物科学研究所, 农作物基因资源与基因改良国家重大科学工程, 北京 100081;2.东北农业大学生命科学学院, 哈尔滨 150030
CIT2基因对突变酿酒酵母木糖利用影响的研究
李维维1,2, 张 骥1, 陈朝儒1, 王 智1, 曲娟娟2*, 顿宝庆1*, 李桂英1, 路 明1
1.中国农业科学院作物科学研究所, 农作物基因资源与基因改良国家重大科学工程, 北京 100081;2.东北农业大学生命科学学院, 哈尔滨 150030
木糖利用困难是秸秆燃料乙醇产业化的制约因素,改造酿酒酵母使其能够代谢木糖生成乙醇是当前研究的热点之一。为了研究CIT2基因对遗传改良酿酒酵母C5D-P-M菌株中葡萄糖与木糖共发酵过程中木糖利用的影响,设计引物并应用重叠PCR技术获得敲除CIT2基因的敲除组件,通过同源重组的方法将C5D-P-M中的CIT2基因敲除得到突变菌株C5D-P-M-CIT2Δ。通过对出发菌株和突变菌株的生长速率、葡萄糖利用率、木糖利用率及乙醇产量进行研究,结果表明突变菌株C5D-P-M-CIT2Δ的上述指标比出发菌株C5D-P-M均有一定程度的提高。因此推测,酿酒酵母中CIT2基因是影响木糖利用的因素之一,CIT2基因的敲除可提高酿酒酵母的木糖利用率。
酿酒酵母;CIT2基因;基因敲除;木糖代谢
能源短缺、气候变化是目前全世界共同面临的重大问题之一[1]。因此,世界各国纷纷积极开展新能源、可再生生物质能的研究与开发[2],其中以燃料乙醇的研究开发最为成功,也是最可能的替代能源之一[3,4]。
我国人口众多,土地资源相对贫乏,用紧缺的粮食生产乙醇燃料受条件限制。然而资源丰富、价格低廉的农作物秸秆类纤维素生物质尚未得到有效充分利用与开发。秸秆类纤维素物质具有木质纤维复合物结构,木质纤维素的组成和结构非常复杂,主要由纤维素、半纤维素和木质素组成。半纤维素构成了秸秆类纤维素物质中的相当部分,约为30%,其水解产物是D-木糖类的五碳糖[5~7],其中木糖的有效利用是乙醇产业化利用的重要限制因素之一[8]。
生产燃料乙醇用到的最主要的微生物是酿酒酵母,但是酿酒酵母不能直接代谢利用木糖,因其没有专一性代谢木糖生成木酮糖的酶系,并且存在细胞膜上木糖跨膜运输障碍[9],只有在酿酒酵母中引入木糖代谢流[10]并利用木酮糖的代谢酶系,将其代谢分解随后进入磷酸戊糖途径,才能进入其本身的代谢流,解决酿酒酵母中葡萄糖与木糖共发酵过程中木糖转运系统产生的影响[11~13]。目前已知在全基因组测序的酿酒酵母S288C中,拥有DUP多基因家族[14]。酿酒酵母的DUP基因家族包括23个成员,其中可分为两个亚族包括DUP240和DUP380。DUP基因家族编码的部分蛋白能够促进膜蛋白转运过程[15]。DUP240有3个保守区域C1、C2、C3和两个预测横跨膜结构H1和H2,并且在Dup380蛋白质序列中观察到一个直接重复的C1-H1-H2-C2模块[16]。DUP380亚家族由11个分别命名为COS1-COS11的基因组成,还包括2个假基因,所有这些基因连同与其相关的9个基因残基都集中分布在酵母16条染色体的端粒区域。研究表明,DUP240家族蛋白均与细胞膜有关,他们具有不同的亚细胞定位而且没有多余的互作因子[17]。这些DUP家族蛋白可能均在细胞膜运输中起作用[18]。
COS12蛋白(YGL263W )为DUP380家族成员,源于酿酒酵母ATCC204508/S288c 菌株。蛋白序列全长为380个氨基酸,并且已经从蛋白水平上得到证实[19,20]。该蛋白位于细胞膜上,为跨膜蛋白,COS12与物质的跨膜运输有关[21],但目前为止关于其在调控木糖利用机制方面的研究未见报道。本研究利用C5D-P-M菌株构建COS12的缺失突变株C5D-P-M-CIT2Δ,研究共生长特性,以期揭示CIT2基因对酿酒酵母木糖利用的影响。
1 材料与方法
1.1 材料
1.1.1 实验菌株及载体 质粒pUG6、pSH65由实验室保存,酿酒酵母菌株C5D-P-M(表型为MatAura3-52,转入含有携带TPI-PxylA基因的质粒pYES2,能够代谢木糖)由本实验室构建。
1.1.2 酶类及其他生化试剂TaqDNA聚合酶购自TaKaRa公司;RNA提取试剂盒RNeasy©Mini Kit (50) Cat.NO.74104、TAKARA荧光定量PCR试剂盒、反转录试剂盒Omniscrippt©Reverse Transcription购自QIAGEN公司。
1.1.3 培养基配方 大肠杆菌培养基LB:1%蛋白胨,0.5%酵母提取物,l% NaCl,pH 7.0。
酵母培养基YPD:1%酵母提取物,2%蛋白胨,2%葡萄糖,平板添加2%琼脂。
选择培养基SC:0.67%YNB,2%葡萄糖,平板添加2%琼脂。
酿酒酵母诱导表达YPG培养基:1.0%酵母膏,2.0%蛋白胨,1.34% YNB,4×10-5%生物素,1.0%甘油;10.0% 100 mmol/L pH 6.0磷酸盐缓冲液,水1 000 mL。
1.2 方法
1.2.1 酿酒酵母C5D-P-M中CIT2基因的敲除①敲除组件引物设计。根据已知的CIT2基因和质粒pUG6的核甘酸序列,设计引物见表1。

表1 CIT2基因敲除及验证引物Table 1 CIT2 gene knockout and verified primers.
②敲除组件的PCR合成。以pUG6质粒为模板,用敲除组件引物进行PCR扩增敲除组件。PCR反应体系(20 μL):2 μL 10×ExTaqBuffer,0.4 μL PCR Forward Primer(10 μmol/L),0.4 μL PCR Reverse Primer(10 μmol/L),0.4 μL 的dNTP Mix(5mmol/L each dNTP),0.1 μL的pUG6模板,0.1 μL 的ExTaq酶,0.1 μL ddH2O。
PCR反应程序为:99℃盖子预热, 94℃预变性5 min;94℃ 45 s,52℃ 45 s,72℃ 1 min 20 s,25个循环;72℃ 10 min,4℃无限延伸。
③敲除组件连接pGEM-T载体。连接体系如下:目的PCR片段(可变),pZeroBack载体(约25 ng/μL)1 μL,2×Reaction Buffer 5 μL,T4 DNA Ligase(5 U/μL)0.5 μL,ddH2O补足到10 μL。轻轻弹动离心管以混匀内容物,短暂离心3~5 s,将混合反应液放置室温(22℃)反应5 min,反应结束后,将离心管置于冰上。取部分连接产物加到50~100 mL DH5α感受态细胞中,轻弹混匀,冰浴30 min。将离心管置于42℃恒温90 s,取出管后立即置于冰浴中放置2~3 min,期间不要摇动离心管。向离心管中加入250~500 mL 37℃预热的LB(不含抗生素)培养基,150 r/min 37℃振荡培养45 min。目的是使质粒上相关的抗性标记基因表达,使菌体复苏。将离心管中的菌液混匀,吸取100 mL加到含氨苄青霉素的LB固体琼脂培养基上,用无菌的弯头玻璃棒轻轻的将细胞均匀涂开。待平板表面干燥后,倒置平板,37℃培养12~16 h。
1.2.2 突变菌株C5D-P-M-CIT2Δ的构建 出发菌株化学转化pSH65及Kanr基因的切除:活化转进敲除组件的酿酒酵母C5D-P-M,用灭菌去离子水清洗两遍后加入1 mL灭菌去离子水取100 μL备用;鲑鱼精50 μL放入沸水中煮10 min备用;PEG3350取240 μL及LiAC36 μL加入以上酿酒酵母与鲑鱼精和敲除组件涡悬混匀后放入30℃水浴中30 min,再放入42℃水浴45 min;加入1 mL YPD培养基于30℃ 120 r/min培养1 h;取20 μL涂于含有抗生素腐草霉素的YPD平板上48 h左右观察:将转进pSH65的菌株用YPG培养基培养4~7 h,诱导质粒pSH65上Cre基因表达,切除基因组上Kanr基因,去除抗性;用YPD培养基传代丢失酿酒酵母中质粒pSH65,得到CIT2缺失的C5D-P-M-CIT2Δ。
1.2.3 突变菌株木糖相关基因的转录水平荧光定量分析 以试剂盒所提RNA反转录的cDNA稀释100倍为模板做荧光定量PCR。按照TaKaRa公司的荧光定量试剂盒说明配制反应液及操作。
利用Primer Express 3.0软件所设计的9对引物如表2所示。分别是参与三羧酸循环的ACO1(乌头酸酶)基因、TCA循坏中的关键酶FUM1(延胡索酸酶)基因、在糖酵解第二步中GND1(6-磷酸葡萄糖酸脱氢酶)基因、三羧酸循环中重要的限速酶IDH2(异柠檬酸脱氢酶)基因、在三羧酸循环中提供能量的MDH1(苹果酸脱氢酶)基因、TCA循环中唯一一个整合于膜上的多亚基酶SDH1(琥珀酸脱氢酶,是连接氧化磷酸化与电子传递的枢纽之一,为线粒体的一种标志酶)基因、催化糖酵解第二部分开始的TDH3(3-磷酸甘油醛脱氢酶)基因、木糖代谢进入到戊糖磷酸途径后的TKL1(转酮醇酶)基因。

表2 内参肌动蛋白及差异基因荧光定量引物Table 2 Fluorescence quantitative primer of actin and differentially expressed genes.
1.2.4 突变菌株生理生化分析 ①生长速率测定。从平板上挑取C5D-P-M与C5D-P-M-CIT2Δ单菌落分别接入酿酒酵母营养缺陷型选择培养基SC中,于30℃ 160 r/min培养,至培养基OD值约为1.0时,用分光光度计测定其准确OD值。取10个已灭菌的50 mL三角瓶,分别装入10 mL液体酿酒酵母营养缺陷型选择培养基SC,将C5D-P-M与C5D-P-M-CIT2Δ各接入5个三角瓶中,每个三角瓶中接入3×108个细胞(OD值为1.0时酿酒酵母细胞个数约为3×107个,通过所测定OD值可计算出来具体每种菌株所需接种体积),每种菌株设置5个平行,菌体浓度OD600,分别取0 h、3 h、6 h、9 h、12 h、14 h、22 h、28 h、34 h、40 h培养液测OD600值。
②剩余葡萄糖含量测定。从平板上挑取C5D-P-M与C5D-P-M-CIT2Δ分别接入酿酒酵母营养缺陷型选择培养基SC中,于30℃ 160 r/min培养,至培养基OD值约为1.0时用分光光度计测定其准确OD值。取10个已灭菌50 mL三角瓶,分别装入10 mL液体酿酒酵母营养缺陷型选择培养基SC,将C5D-P-M与C5D-P-M-CIT2Δ各接入5个三角瓶中,每个三角瓶中接入3×108个细胞(OD值为1.0时酿酒酵母细胞个数约为3×107个,通过所测定OD值可计算出每种菌株所需接种体积),每种菌株设置5个平行,菌体浓度OD600,在0 h 、3 h、6 h、9 h、12 h、15 h、23 h、31 h、39 h、47 h、55 h、63 h、71 h分别取培养液1.5 mL。发酵样品用0.45 μm醋酸纤维滤膜过滤,滤液放于-80℃冰箱保存用于后续的实验。发酵乙醇的产量用高效液相色谱分析仪(HPLC)进行分析。色谱分离柱为BioRad公司离子色谱柱(300 mm×7.8 mm,9 μm),保护柱为Micro-Guard cation H(30 mm×4.6 mm),流动相为4~12 mmol/L H2SO4,流速为0.5 mL/min,折光示差检测器SHO-DEX RI-101进行检测。
③剩余木糖含量测定。从平板上挑取C5D-P-M与C5D-P-M-CIT2Δ分别接入酿酒酵母营养缺陷型选择培养基SC中,于30℃ 160 r/min培养,至培养基OD值约为1.0时用分光光度计测定其准确OD值。取10个已灭菌50 mL三角瓶,分别装入10 mL液体酿酒酵母营养缺陷型选择培养基SC,将C5D-P-M与C5D-P-M-CIT2Δ各接入5个三角瓶中,每个三角瓶中接入3×108个细胞(OD值为1.0时酿酒酵母细胞个数约为3×107个,通过所测定OD值可计算出来具体每种菌株所需接种体积),每种菌株设置5个平行,菌体浓度OD600,在0 h 、3 h、6 h、9 h、12 h、15 h、23 h、31 h、39 h、47 h、55 h、63 h、71 h分别取培养液1.5 mL。发酵样品用0.45 μm醋酸纤维滤膜过滤,滤液放于-80℃冰箱保存用于后续的实验。发酵乙醇的产量用高效液相色谱分析仪(HPLC)进行分析。色谱分离柱为BioRad公司离子色谱柱(300 mm×7.8 mm,9 μm),保护柱为Micro-Guard cation H(30 mm×4.6 mm),流动相为4~12 mmol/L H2SO4,流速为0.5 mL/min,折光示差检测器SHO-DEX RI-101进行检测。
2 结果与分析
2.1 酿酒酵母C5D-P-M中CIT2基因的敲除
通过PCR方法获得敲除组件(1 808 bp),扩增测序分析,证实获得的序列与理论设计引物序列一致。
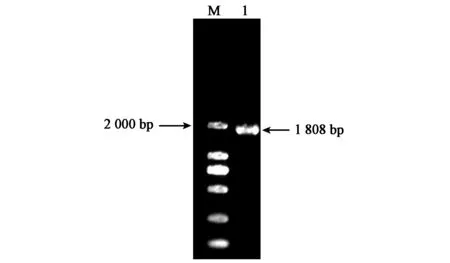
图1 敲除组件PCR电泳对照图Fig.1 Knockout component PCR electrophoretogram.M:DL 2 000; 1:敲除组件1 808 bp
2.2 荧光定量PCR测定C5D-P-M-CIT2Δ菌株木糖代谢相关基因的调控作用
荧光定量PCR验证结果如图2所示,其中有6个基因包括ACO1、GND1、IDH2、SDH1、TDH3、TKL1在C5D-P-M-CIT2Δ中比在C5D-P-M中表达上调,2个基因包括FUM1、MDH1在C5D-P-M-CIT2Δ中比在C5D-P-M中表达下调。表达上调的基因在一定程度上可以使发酵木酮糖的乙醇得率提高,而表达下调的基因则对糖代谢的推动作用不明显甚至起抑制作用。
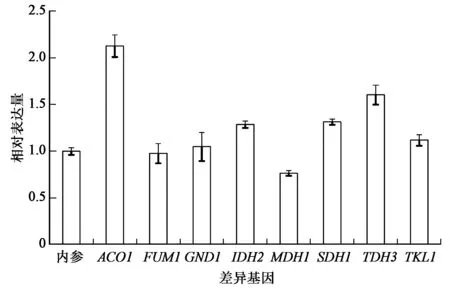
图2 荧光定量PCR结果Fig.2 Fluorescence quantitative PCR result.
2.3 工程菌株生理生化的测定
2.3.1 生长速率结果 C5D-P-M与C5D-P-M-CIT2Δ两株菌的生长速率如图3所示。其中同一时间C5D-P-M-CIT2Δ比C5D-P-M生长速度略快,尤其在对数生长期时C5D-P-M-CIT2Δ比C5D-P-M的生长速率更快,到达稳定期生长速率基本持平。在整个发酵过程中,突变菌株平均增速是出发菌株的1.272倍,适应期和对数生长期的增速明显。
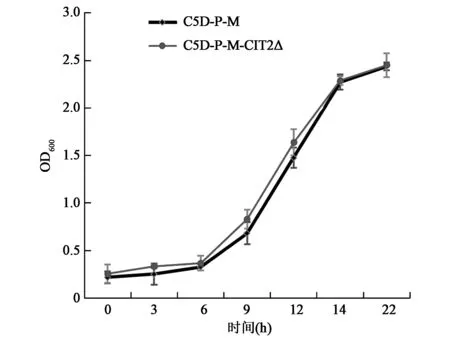
图3 C5D-P-M-CIT2Δ与C5D-P-M生长速率比较Fig.3 The growth rate of C5D-P-M-CIT2Δ compared with C5D-P-M.
2.3.2 剩余葡萄糖含量比较结果 C5D-P-M与C5D-P-M-CIT2Δ两株菌的剩余葡萄糖量比较如图4所示。其中,在适应期突变菌株C5D-P-M-CIT2Δ的剩余葡萄糖量略低于出发菌株;在对数期的12~31 h内突变菌株的剩余葡萄糖量明显低于出发菌株,且差异显著,尤其是在15~23 h内差异是极显著的;随着菌株进入平稳期,两株菌的剩余葡萄糖量的差异越来越小,直到葡萄糖利用完全。

图4 剩余葡萄糖含量Fig.4 Residual glucose quantity.
2.3.3 剩余木糖含量比较结果 C5D-P-M与C5D-P-M-CIT2Δ两株菌的剩余木糖量比较如图5所示。在适应期两株菌的木糖含量随着时间的延长都在减少,其中突变菌株C5D-P-M-CIT2Δ的木糖含量明显低于出发菌株C5D-P-M,且差异显著;进入对数期后两株菌的剩余木糖含量还是在不断减少,突变菌株的剩余木糖含量始终低于出发菌株,但差异越来越小。

图5 剩余木糖含量Fig.5 Residual xylose quantity.
3 讨论
木糖异构酶是木糖利用中的关键酶[22]。本研究利用转入木糖异构酶基因的重组酿酒酵母C5D-P-M,以DUP240家族中的CIT2基因为研究对象,通过基因敲除技术将CIT2基因去除,获得缺失CIT2基因的酿酒酵母C5D-P-M-CIT2Δ后,以出发菌株C5D-P-M为对照,研究其生长速率、剩余葡萄糖含量及剩余木糖含量生理生化指标及进行荧光定量PCR分析。在生长速率测定中前期选择每2 h取样一次,后期每隔12 h取样一次,根据所测数据做图所得,在接种量相同的情况下,已经敲除CIT2基因的酿酒酵母C5D-P-M-CIT2Δ比未敲除基因的对照菌株C5D-P-M增速明显,且实验菌株是对照菌株前期每2 h平均增速的3.2倍,后期平均增速为1.6倍,在整个发酵过程中,平均增速为1.3倍,前期增速约是后期的2倍,前期增速明显大于后期。由此可知当敲除酿酒酵母中CIT2基因后,加强了酿酒酵母本身的糖运输或糖代谢过程,当酿酒酵母有充足的能源时,其生长速率明显提高,但当糖运输或糖代谢达到一定水平时其生长速率增加相对缓慢。
根据剩余葡萄糖含量可知两株菌对葡萄糖的利用率都是100%,但是葡萄糖要比木糖先利用完,这在葡萄糖与木糖共发酵过程中是普遍存在的[20],在48~60 h之间完全用完。实验菌株C5D-P-M-CIT2Δ比对照菌株C5D-P-M利用葡萄糖的效率要略高,这是因为在敲除CIT2基因后,实验菌株对糖的运输能力比对照菌株略有加强,因此带动了糖代谢的加速进行,最终表现在时间上就是实验菌株比对照菌株到48 h对葡萄糖的利用量多约0.2 g/mL。木糖剩余量与葡萄糖剩余量在趋势上是相同的,总体都是实验菌株比对照菌株利用的能力要略强,说明到后期两株菌对木糖的利用率是相当的,由此可知酿酒酵母中CIT2基因的敲除主要影响的是糖运输过程,并且此基因对糖运输过程是抑制与阻碍作用,对于糖代谢过程是受糖运输过程影响,当有丰富糖源供给时会增强糖代谢流向下游,但当糖代谢过程达到一定程度后糖运输对糖代谢的推动作用就会不明显。综上所述CIT2基因对酿酒酵母利用木糖具有负调控作用。
根据Thomas等[23]进行的研究结果,针对酿酒酵母中糖代谢过程中所涉及到的相关8个基因进行荧光定量PCR分析,所得结果中6个相关基因ACO1、GND1、IDH2、SDH1、TDH3、TKL1的表达量均增加。根据这6个基因表达量的提高可知在糖运输作用加强后,酿酒酵母本身的糖代谢过程的底物量充足,促使其代谢过程中相关基因的表达加强,由于木糖代谢的加入,使得糖酵解本身向戊糖磷酸途径加多,与木糖代谢一致转化为3-磷酸甘油,因此TDH3(3-磷酸甘油醛脱氢酶)的表达量增加,随之加强了糖代谢过程,转向乙醇发酵。
[1] Cheng J J,Timilsina G R. Status barriers of advanced biofuel technologies[J]. Renew. Energy,2011,36:3541-3549.
[2] Tommy D,Uffe J,Jorgen E O,etal.. Looking at biofuels and bioenergy[J]. Science,2006,312(23):1743-1748.
[3] Farrell A E, Plevin R J, Turner B T,etal.. Ethanol can contribute to energy and environmental goals[J]. Science,2006,311(5760):506-508.
[4] Hahn-Hägerdal B, Linden T, Senac T,etal.. Ethanolic fermentation of pentoses in lignocellulose hydrolysates[J]. Appl. Biochem. Biotechnol., 1991,28-29:131-144.
[5] 崔 涛,陈 波,刘咸安. D-木糖操纵子[J]. 微生物学通报,1991,18(5):315-316.
[6] Zaldivar J, Nielsen J, Oslsson L. Fuel ethanol production from lignocelluloses: a challenge for metabolic engineering and process integration[J]. Appl. Microbiol. Biotechnol.,2001,56:17-34.
[7] Rodney J B,Nancy N,Bruce S D. Fermentations with new recombinant organisms[J]. Biotechnol. Prog.,1999,15:867-875.
[8] Usher J, Balderas-Hernandez V, Quon P,etal.. Chemical and synthetic genetic array analysis identifies genes that suppress xylose utilization and fermentation inSaccharomycescerevisiae[J]. G3(Bethesda),2011,1:247-258.
[9] Stephen R H,Joseph O R,Kenneth M B,etal.. Automated yeast transformation protocol to engineer saccharomyces cerevisiae strains for cellulosic ethanol production with open reading frames that express proteins binding to xylose isomerase identified using a robotic two-hybrid screen[J]. J. Labor. Automat., 2009,8:200-212.
[10] Prihardi K,Kazuo T,Shuzo T. Enhancement of xylose uptake in 2-deoxyglucose tolerant mutant ofSaccharomycesevervisiae[J]. J. Biosci. Bioengin., 2011,111(5):557-563.
[11] Ronald E H,Nasib Q,Stephen R H,etal.. Expression of a heterologous xylose transport in aSaccharomycescerevisiaestrain engineered to utilize xylose improves aerobic xylose consumption[J]. Appl. Microbiol. Biotechnol.,2008,80:675-684.
[13] Utrilla J, Licona-Cassani C, Marcellin E,etal.. Engineering and adaptive avolution ofEscherichiacolifor D-lactate fermentation reveals GatC as a xylose transporter[J]. Metabolic Engin.,2012,14:469-476.
[14] Nei M, Gu X, Sitnikova T,etal.. Evolution by the birth-and-death process in multigene families of the vertebrate immune system[J]. Proc. Natl. Acad. Sci. USA,1997,94:7799-7806.
[15] Carla F M,David R,Nicolas Z,etal.. TheSaccharomycescereviaiaeYFR041C/ERJ5 gene encoding a type I membrane protein with a J domain is required to preserve the folding capacity of the endoplasmic reticulum[J]. Biochim. Biophys. Acta,2007,1773:232-242.
[16] Laurence D, Be′ne′dicte W, Ve′ronique L L,etal.. An evolutionary scenario for one of the largest yeast gene families[J]. Trends Genit.,2006,22:10-15.
[17] Poirey R, Despons L, Leh V,etal.. Functional analysis of theSaccharomycescerevisiaeDUP240 multigene family reveals membrane-associated proteins that are not essential for cell viability[J]. Microbiology,2002,148:2111-2123.
[18] Kasavi C, Eraslan S, Oner E T,etal.. An integrative analysis of transcriptomic response of ethanol tolerant strains to ethanol inSaccharomycescerevisiae[J]. Mol. BioSys. Mol. BioSyst., 2016, 12:464-476.
[19] Spode I, Maiwald D, Hollenberg C P,etal.. ATF/CREB sites present in sub-telomeric regions ofSaccharomycescerevisiaechromosomes are part of promoters and act as UAS/URS of highly conserved COS genes[J]. J. Mol. Biol., 2002,319(2):407-420.
[20] Wirth B, Louis V L, Potier S,etal.. Paleogenomics or the search for remnant duplicated copies of the yeast DUP240 gene family in intergenic areas[J]. Mol. Biol. Evol., 2005,22:1764-1771.
[21] Despons L, Wirth B, Louis V L,etal.. An evolutionary scenario for one of the largest yeast gene families[J]. Trends Genet., 2006,22(10),10-15.
[22] Marko K, Harry R H, Ann K S,etal.. High-level functional expression of a fungal xylose isomerase: the key to efficient ethanolic fermentation of xylose bySaccharomycescerevisiae[J]. Yeast Res., 2003,4:69-78.
[23] Tomas W J, Igor V G, Jane G,etal.. Genome sequence of the lignocellulose-bioconverting and xylose-fermenting yeast Pichia stipitis [J]. Nature Biotechnol., 2007,25:319-326.
Research on the Effects ofCIT2 Gene on Xylose Utilization of the RecombinantSaccharomycescerevisiae
LI Wei-wei1,2, ZHANG Ji1, CHEN Chao-ru1, WANG Zhi1, QU Juan-juan2*, DUN Bao-qing1*, LI Gui-ying1, LU Ming1
1.CropGeneticResourcesandGeneticImprovementofMajorNationalScienceandEngineering,InstituteofCropScience,ChineseAcademyofAgriculturalSciences,Beijing100081,China;2.College of Life Science, Northeast Agricultural University, Harbin 150030, China
Xylosefermentationisoneofthebottleneckofcellulosicethanolindustry.ThetransformationofSaccharomyces cerevisiaeforxylosemetabolismhasbecomeoneoftheresearchhotspotatpresent.InordertostudytheinfluenceofCIT2geneonxyloseutilizationofSaccharomyces cerevisiaestrainC5D-P-Mforglucoseandxyloseco-fermentation,theCIT2genedisruptioncassetteproducedbyoverlappingPCRtechnology,transformedintoC5D-P-MforknockoutofCIT2byhomologousrecombinationandmutantstrainC5D-P-M-CIT2Δwasgainedfinally.IncontrastwithC5D-P-M,thegrowthrate,glucoseutilization,xyloseutilization,andethanolproductionofC5D-P-M-CIT2Δhadimprovedtosomeextent.TheresultsuggestedCIT2geneisonefactorthatcaninfluencethexyloseutilizationanditsknockoutcanimprovexyloseutilizationofSaccharomyces cerevisiae.
Saccharomyces cerevisiae; CIT2gene;geneknockout;xylosemetabolism
2016-07-20; 接受日期:2016-09-30
公益性行业(农业)科研专项(201503135-16)资助。
李维维,硕士研究生,研究方向为微生物学。E-mail: zhangji1140@126.com。*通信作者:曲娟娟,教授,博士生导师,研究方向为微生物遗传育种。E-mail:juanjuanqu@126.com;顿宝庆,副研究员,硕士生导师,主要从事生物质能源及秸秆的综合利用研究。E-mail:dunbaoqing@caas.cn
10.3969/j.issn.2095-2341.2016.06.11
