辣椒杂交种与亲本苗期叶片基因差异表达分析
2016-12-19徐小万王恒明何水林官德义
徐小万,李 颖,王恒明,李 涛,何水林,官德义
(1.广东省农科院蔬菜研究所,广东 广州 510640;2. 福建农林大学作物科学学院,福建 福州 350002)
辣椒杂交种与亲本苗期叶片基因差异表达分析
徐小万1,李 颖1,王恒明1,李 涛1,何水林2,官德义2
(1.广东省农科院蔬菜研究所,广东 广州 510640;2. 福建农林大学作物科学学院,福建 福州 350002)
目前辣椒杂种优势的利用已取得很大成效,但是辣椒杂种优势的分子机理仍然不明。辣椒杂种优势的分子机理是一个重要的生物学和农学课题。为比较分析苗期辣椒杂交种(D6×D7,D7×D6)及其亲本(D6、D7)的成熟功能叶转录谱,把4个功能叶cDNA 文库在 Illumina HiSeq 2000 平台进行 RNASeq 分析。通过转录组测序,共得到0.6 亿条原始的 reads,经过筛选获得 0.44 亿条高质量reads(100 bp)。基因存在与缺失变异统计结果表明,单亲表达一致型有1068个(6.20%)和780个(4.56%)基因分别在正交(D6×D7)和反交(D7×D6)中被检测到,在4种基因存在与缺失变异类型中所占比例比较高。对双亲表达量不同的基因作进一步表达模式分析,结果表明,杂种(正、反交)与双亲相比,大部分基因呈均非加性表达,加性表达基因比例不足8%,在非加性表达基因中,超显性表达的基因占大多数,正、反交分别为66.08%和62.96%,这与偏单亲的显性模型分析结论相一致。
辣椒;杂种优势;转录组;RNA-seq;
杂种优势是指遗传结构不同的两个群体(或个体)杂交所产生的F1代在生产、生活、繁殖和适应性等方面优于双亲均值或超过两个亲本的现象[1]。杂交育种是根据品种的选育目标选配亲本,通过杂种优势群的划分、自交系的培育、对自交系一般配合力和特殊配合力的检测等手段,获得新品种的重要育种途径之一,但执行起来不仅费力费时而且育种成本也很高。如果杂种优势被分子工具所预测,进而快速筛选出强优势组合,将大大提高杂交育种的效率,这也是育种学家对杂种优势机理非常感兴趣的原因[2-5]。然而杂种优势的研究虽有百余年的历史,但其根本机理仍然未知[6]。
辣椒是世界上最重要的蔬菜作物之一,既可鲜食,又是重要的调味料[7]。辣椒杂种优势分子机理的探索已有约50年的历史,特别是近年来各种分子标记技术在辣椒群体划分及遗传距离研究中取得了一定的进展[8-16]。学者们利用上述各种分子标记对辣椒杂种优势预测进行了大量研究,然而其结果却不尽相同。辣椒杂种优势机理是一个极其复杂的问题,应该从表型、生理生化、遗传学、基因组学以及转录组学等层面开展研究。从基因表达的角度对作物杂种优势展开相关探索,如模式植物水稻[17-19]、玉米[20-22]和拟南芥[23]已有成功的范例,但从基因表达层面来分析辣椒杂种优势机理仍处于空白状态。本研究采用RNA-Seq分析辣椒杂交种F1(正反交)与双亲功能叶的基因差异表达,可为认识辣椒杂种优势分子机理带来新的切入点。
1 材料与方法
1.1 试验材料
选取杂种优势强的辣椒组合(D6×D7)、反交组合(D7×D6)以及其双亲为试材。4个辣椒材料的种子播种于广东省农科院蔬菜所白云实验基地。田间种植 4周后,取第6片真叶为样品立即浸入液氮,带回实验室置于-80℃备用。
1.2 试验方法
1.2.1 数字表达谱测序及基因表达量计算 用改良的CTAB 法分别抽提上述4个辣椒材料的总 RNA,利用Oligo (dT)富集得到 mRNA,将其分别打断成 200 nt,随机引物六聚体反转录合成 cDNA,经末端修复、加 polyA 和接头后PCR扩增,最后进行 Illumina 测序。本研究共进行4个数字表达谱测序。将 Clean reads 比对到参考基因(尊辣1号)[24],最终获得 4 个样本的基因表达谱。使用 RPKM 法(Reads Per Kb per Million reads)[25]计算基因表达量,得到的基因表达量可直接用于比较不同样本间的基因表达差异。
1.2.2 基因表达模式划分 杂种和双亲相比,基因差异表达呈现以下5种模式:双亲共沉默型、亲本特异表达型、杂种特异表达型、单亲表达一致型和双亲及杂种共表达型。前4种模式属基因表达质的差异,即存在与缺失变异,而第5种模式则属基因表达量的差异。
杂种与双亲相比,另一种方法可依据A值划分为高亲、中亲和低亲3大类7小类[26]:

式中,high parent、F1、low parent分别为表达量高的亲本、杂交种和表达量低的亲本的表达量。高亲有2个小类 (A值<0.0,A值为0~0.2),中亲有3个小类(A值为0.2~0.4、0.4~0.6、0.6~0.8),低亲有2个类别(A值为0.8~1.0、A值>1.0)。
1.2.3 基因在双亲中表达量不同时基因表达模式划分 对双亲表达量不同的基因作进一步的表达模式分析,d/|a|的计算公式如下:

式中,F1为杂种的表达量,μ为双亲表达量的均值,P1为亲本1的表达量。依据d/|a|值可划分为负超显性、负显性、部分显性(负)、加性、部分显性(正)、正显性和正超显性7类表达模式,其d/|a|值分别为(-∞,-1.2)、[-1.2,-0.8)、[-0.8,-0.2)、[-0.2,0.2]、(0.2,0.8]、(0.8,1.2]、(1.2,+∞)。
1.2.4 差异基因的划分标准及Pathway 显著性富集分析 差异表达基因为 FDR≤0.05且倍数差异不低于 2 倍的基因。Pathway 显著性富集分析以 KEGG pathway 为单位,应用超几何检验,找出与整个基因组相比较后差异表达基因中显著性富集的 Pathway。FDR≤0.05 的Pathway 定义为在差异表达基因中显著富集的Pathway。通过 Pathway显著性富集能推定差异表达基因可能参与的最主要生化代谢途径和信号转导途径。
2 结果与分析
2.1 与辣椒基因组数据比对
为了分析苗期辣椒杂交种及其亲本的成熟功能叶转录谱,我们将准备好的功能叶cDNA文库在 Illumina HiSeq 2000 平台进行 RNASeq 分析。通过转录组测序,共得到0.6 亿条原始的 reads,经过筛选获得 0.44 亿条高质量的reads(100 bp)用于后续分析。使用Tophat软件将 Clean reads比对到辣椒(尊辣1号)参考基因序列[24],比对上的比例在 83.74%~85.43%之间,唯一比对上的比例在70.32%~79.69%之间。将4个样品 reads 比对到辣椒参考序列(Pepper.v.1.5),65.82%~67.13%的 reads 比对到外显子区域,2.49%~2.96%的 reads 比对到内含子区域,30.36%~31.57% 的 reads 比对到基因间区域(表1)。

表1 RNA-seq data比对统计
2.2 基因表达模式划分
2.2.1 基因存在与缺失变异统计 统计结果(表2)发现,正交组合D6×D7中共有17 217个基因表达,其中有15 128个(87.87%)在双亲和杂种中都表达;反交组合D7×D6中共有17 096个基因表达,其中有14 927(87.31%)个基因在双亲和杂种都表达。相比较而言,反交组合D7×D6中比正交组合少约120个基因;在4种存在与缺失变异类型中,单亲表达一致型和杂种特异表达型属于显性模型。

表2 基因存在与缺失变异统计
2.2.2 基于表达量的高亲、中亲、低亲分析 根据分类方法,有9078、6254个基因分别在杂种D6×D7和D7×D6以高亲模式表达;有3419、3443个基因分别在杂种D6×D7 和D7×D6以中亲模式表达;有4816和7616个基因分别在D6×D7和D7×D6以低亲模式表达。 从图1可知,图两侧的表达量比较高,说明偏单亲的显性模型是主要的,暗示优良的显性基因在杂种的聚合对杂种优势也是有贡献的。
2.3 基因在双亲中表达量不同时基因表达模式划分
对双亲表达量不同的基因作进一步的表达模式分析,结果(表3)表明,杂种(正交、反交)与双亲相比,大部分基因均呈非加性表达,加性表达基因比例不足8%,在非加性表达基因中,超显性表达的基因占大多数,正交、反交分别为66.08%和62.96%,这种分析方法恰好与图1的分析结论相一致;而显性表达的基因比例较低,正交、反交分别为12.50%和13.82%;部分显性表达的基因居中。在超显性表达的基因中,正交、反交杂种F1,正超显性表达个数约大于负超显性。

图1 基于表达量的高亲、中亲、低亲分析结果
2.4 两两比较差异基因表达分析及Pathway 显著性富集分析结果
用RPKM值估计基因的表达水平,RPKM值的变化范围是0~104。以FDR ≤ 0.05且│log2 (FC)│≥ 2 为标准,在亲本D6和D7间检测到351个差异表达基因,仅有17个基因在杂种D6×D7和 D7×D6中存在差异表达。亲本与杂种间的差异表达基因数为197~342个。亲本与杂种间的差异基因表达个数小于双亲间差异基因表达个数,但大于正反交杂种间差异表达基因个数(表4)。杂种与母本的差异基因表达数大于杂种与父本间的差异基因表达数。

表3 基于双亲表达量不一致的杂种基因表达模式分析
利用基因功能注释(Gene ontology,GO)将DEGs进行功能分类。我们将正交D6×D7和反交 D7×D6两个杂交种的 DEGs分类到 36 个功能亚范畴(图略),在生物途径范畴中,氧化还原过程和转录调控、基因调控所占比例最大;在分子功能范畴中,连接和催化过程所占比例最大;在细胞成分范畴中,细胞和细胞部分所占比例最大。
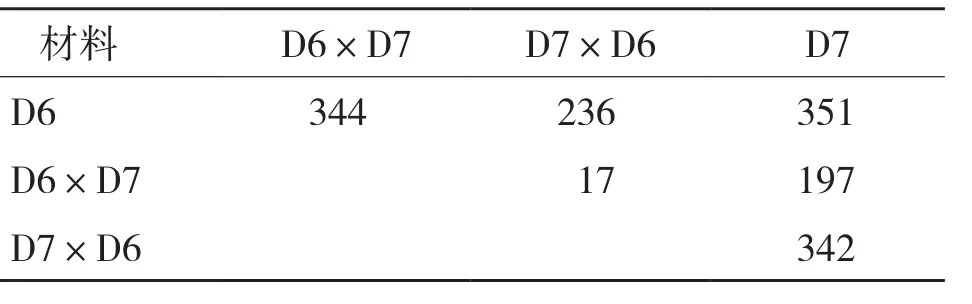
表4 4个基因型辣椒差异表达基因两两比较间的差异基因表达数
进一步统计分析了正交D6×D7和反交 D7 ×D6两个杂交种的 DEGs在生物途径范畴中重要的GO类别,这些 GO 类别为分析正交和反交可能与杂种优势有关的重要生物学途径提供了线索。几个重要的 GO 类别,如氧化还原、转录调控、蛋白质折叠、真菌防御反应、过氧化氢反应和应对冷热反应等在两个杂交种都有富集现象,说明正交D6×D7和反交D7×D6两个杂交种可能需要相同的生物学过程来维持叶片功能。然而,正反交两个杂交种也存在明显差异的 GO 类别,例如电子传递、光呼吸、调控植株过敏反应和MAPK级联途径类别只在正交D6× D7杂交种富集。
3 结论与讨论
尽管植物杂种优势在育种中已经被广泛应用,对农业有重要作用,但是人们对作物杂种优势的分子机理尚不清楚。辣椒杂种优势分子机理的探索已有约50年历史,但从基因表达层面来分析辣椒杂种优势机理仍处于空白状态。本研究应用 RNA-Seq 技术分析了正反交辣椒的转录谱,共检测到 0.44 亿条高质量的、100 bp大小的 reads。研究结果表明,反交组合D7×D6中共有17 096个基因表达,较正交组合少约120个基因;有1068个(6.20%)和780个(4.56%)单亲表达一致型基因分别在正交和反交中被检测到;有331个(1.92%)和211个(1.23%)杂种特异表达型基因分别在正交和反交中表达。已有研究表明,单亲表达一致型与棉花所有产量性状呈正相关[28],杂种特异表达型与小麦杂种优势显著正相关[27]。在4种存在与缺失变异类型中,杂种特异表达型和单亲表达一致型属于显性模型,相关类基因所富集的代谢通路是杂种优势分子机理研究的重点。
对玉米[21]、苜蓿[29]、日本落叶松[30]研究表明,杂种中呈非加性表达模式的基因比例比较大。进一步分析表明,在肯定非加性表达在杂种优势形成中起主要作用的例子中,有研究认为超显性表达模式是杂种优势产生的原因[6,29]。本研究在非加性表达基因中,超显性表达的基因占大多数,正、反交分别为66.08%和62.96%,而且在超显性表达的基因中,正、反交杂种F1,正超显性表达的基因个数约大于负超显性。这与在拟南芥[23]、苜蓿[29]、水稻[18,31-32]和玉米[33-35]等植物中许多基因呈现这种表达模式结果相一致。
本研究中4个样品基因的表达量为一个数量性状,杂种与双亲相比,表达模式分为3大类7个小类,其中偏单亲的显性表达模式是主要的,暗示优良的显性基因在杂种中的聚合对杂种优势也有贡献,相似的结论在水稻[19]杂种优势研究中也已发现。本研究中,正、反交杂种与母本的差异基因表达数(344,342)大于杂种与父本间的差异基因表达数(197,236),这与母本对玉米杂种转录组的影响大于父本的结论相一致[20]。在双亲与杂种间差异表达的基因个数所占比例并不是很大,即只有一小部分基因的表达变化导致了杂种表型的变化[36]。对于理解杂种优势的分子生物学基础,需要进一步探讨差异表达基因在杂种优势形成中的作用[37]。本研究利用基因功能注释GO将差异表达基因进行功能分类,这些 GO 类别为分析正交和反交可能与杂种优势有关的重要生物学途径提供了线索。几个重要的 GO 类别富集在正反交杂种中,如转录调控在水稻[31],防御与逆境反应在超级稻[37]杂种优势研究中相一致。然而,在正反交两个杂交种也存在明显差异的 GO 类别,例如电子传递、光呼吸、调控植株过敏反应和MAPK级联途径类别只在正交D6×D7杂交种富集,这些GO 类别将是下一步研究的重点。
[1]桑世飞,王会,梅德圣,等. 利用全基因组SNP芯片分析油菜遗传距离与杂种优势的关系[J].中国农业科学,2015,48(12):2469-2478.
[2]Goff S A. A unifying theory for general multigenic heterosis:energy efficiency,protein metabolism,and implications for molecular breeding[J]. New Phytologist,2011,189(4):923-937.
[3]桂君梅,王林友,范小娟,等. 基于InDel分子标记的籼粳杂交稻与粳粳杂交稻的杂种优势比较研究[J]. 中国农业科学,2016(2):219-231.
[4]李宏,黄道强,赖穗春,等. 杂种优势预测方法存在问题及一种有效的解决方案[J]. 广东农业科学,2014,41(19):10-12.
[5]郭战勇,吕盼晴,张向歌,等. 利用单片段代换系的测交群体定位玉米籽粒性状杂种优势位点[J]. 中国农业科学,2016,49(4):621-631.
[6]崔会会,项超,石英尧,等. 杂种优势形成的表观遗传学研究进展[J]. 植物遗传资源学报,2015,16(5):933-939.
[7]邹学校.辣椒遗传育种学[M].北京:科学出版社,2009:1-13.
[8]Sood S,Kumar N. Genetic estimates of fruityield and its component traits in bell pepper(Capsicum annuum L. var. grossum Sendt.)[J]. Sabrao Journal of Breeding & Genetics,2011,43:122-129.
[9]Sood S,Kumar N,Chandel K S,et al. Determination of genetic variation for morphological and yield traits in bell pepper(Capsicum annuum var. grossum)[J]. Indian Journal of Agriculturalences,2011,81(7):590-594.
[10]Kumar R L,Sridevi O,Kage U,et al. Heterosis studies in Chilli(Capsicum annuum L.)[J]. Plant Gene & Trait,2014,4(8):40-43.
[11]Hou J Z,Wang L L. Early forecast of physiological indexes of heterosis in pepper seedling[J]. Journal of Gansu Agricultural University,2009,44(5):44-49.
[12]Ryzhova N N,Kochieva E Z. Analysis of microsatellite loci of the chloroplast genome in the genus Capsicum(Pepper)[J]. Russian Journal of Genetics,2004,40(8):892-896.
[13]Bhadragoudar M R,Patil C G. Assessment of genetic diversity among Capsicum annuum L. genotypes using RAPD markers[J]. African Journal of Biotechnology,2011,10(76):17477-17483.
[14]Yumnam J S,Tyagi W,Pandey A,et al. Evaluation of genetic diversity of chilli landraces from north eastern India based on morphology,SSR markers and the Pun1 locus[J]. Plant Molecular Biology Reporter,2012,30(6):1470-1479.
[15]Thul S T,Darokar M P,Shasany A K,et al. Molecular profiling for genetic variability in Capsicum species based on ISSR and RAPD markers[J]. Molecular Biotechnology,2012,51(2):137-147.
[16]Patel A S,Sasidharan N,Vala A G,et al. Genetic relations in Capsicum annuum L. cultivars through microsatellite markers:SSR and ISSR[J]. Electronic Journal of Plant Breeding,2011,2(1):67-76.
[17]Huang S,Zhang Y,et al. Genome-wide transcriptome profiles of rice hybrids and their parents[J]. International Journal of Molecular Sciences,2014,15(11):20833-20845.
[18]He G,Deng X W. Global epigenetic and transcriptional trends among two rice subspecies and their reciprocal hybrids.[J]. Plant Cell,2010,22(1):17-33.
[19]Zhai R,Yue F,Wang H,et al. Transcriptome analysis of rice root heterosis by RNASeq[J]. Bmc Genomics,14:19.DOI:10.1186/1471-2164-14-19.
[20]Paschold A,Jia Y,Marcon C,et al. Complementation contributes to transcriptome complexity in maize(Zea mays L.) hybrids relative to their inbred parents.[J]. Genome Research,2012,22(12):2445-2454.
[21]Li H,Liu T,Cao Y,et al. Transcriptomic analysis of maize mature embryos from an elite maize hybrid Zhengdan958 and its parental lines[J]. Plant Growth Regulation,2015,76(3):315-325.
[22]Ding H,Qin C,Luo X,et al. Heterosis in early maize ear inflorescence development:a genome-wide transcription analysis for two maize inbred lines and their hybrid[J]. International Journal of Molecular Sciences,2014,15(8):13892-13915.
[23]Fujimoto R,Taylor J M,Shirasawa S,et al. Heterosis of Arabidopsis hybrids between C24 and Col is associated with increased photosynthesis capacity[J]. Proceedings of the National Academy of Sciences,2012,109(18):7109-7125.
[24]Kim S,Park M,Yeom S I,et al. Genome sequence of the hot pepper provides insights into the evolution of pungency in Capsicum species[J]. Nature Genetics,2014,46(3):270-278.
[25]Mortazavi A,Williams B A,Mccue K,et al. Mapping and quantifying mammalian transcriptomes by RNA-Seq[J]. Nature Methods,2008,5(7):621-628.
[26]Trapnell C,Pachter L,Salzberg S L. TopHat:discovering splice junctions with RNA-Seq[J]. Bioinformatics,2009,25(9):1105-1111.
[27]Wu L M,Ni Z F,Wang Z K,et al. Relationship between differential expression patterns of multigene families and heterosis in a wheat diallel crosses[J]. Acta Genetica Sinica,2001,28(28):256-266.
[28]Xing C Z,Zhao Y L,Shu-Xun Y U,et al. Relationship between gene differential expression of leaves in full opening flower stages of hybrids & their parents and heterosis in pest-resistant cotton[J]. Acta Genetica Sinica,2006,33(10):948-956.
[29]Li X,Wei Y,Dan N,et al. Comparative gene expression profiles between heterotic and nonheterotic hybrids of tetraploid Medicago sativa[J]. BMC Plant Biology,2009,9(1):107.
[30]Li A,Fang M D,Song W Q,et al. Gene expression profiles of two intraspecific Larix lines and their reciprocal hybrids[J]. Molecular Biology Reports,2012,39(4):3773-3784.
[31]Zhang H Y,He H,Chen L B,et al. A Genome-Wide Transcription Analysis Reveals a Close Correlation of Promoter INDEL Polymorphism and Heterotic Gene Expression in Rice Hybrids[J]. Molecular Plant,2008,1(5):720-731.
[32]Huang Y,Zhang L,Zhang J,et al. Heterosis and polymorphisms of gene expression in an elite rice hybrid as revealed by a microarray analysis of 9198 unique ESTs[J]. Plant Molecular Biology, 2006,62(4):579-591.
[33]Swanson-Wagner R A,Jia Y,Decook R,et al. All possible modes of gene action are observed in a global comparison of gene expression in a maize F1 hybrid and its inbred parents[J]. Proceedings of the National Academy of Sciences of the United States of America,2006,103(18):6805-6810.
[34]Stupar R M,Springer N M. Nonadditive expression and parent-of-origin effects identified by microarray and allele-specific expression profiling of maize endosperm[J]. Plant Physiology,2007,145(2):411-425.
[35]Uzarowska A,Keller B,Piepho H P,et al. Comparative expression profiling in meristems of inbred-hybrid triplets of maize based on morphological investigations of heterosis for plant height[J]. Plant Molecular Biology,2007,63(1):21-34.
[36]许晨璐. 落叶松基因挖掘及基因差异表达与杂种优势形成机制探讨[D]. 北京:中国林业科学研究院,2013.
[37]许晨璐,孙晓梅,张守攻. 基因差异表达与杂种优势形成机制探讨[J]. 遗传,2013,35(6):714-726.
[38]Ge X,Chen W,Song S,et al. Transcriptomic profiling of mature embryo from an elite superhybrid rice LYP9 and its parental lines[J]. BMC Plant Biology,2008,8(1):1-20.
(责任编辑 崔建勋)
Differentially expressed gene profile of hot pepper seedling leaves between hybrid and its parental lines
XU Xiao-wan1,LI Ying1,WANG Heng-ming1,LI Tao1,HE Shui-lin2,GUAN De-yi2
(1.Vegetable Research Institute,Guangdong Academy of Agricultural Sciences,Guangzhou 510640,China;2.College of Crop Science,Fujian Agriculture and Forestry University,Fuzhou 350002,China)
In spite of the great success in utilizing heterosis in hot pepper (Capsicum annuum L.) breeding and production,the underlying molecular mechanisms are still not well-understood,which need to be resolved as they are important subjects in basic biology and agronomy. In this study,mature leaf transcriptomes from the F1 hybrids(D6×D7 and D7×D6) and their parental lines (D6,D7) were compared. cDNA libraries were prepared using the sixth true leaves on plants at seedlings followed by RNA-Seq on a Illumina HiSeq 2000 sequencer. RNA-Seq generated 0.6 billion raw reads,and 0.44 billion high-quality reads (100bp) were obtained after the filtering process. Statistical analysis of genes with presence/ deletion variations showed that there are 1068 (6.20%) and 780 (4.56%)genes in the “single parent express consistent type” in the direct (D6×D7) and reciprocal (D7×D6) F1 hybrids,respectively. A large portion of the transcriotomes in the four pepper genotypes belong to this type of genes. Gene expression profiling analysis indicates that in F1 hybrids (direct and reciprocal crosses),more genes fit into the nonadditive expression type compared to the parents. Less than 8% of the genes belong to the the additive expression type. In the non-additive expression type,a large number of the genes fit into the epistatic dominance expression pattern;it is 66.08% and 62.96% in direct and reciprocal F1 hybrids,respectively. The expression type of these genes fits into the partial single parent dominance type.
hot pepper;heterosis;transcriptome;RNA-seq
S641.303.6
A
1004-874X(2016)11-0036-07
2016-10-25
国家自然科学基金(31301776);广东省科技计划项目(2015B0202009,2016B070701010);广州市科技计划项目(201510010119)
徐小万(1975-),男,博士,副研究员,E-mail:xxw7505@163.com
李颖(1963-),女,研究员,E-mail:ly38469@163.com
徐小万,李颖,王恒明,等. 辣椒杂交种与亲本苗期叶片基因差异表达分析[J].广东农业科学,2016,43(11):36-42.
