糖蛋白质组学中基于化学反应的富集方法研究进展
2016-12-14包慧敏谢力琦陆豪杰
包慧敏, 谢力琦, 陆豪杰
(复旦大学化学系, 生物医学研究院和老年医学研究中心 上海 200433)
糖蛋白质组学中基于化学反应的富集方法研究进展
包慧敏, 谢力琦, 陆豪杰*
(复旦大学化学系, 生物医学研究院和老年医学研究中心 上海 200433)
蛋白质糖基化是一种广泛存在的重要的蛋白质翻译后修饰,糖基化肽在总酶解肽中占的比例不超过5%,这使得糖肽的分离富集成为糖蛋白质组学研究发展的关键技术之一。在诸多糖蛋白质组富集技术中,化学方法是富集技术的主导,本文从化学反应的角度介绍糖基化蛋白质富集的技术进展。富集过程按照连接和释放分别讨论,在连接过程中,重点介绍硼酸化学法、肼化学法、胺化学法和肟点击法;在释放过程中,以N-糖蛋白的酶释放法和O-糖蛋白的β-消除法为主导,一并介绍了最新的氧化断裂释放的化学法。最后,讨论总体富集策略的发展现状。该文以糖蛋白富集的共价反应为核心,分析不同方法的优缺点以及各技术在糖蛋白质组学研究中的应用和贡献。
糖蛋白质组;蛋白质糖基化;糖蛋白;富集;连接/释放;共价反应;综述
蛋白质糖基化在细胞间的识别、信息交流和传递中发挥重要的生物学功能。糖基化蛋白质在病原对宿主的感染、癌症的发生和转移等生物过程中起重要的作用[1,2]。蛋白质糖基化的异常和肿瘤的发生、发展密切相关,美国食品药品监督管理局(FDA)批准用在临床上的肿瘤标志物中有一半以上是糖蛋白[3]。甲胎蛋白(AFP)就是一种被广泛应用于肝癌早期临床诊断的血清糖蛋白,肝癌病人甲胎蛋白糖链上的核心岩藻糖含量显著增加[4,5]。已有研究表明,以蛋白质的核心岩藻糖化水平作为疾病诊断标志物比蛋白质水平更灵敏、更具特异性[6]。此外,用于前列腺癌诊断的前列腺特异性抗原(PSA)、用于胰腺癌监测的癌抗原19-9(CA19-9)、用于乳腺癌监测的癌抗原15-3(CA15-3)、癌抗原CA27-29和CA-125等都是糖基化蛋白质[7]。
哺乳动物蛋白质中超过50%发生了糖基化,但糖基化肽却仅占蛋白质酶解肽的2%~5%[8],加上糖链的微观不均一性以及在检测过程中非糖肽对糖肽质谱信号的抑制,这些都限制了糖蛋白检测的灵敏度[9]。将糖肽从复杂体系中高选择性地分离富集出来并达到质谱可以检测的水平是糖蛋白质组研究首先要解决的一个基础问题。
目前已经发展了多种糖蛋白的富集方法,按照作用原理可以分为通过非共价键连接的方法和通过化学反应形成共价键连接的方法。通过非共价键连接的方法包括亲和法(凝聚素法和免疫法)和亲水法。凝集素本身的特性使凝聚素法不具备普适性,且作用较弱[10,11]。免疫法利用抗原抗体结合的专一性,主要是在含有O-GlcNAc(O连接的N-乙酰葡糖胺)的糖蛋白或糖肽中应用,该方法缺乏通用性[12,13]。亲水法利用糖链多羟基的亲水性进行分离富集,由于肽段亲水基团的干扰,不可避免地存在非特异性吸附[14,15]。以上非共价连接方法的共同特点是选择性不好或普适性不够;而共价连接法利用糖基本身的活性基团(多羟基或氧化后的醛基)与固定相上的功能基团形成的共价键为作用力,结合牢固,可承受强烈的洗涤条件,因而在富集过程中有更高的选择性。
通过化学反应形成共价键进行富集的方法包括硼酸化学法、肼化学法、胺化学法和肟点击法。其中硼酸化学法利用糖链的顺式邻二醇结构可形成环状二酯进行富集,有条件温和、反应可逆和不破坏糖链结构的特点;其他3类方法均需要将糖肽通过高碘酸钠预氧化产生醛基,利用醛基的反应活性进行富集。由于共价连接法有高选择性的突出优点,加上和新型材料的兼容性,有越来越多的商品化试剂涌现,共价连接法富集是糖蛋白质组学研究中最活跃的领域。共价连接法包括共价连接肽链和共价连接糖链,共价连接糖链居多,是本文重点讨论的内容。
1 共价反应连接法
糖蛋白富集包括两个关键过程:糖链的连接和释放。糖链上特殊的邻位多羟基结构,是糖蛋白富集中共价反应连接法的基础。在连接过程中,根据糖链与外源功能基团的共价反应类型分为:硼酸化学法、肼化学法、胺化学法和肟点击化学法。富集过程中共价连接反应原理见图1。其中,硼酸化学法普适性强,能特异性富集所有顺式邻二醇结构的糖。肼化学法、胺化学法和肟点击法这3种方法都需要预氧化,将多羟基结构通过氧化反应转变为反应活性更高的醛基,醛基可以与酰肼发生肼腙反应(肼化学法),醛基可以与酰胺/苯胺发生胺化反应(胺化学法),醛基还可以与羟胺生成肟(肟点击法)。所有的共价连接法在连接过程中对N-糖、O-糖等均没有明显的歧视效应。
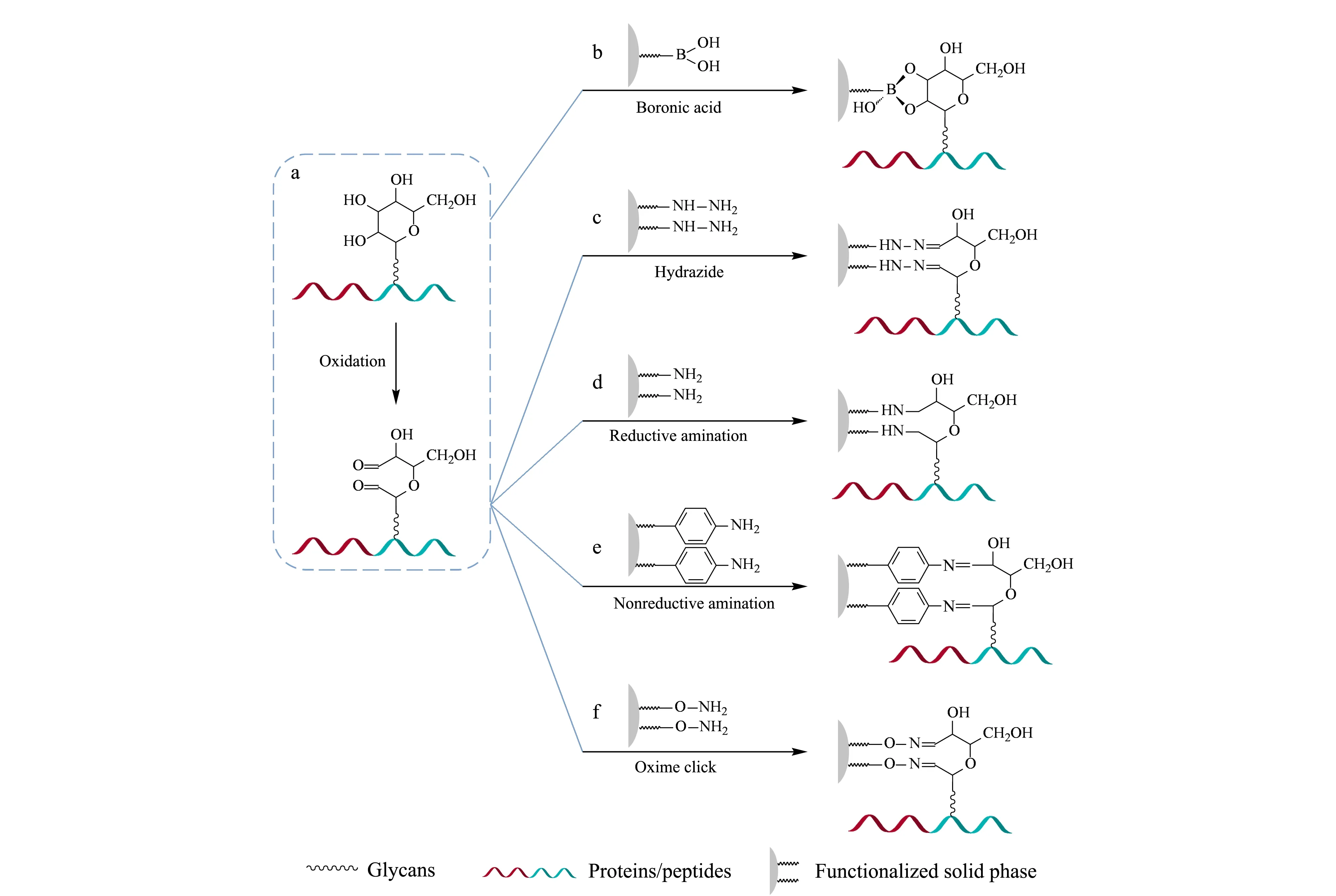
图 1 糖蛋白富集过程中共价连接反应原理图Fig. 1 Schematic diagrams of covalent reactions for conjugation during glycoproteins enrichment a. oxidation of cis-diol groups on glycans to aldehyde groups; b-f. covalent coupling glycoproteins to solid phase. Chemical conjugation methods include (b) boronic acid chemistry between cis-diol groups and boronic acids, and covalent reactions between aldehyde groups and the other functionalized groups, such as (c) hydrazide, (d) reductive amination, (e) nonreductive amination and (f) oxime click chemistry.
1.1 硼酸化学法
硼酸化学法利用硼酸基与糖链上的顺式二羟基的可逆结合进行富集,在碱性条件下,硼酸基与邻位顺式二醇发生酯化反应形成环状二酯;当溶液调至酸性条件下时,反应逆向进行,环状二酯发生水解释放出完整糖肽[16]。该二酯的解离常数大约在10-3~10-4数量级,据硼酸衍生物和反应底物的不同而异[17]。可逆反应结合常数低,伴随着相对低的选择性,将硼酸固定修饰在低吸附的固相表面将有助于该问题的解决。目前,有大量工作围绕硼酸功能化新材料作为固相载体辅助糖蛋白的富集进行,均取得不错的效果。
磁性纳米颗粒拥有大的磁效应系数,在溶液中容易分离,是一种理想的糖肽分离富集基质。本课题组[18]成功地合成了硼酸功能化的“核-卫星”结构的新材料,以Fe3O4@SiO2为“核”,以硼酸功能化的金纳米粒子Au@B(OH)2为“卫星”,中间以长的有机链相连。金纳米颗粒增加固相表面积,长的有机链减少了空间阻力,抑制了非特异性结合。将这一硼酸修饰的材料用于结肠癌组织中糖肽的富集,鉴定到155个糖蛋白,194个糖基化位点,其中有165个糖基化位点为首次报道(占85.1%)。由于合成材料表面不可能完全被硼酸基团覆盖,所以材料表面非特异性吸附很难避免。进一步利用聚甲基丙烯酸甲酯修饰的纳米颗粒竞争性地吸附非糖肽,通过两种不同修饰表面的纳米颗粒的协同作用,极大地提高了糖蛋白的选择性,简化了洗涤的步骤,糖肽的回收率提高到90%[19]。同时,也有报道将硼酸修饰到磁性碳纳米管[20]、两性离子包被的核壳结构的磁纳米颗粒[21]上。介孔纳米颗粒由于其具有高比表面积、大的孔容、均匀的孔径分布等优势被用于蛋白质/肽的吸附,发展了两步法合成硼酸功能化的介孔硅材(FDU-12-GA)并用于糖肽富集。将糖肽的检测限降低了两个数量级,大表面积和空间限域效应加速了反应的发生,上样时间缩短到15 min[22]。更进一步,硼酸嫁接介孔材料MCM-41利用硼酸的选择性和材料体积排阻的特点在糖肽富集中展现了优良的选择性(糖肽和非糖肽的物质的量比为1∶100)、高检测灵敏度(fmol)、大结合容量(40 mg/g)和高回收率(高达88.10%)[23]。
树枝状材料有多位点和亲水的特点,Wang等[24]将其功能化衍生,发现硼酸嫁接在树枝状材料上显示了很多的优点,如:有很好的酸解离常数,可达到10-5~10-6mol/L,比单个硼酸键合高出3~4个数量级;HRP(horseradish peroxidase)的结合容量可达(0.41±0.06) μmol/g,鉴定到9个糖基化位点,检出限达到10-14mol/L;结合/解离过程快,结合过程为1 min,解离过程为5 min,而通常硼酸粒子富集过程需1 h;对寡糖耐受。为了兼容体内糖蛋白存在的近中性环境,避免硼酸法在碱性条件下富集引起的糖蛋白降解。Zhang等[25]合成磁性核壳微球Fe3O4@PAA-AOPB,壳上修饰功能化苯硼基团,该基团的pKa接近7,可以与顺式二醇形成五元环酯,该硼酸功能化的微球展现了对糖蛋白的高选择性的优点(HRP和牛血清白蛋白(BSA)的质量比达1∶80)。
刘震课题组[26]长期从事硼亲和材料用于分离和分子识别的研究,他们总结出硼酸的亲和力与硼酸键的pKa之间的规律。通常硼酸键pKa低,硼酸亲和过程需要相对低的pH条件,同时具有较高的亲和力。另外,硼酸的亲和力与硼酸连接的配基结构以及硼酸修饰的支持介质有关,它们共同决定了亲和反应过程中与硼酸亲和作用力相竞争的二级作用力(氢键、离子键、疏水作用)的强弱,最终反映在硼酸的亲和能力上。他们总结了低pKa的硼酸结构类型,做了糖类物质的高效硼亲和固相标记[27],并发展了新的硼酸亲和方法用于糖蛋白的测定[28,29]。他们的工作对功能化硼酸配基的结构给出了一定的理论指导,对现有工作结果给出了很好的解释。目前硼酸化方法能够在接近中性的条件下实现与顺式二醇的反应,然而糖蛋白中有很多伯胺基团的存在,因此该方法离实际糖蛋白的富集还有很大的距离。
1.2 肼化学法
肼化学法和硼酸化学法同样是利用糖蛋白糖环上的邻二醇结构进行富集,不同的是肼化学法需要先将糖链上邻二羟基的结构氧化成醛基,醛基与肼基发生共价反应生成腙键,故又称肼腙法。肼化学反应法中的功能基团是肼基,可以是苯肼或酰肼,通过酰肼富集N-糖蛋白的选择性可达90%[30]。
2003年,Zhang等[31]使用此反应建立了N-糖蛋白固相萃取(solid phase extraction ofN-linked glycopeptides, SPEG)法,将肼化学为基础的方法用于N-糖肽富集。肼化学法可以结合不同的固定相,比如肼磁珠[32]、金纳米颗粒[33]、聚合物[34]、树枝状聚合物[35],已发展了一系列功能化材料用于糖蛋白的选择性富集。本课题组[34]设计并合成了肼基功能化的磁纳米材料(Fe3O4@PMAH),通过聚合物包被磁纳米颗粒,表面羧基与己二酸二酰肼形成肼功能化的磁球。丰富的肼基团可以高效并特异地富集糖肽,磁核便于溶液中的分离,亲水的聚合物表面能够减少非特异性吸附。为满足低丰度痕量糖蛋白的检测,需要发展更加灵敏、快速的富集新方法。本课题组[35]设计合成了肼功能化的树枝状材料,结合过滤辅助(FASP)技术用于高灵敏和高选择性富集N-糖肽的研究。树枝状聚合物(PAMAM)可以溶解于水相,并且表面拥有丰富的反应基团,因此在保证满足低丰度糖肽高效富集的同时,均相溶液反应保证了富集的重复性。将该方法应用于人血清样品中糖肽的分析,成功检测到158条糖肽,鉴定到60个不同的糖蛋白。
肼化学法和肽N糖酰胺酶F(PNGase F)结合富集N-糖蛋白在糖蛋白质组学中的应用最为广泛[36,37],也可以结合其他酶选择性地释放特定的糖肽。肼化学法也可通过改变氧化反应的条件,选择性氧化唾液酸的侧链[38]、甘露糖片段的顺式邻二醇、葡萄糖片段的反式邻二醇、O-GlcNAc蛋白质的乙酰葡糖氨基[39],实现对特定结构糖肽/糖蛋白的富集。肼化学反应法有反应专一性高、无位点偏向性、共价腙键稳定的优点;但肼化学法富集属于多步骤反应,条件不易控制,且操作较复杂,还有耗时较长的缺点。
1.3 胺化学法
胺化学法反应的功能基团是氨基,糖链上经过氧化后的醛基与固相载体上的氨基反应。胺化反应的关键步骤是羰基和氨基在酸性条件下生成Schiff碱,因Schiff碱的稳定性较差,通常是通过NaBH4还原为稳定的C-N单键最终形成稳定的仲胺,称为还原胺化法[40];或通过与能产生共轭的氨化物反应,比如苯胺,可以增强产物的稳定性,称为非还原胺化法。
和肼化学法相比,还原胺化减少了质谱分析前脱盐的步骤,可以简化操作。本课题组[41]设计并通过一步法合成了氨基功能化的磁性纳米颗粒,用于N-糖基化肽的富集,缩短提取时间到4 h,糖肽的检测灵敏度提高了2个数量级。在5 μL的人血清中鉴定到111个N-糖基化位点和108条糖基化肽,并成功将核酸酶B从牛血清中富集出来。在实际样品中,胺化反应可能会存在肽上伯胺与醛基的副反应,使非糖肽竞争性地抑制氨基磁珠对糖肽的富集。根据胺化机理,胺化反应要先形成Schiff碱为中间态,而在还原之前形成Schiff碱的过程是可逆的,大量存在的非糖肽会竞争性地和氧化后的醛基结合。邹汉法课题组[42]通过预先对肽链进行二甲基化封闭再用胺化学法富集糖肽,大幅度地提高了糖肽富集的效果,N-糖基化位点鉴定数量提高了110%(从126个到264个),对于N端是Ser/Thr的N-糖肽,则从2个增加到25个。
胺化反应中间体Schiff碱不稳定,不同于脂肪胺的是芳香胺与醛基反应产生的Schiff碱由于形成大的共轭体系可以增加其稳定性,不需要将C=N双键还原为C-N单键,而且共轭的Schiff碱在宽的pH范围内稳定[43],可以进一步简化操作。用苯胺代替脂肪胺与寡糖链的还原端生成稳定的胺化产物,该方法已经成功地用于糖链衍生的质谱鉴定中[44]。本课题组[45]设计并合成了苯胺功能化的磁纳米材料,将非还原胺化反应引入糖肽的富集中,发展了新的N-糖蛋白分离富集方法。
胺化学法与肼化学法类似,无偏向性,通过探索适合的含有共轭基团的试剂、发展非还原胺化,减少反应步骤,简化操作。胺化学法是近几年新发展的糖蛋白分离富集方法,利用胺化学法对糖蛋白大规模普适性分离富集的应用还有待进一步发展。
1.4 肟点击化学法
肟化反应是含有羰基的化合物(醛、酮类)与羟胺作用生成含有肟基C=NOH的反应。该反应是典型的亲核加成反应,经过加成消除历程。和酰肼比较,羟胺由于α-效应具有更强的亲核能力[46]。加上条件温和、产物稳定的优势,肟点击化学被用于很多领域,在糖组学上已经用于糖还原端标记的糖印迹[47]。受糖印迹的启发,肟点击化学法被用于唾液酸化糖肽的富集[48]。
Zeng等[49]利用唾液酸侧链的多羟基容易被氧化的特征,发展肟点击的方法标记唾液酸化蛋白质,并用于活细胞表面唾液酸化蛋白质的标记和示踪。步骤为:高碘酸钠温和氧化唾液酸产生醛基,苯胺催化与带有生物素标签的羟胺生成稳定肟键,最后,链酶亲核素富集细胞表面唾液酸化的糖蛋白。苯胺催化可以加速肟键生成,只需要低浓度的羟胺-生物素试剂,在中性条件下就可以标记细胞表面绝大部分唾液酸化的糖蛋白,同时不改变细胞活性。最近,发展了羟胺功能化的生物素作为连接分子,针对含有特定糖链(比如末端是唾液酸或半乳糖)的糖蛋白,通过肟化学法连接上生物素,最后通过商品化的链酶亲核素树脂分离富集,糖蛋白富集获得高的特异性[50]。
以上工作集中在标记示踪,本课题组[51]将肟点击与新材料结合,发展了用于N-糖肽富集的方法。首先设计并合成了羟胺功能化的磁性纳米颗粒,然后将氧化后的糖链通过肟化学连接。该方法富集过程在1 h以内,灵敏度提高到fmol水平,体现了良好的选择性(糖肽和非糖肽的物质的量比为1∶100)和重复性(变异系数CVs<20%),同时运用该方法成功将人类大肠癌患者血清中的N-糖基化蛋白质分离出来。
综上所述,硼酸化学法反应可逆、温和,适合对完整糖蛋白的分析,但选择性不高。醛基为基础的肼化学法、胺化学法和肟化学法虽然有预氧化步骤,增加了实验时间和操作的复杂度,但通过氧化产生的高活性醛基大大提高了糖肽富集的选择性和特异性。结合不同特点的新材料,发展新的方法,可以满足不同类型样品的研究需要。
2 糖蛋白释放方法
蛋白质主要的糖基化类型有两种:一种是N-糖基化,糖基片段通过氮原子链接在天冬酰胺的侧链上,PNGase F酶可以特异地切割天冬酰胺和糖链之间的连接;另一种为O-糖基化,糖基片段通过氧原子链接在丝氨酸或苏氨酸的侧链上,O-糖基化蛋白质在碱性条件下易发生β-消除反应。对于这两类糖基化蛋白质,富集后的释放原理见图2。
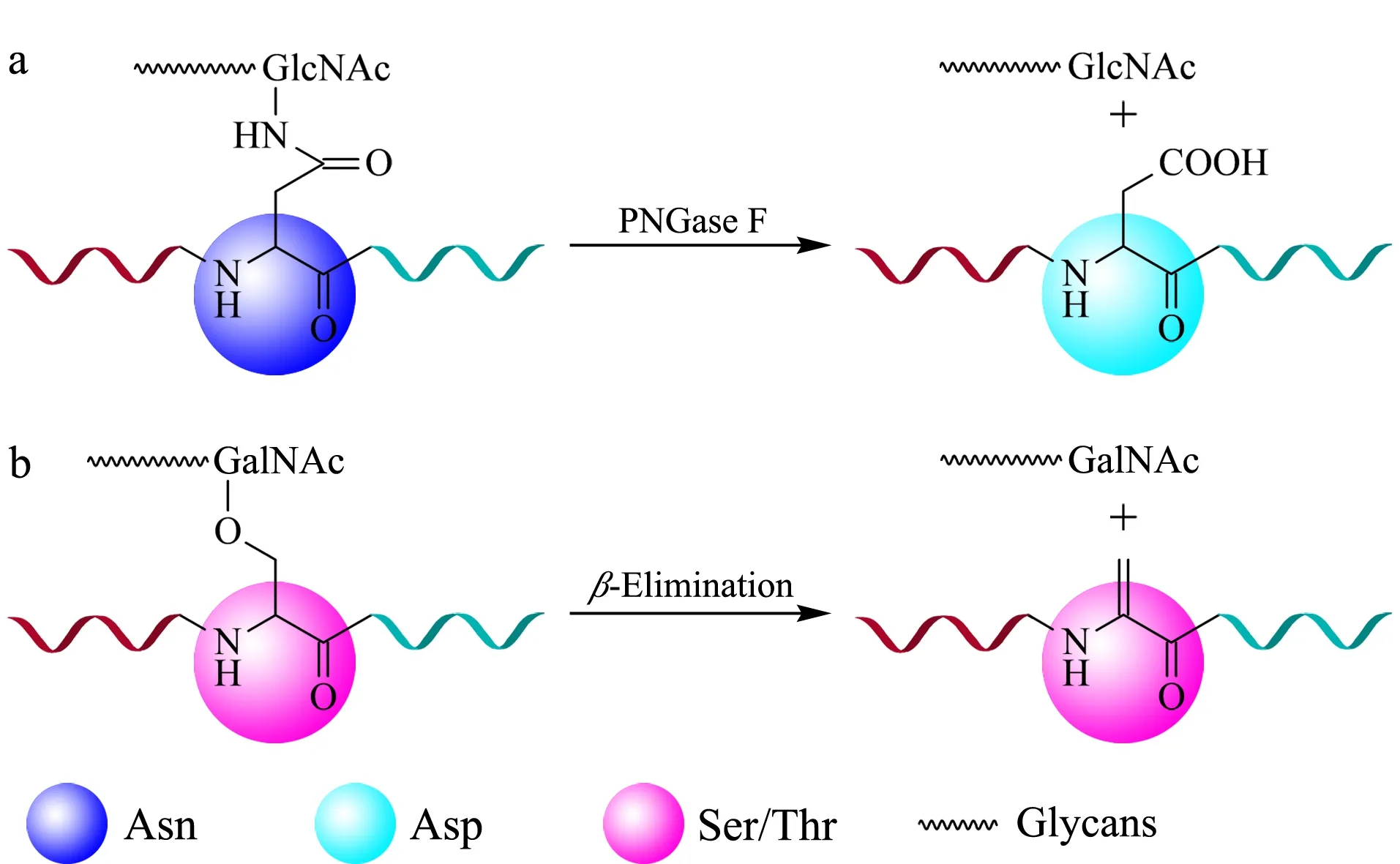
图 2 糖蛋白富集后的糖链或蛋白质的释放方法示意图Fig. 2 Schematic diagrams of the released glycans or proteins after glycoproteins enrichment a. N-glycosidase F (PNGase F) for N-glycoproteins, b. β-elimination to O-glycoproteins. GlcNAc: N-acetylglucosamine.
2.1N-糖蛋白的PNGase酶切法
酶切的方法可以用来分离糖链和肽链,常用的是PNGase和糖苷酶(glycosidase)两类酶,糖苷酶包括糖苷内切酶(endoglycosidase, Endo)和糖苷外切酶(exoglycosidase, Exo)。
PNGase F是目前普遍使用的鉴定N-糖基化位点的酶,在糖基化的研究中应用广泛。杨芃原课题组[52,53]将PNGase F酶结合同位素标记用于糖基化位点的定量研究。利用Endo不同亚型对糖型的专一性可以鉴定糖基化位点以及糖链结构信息。将Endo H与PNGAse F酶结合使用,从大鼠肝脏组织中鉴定到1 063个高甘露糖型和杂合型N-糖基化位点及相应的560个N-糖蛋白,其中大部分位点为首次发现[54]。钱小红课题组[55,56]采用可特异性酶切复杂型核心岩藻糖结构的Endo F3酶从人血浆样品中发现了超过100个含核心岩藻糖的糖基化位点,并发展了相应的MRM定量技术。对于过于复杂的糖链,将多种糖苷外切酶组合,即可分析糖链的组成、序列、连接和构象。促红细胞生成素和人血浆铜蓝蛋白的N-糖链结构得到精细解析就是成功的例子[57,58]。
2.2O-糖蛋白的β-消除法
O-糖蛋白缺乏如PNGase F这样具有普适性的酶,O-糖基化蛋白质研究的发展受到一定的限制。在对O-糖蛋白分离富集的研究中,β-消除米氏加成(β-elimination/Michael addition reaction)法的应用最为广泛。β-消除米氏加成法利用O-链接的糖蛋白在碱性条件下容易发生β-消除反应形成双键,而反应生成的α、β不饱和双键易与亲核基团发生加成反应,在O-链接的位点处修饰上连接基团,并利用连接基团的特有性质来选择性富集目标糖蛋白或糖肽。
Wells等[59]第一次将此方法从磷酸化肽的分析中迁移到O-糖肽的分离富集中,首次利用O-糖蛋白在碱性条件下发生β-消除反应产生不饱和双键,然后再加入二硫苏糖醇(DTT)或生物素戊胺(BAP)等亲电试剂进行米氏加成反应,然后通过巯基亲和柱或生物素亲和柱对标记了的O-糖肽进行亲和富集。他们发现在温和的β-消除反应中O-糖肽要比磷酸化肽更容易发生反应,而且可以通过磷酸化酶的处理消除磷酸化肽的干扰。实验不仅对合成的O-糖肽进行了成功的分析,而且对从鼠脑中纯化的Synapsin Ⅰ、核孔复合物中的Lamin B receptor及nucleo porin Nup155进行了糖基化位点分析,还预测如果使用同位素标记的DTT可以进行定量分析。本课题组[60]使用氨水、甲基胺、二甲基胺3种亲核试剂对β-消除后的O-糖肽进行米氏加成,对比了3种试剂的反应效果,并利用LC-MS测定了O-糖肽的糖基化位点。在此方法的反应条件下,磷酸化蛋白质或烷基化的半胱氨酸也容易发生消除加成反应,可以经过磷酸化酶处理或对照反应来消除干扰。另外,比如烷基化的半胱氨酸也会同时发生反应,需要另外一些对照实验来降低假阳性。
2.3 次氯酸钠消除法
寻找化学试剂、通过化学反应氧化释放糖链的研究一直在进行。N-溴代丁二酰亚胺(NBS)作为一种温和的氧化试剂,可以对糖肽氧化脱羧,当肽链较短时,通过氧化断裂可以得到完整糖链[61]。最近,Song等[62]报道了一种氧化释放糖链(oxidative release of natural glycans,ORNG)的新方法。利用次氯酸钠氧化,从N-糖蛋白和O-糖蛋白以及糖脂上释放出糖链,分别获得自由的还原态N-糖和被氧化为酸的O-糖,鞘糖脂则产生糖腈。
之前,次氯酸钠已经被用于蛋白质的降解,可以高效、选择性地降解天然糖复合物中糖苷的配基部分[63]。次氯酸钠可以帮助溶解酵母细胞壁并促进葡聚寡糖的提取[64],还能降解黏多糖(主要是葡糖氨基葡聚糖)[65,66]。目前尚没有次氯酸钠对糖蛋白的降解方面的系统研究。作为化学试剂,次氯酸钠的氧化没有选择性,凡样品中含有伯胺(比如肝磷脂、硫酸肝素)或含有巯基、碳碳双键等结构,均会被部分氧化降解。通过加化学试剂氧化释放糖链,此法可以与共价连接肽链的方法配合使用,作为糖链释放的方法之一,糖蛋白质组学研究中也迫切需要一种无歧视的糖链释放方法。
3 糖蛋白的富集策略
3.1 “top down”与“bottom up”策略
利用化学反应法对糖蛋白的分离富集进行研究的策略可分为“top-down”(捕获糖蛋白)和“bottom-up”(捕获糖肽)。硼酸化学法多采用“top down”策略,Sparbier等[67]利用硼酸修饰的磁珠对人血清中的糖蛋白进行了富集,证明了硼酸富集法和凝集素富集法的互补性。目前肼化学反应法多采用“bottom up”策略。Sun等[68]认为“top down”策略会受到蛋白质溶解性差和空间位阻的影响,而且被捕获蛋白质经酶解后的样品仍然很复杂,因而“bottom up”策略富集效果更好。他们做了5个标准糖蛋白糖肽的富集实验证明自己的观点。
3.2 糖蛋白共价连接和释放的整体策略
糖蛋白通过共价连接和释放可以用于富集N-糖肽或N-糖链,固相萃取法富集糖蛋白的两种流程见图3,首先通过醛基的共价反应将糖蛋白连接在固相介质上,PNGase F酶切释放N-糖肽或N-糖链进行分析。
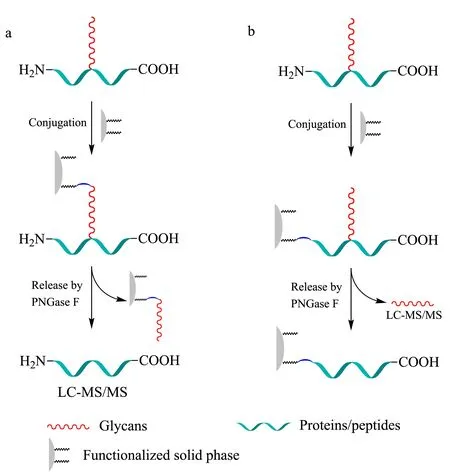
图 3 糖蛋白固相萃取富集策略Fig. 3 Strategies of solid-phase extraction for glycoproteins a. glycoproteins are conjugated to solid phase by glycans. N-linked peptides are then released by PNGase F and analyzed by LC-MS/MS. b. glycoproteins are conjugated to solid phase by N-terminal of peptides. N-glycans are then released by PNGase F and analyzed by LC-MS/MS.
Zhang等[31,69]发展了SPEG法,该方法分为4步:(1)高碘酸氧化糖链上的顺式二羟基为醛基;(2)醛基和肼树脂上的肼基反应生成腙;(3)胰蛋白酶处理固定在肼珠上的糖蛋白,洗去非糖基化的肽段;(4)PNGase F酶解处理,释放特异性结合在肼珠上的N-糖肽。这是肼化学法富集N-糖肽的经典方法。
之后,Zhang课题组[70]发展了一系列技术,丰富了蛋白糖链和糖蛋白的富集以及定量方法。其中以胺化反应为基础,利用商品化的氨基树脂,发展了GIG(glycoprotein immobilization for glycan extraction)方法富集糖链,通过PNGase F释放N-糖链,羟胺释放O-糖链,实现全部糖链的分析。在唾液酸化糖链的定量研究中,通过选择性氧化唾液酸产生醛基,苯胺同位素试剂作为标签连接在唾液酸端,通过质谱峰对唾液酸化蛋白质进行定量分析[71]。随后将该法用于对CHO细胞的唾液酸化N-糖的定量研究,证实建立方法的实际应用价值[72,73]。同样,以胺化反应为基础发展了糖肽的标记试剂QUANTITY(quaternary amine containing isobaric tag for glycan)[74],可供4组样品定量,并考察了作为定量方法的各项指标[75]。
前面提过的SPEG法是基于肼腙反应的原理建立的完整N-糖肽富集技术路线,目前该方法得到了广泛应用[76,77]。最近建立的NGAG(N-linked glycans and glycosite-containing peptides)法[78]可以同时获得N-糖蛋白、N-糖基化位点、N-糖链的信息,为全面系统研究N-糖蛋白提供了技术路线。
与此同时,该课题组[79]还发展了针对糖链的肼腙可逆萃取(reversible hydrazine solid-phase extraction, rHSPE)法。在肼树脂上,于甲醇-乙酸(85∶15, v/v)溶液中将糖链固定,在体积分数为10%的甲酸水溶液中水解腙键释放所有糖链,并应用该方法从血清中富集经过PNGase F酶切得到的N-糖链。该方法利用肼腙法的无选择富集和酸性条件可逆释放糖链的特征,发展了糖肽可逆富集的新方法。
3.3 特殊糖蛋白的富集策略
3.3.1 末端是唾液酸的糖蛋白的富集
唾液酸化的糖蛋白广泛参与人体的生理、病理过程。唾液酸通常位于糖蛋白的糖基化非还原末端,是糖基化修饰的终末步。通过改变氧化的条件,可实现选择性地氧化唾液酸化的糖链/糖肽,具体方法为:选用温和的条件(0~4 ℃, 1~2 mmol/L高碘酸钠,<10 min)选择性地氧化唾液酸侧链的多羟基为醛基;富集后,酸性条件下可以选择性地水解断裂唾液酸的糖苷键,释放得到去唾液酸的糖肽。Nilsson等[80]通过低温度和低氧化剂浓度选择性地将唾液酸的甘油侧链氧化,肼固相载体富集加上酸水解糖苷键,富集唾液酸化的糖肽/糖蛋白获得成功。
3.3.2O-GlcNAc修饰蛋白质的富集
O-GlcNAc修饰的蛋白质是一类特定的单糖糖基化蛋白质,由O-GlcNAc转移酶和O-GlcNAc糖苷酶调控。Klement等[81]将SPEG方法拓展到O-GlcNAc修饰的蛋白质的研究中,不同的是肼腙法富集后使用羟胺水解切断腙键的方法释放糖肽。在果蝇蛋白酶体复合物的研究中,通过富集鉴定到4个O-GlcNAc蛋白质并能指认O-GlcNAc修饰位点。方法分为3步:(1)高碘酸钠氧化O-GlcNAc蛋白糖环上的反式二醇(氨基葡萄糖3,4位),氧化开环生成醛基;(2)酰肼树脂捕获O-糖蛋白,结合胰蛋白酶酶解去除非糖肽;(3)在羟胺的条件下发生腙键断裂,释放出利于质谱分析的开环糖肟衍生物。该法主要的不足在于O-GlcNAc活性低,需要使用剧烈的衍生化条件,会导致侧链副反应发生,比如位于N端的Ser/Thr残基的氧化。同时,带有O-GlcNAc肽段释放的特异性和释放效率均不能满足大规模O-GlcNAc蛋白的鉴定要求,但仍然是现有方法的很好补充。
3.3.3 N端N-糖基化肽的富集
N-糖基化发生在肽段N端的糖肽(PGANs)在所有胰蛋白酶酶解的N-糖肽中仅占约10%,常规的方法很难鉴定到这个位点的糖基化。张玉奎课题组[82]发展了新的方法,通过琥珀酰化选择性标记肽链N端,以此辅助去糖基化酶发挥作用,从而实现PGANs的检测。该课题组[83-85]还在糖肽富集的工作中做了很多整体柱,充分发挥了化学反应在糖蛋白研究中的特长。
3.3.4 富集方法的联合使用
为了获得更加丰富的糖蛋白信息,将不同的富集技术联合使用,这在糖蛋白质组表达谱的深度挖掘中发挥着明显的作用。肼化学法与传统的凝集素富集结合,从小鼠组织中鉴定到2 352个糖蛋白和6 367个N-糖基化位点[86]。肼化学法与亲水法(HILIC)结合在人肝组织中鉴定到14 480个N-糖肽,对应2 210个N-糖基化蛋白质,鉴定到4 783个N-糖基化位点[87]。
4 展望
目前,虽然估计有超过50%的蛋白质发生了糖基化,但现有的数据库条目中只有约10%注释为糖蛋白,这也反映了蛋白质糖基化研究的难度和意义。目前尚没有一种技术可以富集细胞中所有的糖蛋白,已有的富集方法如凝集素亲和层析法、共价连接固相捕获法等已得到了很好的应用,但要实现更全面、更低丰度糖蛋白的鉴定和分析,有赖于富集新方法和新技术的不断开发以及与质谱技术的更好结合。相信随着各项分析技术的不断成熟,糖蛋白质组将会在表达蛋白质组的进一步完善、疾病蛋白质组生物标志物的发现及药物靶点的研究中发挥更大的作用。
[1] Ohtsubo K, Marth J D. Cell, 2006, 126: 855
[2] Walsh G, Jefferis R. Nat Biotechnol, 2006, 24: 1241
[3] Peracaula R, Barrab S, Sarrats A, et al. Dis Markers, 2008, 25(4/5): 207
[4] Comunale M A, Lowman M, Long R E, et al. J Proteome Res, 2006, 5(2): 308
[5] Johnson P J. J Gastroen Hepatol, 1999, 14: S32
[6] Drake R R, Schwegler E E, Malik G, et al. Mol Cell Proteomics, 2006, 5(10): 1957
[7] Zhang S Z. Glycobiology and Biological Engineering. Beijing: Tsinghua University Press, 2002: 97
张树政. 糖生物学和糖生物工程. 北京: 清华大学出版社, 2002: 97
[8] Haltiwanger R S, Lowe J B. Annu Rev Biochem, 2004, 73: 491
[9] Kolli V, Schumacher K N, Dodds E D. Bioanalysis, 2015, 7: 113
[10] Vosseller K, Trinidad J C, Chalkley R J, et al. Mol Cell Proteomics, 2006, 5: 923
[11] Qiu R, Regnier F E. Anal Chem, 2005, 77: 2802
[12] Comer F I, Vosseller K, Wells L, et al. Anal Biochem, 2001, 293: 169
[13] Ball L E, Berkaw M N, Buse M G. Mol Cell Proteomics, 2006, 5: 313
[14] Ding W, Hill J J, Kelly J. Anal Chem, 2007, 79: 8891
[15] Zhao J, Simeone D M, Heidt D, et al. J Proteome Res, 2006, 5: 1792
[16] Ongay S, Boichenko A, Govorukhina N, et al. J Sep Sci, 2012, 35: 2341
[17] Liu X C. Chinese Journal of Chromatography, 2006, 24(1): 73
刘小川. 色谱, 2006, 24(1): 73
[18] Zhang L J, Xu Y W, Yao H L, et al. Chem-Eur J, 2009, 15: 10158
[19] Wang Y L, Liu M B, Xie L Q, et al. Anal Chem, 2014, 86: 2057
[20] Ma R N, Hu J J, Cai Z W, et al. Nanoscale, 2014, 6: 3150
[21] Chen Y J, Xiong Z C, Zhang L Y, et al. Nanoscale, 2015, 7: 3100
[22] Xu Y W, Wu Z X, Zhang L J, et al. Anal Chem, 2009, 81: 503
[23] Liu L T, Zhang Y, Zhang L, et al. Anal Chim Acta, 2012, 753: 64
[24] Wang H Y, Bie Z J, Lu C C, et al. Chem Sci, 2013, 4: 4298
[25] Zhang Y T, Ma W F, Li D, et al. Small, 2014, 10: 1379
[26] Li D J, Chen Y, Liu Z. Chem Soc Rev, 2015, 44: 8097
[27] Pan X H, Chen Y, Zhao P X, et al. Angew Chem Int Ed, 2015, 54: 6173
[28] Li H Y, Wang H Y, Liu Y C, et al. Chem Commun, 2012, 48: 4115
[29] Ye J, Chen Y, Liu Z. Angew Chem Int Ed, 2014, 53: 10386
[30] Cao J, Shen C P, Wang H, et al. J Proteome Res, 2009, 8: 662
[31] Zhang H, Li X J, Martin D B, et al. Nat Biotechnol, 2003, 21: 660
[32] Zou Z Q, Ibisate M, Zhou Y, et al. Anal Chem, 2008, 80: 1228
[33] Tran T H, Park S Y, Lee H, et al. Analyst, 2012, 137: 991
[34] Zhang L J, Jiang H C, Yao J, et al. Chem Commun, 2014, 50: 1027
[35] Liu L T, Yu M, Zhang Y, et al. ACS Appl Mater Interfaces, 2014, 6: 7823
[36] Sun B Y, Ranish J A, Utleg A G, et al. Mol Cell Proteomics, 2007, 6(1): 141
[37] Wollscheid B, Bausch-Fluck D, Henderson C, et al. Nat Biotechnol, 2009, 27(4): 378
[38] Nilsson J, Ruetschi U, Halim A, et al. Nat Methods, 2009, 6: 809
[39] Klement E, Lipinszki Z, Kupihar Z, et al. J Proteome Res, 2010, 9: 2200
[40] Abdel-Magid A F, Carson K G, Harris B D, et al. J Org Chem, 1996, 61: 3849
[41] Zhang Y, Kuang M, Zhang L J, et al. Anal Chem, 2013, 85: 5535
[42] Zhang Z, Sun D G, Cong Y T, et al. J Proteome Res, 2015, 14: 3892
[43] Kaneshiro K, Fukuyama Y, Iwamoto S, et al. Anal Chem, 2011, 83: 3663
[44] Snovida S I, Chen V C, Perreault H. Anal Chem, 2006, 78: 8561
[45] Zhang Y, Yu M, Zhang C, et al. Chem Commun, 2015, 51: 5982
[46] Pauloehrl T, Delaittre G, Bruns M, et al. Angew Chem Int Ed, 2012, 51: 9181
[47] Nishimura S I, Niikura K, Kurogochi M, et al. Angew Chem Int Ed, 2005, 44: 91
[48] Kurogochi M, Amano M, Fumoto M, et al. Angew Chem Int Ed, 2007, 46: 8808
[49] Zeng Y, Ramya T N C, Dirksen A, et al. Nat Methods, 2009, 6: 207
[50] Ramya T N C, Weerapana E, Cravatt B F, et al. Glycobiology, 2013, 23: 211
[51] Zhang Y, Yu M, Zhang C, et al. Anal Chem, 2014, 86: 7920
[52] Zhang W, Cao W Q, Huang J M, et al. Analyst, 2015, 140: 1082
[53] Zhang W, Wang H, Tang H L, et al. Anal Chem, 2011, 83: 4975
[54] Zhang W, Wang H, Zhang L, et al. Talanta, 2011, 85(1): 499
[55] Zhao Y, Jia W, Wang J F. Anal Chem, 2011, 83: 8802
[56] Jia W, Lu Z, Fu Y, et al. Mol Cell Proteomics, 2009, 8(5): 913
[57] Llop E, Gallego R G, Belalcazar V, et al. Proteomics, 2007, 7(23): 4278
[58] Harazono A, Kawasaki N, Itoh S, et al. Anal Biochem, 2006, 348(2): 259
[59] Wells L, Vosseller K, Cole R N, et al. Mol Cell Proteomics, 2002, 1: 791
[60] Zheng Y F, Guo Z H, Cai Z W. Talanta, 2009, 78: 358
[61] Ashline D J, Yu Y, Lasanajak Y, et al. Mol Cell Proteomics, 2014, 13: 2961
[62] Song X Z, Ju H, Lasanajak Y, et al. Nat Methods, 2016, 13(6): 528
[63] Hawkins C L, Davies M J. Biochem J, 1998, 332: 617
[64] Ohno N, Uchiyama M, Tsuzuki A, et al. Carbohydr Res, 1999, 316: 161
[65] Rees M D, Hawkins C L, Davies M J, et al. J Am Chem Soc, 2003, 125: 13719
[66] Rees M D, Davies M J. J Am Chem Soc, 2006, 128: 3085
[67] Sparbier K, Wenzel T, Kostrzewa M. J Chromatogr B, 2006, 840: 29
[68] Sun B Y, Ranish J A, Utleg A G, et al. Mol Cell Proteomics, 2007, 6: 141
[69] Tian Y, Zhou Y, Elliott S, et al. Nat Protoc, 2007, 2: 334
[70] Yang S, Li Y, Shah P, et al. Anal Chem, 2013, 85(11): 5555
[71] Shah P, Yang S, Sun S S, et al. Anal Chem, 2013, 85(7): 3606
[72] Yin B J, Gao Y, Chung C Y, et al. Biotechnol Bioeng, 2015, 112(11): 2343
[73] Lin N, Mascarenhas J, Sealover N R, et al. Biotechnol Prog, 2015, 31(2): 334
[74] Yang S, Wang M Y, Chen L J, et al. Scientific Reports, 2015, 5: 17585
[75] Yang S, Yuan W, Yang W M, et al. Anal Chem, 2013, 85(17): 8188
[76] Zhou Y, Aebersold R, Zhang H. Anal Chem, 2007, 79(15): 5826
[77] Zou Z Q, Ibisate M, Zhou Y, et al. Anal Chem, 2008, 80(4): 1228
[78] Sun S S, Shah P, Eshghi S T, et al. Nat Biotechol, 2016, 34: 84
[79] Yang S J, Zhang H. Anal Chem, 2012, 84(5): 2232
[80] Nilsson J, Ruetschi U, Halim A, et al. Nat Methods, 2009, 6: 809
[81] Klement E, Lipinszki Z, Kupihar Z, et al. J Proteome Res, 2010, 9: 2200
[82] Weng Y J, Sui Z G, Jiang H, et al. Scientific Reports, 2015, 5: 9770
[83] Jiang H, Yuan H M, Qu Y Y, et al. Talanta, 2016, 146: 225
[84] Wu C, Liang Yu, Zhao Q, et al. Chem-Eur J, 2014, 20(28): 8737
[85] Liang Y, Wu C, Zhao Q, et al. Anal Chim Acta, 2015, 900: 83
[86] Zielinska D F, Gnad F, Wisniewski J R, et al. Cell, 2010, 141: 897
[87] Zhu J, Sun Z, Cheng K, et al. J Proteome Res, 2014, 13: 1713
Research advances of enrichment approaches based on chemical reactions in glycoproteomics
BAO Huimin, XIE Liqi, LU Haojie*
(DepartmentofChemistry,InstitutesofBiomedicalSciencesandResearchCenteronAgingandMedicine,FudanUniversity,Shanghai200433,China)
Protein glycosylation is one of the most common and important post-translational modifications (PTMs) in mammalian cells. Glycopeptides usually exist at relatively low abundance (less than 5%) compared with non-glycosylated peptides. So, enrichment approaches are important for the analysis of glycoproteome. Approaches based on chemical reactions are the main trend, here we discuss them from chemical viewpoint. There are two main processes for enrichment of glycoproteins, glycoprotein conjugation and the release from solid phase. In conjugation process, we introduce four types of chemical reactions: one is boronic acid chemistry based on reaction between boronic acid andcis-diol groups on free glycans, and the other three are covalent reactions between aldehyde groups and functionalized groups, hydrazide for hydrazone ligation, alkylamine for reductive amination, aniline for nonreductive amination and aminooxy group for oxime click chemistry. In release process,N-glycosidase (PNGase) F forN-glycosylated proteins,β-elimination reaction forO-glycosylated proteins and a bleach method published recently are introduced, and the oxidative releasing of natural glycans is also introduced here. In addition, analysis strategies forN-/O-glycoproteins are discussed, including detection in glycoproteins and site-specific glycan occupancies. This review mainly focuses on the currently available chemical approaches based on covalent reactions for enrichment of glycoproteins, as well as new materials derivatized with different functional groups. We compare the advantages and disadvantages of different methods and try to evaluate the contribution and future trend based on authors viewpoint.
glycoproteome; protein glycosylation; glycoprotein; enrichment; conjugation/release; covalent reaction; review
10.3724/SP.J.1123.2016.09003
2016-09-01
国家重点研发项目(2016YFA0501303);国家自然科学基金(20905014,21335002); 上海市自然科学基金(14ZR1403600);上海市老年医学临床重点实验室建设项目(13dz2260700).
Foundation item: National Key Research and Development Program (No. 2016YFA0501303); National Natural Science Foundation of China (Nos. 20905014, 21335002); Shanghai Natural Science Foundation (No. 14ZR1403600); Shanghai Key Laboratory of Clinical Geriatric Medicine Construction Project (No. 13dz2260700).
O658
A
1000-8713(2016)12-1145-09
邹汉法研究员纪念专辑(上)·专论与综述
* 通讯联系人.Tel:(021)54237618,E-mail:luhaojie@fudan.edu.cn.
