海洋Pseudomonasaeruginosa sp. DES-3的鉴定以及基因ap02对其产蛋白酶的影响研究*
2016-12-09陈俊海肖桂龙蒋承建
刘 瑶,陈俊海,肖桂龙,孟 灿,曾 蓉,武 波,蒋承建,3*
>(1.亚热带农业生物资源保护与利用国家重点实验室,广西 南宁 530004;2.广西大学 生命科学与技术学院,广西 南宁 530004;3.国家海洋局 海洋生物活性物质与现代分析技术重点实验室,山东 青岛 266061)
海洋Pseudomonasaeruginosasp. DES-3的鉴定以及基因ap02对其产蛋白酶的影响研究*
刘 瑶1,2,陈俊海1,2,肖桂龙1,2,孟 灿1,2,曾 蓉1,2,武 波1,2,蒋承建1,2,3*
>(1.亚热带农业生物资源保护与利用国家重点实验室,广西 南宁 530004;2.广西大学 生命科学与技术学院,广西 南宁 530004;3.国家海洋局 海洋生物活性物质与现代分析技术重点实验室,山东 青岛 266061)
海洋是地球上最大的生态系统,其多样独特的生态环境造就了其有别于其他环境的微生物资源。本研究利用蛋白酶的活性筛选策略,从辽宁省营口市某海洋淤泥中分离得到一株产蛋白酶的优势菌株DES-3。通过微生物形态学特征、生理生化性质和16S rDNA基因鉴定以及系统发育进化关系的分析,将该菌株鉴定为铜绿假单胞菌属,命名为Pseudomonasaeruginosasp. DES-3。其胞外蛋白酶的最适反应温度和最适pH分别为55 ℃和9.0。将来自于碱性污染土壤宏基因组文库的编码碱性蛋白酶基因ap02与广宿主表达载体pBBR1MCS-5连接,转化至野生型菌株P.aeruginosasp. DES-3细胞中,成功构建了基因工程菌株P.aeruginosasp. DES-3/pBBR1MCS-5-ap02。基因工程菌株的最适反应温度为60 ℃,较野生型菌株提高了5 ℃。本研究构建基因工程菌株的策略可为菌株的改造和相应工业应用提供一定的技术参考。
海洋微生物;碱性蛋白酶;Pseudomonasaeruginosasp.;基因工程菌株
海洋是地球上最大的生态系统,体积在整个生物圈超过90%,面积占地球表面积的71%,其包含的微生物占全球初级生产者的50%左右[1]。从2000年开始,科学家进行了海洋生物普查(Census of Marine Life, CoML),旨在评估和解释海洋生物多样性、分布和丰富程度的国际性研究。报告指出,海洋微生物包括细菌域、古菌域、真核生物和病毒在内的多个类群,初步估计有2亿~10亿个物种[2]。
碱性蛋白酶广泛存在于细菌、霉菌和放线菌中,可用于洗涤、纺织、食品、医药、环保和生命科学等领域,其中洗涤剂行业的应用最为广泛。从海洋环境中筛选高产蛋白酶菌株已成为国内外研究热点。1960年,Dane首次从地衣芽孢杆菌Bacilluslicheniformis中分离得到碱性蛋白酶,直至现在也仍是产碱性蛋白酶最合适的微生物之一[3]。1972年,Kato等[4]从海洋嗜冷杆菌Psychrobacter中分离到一个新型碱性蛋白酶,至此大量碱性蛋白酶被陆续从海洋微生物中获得。1995年,Ogino等[5]筛选到产碱性蛋白酶且耐有机溶剂的铜绿假单胞菌PseudomonasaeruginosaLST-03。1998年,作为清洁剂添加剂的碱性蛋白酶是从与海洋中的船蛆共生的细菌中分离获得的[6]。2009年,Haddar等[7]从海水中得到菌株BacillusmojavensisA21,纯化的两种碱性蛋白酶BM1和BM2鉴定为丝氨酸蛋白酶类,在非离子表面活性剂中有很好的稳定性,与商业的液体或固体清洁剂能很好的相容。2014年,Anjali等[8]从印度海岸分离出36株产蛋白酶的细菌,其中编号P15的菌株鉴定为新种Bacillustequilensis,其产生的碱性蛋白酶在分配系数从-0.76~8.8的有机溶剂中均有很好的耐受性,而且能有效去除面料上的血迹、动物皮毛和摄影胶片中的明胶。
本研究对辽宁省营口市某海洋淤泥进行产蛋白酶的菌株分离筛选,通过微生物形态学特征、生理生化性质和16S rDNA基因鉴定以及系统发育进化关系等分析,完成了菌株的分子生物学鉴定。将来自于宏基因组文库的碱性蛋白酶基因ap02与广宿主表达载体连接,转化至野生型菌株P.aeruginosasp. DES-3中,成功构建了基因工程菌株P.aeruginosasp. DES-3/pBBR1MCS-5-ap02。本研究可为野生型的菌株改造和工业应用提供技术参考。
1 材料和方法
1.1 菌株的来源
表达宿主E.coliBL21(DE3)pLysS和表达载体pET-32a(+)、pMP2444、pBBR1MCS-5均为广西大学生命科学与技术学院资源与环境微生物课题组保存。
1.2 环境样品
本课题实验采用的环境样品辽宁营口市某海洋淤泥(122°09′30″E,40°21′39″N),常温采集,环境pH为弱碱性。
1.3 培养基
菌株活化和发酵采用LB培养基[9];产蛋白酶的菌株筛选采用LA固体培养基添加2%的脱脂牛奶。
1.4 产蛋白酶菌株的筛选
称取适量的环境样品制备悬浮液,37 ℃振荡培养24 h。梯度稀释后,根据蛋白酶的活性筛选策略,筛选优势菌株。
1.5 碱性蛋白酶活力的测定
利用蛋白酶制剂[10]中的福林酚法测定蛋白酶活力,参照文献[11]进行。
1.6 扫描电镜
选取固体培养基上的单菌落,经菌体收集、固定和临界点干燥以及离子溅射金等一系列预处理,然后在扫描电子显微镜下镜检。具体操作参照文献[12]进行。
1.7 分子鉴定和系统发育分析
16S rDNA基因鉴定中的DNA提取和PCR扩增参照文献[13]进行。将PCR产物送至华大基因有限公司测序,对测序后的16S rDNA序列在GenBank数据库进行序列比对,用软件MEGA 6.0分析系统进化关系。
1.8 基因ap02对菌株DES-3的产酶特征影响的测定
编码碱性蛋白酶的基因ap02来自于碱性污染土壤宏基因组文库[14]。将基因ap02与表达载体pMP2444、pBBR1MCS-5用BamH Ι和EcoR Ι双酶切,纯化后用T4连接酶在16 ℃过夜连接,然后用电转化方法转化至以0.3 mol/L蔗糖溶液为电转化介质的野生菌株DES-3感受态细胞中,构建基因工程菌株。将工程菌和野生菌在30 ℃发酵30 h,将发酵液稀释一定倍数,分别在不同温度(50,55,60,65以及70 ℃)和pH=9.0的条件下,测定蛋白酶活。
2 结果与分析
2.1 产蛋白酶菌株的筛选结果
基于蛋白酶菌株的活性筛选策略,经分离纯化和活性检测,得到6株产蛋白酶的优势菌株,对6株菌株进行产水解圈时间的统计,并进行了粗酶活力的测定,平行实验3次结果如表1所示。分析数据显示菌株C1在培养3 h后就产生了清晰的水解圈,相同条件下,发酵液酶活力最高。选择菌株C1作为目标菌株,命名为DES-3。

表1 6株活性菌株的D/d值和发酵液酶活Table 1 The D/d value and protease activity of the six primary strains
2.2 菌株DES-3的生理生化特征
挑取单菌落进行镜检,革兰氏染色呈阴性。扫描电镜下(图1),菌株DES-3为细长杆菌,长短不一,成对或成短链状排列,菌株一端有鞭毛,运动,无芽孢,无荚膜。在肉汤培养基上培养20 h产生粉橙色的半透明圆形菌落。V-P实验和甲基红实验均为阴性,能产生脓青素,不能利用柠檬酸盐。
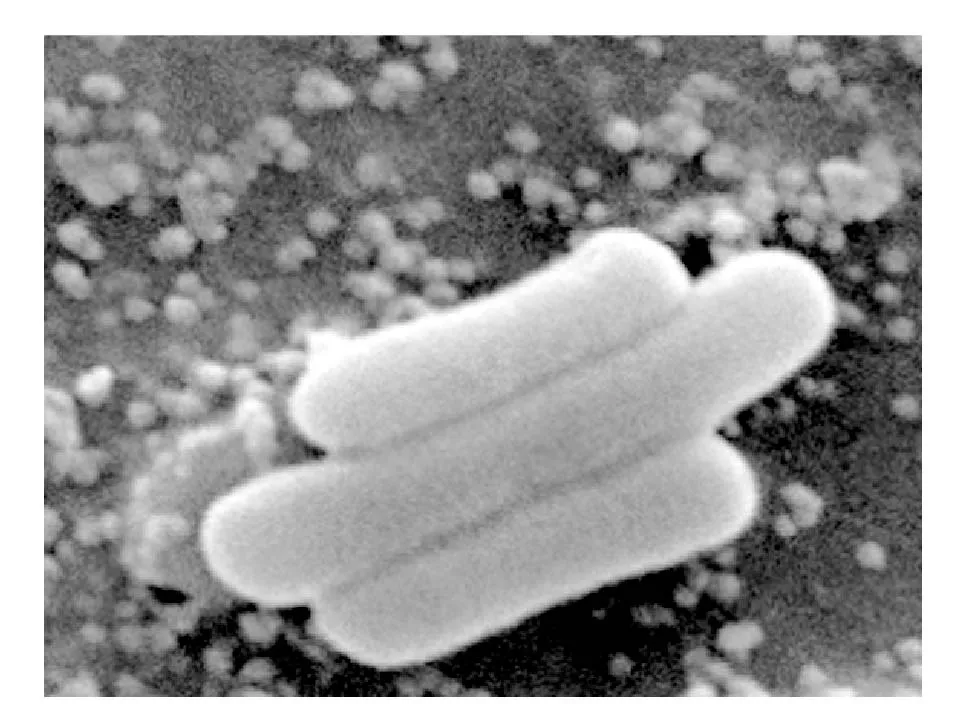
图1 菌株DES-3的扫描电镜图Fig.1 The morphology of DES-3 in the scanning electron microscope
2.3 活性菌株的系统发育分析
利用细菌通用引物27F/1492R对菌株DES-3的16S rDNA基因进行扩增,测序获得的16S rDNA基因序列约1.5 kb,在GenBank数据库进行序列比对,与菌株PseudomonasaeruginosaPAO1(NR_074 828.1)的一致性较高,为99%,用软件MEGA 6.0以邻位相接算法构建系统进化树(图2)。系统进化关系分析显示,菌株DES-3与铜绿假单胞菌Pseudomonasaeruginosa的亲缘关系最接近,其进化关系趋于一致,初步将菌株DES-3鉴定为铜绿假单胞菌属,菌株被命名为P.aeruginosasp. DES-3。
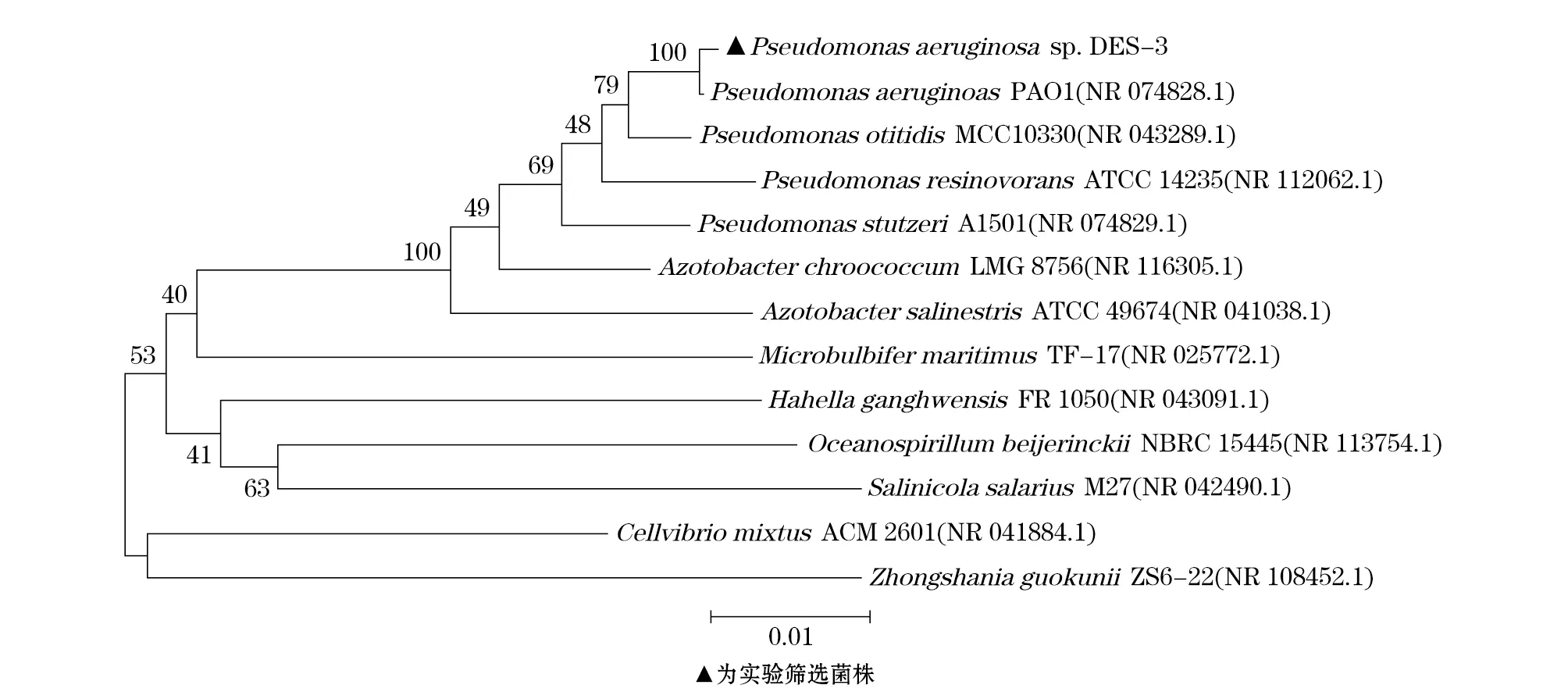
图2 菌株P. aeruginosa sp. DES-3的系统进化关系的分析Fig.2 The phylogenetic analysis of P. aeruginosa sp. DES-3
2.4 菌株DES-3蛋白酶活力测定
2.4.1 最适温度的测定
以1%的比例将菌株DES-3转接到LB培养基中,30 ℃,200 r/min摇床培养30 h,离心取上清,稀释一定倍数后测定蛋白酶活力。实验中以pH 7.5的磷酸缓冲液作为反应缓冲液,设定梯度温度,测定不同温度下的酶活,将测得的最高酶活力设为100%,计算其他温度下的相对酶活。由图3可知,菌株DES-3所产蛋白酶的最适反应温度为55 ℃。

图3 P. aeruginosa sp. DES-3 蛋白酶的最适温度Fig.3 The optimal temperature of protease from P. aeruginosa sp. DES-3
2.4.2 最适pH的测定
取处理好的发酵上清液,设定反应温度为55 ℃,在不同pH值的缓冲液体系中测定酶活力。由图4可知,该蛋白酶的最适反应为pH=9.0,在pH 7.0~10.0的范围内,相对酶活均在80%以上,推测该蛋白酶为碱性蛋白酶。

图4 P. aeruginosa sp. DES-3蛋白酶的最适pHFig.4 The optimal pH of protease from P. aeruginosa sp. DES-3
2.5 碱性蛋白酶基因ap02的分析
碱性蛋白酶基因ap02来自于课题组前期所构建的碱性污染宏基因组文库[14]。该基因全长1 146 bp,编码381个氨基酸,蛋白分子量为39.46 kDa,等电点为8.91。Blast结果显示,在氨基酸水平上与Salinibacillusaidingensis(登录号为WP_044 156 525.1)的peptidase序列一致性为100%。对其功能结构域进行分析,推测该蛋白ap02可能属于肽酶S8超家族或SB宗,为丝氨酸蛋白酶类。对ap02蛋白的蛋白酶活性检测发现,该酶的最适反应温度为65 ℃,最适作用pH=9.0。
2.6 基因ap02对菌株DES-3产酶活性的影响
将基因ap02与广宿主表达载体pMP2444和pBBR1MCS-5连接,转化至原始菌株P.aeruginosasp. DES-3电转感受态细胞中,构建基因工程菌株。实验中在最适反应pH=9.0的条件下,分别将野生菌与工程菌的发酵液稀释相同倍数,在不同温度50,55,60,65和70 ℃条件下,进行酶活力检测,以测得的最高酶活力为100%。
由图5可知,野生型菌株DES-3的最适反应温度为55 ℃,工程菌株P.aeruginosasp. DES-3/pMP2444-ap02的最适反应温度为55 ℃,工程菌株P.aeruginosasp. DES-3/pBBR1MCS-5-ap02的最适温度增加至60 ℃,较野生型菌株提高了5 ℃。
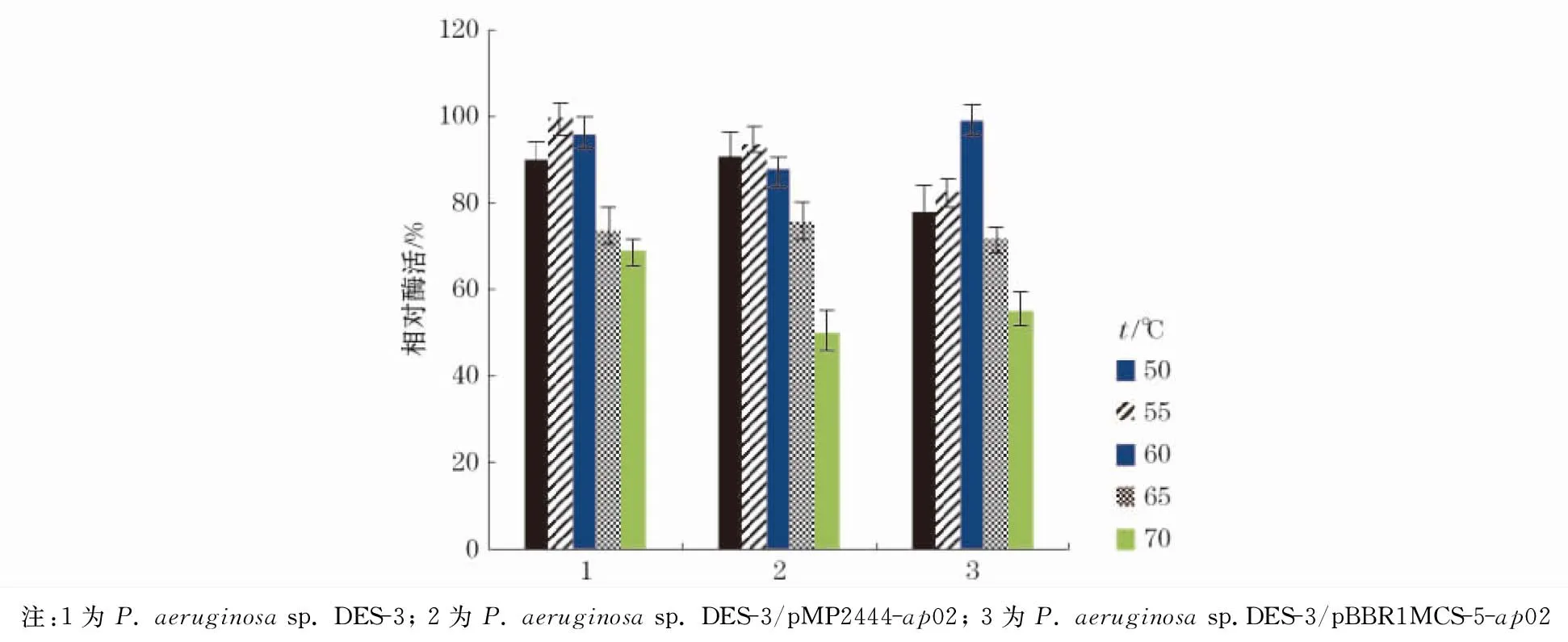
图5 温度对原始菌株和基因工程菌株酶活力的影响Fig.5 Effect of temperature on the enzyme activity of the original and genetically engineered strains
3 结 论
蛋白酶的经济价值较高,但蛋白酶的最适反应条件(温度和pH等)与实际应用条件存在差别,导致酶的催化效率降低。本研究尝试从辽宁海洋淤泥中筛选得到一株高产蛋白酶菌株,经形态学特征和分子生物学鉴定属于铜绿假单胞菌属,暂命名为P.aeruginosasp. DES-3,测定其产生的蛋白酶最适反应温度为55 ℃,最适反应pH=9.0。
目前,蛋白酶的研究已达到分子研究水平,利用基因工程手段改造蛋白酶产生菌,筛选高产酶活和具有极端酶学性质的菌株是未来的发展趋势[15]。Tao课题组利用反向PCR扩增得到能高效结合重金属离子的PCs基因,将其转化至Pseudomonasputida感受态细胞中,重组菌KT2440-SpPCS表现出对金属离子Cd和Ag以及Hg具有较好的耐受性[16]。邱吉国将nox基因与广宿主表达载体连接,导入到PseudomonasputidaKT2444中,重组菌获得了尼古丁降解活性[17]。受此启发,将ap02基因与广宿主表达载体连接,转化至P.aeruginosasp. DES-3感受态细胞中,将原始菌与工程菌在不同温度下反应。结果表明,重组菌株P.aeruginosasp. DES-3/pBBR1MCS-5-ap02的最适反应温度为60 ℃,较野生型菌株提高了5 ℃。
本研究探索了海洋淤泥中的产蛋白酶微生物资源,尝试将ap02基因导入到野生型菌株中,成功构建了两株基因工程菌株,工程菌P.aeruginosasp. DES-3/pBBR1MCS-5-ap02较野生型菌株的最适温度提高了5 ℃。本研究为蛋白酶基因工程菌株的改造和工业应用提供了数据参考和技术支持。
致谢:感谢广西大学生命科学与技术学院提供完善的技术平台,感谢蒋承建教授对本文实验的悉心指导和技术支持,感谢资源与环境微生物课题组全体成员的帮助与合作。
[1] LAURO F, MCDOUGALD D, THOMAS T, et al. The genomic basis of trophic strategy in marine bacteria[J].Proceedings of the National Academy of Sciences of USA, 2009, 106(37): 15527-15533.
[2] JACK A G, CHRISTOPHER L D. Microbial metagenomics: beyond the genome[J].Annual Review of Marine Science, 2011, 3: 347-371.
[3] CHEN Z, KIM S K. Research and application of marine microbial enzymes: status and prospects[J].Marine Drugs, 2010, 8(6): 1920-1934.
[4] ZHANG L X, AN R, WANG J P, et al. Exploring novel bioactive compounds from marine microbes[J].Current Opinion in Microbiology, 2005, 8(3): 276-281.
[5] OGINO H, MIYAMOTO K, ISHIKAWA H. Organic-solvent-tolerant bacterium, which secretes organic-solvent-stable lipolytic enzyme[J].Applied and Environmental Microbiology, 1995, 60(10): 3884-3886.
[6] GREENE R V, GRIFIN H L, COTTAL M A. Utility of alkaline protease from marine shipworm bacterium in industrial cleansing applications[J].Biotechnology Letters, 1996, 18(7): 759-764.
[7] HADDAR A, AGREBI R, BOUGATEF A, et al. Two detergent stable alkaline serine-proteases from Bacillus mojavensis A21: purification, characterization and potential application as a laundry detergent additive[J].Bioresource Technology, 2009, 100(13): 3366-3373.
[8] ANJALI B, VISHAL C, HARESH K, et al. Production and characterization of a solvent-tolerant protease from a novel marine isolateBacillustequilensisP15[J].Annals of Microbiology, 2014, 64(1): 343-354.
[9] SAMBROOK J, FRITSCH E F, MANIATIS T. Molecular cloning: A laboratory manual (2nd)[M].New York: Cold Spring Harbor Laboratory Press, 1989.
[10] Protease preparations:GB/T 723527-2009[S].Beijing: Standards Press of China.蛋白酶制剂法:GB/T 723527-2009[S].北京:中国标准出版社,2009.
[11] ZHANG S Z. Enzyme industry[M].Beijing: Science Press,1984. 张树政. 酶制剂工业[M].北京: 科学出版社, 1984.
[12] XIE J Y, DONG G J, LIU Z Y. Microbial sample preparation method of the scanning electron microscopy[J].Journal of Chinses Electron Microscopy Society, 2005, 24(4): 440. 谢家仪, 董光军, 刘振英. 扫描电镜的微生物样品制备方法[J].电子显微学报, 2005, 24(4): 440.
[13] WEISBERG W G, BARNS S M, PELLETIER D A, et al. 16S ribosomal DNA amplification for phylogenetic study[J].Journal of Bacteriology, 1991, 173(2): 697-703.
[14] JIANG C J, LI S X, LUO F F, et al. Biochemical characterization of two novel β-glucosidase genes by metagenome expression cloning[J].Bioresource Technology, 2011, 102(3): 3272-3278.
[15] DENG J Y. Research progress in microbial alkaline protease[J].Modern Food Science and Technology, 2008, 24(3): 293-296. 邓菊云. 微生物碱性蛋白酶研究进展[J].现代食品科技, 2008, 24(3): 293-296.
[16] YONG X, CHEN Y, LIU W, et al. Enhanced cadmium resistance and accumulation in Pseudomonas putida KT2440 expressing the phytochelatin synthase gene of Schizosaccharomyces pombe[J].Letters in Applied Microbiology, 2013, 58(3): 255-261.
[17] QIU J G. The molecular catabolic mechanism of nicotine byPseudomonassp.HZN6[D].Hangzhou: Zhejiang University, 2014. 邱吉国. 假单胞菌降解尼古丁的分子机制研究[D].杭州: 浙江大学, 2014.
Received: December 4, 2015
Identification of aPseudomonasaeruginosasp. DES-3 From Marine and the Effect of Protease-producing by a Gene ofap02
LIU Yao1,2, CHEN Jun-hai1,2, XIAO Gui-long1,2, MENG Can1,2, ZENG Rong1,2, WU Bo1,2, JIANG Cheng-jian1,2,3
(1.StateKeyLaboratoryforConservationandUtilizationofSubtropicalAgro-bioresources, Nanning 530004, China; 2.CollegeofLifeScienceandTechnology,GuangxiUniversity, Nanning 530004, China; 3.TheKeyLabofMarineBioactiveSubstanceandModernAnalyticalTechnique,SOA, Qingdao 266061, China)
Ocean is the largest ecosystem on the earth, and the microbial resources in the marine environment are significantly different from the others natural eco-systems. In this study, a protease-producing strain DES-3 was obtained by using functional screening strategy from the marine sludge in Yingkou city, Liaoning Province of China. The DES-3 strain was identified as the genus ofPseudomonasaeruginosaand named asPseudomonasaeruginosasp. DES-3 based on the results of microbial morphological characterization, physiological and biochemical properties, and 16S rDNA gene identification and phylogenetic tree analysis. The optimal temperature and pH of the purified protease were 55 ℃ and 9.0, respectively. Furthermore, anap02 gene encoding alkaline protease identified from an alkaline polluted soil metagenomics library, was ligated into a widely host expression vector of pBBR1MCS-5 and transferred into the competition cells ofPseudomonasaeruginosasp. DES-3. Finally, a genetically engineered strain of theP.aeruginosasp. DES-3/pBBR1MCS-5-ap02 was constructed. The enzymatic data indicated that the optimal temperature and pH of theP.aeruginosasp. DES-3/pBBR1MCS-5-ap02 were 60 ℃and 9.0, respectively, the final optimal temperature increased 5 ℃ than that of the originalP.aeruginosasp. DES-3. This study showed a technical reference for the genetic modification of the wild type strain and also provided the potential industrial application.
marine microorganisms; alkaline protease;Pseudomonasaeruginosasp.; genetically engineered strain
2015-12-04
广西自然科学基金项目——一个新的胰蛋白酶基因的分子结构特征和生物学功能研究(2015GXNSFAA139050);国家海洋局海洋生物活性物质与现代分析技术重点实验室项目——海洋胰蛋白酶新基因的分子结构特征和功能研究(MBSMAT-2015-02)
刘 瑶(1990-),女,湖北天门人, 硕士,主要从事环境资源与微生物方面研究.E-mail:lyao@6677@126.com
*通讯作者:蒋承建(1980-),男,广西桂林人,教授,博士,博士生导师,主要从事新酶的结构和功能方面研究.E-mail:jiangcj0520@gmail.com
Q344+.3
A
1671-6647(2016)04-0553-07
10.3969/j.issn.1671-6647.2016.04.011
