鸱鸮科27种鸟类COⅠ序列变异及系统发育分析
2016-11-18康慧丁鹏李波
康慧丁鹏李波
(1.东北林业大学,哈尔滨,150040;2.国家林业局野生动植物检测中心,哈尔滨,150040)
鸱鸮科27种鸟类COⅠ序列变异及系统发育分析
康 慧丁 鹏李 波1,2*
(1.东北林业大学,哈尔滨,150040;2.国家林业局野生动植物检测中心,哈尔滨,150040)
鸱鸮科;
(1.Northeast Forestry University,Harbin,150040,China;2.State Forestry Administration Dectecting Center of Wildlife,Harbin,150040,China)
鸱鸮科(Strigidae)鸟类是一类适应夜行性生活的猛禽,隶属于鸟纲鸮形目(Strigiformes)。该科鸟类多栖息于森林中,主要捕食鼠类,处于食物链的顶层,对控制鼠害和维护生态平衡有着不可替代的作用[1-2]。目前鸱鸮科鸟类的种属分类仍存在一些争议[3]。例如,渔鸮属(Ketupa)和雪鸮属(Nyctea)是否应并入雕鸮属(Bubo)。渔鸮属鸟类为大型鸮类,形态上与雕鸮相似,有学者将该属并入雕鸮属[4]。但郑作新等基于渔鸮属鸟类专一化的食性,以及跗跖部分甚至全部裸出,跖底具刺突等形态特征,认为它应为独立于雕鸮属外另一个属[5]。但新近基于细胞色素B(Cytb)基因的系统发育研究则支持将它作为一个亚属置于雕鸮属内[6-7]。类似的情况也出现在雪鸮属。传统分类学认为它是一单型属,下辖一单型种雪鸮(Nycteascandiaca)[5,8]。而基于线粒体和核DNA(Cytb和RAG-1基因)的系统发育研究也支持将它并入雕鸮属[9]。
四川林鸮(Strixdavidi)是我国唯一特有的鸮类,分布区狭窄,包括青海省的班玛,四川省的宝兴、松潘和巴塘,甘肃省的莲花山[10-11]。四川林鸮形态上与长尾林鸮(Strixuralensis)相似,曾长期被认为是它的一个亚种(Strixuralensisdavidi)。20 世纪 90 年代后,学者们认为青藏高原的抬升使得四川林鸮与长尾林鸮实现了完全的地理隔离,建议将四川林鸮列为一个独立种[6-7,9,12-16]。国际鸟类联盟(Bird Life International)还将四川林鸮列为易危种(vulnerable)。目前对于该物种的分类学的研究还甚少,仅见零星报道[11,16-19]。缺乏利用分子标记将其与长尾林鸮及林鸮属其他鸟类进行综合比较的研究。
DNA 条形码是用于物种鉴别和进化研究的线粒体 DNA部分序列,目前通用的是细胞色素C氧化酶亚基I(cytochrome oxidase subunit I,CO I)基因648 bp片段[20-21]。CO I基因具有在保证物种间足够变异的同时,种内变异又相对保守,且很容易被通用引物扩增等特点。已经广泛应用于有争议物种分类地位的重新认定、发现新种和隐存种、重建物种和高级阶元的演化关系、种群遗传分析等领域[22]。目前也被广泛应用在许多鸟类,包括鸮形目鸟类的分子系统学研究中[19,23-30]。
通过测定中国产的红角鸮(Otussunia)、雕鸮(Bubobubo)和长尾林鸮的CO I基因部分序列,结合 GenBank 中查询整理的四川林鸮以及鸱鸮科其他鸟类的CO I基因序列,研究鸱鸮科10个属27种鸟类CO I基因的变异及系统发育情况,为鸱鸮科鸟类的系统发育和分类提供一定的理论依据。
1 材料与方法
1.1 材料
本研究所选19只中国产的鸱鸮科鸟类(红角鸮4只、雕鸮2只、长尾林鸮13只)肌肉样本均来自国家林业局野生动植物检测中心。同时从 GenBank 中下载鸱鸮科其他鸟类的CO I基因同源序列,去除同一产地相同序列,得到47条有效序列(表1)。为给系统发育分析提供外群,选择中国产的金雕(Aquilachrysaetos)、松雀鹰(Accipitervirgatus)、灰背隼(Falcocolumbarius)肌肉样本检测CO I基因部分序列。
1.2 PCR扩增与测序
肌肉样本经过初步处理后利用蛋白酶K消化,后用基因组DNA提取试剂盒(AxyPrep Biosciences,杭州)提取纯化总DNA。PCR引物选择BirdF1/BirdR1[28]扩增CO I基因部分序列。PCR反应在50 μL体系中进行,包括10×PCR Buffer(Mg2+plus)、dNTP Mixture 2.5 mM、r Taq酶5 U(Takara,大连)、上下游引物各200 mmol/L和DNA模板50~100 ng。扩增采用PE-9700型和2400型DNA扩增仪(PE,美国),反应程序为94℃/60 s,(94℃/60 s,47℃/45 s,72℃/1.5 min)×30 cycle,72℃/5 min。扩增产物置于1.5%的琼脂糖凝胶上100 V稳压电泳45 min。紫外灯下切下目的条带,用AxyPrep DNA凝胶回收试剂盒纯化回收后,将样品送至哈尔滨博仕生物有限公司,采用原扩增引物进行双向测序。
1.3 数据分析
测序完成后,所得的每条序列利用DNAstar 软件包中的 SeqMen 软件进行校对,结合测序峰图,舍弃两端低信号序列,对序列内部位点进行人工校对。将所得序列用软件Clustal W进行分析比对,确定变异位点及单倍型。利用MEGA 5.02基于Gamma分布的K2P模型(Kimura 2 parameter)计算序列间遗传距离(自引导的重复数为1 000)[31],统计鸱鸮科鸟类种内变异最大值、种间变异最小值。
利用Mrmodeltest 2.3[32]依据AIC(Akaike Information Criterion)标准选择最佳的核苷酸替换模型GTR+I+G,用MrBayes 3.1.2[33]基于该模型构建系统发育树(BI)。主要参数包括:将Mcmc分析运行2 000 000代,记录每100代生成的树,舍弃最先生成的25%的树,并用剩余的树根据50%多数原则统计各分支后验概率(posterior probabilities,PP)。为验证系统发育树的拓扑结构,利用PAUP 4.0b10[34]构建MP(Maximum Parsimony)树,且系统树各分支的自举检验值(bootstrap)由1 000次重复检验获得。2种方法的系统发育分析都以金雕、松雀鹰、灰背隼CO I基因同源序列作为外群。
2 结果
2.1 鸱鸮科鸟类种内和种间变异
实验测得19条长663 bp鸱鸮科鸟类(红角鸮、雕鸮、长尾林鸮)CO I基因部分序列,经分析为10个单倍型(表1),以及3个外群的同源序列。结合GenBank中的鸱鸮科鸟类CO I基因序列,由于部分序列长度存在差异,取最大公约数后得到620 bp,数据处理最后得到鸱鸮科10个属27种鸟类40个单倍型。经计算,鸱鸮科鸟类种内变异最大值和种间变异最小值如柱状图所示(图1)。鸱鸮科鸟类种内变异最大的是乌林鸮(Strixnebulosa)(2.47%),显著小于林鸮属种间变异的最小值(不将四川林鸮视为独立种时,灰林鸮(Strixaluco)与长尾林鸮间8.50%)。其中,例外的是四川林鸮,它与长尾林鸮的种间变异为1.10%~2.10%,其最小值小于长尾林鸮的种内变异最大值(1.18%,如将四川林鸮视为长尾林鸮亚种,该值为1.28%)。雕鸮种内变异最大值(0.79%)也显著小于雕鸮属种间变异的最小值(雪鸮与美洲雕鸮间7.99%)。角鸮属种间变异最小的是日本领角鸮(Otussemitorques)与领角鸮(Otuslettia)间(0.80%),虽显著大于红角鸮种内变异的最大值(0.22%),但小于林鸮属和耳鸮属内部分种内变异的最大值。
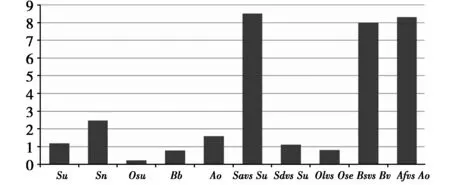
图1 鸱鸮科鸟类种内变异最大值和种间变异最小值Fig.1 Max.intraspecific variations and Min.interspecific variations in StrigidaeSa:灰林鸮Strix aluco;Su:长尾林鸮Strix uralensis;Sn:乌林鸮Strix nebulosa;Sd:四川林鸮Strix davidi;Osu:红角鸮Otus sunia;Ose:日本领角鸮Otus semitorques;Ol:领角鸮Otus lettia;Bb:雕鸮Bubo bubo;Bs:雪鸮Bubo scandiaca;Bv:美洲雕鸮Bubo virginianus;Af:短耳鸮Asio flammeus;Ao:长耳鸮Asio otus
2.2 鸱鸮科鸟类系统发育树
运用2种方法构建鸱鸮科27种鸟类系统发育树,不同方法构建的系统发育树除置信度不同外,拓扑结构大致相同(图2)。鸱鸮科27种鸟类各自的CO I基因单倍型都聚在一起。除美洲角鸮(Otusflammeolus)外(它与鹰鸮Ninoxscutulata聚为1支,BI树置信度为91%,MP树不存在该分支,遗传距离分析也不支持该分支的存在),26种鸟类的CO I基因单倍型都是同属的优先聚类。在林鸮属中,四川林鸮与欧亚大陆分布(包括来自俄罗斯、挪威、瑞典、中国吉林)的长尾林鸮先聚为一支(BI树和MP树置信度分别为100%和93%),然后是日本分布的长尾林鸮(BI树和MP树置信度均为100%)。与长尾林鸮亲缘关系最近的是灰林鸮(BI树和MP树置信度分别为100%和84%),然后是乌林鸮与褐林鸮(Strixleptogrammica)组成的小支(BI树置信度为93%),最后是横斑林鸮(Strixvaria)和斑林鸮(Strixoccidentalis)组成的小支(BI树置信度为96%)。在雕鸮属4种鸟类中,最先分化出来的是毛腿渔鸮(Ketupablakistoni)(BI树和MP树置信度分别为97%和72%),然后是雕鸮(BI树和MP树置信度分别为99%和55%),最后是亲缘关系较近的雪鸮与美洲雕鸮(Bubovirginianus)(BI树和MP树置信度分别为49%和55%)。
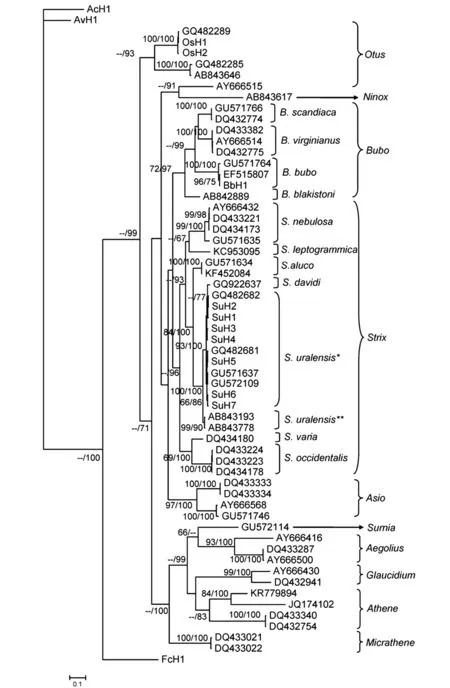
图2 基于CO I基因单倍型构建的鸱鸮科鸟类系统发育树Fig.2 Bayesian phylogenetic tree of CO I gene sequences in Strigidae分支上的数字分别表示MP树的自举值和BI树的后验概率(低于60%的数值没有显示),*表示来自欧亚大陆的长尾林鸮、**表示来自日本的长尾林鸮,金雕(Ac H1)、松雀鹰(Av H1)、灰背隼(Fc H1)CO I基因同源序列作为外群
3 讨论
通过对超过640种鸟类的研究,Hebert等提出鸟类DNA条形码的阈值为 2.7%,即种内个体间K2P遗传距离应小于 2.7%[27,30,35-36]。研究涉及的鸱鸮科27种鸟类种内个体间K2P遗传距离都符合该阈值。计算还得出日本领角鸮和领角鸮种间变异比较小,甚至小于林鸮属和耳鸮属内部分种内变异的最大值。尽管同属的红角鸮种内最大变异也相对较小,但还是不能排除因单倍型数据太少而低估种间变异的可能性。另外,根据“10×规则”,即一个群体的差异超过平均种内差异10倍[22,37],雕鸮属是符合该规则,而林鸮属、角鸮属、耳鸮属均低于该规则(分别是3.4、3.6、5.2倍)。这可能与该规则的适应性相关,且已有学者认为在鸟纲中仅用单一的标准是不可行的[24,36]。
König等[6-7]基于形态相近和利用线粒体Cytb基因重建的系统发育关系,认为应将渔鸮属作为亚属并入雕鸮属。基于CO I基因的系统发育分析表明,在雕鸮属中4种鸟类的分化关系是:(毛腿渔鸮,(雕鸮,(雪鸮,美洲雕鸮))),进一步从遗传学角度支持了上述观点。此外,雪鸮全身羽毛白色,环北极地区分布,曾被认为是单型属[5,8]。而基于线粒体和核DNA重建的鸮形目鸟类系统发育关系,认为雪鸮与美洲雕鸮关系相近,二者的分歧时间发生在距今约四百万年前,因此雪鸮属也应归于雕鸮属下的一个亚属[9]。研究基于CO I基因的系统发育分析也得出了类似的结果。
四川林鸮的分类地位存在争议:是独立种还是长尾林鸮的一个亚种。K2P遗传距离分析表明,四川林鸮与长尾林鸮的分歧小于长尾林鸮种内的最大变异,明显违背了DNA条形码关于种的认定规则[22,35,38]。系统发育分析表明,四川林鸮处于日本与欧亚大陆分布的长尾林鸮之间。或者说相对于欧亚大陆长尾林鸮种群,日本的长尾林鸮较四川林鸮分歧更大。这进一步证实,现有的研究支持将四川林鸮作为长尾林鸮的一个地理亚种。由于线粒体DNA是母系遗传,单一CO I基因的分析、缺乏核基因的联合分析,某种程度上弱化了上述分子证据。在青藏高原抬升后,四川林鸮逐步与大陆分布的长尾林鸮种群发生分化,其分类地位的最终确定还需要相关生态生物学及遗传学方面的深入研究证据[39]。
致谢:样品采集中,得到国家林业局野生动植物检测中心白素英、王震、马跃老师的大力协助,在此表示衷心感谢!
[1] 常家传.鸟类学[M].哈尔滨:东北林业大学出版社,1994:62-63.
[2] 郑光美.鸟类学[M].北京:北京师范大学出版社,1995:453.
[3] 朱磊,孙悦华,胡锦矗.中国鸮形目鸟类分类现状[J].四川动物,2012,31(1):170-175.
[4] Amadon D,Bull J L,Marshall J T,et al.Hawks and owls of the world:a distributional and taxonomic list[C].Western Foundation of Vertebrate Zoology,1988:295-347.
[5] 郑作新,冼耀华,关贯勋.中国动物志:鸟纲 第六卷[M].北京:科学出版社,1991:141-150.
[6] König C,Weick F,Becking J H.Owls:a guide to the owls of the world[M].East Sussex:Pica Press,1999:197-426.
[7] König C,Weick F.Owls of the world[M].2nd Ed.Christopher Helm,2008:34-486.
[8] 赵正阶.中国鸟类手册:上卷 非雀形目[M].长春:吉林科学技术出版社,1995:662 -700.
[9] Wink M,El-Sayed A A,Sauer-Gürth H,et al.Molecular phylogeny of owls(Strigiformes)inferred from DNA sequences of the mitochondrial cytochrome b and the nuclear RAG-1 gene[J].Ardea,2009,97(4):581-591.
[10] 郑作新.中国鸟类种和亚种分布名录[M].北京:科学出版社,2000:59-63.
[11] 孙悦华,潘超.我国的鸮形目鸟类[J].生物学通报,2001,36(3):3-4.
[12] 马敬能,菲利普斯,何芬奇.中国鸟类野外手册[M].长沙:湖南教育出版社,2000:97-109.
[13] 郑作新.中国鸟类种和亚种分布名录[M].北京:科学出版社,2000:59 -63.
[14] 郑光美.中国鸟类分类与分布名录[M].北京:科学出版社,2005:116-126.
[15] Weick F.Owls(Strigiformes):annotated and illustrated checklist[M].Springer-Verlag Berlin Heidelberg,2006:7-244.
[16] 雷富民,卢汰春.中国鸟类特有种[M].北京:科学出版社,2006:368-370.
[17] Scherzinger W,Fang Y.Field observations of the Sichuan wood owlStrixuralensisdavidiin western China[J].Acrocephalus,2006,27:215-224.
[18] 方昀,孙悦华,Scherzinger W.甘肃莲花山四川林鸮初步观察[J].动物学杂志,2007,42(2):146-147.
[19] Cai Y,Yue B,Jiang W,et al.DNA barcoding on subsets of three families in Aves[J].Mitochondrial DNA,2010,21(3-4):132-137.
[20] Savolainen V,Cowan R S,Vogler A P,et al.Towards writing the encyclopedia of life:an introduction to DNA barcoding[J].Philosophical Transactions of the Royal Society B:Biological Sciences,2005,360(1462):1805-1811.
[21] Hebert P D N,deWaard J R,Landry J.DNA barcodes for 1/1000 of the animal kingdom[J].Biology Letters,2010,6:359-362.
[22] Hajibabaei M,Janzen D H,Burns J M,et al.DNA barcodes distinguish species of tropical Lepidoptera[J].Proceedings of the National Academy of Sciences of the United States of America,2006,103(4):968-971.
[23] Rawankar A S,Wagh G A,Wadatkar J S.DNA barcoding and phylogenetic analysis ofTytoalba,OtusbakkamoenaandAthenebramafrom Indian subcontinent[J].International Journal of Zoology and Research(IJZR),2015,5(5):7-14.
[24] 蔡延森,张修月,岳碧松,等.我国8种猛禽的 DNA条形码技术研究[J].四川动物,2009,28(3):334-340.
[25] Johnsen A,Rindal E,Ericson P G P,et al.DNA barcoding of Scandinavian birds reveals divergent lineages in trans-Atlantic species[J].Journal of Ornithology,2010,151(3):565-578.
[26] Saitoh T,Sugita N,Someya S.DNA barcoding reveals 24 distinct lineages as cryptic bird species candidates in and around the Japanese Archipelago[J].Molecular Ecology Resources,2015,15(1):177-186.
[27] Kerr K C R,Stoeckle M Y,Dove C J,et al.Comprehensive DNA barcode coverage of North American birds[J].Molecular Ecology Notes,2007,7(4):535-543.
[28] Kerr K C R,Birks S M,Kalyakin M V,et al.Filling the gap - COI barcode resolution in eastern Palearctic birds[J].Frontiers in Zoology,2009,6:29.
[29] Schindel D,Stoeckle M,Milensky C,et al.Project description:DNA barcodes of bird species in the National Museum of Natural History,Smithsonian Institution,USA[J].ZooKeys,2011,152:87-91.
[30] Yoo H S,Eah J Y,Kim J S,et al.DNA barcoding Korean birds[J].Molecules and Cells,2006,22(3):323-327.
[31] Tamura K,Peterson D,Peterson N,et al.MEGA5:molecular evolutionary genetics analysis using maximum likelihood,evolutionary distance,and maximum parsimony methods[J].Molecular Biology and Evolution,2011,28(10):2731-2739.
[32] Nylander J A.MrModeltest v2(Program distributed by the author,Evolutionary Biology Centre,Uppsala University,Uppsala,Sweden)[J].2004.
[33] Ronquist F,Huelsenbeck J P.MrBayes 3:bayesian phylogenetic inference under mixed models[J].Bioinformatics,2003,19(12):1572-1574.
[34] Swofford D L.PAUP:Phylogenetic analysis using parsimony(* and other methods),Version 4[M].Sunderland MA:Sinauer Associates,2003.
[35] Hebert P D N,Stoeckle M Y,Zemlak T S,et al.Identification of birds through DNA barcodes[J].PloS Biology,2004,2(10):e312.
[36] Tavares E S,Baker A J.Single mitochondrial gene barcodes reliably identify sister-species in diverse clades of brids[J].BMC Evolutionary Biology,2008,8(1):81.
[37] Hebert P D N,Penton E H,Burns J M,et al.Ten species in one:DNA barcoding reveals cryptic species in the neotropical skipper butterflyAstraptesfulgerator[J].Proceedings of the National Academy of Sciences of the United States of America,2004,101(41):14812-14817.
[38] Janzen D H,Hajibabaei M,Burns J M,et al.Wedding biodiversity Inventory of a large and complex Lepidoptera fauna with DNA barcoding[J].Philosophical Transactions of the Royal Society of London B:Biological Sciences,2005,360(1462):1835-1845.
[39] 孙悦华,Scherzinger W,刘迺发,等.四川林鸮在甘肃的新分布[J].动物学报,2001,47(4):473-475.
表1 鸱鸮科27种鸟类COⅠ基因序列
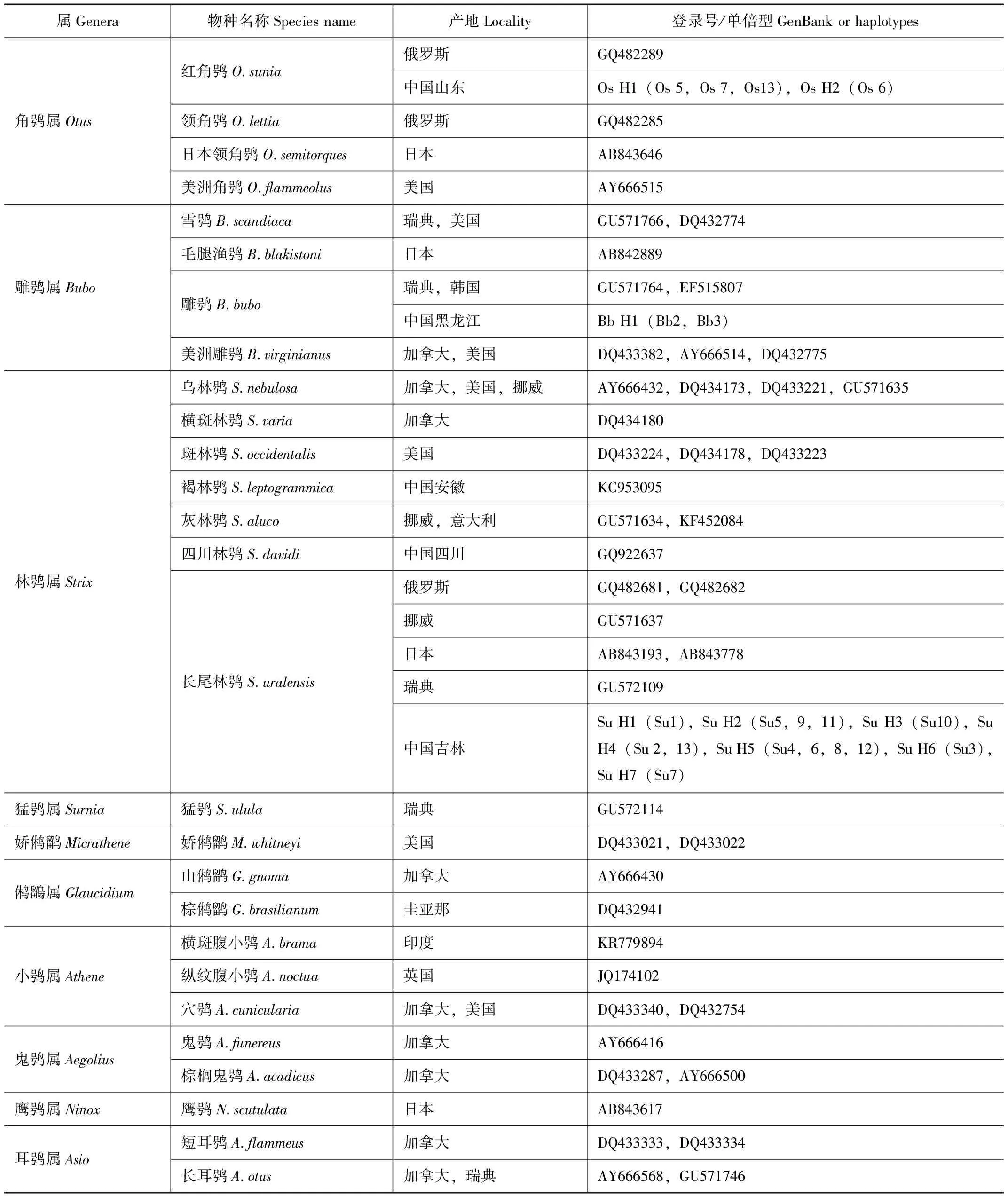
Tab.1 COⅠgene sequences of 27 species of Strigidae birds
Strigidae;
COI Sequence Variations and Phylogenetic RelationshipsAmong 27 Species of Strigidae Birds
Kang Hui1Ding Peng1Li Bo1,2*
In the taxonomy of the avian family Strigidae,there remain several controversial issues.For instance,should the generaKetupaandNycteabe incorporated into the genusBubo? ShouldStrixdavidiremain a species or should it be classified as a subspecies ofS.uralensis? mtDNA cytochrome oxidase subunit I(CO I)is an important gene in DNA barcoding and has widely been used for species identification and phylogenetic studies of birds.In this study we sequenced the CO I genes ofOtussunia,B.buboandS.uralensis from China.We used these sequences in combination with homologous sequences from GenBank to analyze the COI sequence variations and phylogenetic relationships of 27 species of the Strigidae.Interspecific variation was greater than intraspecific variation for most strigid species.Congeneric species were always clustered in phylogenetic analyses.Sequence variations and phylogenetic relationships supported the position thatKetupaandNycteabelong toBubo.With respect toS.uralensisin Eurasia,S.uralensisin Japan divided more than didS.davidi.The results of phylogenetic analysis confirmed thatS.davidiis a subspecies ofS.uralensis.This study provided new molecular evidence for defining phylogenetic relationships and revising the taxonomy of the Strigidae.
稿件运行过程
2016-05-19
修回日期:2016-06-21
发表日期:2016-11-10
四川林鸮;
CO I基因;
遗传距离;
系统发育
Strixdavidi;
COⅠgene;
Genetic distance;
Phylogeny
Q953
A
2310-1490(2016)04-351-06
鸱鸮科鸟类的种属分类仍存在一些争议。例如:渔鸮属和雪鸮属是否应并入雕鸮属,四川林鸮是一独立种还是长尾林鸮的一个亚种?线粒体 DNA 中细胞色素 C 氧化酶亚基I(CO I)基因是 DNA 条形编码的主要基因,被广泛应用于鸟类的物种鉴定和系统发育研究。通过测定中国产红角鸮、雕鸮和长尾林鸮COⅠ基因的序列,并结合 GenBank中鸱鸮科鸟类的同源序列,本文对27 种鸟类进行了序列变异和系统发育分析。结果显示,鸱鸮科鸟类多数是种间变异大于种内变异,该科鸟类几乎都是同属的优先聚类。序列分歧和系统发育分析结果支持将渔鸮属和雪鸮属置于雕鸮属下。同时,相对于欧亚大陆长尾林鸮种群,日本的长尾林鸮较四川林鸮分歧更大。系统发育分析结果也支持四川林鸮是长尾林鸮的一个亚种的观点。研究为进一步明晰鸱鸮科鸟类的系统发育和分类提供了新的分子证据。
东北林业大学大学生创新项目资助(201410225071)
康慧,女,23岁,硕士研究生;主要从事野生动物遗传育种与繁殖技术研究。E-mail:1715636570@qq.com
*通讯作者:李波,E-mail:libo_770206@126.com
