基因组选择一步法理论及应用研究进展
2016-11-12潘荣杨陈赞谋李加琪
潘荣杨,张 哲,高 宁,陈赞谋,李加琪,张 豪
(华南农业大学动物科学学院/国家生猪种业工程技术研究中心/广东省农业动物基因组与分子育种重点实验室,广东 广州 510642)
基因组选择一步法理论及应用研究进展
潘荣杨,张 哲,高 宁,陈赞谋,李加琪,张 豪
(华南农业大学动物科学学院/国家生猪种业工程技术研究中心/广东省农业动物基因组与分子育种重点实验室,广东 广州510642)
基因组选择(Genomic selection,GS)是一种新兴的畜禽遗传评定方法,较传统方法有明显优势,近十几年来成为畜禽遗传评定研究热点。但基因分型成本较高,实际育种过程中不可能对所有个体进行基因型检测,致使某些经济价值较小物种实施GS受限。一步法(Single step procedure)有效解决了这个问题。一步法既能将全基因组遗传标记用于畜禽遗传评定,又能将未经基因分型的个体全部纳入遗传评定模型,在猪、鸡等群体的基因组选择中应用备受关注。介绍了基因组选择一步法的原理,综述了其应用效果及相关问题等。
基因组选择;遗传评定;一步法
潘荣杨,张哲,高宁,等. 基因组选择一步法理论及应用研究进展[J].广东农业科学,2016,43(9):124-131.
动物育种的最终目的是提高群体的遗传水平,这主要是在个体遗传评定(genetic evaluation)或者育种值估计的基础上通过选择遗传优良的种畜来实现的,因此个体遗传评定是畜禽育种工作的中心任务之一。随着计算机及分子遗传学相关技术的不断进步,遗传评定的方法在不断改进与发展。
20世纪70~80年代,基于表型记录及系谱登记信息进行的选择指数理论和最佳线性无偏预测(best linear unbiased prediction,BLUP)育种值估计理论先后被提出,将畜禽遗传育种的理论和实践带入了一个崭新的发展阶段[1]。BLUP方法利用的信息为表型和系谱记录信息,收集表现较晚性状信息需要花费较长时间,且低遗传力性状的精确性较低,活体难以测量的性状(如胴体性状)实践中较难实施。
近年来,随着分子生物学技术的飞速发展,少量效应显著的遗传标记被用于实施标记辅助选择,即标记辅助BLUP法(marker assisted BLUP,MA-BLUP)[2-3]。但是,对畜禽重要经济性状而言都是数量性状,并且受到多个基因座位的影响。而目前发现并经过功能确认的影响畜禽重要经济性状的基因或标记数量有限,标记辅助选择所用标记往往只能解释较小比例的遗传变异[4-5],因此在育种实践中应用效果不如预期,限制了标记辅助选择在畜禽育种中的推广应用。2001年Meuwissen等提出了具有划时代意义的基因组选择(genomic selection,GS)方法,该方法将全基因组高密度标记信息用于畜禽遗传评定,克服了MA-BLUP的缺陷[6]。随着高通量测序技术的不断发展,标记基因分型成本不断降低。尤其是Schaeffer等指出GS在奶牛的育种中具有巨大的实用价值[7],许多研究者纷纷投入到基因组选择的研究中,并提出了一系列估计育种值的计算方法,基因组选择理论得以飞速发展,在奶牛等育种实践中得到了广泛应用。
1 基因组选择GBLUP和一步法的方法原理
GS利用覆盖全基因组的高密度SNP标记,结合表型记录和(或)系谱信息对个体育种值进行估计。基因组选择的一个基本假设是:在高密度的SNP标记中,每个基因都至少有1个SNP与其处于连锁不平衡(linkage disequilibrium,LD)状态,可用标记间接反映基因的效应[4,6]。从而通过全基因组遗传标记准确预测个体的基因组育种值(genomic estimated breeding values,GEBV)。GEBV能够提高育种值估计准确性,加速遗传进展,减少育种投入而受到畜禽育种界的广泛关注。目前,GEBV的计算方法主要有通过估计标记效应间接计算GEBV的间接法[8-9]和通过构建基因组关系矩阵(genomic relationship matrix,G)直接估计得到GEBV的直接法[10-13]。目前,GS已经在奶牛育种已经得到了广泛应用[14]。
然而,对于实际育种工作中,受到成本、人力、时间等客观因素的限制,难以对所有个体进行基因分型,限制了GS在其他物种中的应用。根据诸多文献报道,我们总结了对经济价值低的物种实施GS的可能途径:(1)低密度SNP策略:采用低密度SNP芯片进而降低成本,由于前期全基因组关联分析(GWAS)研究表明大量座位对某些性状没有影响,进而可根据GWAS结果对不同选择的性状设计特定低密度SNP芯片[15];另外,采用基因填充技术(imputation),把低密度芯片填充成高密度芯片,从而降低高密度芯片基因分型成本[16-17];(2)实施基因组选择一步法(single step procedure):2010年Misztal、Christensen等先后提出一步法,一步法的核心思想是将传统的血缘关系矩阵A矩阵与基因组关系矩阵G矩阵合并,重新构建出新的关系矩阵H矩阵[13,18-20]。一步法既能将全基因组遗传标记用于畜禽遗传评定,又能将未经基因分型检测的个体全部纳入遗传评定模型,个体的信息在参考群体中包括了没有基因型记录个体的表型信息,同时也运用有基因分型个体的信息和没有基因分型个体的信息,允许了部分个体测定基因型即可实施GS。因此,不同程度上克服了GBLUP和传统BLUP的某些缺陷,大大降低对所有个体测定基因分型成本,为猪或家禽育种实施GS提供新的方法。该方法有效地将传统遗传评定体系与基因组选择遗传评定体系进行融合,有研究者将一步法称作SSGBLUP(single step genomic BLUP,SSGBLUP)[21-22]。近年来,一步法应用于猪、鸡等群体基因组选择中而备受关注(如丹育公司、PIC、Topigs等),但在实际应用中也有一些关键问题有待探讨。
1.1 基因组选择GBLUP模型
基因组选择中GBLUP方法模型实质是与传统BLUP模型一致,其模型表示如下:

式中,y是表型性状观察值;b是固定效应向量;u是随机加性遗传效应向量,即育种值向量;X和Z分别为固定效应b和随机效应u的关联矩阵;e为随机残差效应向量,即e~N(0,Iσe2)。
该模型中随机效应u,在传统动物模型BLUP法中,u~N(0,Aσg2),其中A为由系谱构建的分子血缘关系矩阵,σg2为加性遗传方差;而在基因组选择GBLUP法中,u~N(0,Gσg2),其中G为利用全基因组标记构建的基因组关系矩阵,两者的区别只在于关系矩阵的不同。VanRaden提出了构建G矩阵的策略[12],G矩阵被定义为:G = ZZ′/k,式中,Z阵列元素为:0-2pj、1-2pj、2-2pj分别对应SNP基因型A1A1、A1A2、A2A2,而pj为等位基因A2频率。k定义为:

我们可根据混合模型方程组(mixed model equation,MME)求解技术进行求解[23],对于如下方程组:

式中,G-1为基因组关系矩阵G的逆矩阵,而α=σg2/σe2。
1.2基因组选择一步法理论
基因组选择中一步法模型是传统动物模型BLUP法及基因组选择GBLUP法的合并,其模型形式上与BLUP及GBLUP法模型并无区别。其模型(1)式相同,三者区别于u(随机加性遗传效应);在基因组选择一步法中u~N(0,Hσg2),其中H为合并关系矩阵[13,19-20],定义为:
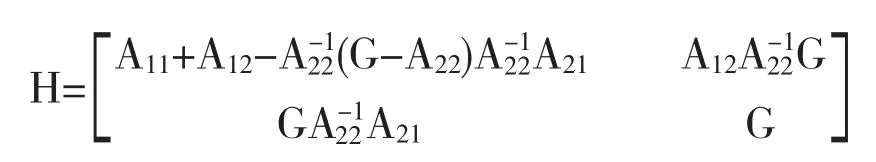
式中,A11和A22分别为系谱群体中无基因分型检测及有基因分型检测的个体间的分子血缘关系矩阵,G为有基因分型个体间的基因组关系矩阵。通过此方式,基因分型记录的亲缘关系与系谱记录的亲缘关系信息便合并到了同一个模型中,基因组遗传评定便可与传统遗传评估方式实现有机整合。而对于一步法的求解,同样可以采用MME方程组求解,只需将G-1矩阵替换为H-1矩阵即可。
如上述所说,通过采用MME即可对一步法模型进行求解,但MME的完整构建需要先实现对H矩阵的求逆计算。一步法中的H矩阵是由基因组关系矩阵G和分子血缘关系矩阵A合并而成,对于庞大的系谱而言,H矩阵的直接求逆计算强度太大。Christensen等和Aguilar等均推导了H-1矩阵的构建方法[18,20],可表示为:

由于G有时为奇异矩阵或者几乎接近奇异矩阵而无法求逆,为此VanRaden提出将G定义为:Gω=(1-ω)G+ωA22,而解决G矩阵无法求逆问题[15]。一步法的H-1则可表示为:

式中,ω为加权因子,表示多基因遗传效应所占的比例。ω的选取主要取决于对应组分所能解释的遗传方差比例,可用方差组分估计的方式确定,或尝试不同的值确定最优参数[24-26]。
此外,一步法中关系矩阵合并的兼容性也引起了研究者们的重视。根据VanRaden构建G策略中的等位基因频率应为基础群的频率,但由于获得基础群等位基因频率非常困难,因此在实际中往往通过对有基因型个体的标记数计算而得。对于分子血缘关系矩阵A而言,系谱的记录可以追索到基础群中,A矩阵能反映所有个体间的血缘关系;但对于基因组关系矩阵G而言,考虑到基因分型过程中的客观问题,往往只选择部分优秀个体(或有代表性个体)进行基因分型,因此G矩阵恰恰只是反映有基因型个体间的基因组关系,这就导致分子血缘关系矩阵A与基因组关系矩阵G存在尺度上的差异[25,27]。这种差异对单性状模型或多性状模型的育种值估计结果都产生一定影响,因此需适当考虑尺度校正,而不同的校正方式先后被研究者们提出。Christensen等综合前人研究报道后提出新的尺度校正公式:

式中,G*为校正后的基因组关系矩阵,而实施的α和β则根据下列公式求得:

这种校正方法在一个丹麦杜洛克猪群体中得以验证,Christensen等[26]证实了这种校正方式的有效性,该种校正方式后的一步法准确性优于原始一步法。
2 一步法的应用及研究进展
2.1一步法的应用效果
一步法能有效解决不同畜种基因组选择实施中可能存在的一些问题,研究者们对该方法展开了不同物种群体研究及方法可行性研究。对于荷斯坦奶牛群体的体细胞评分性状,与传统BLUP方法比较,一步法准确性提高了10%;而间接法的准确性则与一步法接近[18]。Chen等[21-22]的研究结果显示,在胸肌重、腿部分数两个性状中,一步法与传统方法和间接法相比的准确性分别有10%和20%的提高,校正后一步法也明显高于未校正的一步法。Christensen等[26]的研究性状为一个杜洛克猪群体分别对日增重和饲料转化率(FCR),结果多性状模型的一步法准确性最高,单性状一步法次之,一步法与传统方法比较准确性提高了16%,而与GBLUP相比较提高得不明显。Baloche等[28]分析了法国奶山羊的产奶量等性状,一步法的准确性为0.47、间接法为0.43、传统方法为0.32。Lourenco等[29]通过模拟一个肉牛群体,并考虑了加性遗传效应与母体效应是否相关,结果一步法准确性比传统方法和间接法分别提高了2%和17%。此外,许多国家地区也根据国家遗传评定方式或者综合选择指数对各自国家物种展开研究,其中对奶牛群体研究有不少报道,例如Su和Gao等基于北欧综合指数下的多个性状,分别对北欧荷斯坦奶牛及北欧红荷斯坦奶牛群体进行了一步法与多种方法的比较[24,30];Tsuruta等[31]选择美国国家遗传评定的18个荷斯坦牛性状,表明一步法的准确性皆高于传统方法或间接法等其他方法。Guo等[32]基于丹育公司(DanBred company)大白猪和长白猪群体,对窝产仔数和仔猪死亡率两个低遗传力繁殖性状进行遗传评估,无论对有基因分型还是没有基因分型的个体,一步法准确性相比于传统方法大大提高,证实了基因组选择方法对低遗传力性状研究的显著优势。我们汇总了这些研究所使用的物种、性状、准确性、标记数、作者等信息,如表1、表2所示。可见,一步法已经在许多动物中展开了深入研究,其可行性与准确性也得到验证。
2.2一步法的计算软件
软件求解问题常常也是育种者所关注的,一步法求解计算软件逐渐被公布,例如,Misztal研究团队开发的BLUPF90软件(BLUPF90 family of program)[36]和丹麦Madsen研究团队开发的DMU(Derivative free multivariate)软件[37]等均可对一步法进行求解。虽然这些软件各有不用的使用方式和功能,但都是服务于畜禽育种领域。如BLUPF90应用于美国荷斯坦奶牛遗传评定中心,而DMU应用于丹麦猪遗传评定工作等。然而,实际育种工作的数据记录很庞大,如果采用MME的计算方式,则对计算的内存和硬件要求很大,对于大数据通常采用递归算法进行求解计算。Fragomeni等对奶牛大规模数据进行计算策略探讨,提出了一种APY(proven and young animals)递归法计算,解决了一步法在数据庞大(记录数超过10万)时的计算问题[38-40]。
2.3候选群测定基因型大小及测定个体选择
实施基因选择要求个体要基因分型,有了基因分型数据才可根据统计模型准确地计算个体的基因组育种值(GEBV)。然而对于一些经济价值较小的物种(猪或家禽),候选群测定基因型多少,测定个体如何选择、实施某种基因选择方法成为育种者实施基因组选择育种所关心的关键问题[4,41]。在一步法遗传评估理论中,允许了部分个体进行基因分型即可实施基因组选择而大大降低对所有个体基因分型的成本,但是候选群中基因分型大小及测定个体选择的问题,成为了实施基因选择一步法探讨所在(Danish Pig ResearchCentre,2010)。在丹育公司猪遗传研究中心(Pig Research Centre,PRC)根据丹育育种体系结构,初步挑选2 000头最优的杜洛克群体开始实施GS,并且探讨候选群体中基因分型个体的比例,以及基因分型个体如何挑选等诸多问题进行模拟研究。研究结果表明:根据传统EBV挑选出最优的2 000头杜洛克猪群体实施GS,候选群在性能测定后根据EBV挑选40%个体进行基因分型、基因分型个体公母猪比例为1∶3,即可获得与全群个体基因分型而获得的进展[42]。这也提示我们候选群个体不需要全群测定基因型、只要部分个体基因分型且分型个体按传统EBV挑选即可获得GS潜在的最大遗传进展,进而育种者大大节省部分基因分型费用。

表1 一步法在各物种中应用情况

表2 一步法与其他方法在各物种中准确性的比较
2.4一步法在杂种及GWAS应用
杂种评估一直是基因组选择研究领域的热点与难点[14,43]。Christensen等[43]采用模拟数据,尝试将一步法扩展到杂种评估,在不同品系间考虑配子效应(gametic effects),并将其合并到关系矩阵中,验证了一步法在杂种遗传评估的有效性。Lourenco等[44]通过模拟数据和真实数据证实了一步法对杂种群体的遗传评估效果,并论证了基于杂种群体等位基因频率构建基因组关系矩阵才能发挥一步法的优势。随着高通量测序技术日益完善、测序费用的逐渐降低,掀起了全基因组关联分析(GWAS)研究的热潮,而将基因组选择方法应用到GWAS研究也引起研究者们的重视。Wang等[45]将一步法应用到GWAS的研究中,并且在一个肉鸡群体中验证了该方法的可行性。由此可见,一步法已不是简单地对单性状(或单一品种)进行GEBV预测,一步法的研究已逐渐多元化,扩展到多性模型、杂种遗传评估甚至GWAS研究中[46-47]。
3 展望
一步法将基因型信息与系谱信息进行合并,有效地将基因组信息运用到遗传评定中。一步法对于经济价值较低的物种(如猪、鸡),更是提供了新的基因组选择方法途径,使得这些动物实施GS成为可能。目前,一步法已经在国际著名猪育种公司(丹育、PIC、Topigs等)得以应用。Zhang等[11,48]根据前人GWAS的研究,将部分有显著效应的座位信息加入G矩阵中,构建出GA矩阵进而提出了BLUP|GA法(BLUP approach conditional on the Genetic Architecture),与GBLUP相比提高了遗传评定的准确性。根据BLUP|GA法构建的GA矩阵替代G矩阵,生物学先验信息是否使一步法包含的信息更丰富,能否提高准确性有待进一步研究。而近期随着高通量技术的发展,测序成本不断降低,未来基因分型个体数将更多,实施一步法的效果也将更明显。通过不断的改进与完善,一步法的优势将得到充分的利用,并将成为畜禽遗传评估中的主流方法之一。
[1]张沅. 家畜育种学[M]. 北京:中国农业出版社,2001.
[2]Goddard M E. A mixed model for analyses of data on multiple genetic markers[J]. Theor Appl Genet,1992,83(6-7):878-886.
[3]Fernando R L A M. Grossman marker assisted selection using best linear unbiased prediction[J]. Genet Sel Evol,1989,21(4):467-477.
[4]Goddard M E,Hayes B J. Mapping genes for complex traits in domestic animals and their use in breeding programmes[J]. Nature Reviews Genetics,2009,10(6):381-391.
[5]张哲,张勤,丁向东. 畜禽基因组选择研究进展[J]. 科学通报,2011(26):2212-2222.
[6]Meuwissen T H E,Hayes B J,Goddard M E. Prediction of total genetic value using genomewide dense marker maps[J]. Genetics,2001,157(4):1819-1829.
[7]Schaeffer L R. Strategy for applying genomewide selection in dairy cattle[J]. J Anim Breed Genet,2006,123(4):218-223.
[8]Gao N,Li J,He J,et al. Improving accuracy of genomic prediction by genetic architecture based priors in a Bayesian model[J]. BMC Genetics,2015,16(1):1-11.
[9]Meuwissen T H,Solberg T R,Shepherd R,et al. A fast algorithm for BayesB type of prediction of genome-wide estimates of genetic value[J]. Genetics Selection Evolution,2009,41(1):2.
[10]Zhang Z,Liu J F,Ding X D,et al. Best linear unbiased prediction of genomic breeding values using a trait-specific marker-derived relationship matrix.[J]. Plos One,2010,5(9):e12648.
[11]Zhang Z,Ober U,Erbe M,et al. Improving the accuracy of whole genome prediction for complex traits using the results of genome wide association studies[J]. Plos One,2014,9(3):e93017.
[12]VanRaden P M. Efficient methods to compute genomic predictions[J]. J Dairy Sci,2008,91(11):4414-4423.
[13]Misztal I,Legarra A,Aguilar I. Computing procedures for genetic evaluation including phenotypic,full pedigree,and genomic information[J]. J Dairy Sci,2009,92(9):4648-4655.
[14]Meuwissen T H E,Hayes B J,Goddard M E.Genomic selection:A paradigm shift in animal breeding[J]. Animal Frontiers,2016,1(6):6-14.
[15]张勤. 基因组选择[A]. 第十五次全国动物遗传育种学术讨论会专题报告[C]. 杨凌,2009.
[16]Cleveland M A,Hickey J M. Practical implementation of cost-effective genomic selection in commercial pig breeding using imputation[J]. J Anim Sci,2013,91(8):3583-3592.
[17]Zhao Y,Dai J,Bai J. Genotype Imputation for Genome-wide Association Studies[J]. Chinese Journal of Health Statistics,2011,11(7):1-8.
[18]Aguilar I,Misztal I,Johnson D L,et al. Hot topic:A unified approach to utilize phenotypic,full pedigree,and genomic information for genetic evaluation of Holstein final score1[J]. J Dairy Sci,2010,93(2):743-752.
[19]Legarra A,Ducrocq V. Computational strategies for national integration of phenotypic,genomic,and pedigree data in a single-step best linear unbiased prediction[J]. J Dairy Sci,2012,95(8):4629-4645.
[20]Christensen O F,Lund M S. Genomic prediction when some animals are not genotyped[J]. Genetics Selection Evolution,2010,42(1):1-8.
[21]Chen C Y,Misztal I,Aguilar I,et al. Effect of different genomic relationship matrices on accuracy and scale[J]. J Anim Sci,2011,89(9):2673-2679.
[22]Chen C Y,Misztal I,Aguilar I,et al. Genomewide marker-assisted selection combining all pedigree phenotypic information with genotypic data in one step:An example using broiler chickens[J]. J Anim Sci,2010,89(1):23-28.
[23]Henderson C R. Best linear unbiased estimation and prediction under a selection model[J]. Biometrics,1975,31(2):423-447.
[24]Gao H,Christensen O F,Madsen P,et al. Comparison on genomic predictions using three GBLUP methods and two single-step blending methods in the Nordic Holstein population.[J]. Genetics Selection Evolution,2012,44(1):8.
[25]Forni S,Aguilar I,Misztal I. Different genomic relationship matrices for single-step analysis using phenotypic,pedigree and genomic information[J]. Genetics Selection Evolution,2011,43(1):1-7.
[26]Christensen O F,Madsen P,Nielsen B,et al. Single-step methods for genomic evaluation in pigs[J]. animal,2012,6(10):1565-1571.
[27]Vitezica Z G,Aguilar I,Misztal I,et al. Bias in genomic predictions for populations under selection[J]. Genet Res (Camb),2011,93(5):357-366.
[28]Baloche G,Legarra A,Salle G,et al. Assessment of accuracy of genomic prediction for French Lacaune dairy sheep[J]. J Dairy Sci,2014,97(2):1107-1116.
[29]Lourenco D A L,Misztal I,Wang H,et al. Prediction accuracy for a simulated maternally affected trait of beef cattle using different genomic evaluation models[J]. J Anim Sci,2013,91(9):4090-4098.
[30]Su G,Madsen P,Nielsen U S,et al. Genomic prediction for Nordic Red Cattle using one-step and selection index blending[J]. J Dairy Sci,2012,95(2):909-917.
[31]Tsuruta S,Misztal I,Aguilar I,et al. Multipletrait genomic evaluation of linear type traits using genomic and phenotypic data in US Holsteins[J]. J Dairy Sci,2011,94(8):4198-4204.
[32]Guo X,Christensen O F,Ostersen T,et al. Improving genetic evaluation of litter size and piglet mortality for both genotyped and nongenotyped individuals using a single-step method[J]. J Anim Sci,2015,93(2):503.
[33]Koivula M,Strandén I,Su G,et al. Different methods to calculate genomic predictions-Comparisons of BLUP at the single nucleotide polymorphism level (SNP-BLUP),BLUP at the individual level (G-BLUP),and the one-step approach (H-BLUP)[J]. J Dairy Sci,2012,95(7):4065-4073.
[34]Gray K A,Cassady J P,Huang Y,et al. Effectiveness of genomic prediction on milk flow traits in dairy cattle[J]. Genetics Selection Evolution,2012,44(1):1-6.
[35]Aguilar I,Misztal I,Legarra A,et al. Efficient computation of the genomic relationship matrix and other matrices usedin single-step evaluation[J]. J Anim Breed Genet,2011,128(6):422-428.
[36]Misztal I. Tsuruta S. Strabel. BLUPF90 and related programs (BGF90):In:Proceedings of the 7th World Congress on Genetics Applied to Livestock Production,Montpellier,France,2002[C].
[37]Madsen P. Jensen J. A user's guide to DMU. A Package for analysing multivariate mixed models[Z]. 2000.
[38]Fragomeni B O,Lourenco D A,Tsuruta S,et al. Hot topic:Use of genomic recursions in singlestep genomic best linear unbiasedpredictor(BLUP) with a large number of genotypes[J]. J Dairy Sci,2015,98(6):4090-4094.
[39]Misztal I. Inexpensive Computation of the Inverse of the Genomic Relationship Matrix in Populations with Small Effective Population Size[J]. Genetics,2016,202(2):401-409.
[40]Misztal I,Legarra A,Aguilar I. Using recursion to compute the inverse of the genomic relationship matrix[J]. J Dairy Sci,2014,97(6):3943-3952.
[41]Wolc A,Stricker C,Arango J,et al. Breeding value prediction for production traits in layer chickens using pedigreeor genomic relationships in a reduced animal model[J]. Genet Sel Evol,2011,43:5.
[42]Centre Danish Pig Research. annual report[Z]. 2010:15-17.
[43]Christensen O F,Madsen P,Nielsen B,et al. Genomic evaluation of both purebred and crossbred performances[J]. Genetics Selection Evolution,2013,46(1):1-9.
[44]Lourenco D A,Tsuruta S,Fragomeni B O,et al. Crossbreed evaluations in single-step genomic best linear unbiased predictor using adjusted realized relationship matrices[J]. J Anim Sci,2016,94(3):909-919.
[45]Wang H,Misztal I,Aguilar I,et al. Genomewide association mapping including phenotypes from relatives without genotypes in a single-step(ssGWAS) for 6-week body weight in broiler chickens[J]. Frontiers in Genetics,2014,5:134.
[46]Legarra A,Christensen O F,Aguilar I,et al. Single Step,a general approach for genomic selection ☆[J]. Livestock Science,2014,166(1):54-65.
[47]Misztal I,Aggrey S E,Muir W M. Experiences with a single-step genome evaluation[J]. Poult Sci,2013,92(9):2530-2534.
[48]Zhang Z,Erbe M,He J,et al. Accuracy of wholegenome prediction using a genetic architectureenhanced variance-covariance matrix[J]. G3-Genes Genomes Genetics,2015,5(4):615-27.
(责任编辑 崔建勋)
Research progress in Single step procedure theory and application in genomic selection
PAN Rong-yang,ZHANG Zhe,GAO Ning,CHEN Zan-mou,LI Jia-qi,ZHANG Hao
(College of Animal Science,South China Agricultural University/National Engineering Research Center for Breeding Swine Industry/Provincial Key Lab. of Agro-Animal Genomics and Molecular Breeding,Guangzhou 510642,China)
Genomic selection is an emerging technology of genetic evaluation in livestock and poultry. Studies have reported that genomic selection has more advantages than traditional methods,which make it a hot topic in the field of animal genetic evaluation. However,for species with lower economic importance such as poultry and swine,it is difficult to genotype all individuals. Single-step procedure integrates not only phenotypes and genetic markers of genotyped individuals into genomic evaluation,but also field records of non-genotyped individuals through the pedigree of the population,which is an ideal alternative for genomic evaluation. The application of single step procedure in pig (or poultry) genomic selection has attracted much attention,but there are some key issues to be discussed in practical application. In this paper,the principle of single step procedure,the current status of implementation and related issues of single step method in livestock were reviewed.
genomic selection;genetic evaluation;single step procedure
S813.3
A
1004-874X(2016)09-0124-08
2016-06-15
国家自然科学基金(31200925);国家现代农业产业技术体系专项(CARS-36)
潘荣杨(1989-),男,在读硕士生,E-mail:panrongyang@126.com
张豪(1965-),男,博士,教授,E-mail:zhanghao@scau.edu.cn
