多元PCR技术在毛蚶家系鉴定中的应用*
2016-10-18孔令锋
陈 辰, 李 琪, 孔令锋, 于 红
(中国海洋大学海水养殖教育部重点实验室,山东 青岛 266003)
研究简报
多元PCR技术在毛蚶家系鉴定中的应用*
陈辰, 李琪❋❋, 孔令锋, 于红
(中国海洋大学海水养殖教育部重点实验室,山东 青岛 266003)
为促进毛蚶(Scapharcakagoshimensis)的遗传育种研究及开展其资源的保护工作。本研究用4组微卫星多元PCR体系对5个毛蚶全同胞家系进行了家系鉴定分析。研究表明:4组多元PCR体系中的10个微卫星位点均为高多态性位点,使用1组多元PCR体系的子代模拟鉴定成功率和实际鉴定成功率分别为78%和72%,3组以上多元PCR体系子代的鉴定成功率为100%。研究结果表明,微卫星多元PCR技术能快速、准确地将任意子代鉴定至其所属家系,该技术可用于大批量家系材料分析,具有较好的应用价值。
毛蚶;微卫星标记;多元PCR;家系鉴定
引用格式:陈辰, 李琪, 孔令锋, 等. 多元PCR技术在毛蚶家系鉴定中的应用[J]. 中国海洋大学学报(自然科学版), 2016, 46(8): 18-23.
CHEN Chen, LI Qi, KONG Ling-Feng, et al. Parentage analysis in the ark shell (Scapharcakagoshimensis) based on microsatellite multiplex PCRs[J]. Periodical of Ocean University of China, 2016, 46(8): 18-23.
毛蚶(Scapharcakagoshimensis)是广泛分布在西北太平洋近海的埋栖型贝类,因其肉质肥嫩,营养丰富,具有较高的经济价值,是中国传统的渔业对象[1]。由于在采捕过程中,长期缺乏必要的资源保护意识和行之有效的保护措施,近年来中国毛蚶的自然资源量出现明显的下降,一些重要的毛蚶产区目前已无法形成渔汛[2-3]。为促进我国毛蚶产业的可持续发展,很多地方已经陆续展开了毛蚶苗种人工繁育和底播增殖放流工作[3-4]。
准确掌握亲本和子代的遗传信息是制订科学合理放流计划和评估放流效果的前提条件。与鱼类和甲壳类不同,贝类体表通常存在钙质外壳,限制了物理标记和荧光标记的应用,因而使用分子标记进行家系分析,是目前贝类群体研究和种质资源保护中最为有效的分析工具[5-9]。微卫星标记因其变异程度高,在基因组中分布广泛,易于PCR扩增等特点,是家系分析中常用的分子标记[10-12]。
然而,在实际应用中微卫星标记存在2个突出问题。首先,微卫星标记属于单位点标记[13],通过一个PCR反应,每次只能获得1个样本的1个位点的遗传信息,样本的处理通量较低。应用多元PCR体系,可以一定程度上解决微卫星标记处理通量的问题[14]。其次,虽然微卫星标记理论上符合孟德尔遗传规律,但在一些位点中存在无效等位基因以及与隐性致死基因连锁的情况,使得这些微卫星标记在子代的分离比产生偏离[15-17],从而影响数据分析的准确性。因而,在实际应用前,对于已经开发的微卫星标记进行相关验证是必要的[18]。
目前毛蚶已经开发出微卫星标记[19],但对于标记的验证工作尚未见报道。本文根据前期开发的多元PCR体系[20],利用5个毛蚶全同胞家系对标记进行验证,以期为毛蚶今后的增殖放流效果评估提供可靠的技术支持。
1 材料与方法
1.1 材料
全同胞家系构建于2014年6月在山东省日照欣慧水产育苗有限公司进行。挑选性成熟良好的亲贝,采用阴干加流水刺激的方法,诱导亲贝配子排放,通过人工受精的方式建立了5个毛蚶单对交配家系。受精后72h,使用300目筛绢过滤收集每个家系的D形幼虫。使用胶头滴管将收集到的幼虫,分别转移至1.5mL的离心管中,每个离心管中加入1mL无水乙醇进行固定。此后,每12h更换无水乙醇1次,更换无水乙醇3~4次,以充分置换样本中的水分。亲贝精卵排放后,立即对亲本进行解剖,剪取亲本闭壳肌,转移至1.5mL的离心管中,加入无水乙醇进行固定,处理方法同幼虫。
1.2 DNA提取与微卫星分析
亲本DNA提取使用优化苯酚-氯仿法[21];幼虫DNA提取方法参照Li等[17],使用Chelex树脂方法。选择前期开发的4组微卫星多元PCR体系[20],位点的序列信息见表1。PCR反应体系为10μL,每个反应体系中包括:50~100ng/μL的DNA模板,0.4 U DNA聚合酶,1×PCR Buffer(Mg2+Free),dNTP混合物200mmol/L,MgCl21.5mmol/L,正反向引物各1μL(2~5μmol/L)。为保证多元PCR的扩增效率,在反应体系中加入了强化组分,成分包括30%(w/v)PEG6000,2.5μg/μL BSA,5mol/L Betaine。PCR的反应程序为:94℃ 3min;94℃变性45s,54℃退火60s,72℃延伸75s,35~40个循环;72℃延伸10min,4℃保存。PCR扩增产物经95℃变性3min后,与变性剂等体积混合,使用6%聚丙烯酰胺凝胶电泳分离等位基因,使用弱碱法进行银染显色,选择10bp DNA Lad-der(Invitrogen)作为marker。
1.3 数据分析
使用χ2检验子代中位点的分离情况是否符合孟德尔遗传定律,子代表型期望分离比分别为1∶1,1∶2∶1和1∶1∶1∶1,差异显著性水平设定为0.01。使用CERVUS 3.0软件[22]计算各位点在5个家系中的等位基因数(NA),多态信息含量(PIC),以及位点的非排除能力(NE-1P,NE-2P,NE-PP)。使用FSTAT软件[23]计算每一位点在全部家系样本中的等位基因丰富度(AR),以校正样本数目差异产生的误差。混合5个单对交配家系135个子代的基因型数据,以检验多元PCR技术在家系鉴定中的效率。使用CERVUS 3.0软件进行家系的模拟鉴定以及实际鉴定计算,具体参数设置为:亲本数量10,模拟子代数10000,亲本检测率及位点检测率100%,分型误差率1%,置信水平95%。
2 结果
2.1 微卫星位点的特征
4组微卫星多元PCR体系在5个家系中,每个位点的等位基因数目、等位基因丰富度、多态信息含量和非排除率见表1。10个微卫星位点的等位基因数范围为7(Sk02,Sk03,Sk04)~13(Sk14),平均等位基因数为9.4。等位基因丰富度范围为3.606~4.780,平均等位基因丰富度为4.125。位点的多态信息含量范围为0.727(Sk01)~0.895(Sk09),平均多态信息含量为0.809。
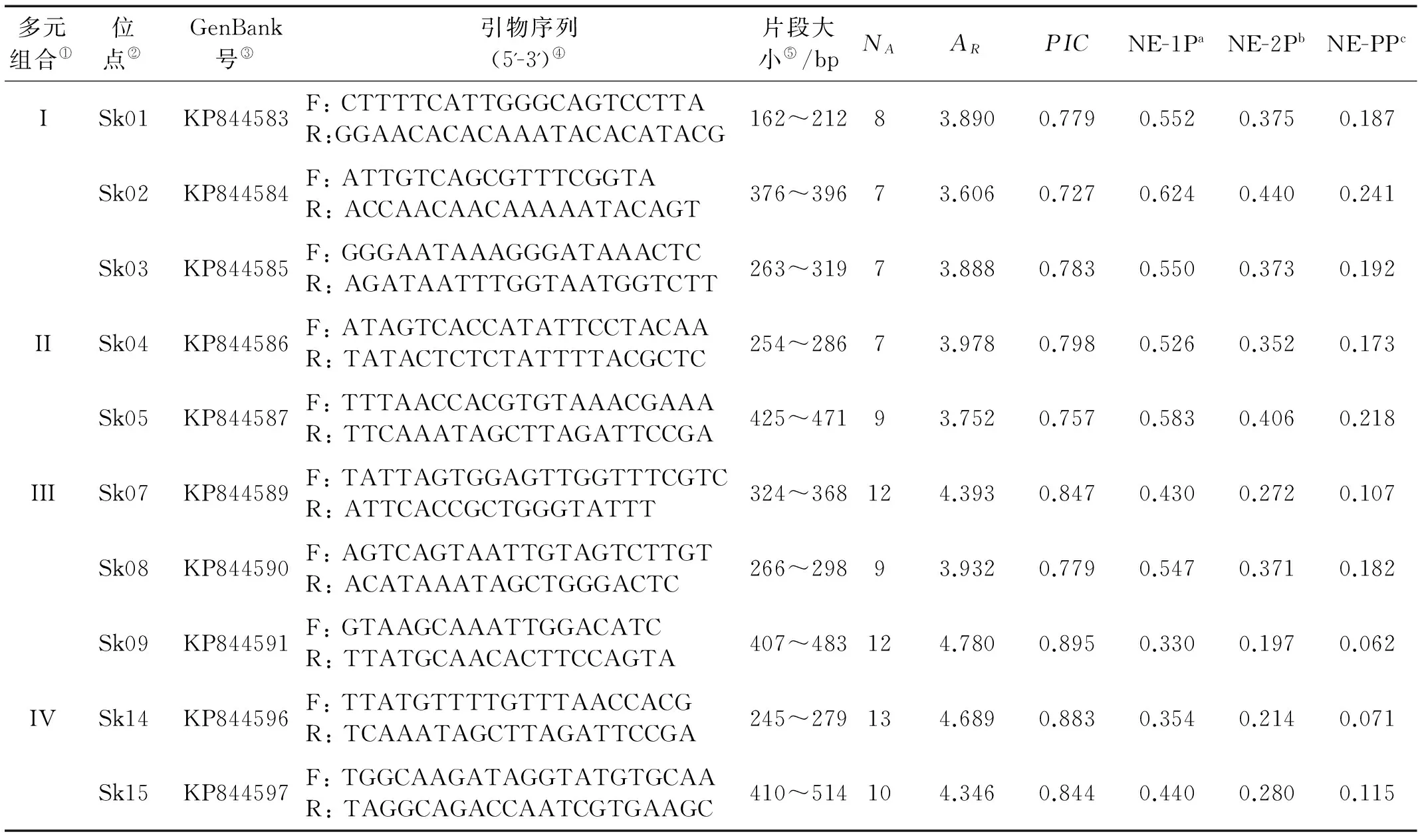
表1 毛蚶4组微卫星多元PCR在5个全同胞家系中的特征
注: 上标a,b,c含义如下,a.单亲本的平均非排除率; b.已知单亲本基因型的平均非排除率; c.两亲本的平均非排除率。
Note: The meaning of superscript a,b,c are as follows. a. Average nonexclusion probability for one candidate parent. b. Average non-exclusion probability for one candidate parent given the genotype of a known parent of the opposite sex. c. Average non-exclusion probability for a candidate parent pair.
①Multiplex panel; ② Locus; ③GenBank accession number; ④Primer sequence (5'-3'); ⑤Size range
2.2 微卫星位点在子代中的分离模式
在获得的50个基因型分离比中(10位点×5个家系),有5个为亲本全部为纯合子产生的基因型(见表2),所获得的子代的基因型与亲本或者完全相同(亲本等位基因相同),或者为同一种杂合状态的基因型(亲本等位基因不同)。对剩余的45个分离模式分析发现,32个分离模式符合孟德尔分离规律;在考虑无效等位基因的情况下,剩余13个基因型中,有2个基因型分离比偏离孟德尔分离定律(F3:Sk04,Sk05;P<0.01)。在5个家系所包含的200个等位基因中(10个亲本×10个微卫星位点×2),无效等位基因频率为9.5%(见表2)。

表2 10个微卫星标记在5个毛蚶全同胞家系中的分离
续表2

家系①多元组合②位点③母本④父本⑤子代⑥期望分离比⑦观测分离比⑧χ2P值⑨F4ISk01162/180162/162162/162∶162/1801∶118∶93.0000.083Sk02376/382384/X376/384∶376/X∶382/384∶382/X1∶1∶1∶14∶7∶11∶45.0770.166Sk03287/X307/X287/307∶287/X∶307/X∶X/X1∶1∶1∶18∶13∶4∶28.5900.035IISk04276/282276/282276/276∶276/282∶282/2821∶2∶110∶8∶95.6670.059Sk05459/465425/445425/459∶445/459∶425/465∶445/4651∶1∶1∶17∶7∶3∶103.6670.300IIISk07332/364324/346324/332∶332/346∶324/364∶346/3641∶1∶1∶19∶9∶6∶25.0770.166Sk08272/296282/290272/282∶272/290∶282/296∶290/2961∶1∶1∶110∶6∶2∶85.3850.146Sk09455/479413/413413/455∶413/4791∶116∶82.6670.102IVSk14474/474456/456456/474126--Sk15265/279279/279265/279∶279/2791∶112∶140.1540.695F5ISk01164/164164/192164/164∶164/1921∶114∶91.0870.297Sk02384/396376/382376/384∶382/384∶376/396∶382/3961∶1∶1∶16∶10∶3∶83.9630.265Sk03285/307301/X285/301∶285/X∶301/307∶307/X1∶1∶1∶15∶11∶7∶18.6670.034IISk04254/254262/274254/262∶254/2741∶18∶173.2400.072Sk05449/449449/449449/449127--IIISk07344/348362/X344/362∶344/X∶348/362∶348/X1∶1∶1∶16∶10∶6∶42.9230.404Sk08270/X284/X270/284∶270/X∶284/X∶XX1∶1∶1∶19∶4∶12∶26.4480.092Sk09457/457425/425457/425124--IVSk14434/514428/428428/434∶428/5141∶112∶140.1540.695Sk15265/X245/X245/265∶265/X∶245/X∶X/X1∶1∶1∶15∶4∶9∶93.0740.380
注:X代表无效等位基因;*代表基因型分离比偏离孟德尔分离定律(P<0.01)。
Note: X, Null allele; *, Genotypic ratios that are not in agreement with Mendelian segregation (P<0.01).
①Family; ②Multiplex panel; ③Locus; ④Dam; ⑤Sire; ⑥Offspring; ⑦Expected segregation ratio; ⑧Observed segregation ratio; ⑨Pvalue
2.3 多元PCR体系的家系鉴定成功率
在5个毛蚶单对交配家系中,4组微卫星多元PCR鉴定结果如图1所示。CERVUS 3.0软件分析结果显示,只使用1组多态性信息含量最高的多元PCR(Panel III),其家系模拟和实际的鉴定结果成功率分别为78%和72%。使用3组以上的多元PCR组合时,子代的亲本鉴定成功率可达100%。

(鉴定成功置信水平为95%。95% confidence interval.)
3 讨论
微卫星标记是一种多态性丰富的共显性标记。根据Bostein等[24]所提出的多态信息含量指标范围,当PIC>0.5时,为高度多态基因位点,PIC<0.25时,则为低多态基因位点。本研究所使用的4组多元PCR体系,位点的PIC均大于0.7,属于高多态基因位点。在实际应用中,选择的标记具有较高的多态性,对于家系分析效率具有重要意义,因为这意味着在位点数目相同的情况下,可以获得更多的遗传信息,从而提高分析结果的准确度[25]。Norris等[26]研究发现,在全部的15个标记中使用其中8个多态性最高位点(平均期望杂合度0.85)获得成功鉴定的子代比例,仅比使用全部标记降低了2%,这样可以大量节省分析时间和基因分型成本。通常,家系鉴定所需要的位点数量,取决于标记的总体信息量,排除能力的高低以及亲本和子代的数量。Bulter等[27]根据实际和模拟数据的计算结果,建议家系分析中应该至少使用6~8个标记。在本研究中,使用2组多元PCR组合,就可达到95%以上的实际鉴定成功率。这可能是与组成这4组多元PCR的10个微卫星标记具有较高的多态信息含量,且家系亲本规模较为有限(5个父本和5个母本)有关。因此,在大批量的家系材料分析中,可能需要更多的标记位点,才能使家系鉴定成功率达到满意的效果。
子代基因型的分离模式显示了本研究中的一些位点存在无效等位基因。无效等位基因是指不能被PCR扩增的等位基因,微卫星标记中存在无效等位基因是一个较普遍的现象,尤其在海洋贝类[28]。本研究结果表明,10个微卫星位点在5个全同胞家系中无效等位基因频率为9.5%,这与其他海洋贝类微卫星标记的研究结果相类似。例如:聂鸿涛等[29]研究结果显示,在皱纹盘鲍12个家系中,12个微卫星标记的无效等位基因频率为10.7%;Li等[30]开发的12个太平洋牡蛎EST微卫星标记中,无效等位基因频率为4.9%;而太平洋牡蛎的非编码区基因组微卫星标记,无效等位基因的频率为11%~22%[31-32]。一般来说,来自EST中的微卫星标记,无效等位基因频率通常会小于来自基因组中的微卫星[33]。偏分离现象也是使用微卫星标记进行海洋贝类家系分析研究中经常会遇到的情况。一般来说,偏分离的产生主要由两方面原因造成,即无效等位基因的存在和合子存活力的差异[28]。本研究结果表明,在考虑无效等位基因的情况下,仍有2个位点的基因型分离偏离孟德尔分离比。这种偏分离情况,可能的原因是基因转换、减数分裂过程中染色体非随机分离,以及同致死的主效基因连锁等形成[15]。
综上所述,微卫星标记是较为理想的分子标记,在水产动物研究的很多领域都有广泛的应用。本研究将开发的微卫星标记多元PCR体系应用于毛蚶的家系鉴定分析,获得较高的家系鉴定成功率,对于毛蚶的遗传多样性分析、遗传育种研究以及种质资源保护具有重要意义。
[1]马云聪. 毛蚶育苗养殖实用技术[M]. 北京: 海洋出版社, 2008: 1-2. Ma Y C. Practical Technologies for Ark Shell (Scapharcakagoshimensis) Seed Breeding [M]. Beijing: China Ocean Press, 2008: 1-2.
[2]边绍新. 丰南区浅海养殖毛蚶现状及发展设想[J]. 河北渔业, 2004, 3: 21-22.
Bian S X. Status and development plan for inshore culture of ark shell (Scapharcakagoshimensis) in Fengnan district [J]. Hebei Fisheries, 2004, 3: 21-22.
[3]付卓, 朱守维. 锦州浅海毛蚶底播增养殖成效调查[J]. 河北渔业, 2008, 2: 40-42.
Fu Z, Zhu S W. Investigation of bottom sowing effects of ark shell (Scapharcakagoshimensis) in Jinzhou city [J]. Hebei Fisheries, 2008, 2: 40-42.
[4]任玉水, 赵炳然. 莱州湾毛蚶苗种健康培育技术初探[J]. 中国水产, 2014, 3: 55-57.
Ren Y S, Zhao B R. Preliminary study on heathy farming of ark shell (Scapharcakagoshimensis) seedlings [J]. China Fisheries Monthly, 2014, 3: 55-57.
[5]Waples R S, Punt A E, Cope J M. Integrating genetic data into management of marine resources: How can we do it better? [J]. Fish and Fisheries, 2008, 9: 423-449.
[6]Palumbi S R. Marine reserves and ocean neighborhoods: The spatial scale of marine populations and their management [J]. Annual Review of Environment and Resources, 2004, 29: 31-68.
[7]Haig S M. Molecular contributions to conservation [J]. Ecology, 1998, 79: 413-425.
[8]Ni L H, Li Q, Kong L F. Microsatellites reveal fine-scale genetic structure of the Chinese surf clamMactrachinensis(Mollusca, Bivalvia, Mactridae) in Northern China [J]. Marine Ecology, 2011, 32: 488-497.
[9]Selkoe K A, Henzler C M, Gaines S D. Seascape genetics and the spatial ecology of marine populations [J]. Fish and Fisheries, 2008, 9: 363-377.
[10]Li R H, Li Q, Wang C L. Sibship reconstruction and effective population size estimation in mass spawning ark shell,Scapharcabroughtoniibased on microsatellite analysis [J]. Genes & Genomics, 2013, 35: 703-708.
[11]Jones O R, Wang J. Molecular marker-based pedigrees for animal conservation biologists [J]. Animal Conservation, 2010, 13: 26-34.
[12]Li Q, Yu H, Yu R H. Genetic variability assessed by microsatellites in cultured populations of the Pacific oyster (Crassostreagigas) in China [J]. Aquaculture, 2006, 259: 95-102.
[13]Sunnucks P. Efficient genetic markers for population biology [J]. Trends in Ecology & Evolution, 2000, 15: 199-203.
[14]Guichoux E, Lagache L, Wagner S, et al. Current trends in microsatellite genotyping [J]. Molecular Ecology Resources, 2011, 11: 591-611.
[15]Li Q, Park C, Kobayashi T, et al. Inheritance of microsatellite DNA markers in the Pacific abaloneHaliotisdiscushannai[J]. Marine Biotechnology, 2003, 5: 331-338.
[16]Li Q, Zheng X D, Yu R H. Inheritance mode of microsatellite loci and their use for kinship analysis in the Pacific oyster (Crassostreagigas) [J]. Chinese Journal of Oceanology and Limnology, 2008, 26: 256-262.
[17]Li Q, Park C, Kijima A. Allelic transmission of microsatellites and application to kinship analysis in newly hatched Pacific abalone larvae [J]. Fisheries Science, 2003, 69: 883-889.
[18]Hellberg M E. Gene flow and isolation among populations of marine animals [J]. Annual Review of Ecology Evolution and Systematics, 2009, 40: 291-310.
[19]Feng Y W, Li Q, Kong L F. Isolation and characterization of 14 polymorphic microsatellite loci in the ark shellScapharcasubcrenata(Bivalvia: Arcidae) [J]. Conservation Genetics, 2009, 10: 1125-1127.
[20]Chen C, Li Q, Yu H, et al. Development and multiplex genotyping of 20 microsatellite markers in the ark shellScapharcakagoshimensis(Mollusca: Bivalvia)[J]. Conservation Genetics Resources, 2015, 7: 917-944.
[21]Li Q, Park C, Kijima A. Isolation and characterization of microsatellite loci in the Pacific abalone,Haliotisdiscushannai[J]. Journal of Shellfish Research, 2002, 69: 883-889.
[22]Kalinowski S T, Taper M L, Marshall T C. Revising how the computer program CERVUS accommodates genotyping error increases success in paternity assignment[J]. Molecular Ecology, 2007, 16: 1099-1106.
[23]Goudet J. FSTAT, a program to estimate and test gene diversities and fixation indices (version 2.9.3)[EB/OL]. [2015-04-20].http://www2.unil.ch/popgen/softwares/fstat. htm. Updated from Gouclet.
[24]Botstein D, Whiter R L, Skolnick M, et al. Construction of a genetic linkage map in man using restriction fragment length polymorphisms [J]. American Journal of Human Genetics, 1980, 32(3): 314-331.
[25]Pearse D E, Crandall K A. BeyondFST: Analysis of population genetic data for conservation [J]. Conservation Genetics, 2004, 5: 585-602.
[26]Norris A T, Bradley D G, Cunningham E P. Parentage and relatedness determination in farmed Atlantic salmon (Salmosalar) using microsatellite markers [J]. Aquaculture, 2000, 182: 73-83.
[27]Butler K, Field C, Herbinger C M, et al. Accuracy, efficiency and robustness of four algorithms allowing full sibship reconstruction from DNA marker data [J]. Molecular Ecology, 2004, 13: 1589-1600.
[28]Reece K S, Ribeiro W L, Gaffney P M, et al. Microsatellite marker development and analysis in the eastern oyster (Crassostreavirginica): Confirmation of null alleles and non-mendelian segregation ratios [J]. Journal of Heredity, 2004, 95: 346-352.
[29]聂鸿涛, 李琪, 孔令锋. 皱纹盘鲍微卫星多重PCR体系构建及其在家系鉴定中的应用[J]. 水产学报, 2013, 37: 207-215.
Nie H T, Li Q, Kong L F. Development of four multiplex PCR panels of microsatellites and application to kinship analysis in the Pacifica balone (Haliotisdiscushannai) [J]. Journal of Fisheries of China, 2013, 37: 207-215.
[30]Li R H, Li Q, Cornette F, et al. Development of four EST-SSR multiplex PCRs in the Pacific oyster (Crassostreagigas) and their validation in parentage assignment [J]. Aquaculture, 2010, 310: 234-239.
[31]Li L, Guo X M, Zhang G F. Inheritance of 15 microsatellites in the Pacific oysterCrassostreagigas: segregation and null allele identification for linkage analysis [J]. Chinese Journal of Oceanology and Limnology, 2009: 74-79.
[32]Mcgoldrick D J, Hedgecock D, English L J, et al. The transmission of microsatellite alleles in australian and north american stocks of the pacific oyster (Crassostreagigas): selection and null alleles [J]. Journal of Shellfish Research, 2000, 19: 779-788.
[33]Li Q, Liu S K, Kong L F. Microsatellites within genes and ESTs of the Pacific oysterCrassostreagigasand their transferability in five otherCrassostreaspecies [J]. Electronic Journal of Biotechnology, 2009, 12: 1-6.
责任编辑朱宝象
Parentage Analysis in the Ark Shell (Scapharca kagoshimensis)Based on Microsatellite Multiplex PCRs
CHEN Chen, LI Qi, KONG Ling-Feng, YU Hong
(The Key Laboratory of Mariculture, Ministry of Education, Ocean University of China, Qingdao 266003, China)
Ark shell (Scapharcakagoshimensis) is a commercially important but dwindling natural resource bivalve species in the shallow coastal waters of the northwestern Pacific. In order to protect and exploit the resource of the species effectively, the inheritance mode of 10 microsatellite markers, which were pooled into 4 multiplex PCR panels, was investigated in newly hatchedS.kagoshimensislarvae from five full-sib families, and the feasibility of these markers for kinship estimation was also examined. The result showed that all of these loci were highly polymorphic. The average polymorphism information content was 0.809. The frequency of null alleles was estimated 9.5%. Two loci (Sk04, Sk05) showed deviation from Mendelian segregation in a family (F3) even though null alleles were considered. Parentage analysis showed that with the most informative multiplex set (Panel III), the simulated and real assignment success could be 78% and 72%, respectively, and 100% of the offspring were correctly allocated to their parents when three or more multiplex PCR panels were used. Our results showed that the four multiplex PCR panels of microsatellites are possible and can be used for rapid and highly efficient parentage assignment inS.kagoshimensis.
Scapharcakagoshimensis; microsatellite locus; multiplex PCR; parentage assignment
海洋公益性行业科研专项(201205023);教育部博士点基金项目(20130132110009)资助
2015-05-04;
2015-07-30
陈辰(1987-),男,博士生。E-mail: chenchen3729@163.com
❋❋通讯作者:E-mail:qili66@ouc.edu.cn
Q347
A
1672-5174(2016)09-018-06
10.16441/j.cnki.hdxb.20150171
Supported by the Grants from National Marine Public Welfare Research Program (201205023); Doctoral Program of Ministry of Education of China (20130132110009)
