企鹅珍珠贝Creb2基因的克隆及表达分析
2016-08-23于非非余祥勇潘珍妮宋娜娜王梅芳
于非非,余祥勇,潘珍妮,宋娜娜,王梅芳
(广东海洋大学 水产学院,广东 湛江 524088)
企鹅珍珠贝Creb2基因的克隆及表达分析
于非非,余祥勇,潘珍妮,宋娜娜,王梅芳
(广东海洋大学 水产学院,广东 湛江 524088)
CREB(cAMP response element binding protein)是一种真核生物中广泛存在的调控因子。为了探讨企鹅珍珠贝(Pteria penguin)Creb基因的序列和表达特征,为进一步阐述Creb的生理功能提供科学依据,本研究利用RACE-PCR技术克隆到企鹅珍珠贝一个Creb基因的全长序列,分析其理化性质和进化地位;利用荧光定量PCR分析Creb基因在各组织中的表达特征。结果表明,该企鹅珍珠贝Creb基因的cDNA全长为1484 bp,其中开放阅读框(ORF)为1071 bp,编码356个氨基酸,5′非编码区为218 bp,3′非编码区为 195 bp,C端包含一个碱性亮氨酸拉链结构(basic region leucin zipper,bZIP)。预测其分子质量为39.49 kD,等电点为4.43。序列比对显示该企鹅珍珠贝Creb基因与欧洲平牡蛎(Ostrea edulis)和太平洋牡蛎(Crassostrea gigas)的Creb2同源性(identity)最高,分别为46.3%和46.1%,故命名为ppCreb2;系统进化树分析结果与传统形态学分类结果相吻合。荧光定量PCR分析显示,ppCreb2在企鹅珍珠贝的各个组织中组成性表达,其中在足中表达量最高,鳃中其次。
企鹅珍珠贝;ppCreb2;基因克隆;表达分析;荧光定量PCR
doi:10.11759/hykx20150824001
CREB(cAMP response element binding protein)即环磷酸腺苷反应元件结合蛋白,是真核生物中广泛存在的一种细胞核内调控因子,通过自身磷酸化实现转录功能的调控[1]。CREB家族蛋白的C端都包含大量碱性氨基酸和一个亮氨酸拉链,被称为碱性亮氨酸拉链结构域 (basic region leucin zipper,bZIP)[2],这一结构域的DNA亲和力很高,能以二聚体的形式与靶基因启动子中的CRE(cAMP response element)位点结合,调控下游多个基因的转录活性,是CREB家族蛋白最保守的区域。作为一种重要的转录调控因子,CREB家族蛋白参与了细胞的增殖[3]、分化[4]和凋亡[5],学习记忆[6],精子发生[7]和肤色决定[8]等多种复杂的生命活动。根据结构和功能的不同,CREB家族被划分为CREB1、CREB2、CREB3和CREB3L4(CREB3-like4)四个亚族。近年来,已在节肢动物[9]、腔肠动物[10]、鱼类[11]、鸟类[12]和哺乳动物[7]等几十个物种中克隆到Creb基因,但在双壳类中研究较少[13]。
企鹅珍珠贝(Pteria penguin)属于软体动物门(Mollusca),瓣鳃纲(Lamellibranchia),翼形亚纲(Ptermorphia),珍珠贝目(Pterioida),珍珠贝科(Pteriidae)[14]。企鹅珍珠贝个体大、生长快、成活率高,其突出特点是分泌珍珠质机能旺盛、珠层厚、色泽奇异,是我国目前唯一能规模养殖的大型珍珠贝,被认为是最具有“振兴南珠”潜能的品种[15]。作为一种调控因子的Creb基因是否也存在于企鹅珍珠贝中,是否参与了企鹅珍珠贝重要生命活动的调控是值得探索的问题。
本研究建立了企鹅珍珠贝全组织的转录组数据库,利用RACE-PCR技术克隆到企鹅珍珠贝Creb2基因的cDNA全长序列,构建系统进化树分析该基因的进化地位;利用荧光定量PCR技术分析Creb2基因在企鹅珍珠贝各组织中的表达差异,旨在为进一步阐述Creb2在企鹅珍珠贝各种生理过程中的作用提供理论依据。
1 材料与方法
1.1 材料
实验所用企鹅珍珠贝采自广东省湛江市雷州市流沙湾养殖群体,体重300~350 g。
1.2 主要试剂
实验所用主要试剂包括SMARTer®RACE 5′/3′Kit (Clontech)、 UNlQ-10柱式Trizol总RNA抽提试剂盒(上海生工)、 RevertAid First Strand cDNA Synthesis Kit(Thermo)、 E.Z.N.A Gel Extraction Kit试剂盒(Omega)、SYBR Select Master Mix(Life technologies)、LA taq(Takara)、PMD18-T(Takara)、琼脂糖(Agarose)等。DEPC、氨苄青霉素等其他试剂为国产分析纯,DH5α感受态细胞购自全式金。
1.3 企鹅珍珠贝Creb全长cDNA的克隆
分别提取企鹅珍珠贝的外套膜、鳃、闭壳肌、消化盲囊、足、精巢、卵巢的RNA,将各组织RNA混合后应用Illumina测序技术进行转录组测序,建立了企鹅珍珠贝全组织的转录组数据库,并进行生物信息学分析。
利用企鹅珍珠贝各组织混合 R N A,按照SMARTer®RACE 5′/3′Kit说明书合成企鹅珍珠贝Creb的3′RACE模板和5′RACE模板。根据企鹅珍珠贝转录组数据库中注释为Creb的unigene序列设计企鹅珍珠贝Creb基因3′RACE的特异性引物Creb-3-F和5′RACE的特异性引物Creb-5-R(表1),3′RACE和5′RACE PCR反应体系为2×SeqAmp Buffer 25 μL,SeqAmp DNA Polymerase 1 μL,5′RACE或3′RACE cDNA模板(100 ng/µL)2.5 μL,10×UPM(Universal Primer Mix,0.2µmol/L)5 μL(表1),Creb-3-F或Creb-5-R(10µmol/L)1 μL,PCR-Grade H2O 15.5 μL。使用touch-down PCR程序扩增:94℃30 s,72℃2 min,5个循环;94℃30 s,70℃30 s,72℃ 2 min,5个循环;94℃30 s,68℃30 s,72℃2 min,25个循环。PCR产物经电泳分离、纯化,与RACE试剂盒中的 Lineareized pRACE vector连接,转化进DH5α感受态细胞中培养过夜,筛选阳性克隆进行测序。经测序后3′RACE序列和5′RACE序列进行拼接,并利用 VecScreen软件(http://www.ncbi.nlm.nih.gov/VecScreen/VecScreen.html)去除载体序列,获得企鹅珍珠贝Creb基因的电子全长。
根据Creb基因的电子全长,设计特异引物Creb-cDNA-F和Creb-cDNA-R(表1),利用LA taq酶克隆Creb基因全长cDNA。PCR产物与PMD18-T载体连接、转化、培养,筛选阳性克隆测序以验证电子全长序列的正确性。
1.4 目的基因的生物信息学分析
通过ORF Finder工具,推导企鹅珍珠贝Creb基因编码的氨基酸序列;通过 Expasy网站(http:// web.expasy.org/compute_pi/)分析其相对分子质量和等电点;经ProtScale软件分析氨基酸序列的疏水性;经SignalP 4.1、TMHMM和NetOGlyc 4.0分别预测其信号肽、跨膜结构及糖基化位点;经SMART对其进行结构预测和功能分析;利用Clustal X(1.8)软件将获得的企鹅珍珠贝Creb基因与其他物种的Creb基因编码氨基酸进行同源比对,用MEGA6软件构建系统进化树。
1.5 荧光定量PCR检测目的基因的组织表达
分别提取企鹅珍珠贝外套膜、鳃、闭壳肌、消化盲囊、足、精巢和卵巢的RNA,反转录合成第一链cDNA,进行荧光定量PCR检测。以β-actin基因作为内参,以企鹅珍珠贝的闭壳肌为对照,采用2-ΔΔCt法计算Creb基因在不同组织中的表达水平,每组设3个重复,组间重复性和差异显著性用SPSS16.0分析。
2 结果与分析
2.1 企鹅珍珠贝Creb基因的克隆和序列分析
采用5′RACE技术获得了1169 bp的Creb 5′序列,采用3′RACE技术获得了678 bp的Creb 3′序列,拼接后获得1484 bp的Creb全长序列。利用CrebcDNA-F和Creb-cDNA-R引物进行PCR验证,获得1328 bp序列,与我们拼接的Creb基因全长序列相吻合。企鹅珍珠贝Creb基因cDNA全长为1484 bp,其中开放阅读框(ORF)为1071 bp,编码356个氨基酸,5′非编码区为218 bp,3′非编码区为 195 bp,包含30 bp 的polyA尾巴,如图1所示。登录号为KT388104。

图1 企鹅珍珠贝Creb基因序列分析Fig.1 Creb cDNA and deduced amino acid sequences of Pteria penguin小写字母代表非编码序列(UTR);大写字母代表开放阅读框(ORF),灰色框代表碱性亮氨酸拉链结构域(276~341 bp)5′UTR and 3′UTR regions are shown in small letters;open reading fragment is shown in capital letters;basic region leucin zipper(bZIP)is shown in gray box(276-341 bp)
2.2 CREB蛋白理化性质预测和分析
经Expasy预测企鹅珍珠贝CREB蛋白分子质量为39.49 kD,等电点为4.43。ProtScale分析疏水性显示,该蛋白的疏水指数从-3.678至2.2,属于亲水性蛋白,约在第164位表现出最强的疏水性,第298位表现出较出最强的亲水性(图2)。经TMHMM 2.0进行跨膜结构域预测,显示没有跨膜区。SignalP4.1预测其不存在信号肽,属于非分泌型蛋白。NetOGlyc4.0分析发现其有3个糖基化位点,分别是第22位、第26位和第108位。SMART软件分析发现,企鹅珍珠贝CREB蛋白的N端富含天冬氨酸(Asp,D)和谷氨酸(Glu,E)等酸性氨基酸,C端富含赖氨酸(Lys,K)和精氨酸(Arg,R)等碱性氨基酸,包含一个典型的碱性亮氨酸拉链结构域(basic region leucin zipper,bZIP)(图1和图3)。

图2 企鹅珍珠贝CREB蛋白的疏水性预测Fig.2 Hydrophobicity prediction of CREB in Pteria penguin“+”代表疏水值,“-”代表亲水值+hydrophobicity;-hydrophobicity

图3 企鹅珍珠贝CREB蛋白质结构预测Fig.3 Prediction of CREB structure in Pteria penguin
2.3 CREB氨基酸序列同源性分析
采用Clustal X软件将企鹅珍珠贝的Creb基因与其他物种的Creb基因编码氨基酸进行多序列比对,结果显示,该基因与欧洲平牡蛎(Ostrea edulis,ADG85255.1)和近江牡蛎(Crassostrea ariakensis,BQ59345.1))的Creb2基因同源性(identity)最高,分别为46.3%和46.1%;与人(Homo sapiens,AAA52071.1)、小鼠(Mus musculus,NP_033846.2)和墨西哥脂鲤(Astyanax mexicanus,XP_007231405.1)的Creb2基因同源性其次,分别为为26.3%、25.7%和24.6%。其中,保守位点主要集中在碱性亮氨酸拉链结构域(图4)。因此将该企鹅珍珠贝Creb基因命名为ppCreb2。
分别选取近江牡蛎、欧洲平牡蛎、光滑双脐螺(Biomphalaria glabrata,ACZ51334.1)creb、静水椎实螺(Lymnaea stagnalis,BAC21159.1)、海兔(Aplysia californica,NP_001191630.1)和墨西哥脂鲤等物种的Creb2基因编码的氨基酸进行聚类分析,如图 5 Neighbor-joining(NJ)树所示:企鹅珍珠贝、太平洋牡蛎和欧洲平牡蛎都属于软体动物瓣鳃纲,其Creb2编码氨基酸聚为一个小的进化分支;光滑双脐螺、静水椎实螺和海兔属于软体动物腹足纲,聚为一个小的进化分支;以上所有的软体动物聚为一个大的进化分支,而墨西哥脂鲤这一外群物种单独列为一个进化分支。CREB2聚类结果与传统的进化分类学相吻合,印证了双壳类瓣鳃纲和腹足纲动物的亲缘关系。


图4 企鹅珍珠贝与其他物种Creb2基因编码氨基酸比对Fig.4 Amino acid alignment of Creb2 in Pteria penguin and other homologues相同的氨基酸以涂黑表示The amino acids showing100%identity are marked with black box

图5 企鹅珍珠贝与其他物种Creb2氨基酸序列的系统进化树(NJ)Fig.5 Phylogenetic tree of Creb2 from Pteria penguin and other homologues
2.4 Creb2基因的组织定量分析
采用荧光定量PCR分析Creb2基因在企鹅珍珠贝各个组织中的表达水平,结果显示(图6),从RNA水平看,ppCreb2基因在企鹅珍珠贝各个组织中都有表达,但表达量有所差异。在足中表达量最高,其次是鳃,在外套膜、闭壳肌、精巢和卵巢中的表达量较低。
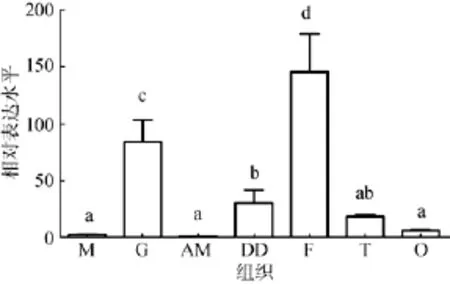
图6 ppCreb基因在企鹅珍珠贝各组织的表达水平Fig.6 Expression of ppCreb in different tissues of Pteria penguinM.外套膜;G.鳃;AM.闭壳肌;DD.消化盲囊;F.足;T.精巢;O.卵巢M:mantle;G:gill;AM:adductormuscle;DD:digestive diverticulum;F:foot;T:testis;O:ovary
3 讨论
1986年,Montrminy等[1]发现许多基因都受cAMP的调控,这些基因的共同特点是启动子和增强子序列中含有一段高度保守的5′-TGACGTCA-3′序列,并命名为cAMP反应元件(cAMP response element,CRE)。随后,他们又分离纯化到一种特异性结合CRE的核蛋白,命名为cAMP应答元件结合蛋白(cAMP response element binding protein)即CREB蛋白[16]。CREB蛋白家族主要包括CREB1、CREB2、CREB3和CREB3L4四个成员。CREB2(cyclic AMP response element binding protein 2),也称ATF4(activating transcription factor 4),是内质网应激通路中的信号转导蛋白,由PERK/elF2α通路中磷酸化的elF2α激活[17]。
本研究从企鹅珍珠贝cDNA文库中克隆了Creb基因全长序列。同源性分析显示,企鹅珍珠贝Creb基因与欧洲平牡蛎Creb2基因具有最高的同源性,为46.3%。结构分析发现,企鹅珍珠贝Creb基因编码氨基酸分为N端和C端两个结构域,其N端富含酸性氨基酸,为酸性激活区域;C端为保守的碱性亮氨酸结构域。不含KID区(激酶诱导结构域),为非跨膜蛋白,这些特点与大多数动物经典的CREB2蛋白结构相似[18-19]。其中N端主要行使转录激活功能,C端主要行使DNA结合和蛋白二聚体化的功能。因此,我们将该企鹅珍珠贝Creb基因鉴定为ppCreb2。这说明Creb2基因在各个物种中是相对保守的,也暗示了ppCreb2基因在贝类的生命活动中起到重要作用。
作为一种重要的转录因子,CREB2参与了多种生命过程。对海兔和果蝇的研究发现,CREB2可抑制记忆贮存和突出可塑性,是一种记忆抑制基因[20-21]。CREB2参与了小鼠造骨细胞早期分化和末期分化,对维持造骨细胞的分化和功能至关重要,CREB2缺陷小鼠出现小眼症状表型[4]。CREB2也可参与内质网应激反应(endoplasmic reticulum stress),调节内质网内稳态体系[22]。还可与多种病毒蛋白相互作用,调节与之结合的病毒蛋白的表达水平[23]。
与CREB2广泛的生理功能相一致的,Creb2在各个组织中普遍表达。对猪的研究发现,Creb2在白色脂肪组织、胃、肾肺、小脑、肝脏和淋巴组织中表达量较高,在心脏和肌肉中表达量较低[24]。我们的研究发现,ppCreb2基因在企鹅珍珠贝足中表达量最高,这可能与Creb2在神经系统中大量表达相一致[20]。作为进化地位比较低的软体动物,其神经系统主要由4对神经节和与之联络的神经构成,其中重要的一对神经节足神经节1就位于足的前部[25]。另外,ppCreb2基因在鳃中表达量也很高。贝类通过鳃直接与水环境接触,营呼吸功能,接触大量病毒和细菌,在鳃中大量表达印证了CREB2蛋白可与多种病毒蛋白相互作用这一功能。因此,我们推测CREB2在企鹅珍珠贝多种生命活动中起到重要作用。
[1] Montrminy M R,Sevarino K A,Wag-ner J A,et al.Identification of a cyclic-AMP responsive element within the rat somatostatin gene[J].Proceedings of the Natural Academic of Science of the United Stated America,1986,83(18):6682-6686.
[2] Shanware N P,ZhanL,HutchinsonJ A,etal.Conserved and distince modes of CREB/ATF transcription factor regulation by PP2A/B56 gamma and genotoxic stress[J].Plos One,2010,5(8):e12173.
[3] Garcia,G E,Nicole A,Bhasharan S,et al.Akt and CREB-mediated prostate cancer cell proliferation inhibition by Nexrutine,a Phellodendron amurense extract[J].Neoplasia,2006,8(6):523-533.
[4] Yang X, Karsenty G. ATR4, the osteoblast accumulation of which is determined post-translationally,can induce osteoblast-specific gene expression in non-osteoblastic cells[J].The Journal of Biological Chemistry,2004,279(45):47109-47114.
[5] Persengiev S P,Devireddy L R,Green M R.Inhibition of apoptosis by ATFx:a novel role for a member of the ATF/CREB familiy of mammalian bZIP transcription factors[J].Genes&Development,2002,16(14):1806-1814.
[6] Kida S,Serita T.Functional roles of CREB as a positive regulator in the formation and enhancement of memory[J].Brain Research Bulletin,2014,105:17-24.
[7] Gomez M, Manzano A, Figueras A, et al.Sertoli-secreted FGF-2 induces PFKFB4 isozyme expression in mouse spermatogenic cells by activation of the MEK/ERK/CREB pathway[J].American journal of Physiology Endocrinology and Metabolism,2012,303(6):E695-707.
[8] Wan P,Hu Y Q,Li H.Regulation of melanocyte pivotal transcription factor MITF by some other transcription factors[J].Molecular and Cellular Biochemistry,2011,354(1-2):241-246.
[9] Lijima K,Zhao L,Shenton C,et al.Regulation of energy stores and feeding by neuronal and peripheral CREB activity in Drosophila[J].PLoS One,2009,4(12):e8498.
[10]Kaloulis K,Chera S,Hassel M,et al.Reactivation of developmental programs: the cAMP-reponse element-binding protein pathway is involved in hydra head regeneration [J].Proceedings ofthe Natural Academic of Science of the United Stated America,2004,101(8):2363-2368.
[11]Rajan K E,Thangaleela S,Balasundaram C.Spatial learning associated with stimulus response in goldfish Carassius auratus:relationship to activation of CREB signalling[J].Fish Physiology and Biochemistry,2015,41(3):685-694.
[12]Guo F,ZhangY,SuL,etal.Breed-dependent transcriptional regulation of phosphoenolpyruvate carboxylase,cytosolic form,expression in the liver of broiler chickens[J].Poultry Science,2013,92(10):2737-2744.
[13]Zhang S M,Coultas K A.Identification and characterization of five transcription factors that are associated with evolutionarily conserved immune signaling pathway s in the schistosome-transmitting snail Biomphalaria glavrata[J].Molecular Immunology,2011,48(15-16):1868-1881.
[14]蔡英亚,张英,魏若飞.贝类学概论[M].上海:上海科学技术出版社,1995:209-210.
Cai Yingya,Zhang Ying,Wei Ruofei.Malacology[M].Shanghai:Shanghai Science and Technology Press,1995:209-210.
[15]毛勇,梁飞龙,余祥勇,等.企鹅珍珠贝游离珠插核效果的初步观察[J].海洋科学,2003,27(11):1-4.
Mao Yong,Liang Feilong,Yu Xiangyong,etal.Preliminary observations on implanation efficiency of cultured round pearls with pearl oyster,Pteria (Magnavicula)penguin roding[J].Marine Sciences,2003,27(11):1-4.
[16]Montrminy M R,Bilezikjan L M.Binding of a nuclear protein to the cycli c-AMP response element of the somatost atin gene[J].Nature,1987,328(6126):175-178.
[17]Ameri K,Harris A L.Activating transcription factor 4[J].Int J Biochem Cell Biol,2008,40(1):14-21.
[18]Qi M,Lei T,Zhou L,et al.Cloning,characterization,chromosomal mapping and tissue transcription analysis of porcine CREB2 and CREB3 genes[J].Folia Biologica,2009,55(4):137-144.
[19]Endo M,Su L,Nielsen T O.Activating transcription factor 2 in mesenchymal tumors[J].Human Pathology,2014,45(2):276-284.
[20]Mohamed,H A,Yao W,Fioravante D,et al.cAMP-response elements in Aplysia crebl,creb2,and Ap-uch promoters:implications for feedback long term memory[J].The Journal of Biological Chemistry,2005,280(29):27035-27043.
[21]Dubnau J,Tully T.Gene discovery in Drosophila:new insights for learning and memory[J].Annual review of Neuroscience,1998,21:407-444.
[22]Lu P D,Harding H P,Ron D.Translation reinitiation at alternative open reading frames regulates gene expression in an integrated stress response[J].The Journal of Cell Biology,2004,167(1):27-33.
[23]Lim C,Sohn H,Gwack Y,et al.Latency-associated nuclear antigen of Kaposi's sarcoma-associated herpesvirus(human herpesvirus-8)binds ATF4/CREB2 and inhibits its transcriptional activation activity[J].The Journal of General Virology,2000,81(pt11):2645-2652.
[24]祁艳梅.猪CREB家族基因的克隆及表达研究的初步探讨[D].武汉:华中农业大学,2009.
QiYanmei.Cloning and preliminary research of porcine CREBs family[D]. Wuhan: HuaZhong Agricultural University,2009.
[25]李霞.水产动物组织胚胎学[M].北京:中国农业出版社,2005:82-83.
LiXia.AquaticAnimalEmbryology[M].Beijing:China Agriculture Press,2005:82-83.
(本文编辑:康亦兼)
Molecular cloning and expression analysis of Creb2 gene from Pteria penguin
YU Fei-fei,YU Xiang-yong,PAN Zhen-ni,SONG Na-na,WANG Mei-fang
(Fisheries College,Guangdong Ocean University,Zhanjiang 524088,China)
Aug.22,2015
Pteria penguin;ppCreb2;Gene cloning;expression analysis;real-time PCR
In this study,in order to analyze the sequence and expression characteristics of the cAMP response element binding protein(Creb)gene in the Pteria penguin,we characterized the full-length cDNA of the Creb gene from the Pteria penguin by the rapid amplification of cDNA ends(RACE)-polymerase chain reaction(PCR).We then used real-time PCR to examine the expression of Creb in different tissues.The results show that the full-length cDNA of Creb was 1484 bp,including a 5'UTR of 218 bp,a 3'UTR of 195 bp,and an open-reading frame of 1071 bp,which encodes a deduced protein of 356 amino acids.The C-terminal region contained a conserved basic region leucin zipper(bZIP).The predicted molecular weight was 39.49 kD,and the isoelectric point was 4.43.The sequence comparison showed that Creb in the Pteria penguin(ppCreb2)shares 46.3%and 46.1%sequence identities with Creb2 in Ostrea edulis and Crassostrea gigas,respectively.The phylogenetic analysis results were consistent with traditional taxonomic analysis.The real-time PCR showed that ppCreb2 was constitutively expressed in all studied tissues(mantle,gill,adductor muscle,digestive diverticulum,foot,testis,and ovary),with higher levels in the foot and gill.
Q341
A
1000-3096(2016)05-0029-07
2015-08-22;
2015-10-20
广东海洋大学优秀青年骨干教师培养项目(20140040);广东海洋大学博士启动基金(E15041);广东省海洋渔业科技推广专项项目(A201308A11)
[Foundation:the Outstanding Young Teacher Foundation of Guangdong Ocean University,No.20140040;Doctoral research project of Guangdong Ocean University,No.E15041;the Technology Extension Foundation of Marine Fishery in Guangdong Province,No.A201308A11]
于非非(1980-),女,山东威海人,讲师,博士,从事贝类分子遗传育种的研究,电话:0759-2383504,E-mail:yufeifei2000@163.com;王梅芳,通信作者,教授,E-mail:pearlang@126.com
