兔粪中微生物区系ERIC-PCR DNA指纹图谱的建立及分析
2016-07-11李云飞秦翠丽王忠泽杨若岚
李云飞,秦翠丽,王忠泽,杨若岚,李 洋
(河南科技大学 食品与生物工程学院,洛阳 471023)
兔粪中微生物区系ERIC-PCR DNA指纹图谱的建立及分析
李云飞,秦翠丽*,王忠泽,杨若岚,李洋
(河南科技大学 食品与生物工程学院,洛阳 471023)
摘要:本研究旨在将ERIC-PCR基因指纹图谱技术应用于兔粪微生物的分析中,建立一种快速分析检测兔肠道菌群变化的方法。通过对PCR反应条件的优化确定适合兔粪微生物的ERIC-PCR反应条件,对比兔粪微生物组和兔粪优势菌的ERIC-PCR DNA指纹图谱差异,从而分析兔粪微生物ERIC指纹图谱的特征。结果建立了适合兔粪样品 ERIC-PCR 分析的反应体系,确定450 bp处条带为双歧杆菌特征条带,1 300和4 000 bp处为大肠杆菌特征条带;经发酵验证,图谱中的相应条带亮度变化趋势与计数平板所测结果相一致(相对亮度R与相对菌落数lg cfu的相关系数为0.967 6)。本研究优化建立了一种快速分析检测兔粪中菌群变化的ERIC-PCR指纹图谱技术,该技术可较好反映兔粪中优势菌含量的变化情况,为兔肠道疾病的预防、诊断和治疗提供依据。
关键词:兔粪;粪便菌群;ERIC-PCR;基因指纹图谱;特征条带
近年来国内外市场对兔产品需求量的提高大大促进了养兔业的发展,且养兔投资少、见效快,是振兴农村经济,解决下岗职工再就业的好门路。但由于家兔盲肠和胃壁都很薄,容易患腹泻等胃肠疾病且很难治愈,这成为困扰养殖户的一大难题[1]。动物肠道微生物被认为是机体的另一个器官,有研究指出,肠道菌群结构的变化与腹泻等疾病直接相关,因此建立快速有效的肠道菌群结构分析方法对于预防、诊断和治疗兔肠道疾病具有重要的意义。
ERIC序列是近十几年来,在肠道细菌中发现的一段长为126 bp的反向重复序列,定位于基因组内可转录的非编码区域或与转录有关的区域,且 ERIC 序列散布在整个基因组中,具有极强的保守性[2]。该技术自从1991年被发明以来得到了广泛的发展,R.K.Mishra等[3]基于ERIC-PCR分析对尖孢镰刀菌分离菌进行基因分型;G.Nath等[4]对二十年间分离的伤寒沙门氏菌做ERIC-PCR对比,分析沙门氏菌的变异情况;何力等[5]以大肠杆菌的ERIC指纹图谱为指示因子对水库中的贝类产品污染进行微生物源示踪;M.Kosek等[6]将ERIC技术与检测能力较强的脉冲凝胶电泳(PFGE)技术同时对比分析指定菌株为同系或不同系,结果表明,ERIC技术的分析结果与PFGE技术相关性达90.4%,且操作简单、重复性好。因此,通过分析扩增出条带的分布、数量和亮度等信息,即可从分子水平对混合样的菌群组成进行鉴定。该技术与16srRNA、RT-qPCR、PCR-DGGE等细菌鉴定方法相比,具有操作简便、检测快捷、成本低的优点。
本试验以兔粪微生物为研究材料,对ERIC指纹图谱技术进行优化,构建出能够代表家兔肠道主要菌群组成的DNA指纹图谱,分离兔粪中的优势菌(双歧杆菌和大肠杆菌)并构建其指纹图谱,从而分析双歧杆菌和大肠杆菌对应的特征条带,从而建立一套能够快速检测分析家兔肠道菌群结构变化的方法。
1材料与方法
1.1主要材料和试剂
粪样采集:从洛阳万寿养殖场挑选90日龄、体重2.4 kg的健康德国花巨兔,无菌操作采集其新鲜粪便放于无菌容器中。养殖场采用3层垂直兔笼单笼饲养,以常规兔用复合颗粒饲料饲喂,定期加喂少量胡萝卜以补充维生素。
主要试剂:Taq酶(5 U·μL-1)、dNTP、DL 5000 DNA Marker、琼脂糖均购于南京诺唯赞生物科技有限公司;扩增引物(ERIC1:5′-ATGTAAGCTCCTGGGGATTCAC-3′,ERIC2:5′-AAGTAAGTGACTGGGGTCAGCG-3′),由上海生工生物工程有限公司合成。
1.2ERIC-PCR反应条件的优化
1.2.1粪样预处理无菌操作取新鲜家兔粪样1粒加入3 mL无菌PBS溶液(0.05 mol·L-1,pH 7.4)中,涡旋混匀5~10 min;2 000 r·min-1离心5 min,收集上清液。此法重复3次后,取上清液置于离心管中,10 000 r·min-1离心3 min,收集菌体细胞;再将细胞用PBS洗涤4次,无菌水洗涤1次,用0.1 mL去离子水重悬[7]。
1.2.2基因组提取参考宫强等[8]设计的改良CTAB法对样品的基因组进行提取。
1.2.3ERIC-PCR反应条件的优化以粪样微生物基因组为模板,使用ERIC引物进行PCR,针对TaqDNA聚合酶、dNTPs、引物和退火温度4个影响因素,设置不同梯度进行优化(表1),找出各自最适合的反应条件。参考文献[3,9],初始反应体系为25 μL:其中10×PCR buffer(含Mg2+15 mmol·L-1) 2.5 μL,dNTP Mixture(10 mmol·L-1)1 μL,上下游引物(20 μmol·L-1)各1 μL,DNA模板2 μL,TaqDNA聚合酶(5 U·μL-1) 0.5 μL,ddH2O 补充至25 μL;初始PCR反应程序:94 ℃预变性5 min;94 ℃变性1 min,56 ℃退火1 min,72 ℃延伸3 min,30个循环;最后72 ℃末端延伸10 min。PCR结束后,取5 μL产物点样于1.5%琼脂糖凝胶,70 V电泳1 h,紫外凝胶成像仪检测拍照。
1.3肠道优势菌的分离及其ERIC指纹图谱的建立1.3.1细菌分离筛选梯度稀释预处理后的粪样菌悬液,分别涂布Beerens平板(培养双歧杆菌)、MCA平板(培养大肠杆菌),Beerens平板放入厌氧罐中,MCA平板放入恒温培养箱中,37 ℃培养48 h,菌落计数并挑取平板上特征较明显且数量较多的菌落,镜检、生理生化鉴定,筛选出双歧杆菌和大肠杆菌,接种相应液体培养基摇瓶发酵。
1.3.2分离菌ERIC指纹图谱的构建用CTAB法提取双歧杆菌、大肠杆菌基因组,用优化的反应体系进行ERIC-PCR;反应产物琼脂糖凝胶电泳,凝胶成像检测,与粪样微生物指纹图谱进行比对分析,找出各自对应的特征条带。
表1ERIC-PCR反应优化影响因素梯度设置
Table 1The optimization design for influencing factors of ERIC-PCR

影响因素Influencefactor梯度变量GradientvariablesTaq聚合酶/μLTaqpolymerase0.40.60.81.01.2dNTPs/μL0.61.01.41.82.2引物/μLPrimer0.51.01.52.02.5退火温度/℃Annealingtemperature4952555860
1.4特征条带的验证及稳定性分析
1.4.1发酵前后粪样中双歧杆菌与大肠杆菌含量变化分析参照文献[10]配制与肠道内容物相近的发酵培养基(配方(g·L-1):牛肉膏 2.4、蛋白胨 10、L-半胱氨酸 0.6、葡萄糖 2.5、酵母膏 5.0、氯化钠 5.0、pH 6.5),接种1%经过预处理的粪样菌悬液,37 ℃厌氧培养48 h;取发酵液用CTAB法提取基因组,用优化的反应体系进行ERIC-PCR,得到发酵菌群ERIC基因指纹图谱,与发酵前的粪样菌群基因指纹图谱进行比对。用Beerens、MCA平板分别对发酵液中的双歧杆菌与大肠杆菌进行菌落计数,并与指纹图谱结果进行对比。
1.4.2图谱分析及数据处理通过凝胶成像仪的图像处理软件Image Lab 3.0对图谱进行处理,以图谱中标准条带DL 5 000 Marker的 1 000 bp条带亮度(对应150 ng的量)为相对标准(R0=1),对其他条带的亮度进行定量,获得相对亮度值R。采用DPS 7.05软件对数据进行统计学分析。
2结果
2.1ERIC-PCR反应体系的优化
ERIC-PCR反应体系中各因素不同梯度设置的ERIC-PCR电泳结果见图1。图1中不同因素梯度的条带亮度不同,说明优化结果不同。
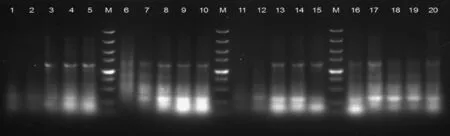
M.DNA相对分子质量标准DL 5000;1~5.Taq酶的浓度梯度依次为0.4、0.6、0.8、1.0和1.2 μL;6~10.引物浓度梯度依次为0.5、1.0、1.5、2.0和2.5 μL;11~15.dNTPs浓度梯度依次为0.6、1.0、1.4、1.8和2.2 μL;16~20.退火温度梯度依次为49、52、55、58、60 ℃M.DL 5000 marker;1-5.Taq polymerase concentration gradient followed by 0.4,0.6,0.8,1.0 and 1.2 μL;6-10.Primer concentration gradient followed by 0.5,1.0,1.5,2.0 and 2.5 μL;11-15.dNTPs concentration gradient followed by 0.6,1.0,1.4,1.8 and 2.2 μL;16-20.Annealing temperature gradient followed by 49,52,55,58 and 60 ℃图1 ERIC-PCR反应条件优化电泳图Fig.1 The electrophoresis figure of the ERIC-PCR reaction condition optimization
2.1.1TaqDNA聚合酶的优化图1表明,Taq酶添加量为1.0 μL最为合适。添加量在0.8 μL以下时条带稀少且亮度弱,添加1.2 μL时亮度很大但杂带较多拖尾较严重。
2.1.2引物浓度的影响图1中6~10泳道表明,加入引物浓度1.5 μL最为合适。加入量为0.5、1.0 μL时条带亮度小且较为模糊;2.0 μL以上时条带亮度大,但底部出现引物二聚体条带,因此以最低引物量产生所需要的结果为好。
2.1.3dNTPs添加量的优化图1中11~15泳道表明,dNTPs添加量为1.4 μL时最好。添加量过高会与Mg2+结合,使体系中游离的Mg2+减少,从而影响Taq酶活性,且增大错配机率;而浓度不足又会影响PCR扩增产量。
2.1.4退火温度对扩增条带的影响图1中16~20泳道表明,当退火温度选用52 ℃时亮度大,效果最好。55 ℃以后条带暗且较稀疏,扩增效率低;温度为49 ℃时条带较模糊有拖尾。
经过对各个影响因素的优化,得到了适合构建兔粪微生物ERIC-PCR DNA指纹图谱的反应体系:25 μL体系,其中10×PCR buffer(含Mg2+15 mmol·L-1) 2.5 μL,dNTP Mixture(10 mmol·L-1) 1.4 μL,上下游引物(20 μmol·L-1)各1.5 μL,DNA模板2.0 μL,TaqDNA聚合酶(5 U·μL-1)1.0 μL,ddH2O 补至25 μL。PCR反应程序:94 ℃预变性5 min;94 ℃变性1 min,52 ℃退火1 min,72 ℃延伸3 min,30个循环;最后72 ℃末端延伸10 min。
2.2兔粪中双歧杆菌和大肠杆菌菌落特征与含量测定
通过平板分离、镜检与生理生化鉴定,获得了兔粪较具代表性的双歧杆菌与大肠杆菌。兔粪中分离的双歧杆菌与大肠杆菌菌落特征及含量测定的平板菌落计数结果见表2。
表2兔粪中分离菌的含量及菌落特征
Table 2Content and colony characteristics of isolated bacteria from rabbit manure
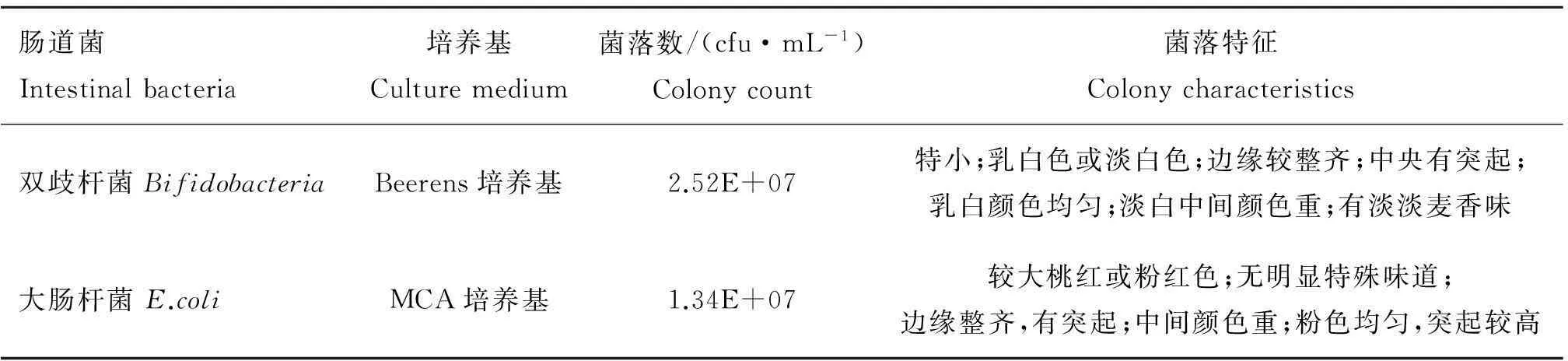
肠道菌Intestinalbacteria培养基Culturemedium菌落数/(cfu·mL-1)Colonycount菌落特征Colonycharacteristics双歧杆菌BifidobacteriaBeerens培养基2.52E+07特小;乳白色或淡白色;边缘较整齐;中央有突起;乳白颜色均匀;淡白中间颜色重;有淡淡麦香味大肠杆菌E.coliMCA培养基1.34E+07较大桃红或粉红色;无明显特殊味道;边缘整齐,有突起;中间颜色重;粉色均匀,突起较高
2.3粪样微生物DNA指纹图谱的建立与特征条带分析
发酵前、后的混合菌指纹图谱与分离菌指纹图谱见图2。图2表明,双歧杆菌有2条ERIC条带,其中在450 bp处有1条明显的特征条带;大肠杆菌有4条ERIC条带,其中1 300与4 000 bp处为较为明显的2条特征条带;并且,双歧杆菌与大肠杆菌的这3条特征条带,均可在粪样菌图谱中找到明显的对应条带。

M.DNA相对分子质量标准DL 5000;1.粪样菌群ERIC基因指纹图谱;2.发酵后菌液ERIC基因指纹图谱;3、4.双歧杆菌ERIC基因指纹图谱;5、6.大肠杆菌ERIC基因指纹图谱M.DL 5000 marker;1.ERIC genetic fingerprint of fecal samples flora;2.ERIC genetic fingerprint of bacteria liquid after fermentation;3,4.ERIC genetic fingerprint of the Bifidobacteria;5,6.ERIC genetic fingerprint of the E.coli图2 有益菌及有害菌特征条带分析图Fig.2 The analysis figure of beneficial bacteria and harmful bacteria characteristic bands
2.4特征条带的稳定性
对分离菌基因组和兔粪微生物基因组进行5次重复扩增,获得的特征条带大小以及粪样指纹图谱中450、1 300 bp和4 000 bp处的相对亮度数据见表3。对亮度数据进行统计分析,可知变异系数CV均比较小(在5%以下),表明确定的条带位置以及图谱中相应条带的相对亮度具有很好的稳定性。
2.5特征条带的验证
双歧杆菌与大肠杆菌发酵前后的特征条带的相对定量值R变化与相对菌落数变化见表4,特征条带相对亮度与相对菌落数的关系见图3。表4中双歧杆菌的R值取450 bp条带的相对亮度值,大肠杆菌R值取1 300和4 000 bp条带相对亮度的平均值。图2中,发酵后ERIC指纹图谱中的条带向低位方向迁移,从表4也可看出,发酵后450 bp处的条带亮度显著提高(相对亮度由0.56提高到1.48),而1 300 bp处的条带仅隐约可见,4 000 bp处的条带基本没有(两条带相对亮度平均值由0.50降低到0.04),说明发酵液中双歧杆菌生长较好、大肠杆菌受到抑制。图3表明,条带亮度变化与相应菌数变化明显呈正相关;对相对菌落数lg cfu和相对亮度R进行回归分析,得到二者的相关系数高达0.967 6,表明特征条带的相对亮度也能较好的表征对应菌在复合样中的含量。
表3特征条带稳定性分析
Table 3The stability analysis of characteristic bands

组别Group特征条带/bpCharacteristicband相对亮度Relativebrightness双歧杆菌Bifidobacteria大肠杆菌E.coli450bp1300bp4000bp1450.161286.804011.560.560.500.512455.241325.784005.650.570.530.523447.621308.763987.490.540.480.484450.171321.973990.180.580.510.535440.361277.724017.560.550.520.50平均值Mean448.711304.214002.490.560.500.51标准差Standarddeviation4.8519.0111.800.010.020.02变异系数/%Coefficientofvariation1.081.460.292.463.553.00

Table4ChangesofBifidobacteriaandE.colicontentbeforeandafterfermentation

类别Category发酵前Beforethefermentation发酵后Afterthefermentation双歧杆菌Bifidobacteria大肠杆菌E.coli双歧杆菌Bifidobacteria大肠杆菌E.coli相关系数Correlationcoefficient相对菌落数lgcfu相对亮度Relativebrightness7.40±0.510.56±0.017.13±0.490.50±0.0210.83±0.521.48±0.014.09±0.750.04±0.020.9676

图3 特征条带相对亮度与相对菌落数的关系Fig.3 The relationship between relative brightness of characteristic bands and lg cfu
3讨论
ERIC序列是肠杆菌基因组中普遍存在的保守序列,在不同菌种间的分布及数量不同。兔粪中菌群组成复杂,PCR反应会扩增出众多大小不一、亮度不同的条带,可以将每个条带看作是1种细菌类群,条带亮度代表该类群的种群数量[11],从而通过获得的条带数目和亮度信息分析混合菌群的多样性以及不同样品间的相似性。
影响ERIC-PCR最终条带亮度的因素众多,模板、Taq酶的活性、PCR反应的条件等等。为了建立适宜兔粪混合菌群的PCR反应体系,本研究对ERIC-PCR的反应条件进行优化,使ERIC引物能够以兔粪微生物基因组为模板扩增出较为完整的指纹图谱。优化结果显示,以兔粪微生物基因组为模板需要较高的Taq酶浓度(1.0 μL)、引物浓度(1.5 μL)和dNTP浓度(1.4 μL),以及较为适中的退火温度(52 ℃),以满足此复杂样品的扩增需要。从图2中可以看出,获得的条带图谱中有几条亮度较大的条带,与粪便中含量较大的几种菌群(双歧杆菌和大肠杆菌)有关。
ERIC指纹图谱技术应用广泛,如菌株的基因分型、微生物污染源示踪等,然而通过确定分离株的特征条带分析其在复合菌群中相应比例的研究很少有报道。双歧杆菌为厌氧的有益菌,大肠杆菌为好氧的条件致病菌且含量多不利于肠道健康,二者本身在肠道微生物中的含量较大,加上生长环境的差异,易于通过控制厌氧环境进行选择性培养。本研究通过对兔粪微生物中的优势菌分离、镜检与生理生化鉴定,获得特征较为明显的双歧杆菌和大肠杆菌菌株;提取分离菌基因组,进行ERIC-PCR扩增,得到的指纹图谱条带较单一且均有较高亮度的特征条带,其中双歧杆菌的特征条带在450 bp处,而大肠杆菌的特征条带有2条,分别在1 300和4 000 bp处,这3条条带在兔粪ERIC指纹图谱中均能找到,且同样为亮度较大的特征条带。
为了验证所确定的3条特征条带的可信度,对兔粪微生物进行了厌氧发酵,选择性地富集了双歧杆菌,分离计数平板测定了两类菌的含量变化,如表3所示,发酵后双歧杆菌含量占据优势,而肠杆菌则只剩下极少数存活。发酵后菌群的ERIC-PCR DNA指纹图谱如图2所示,条带数目趋于单一,450 bp处条带亮度增大,而1 300和4 000 bp处条带亮度微弱,各条带亮度值如表3和图3所示,可以发现,特征条带亮度变化情况与菌落变化情况成正相关(相关系数为0.967 6),表明这几条特征条带亮度可以从很大程度上表征相应菌群的含量。
由于ERIC-PCR指纹图谱影响因素较多,很难保证结果能够与实际的菌群数量完全一致[12],这也是造成图谱分析结果与菌落分析结果并不绝对相符的原因,二者只能反映出菌群结构变化的趋势相似;另外,单从条带的相对亮度值很难找出与菌群浓度间的具体对应关系,需要做进一步研究,如对条带进行序列分析、建立详细的图谱信息数据库等,最大限度的挖掘指纹图谱的潜在信息,充分发挥其指示复杂样品菌群结构的能力。
4结论
本研究优化建立了针对兔粪微生物进行ERIC-PCR的反应体系,获得了兔粪微生物的基因指纹图谱。将此图谱与分离出的双歧杆菌和大肠杆菌ERIC-PCR图谱进行对比,确定双歧杆菌的特征条带在指纹图谱的450 bp处,大肠杆菌特征条带在指纹图谱的1 300与4 000 bp处。通过比较发酵前、后兔粪微生物ERIC指纹图谱的条带亮度和迁移情况,发现其与菌落计数结果趋势相一致,二者相关系数达0.967 6,表明所确定的条带有较高的可信度。因此,通过检测兔粪微生物ERIC指纹图谱中450、1 300和4 000 bp处条带亮度的变化情况,可了解兔粪中双歧杆菌和大肠杆菌的含量变化情况,为兔肠道疾病的预防、诊断和治疗提供依据。
参考文献(References):
[1]郎跃深.肉兔饲养与繁育技术[M].北京:科学技术文献出版社,2009.
LANG Y S.The rearing and breeding techniques of meat rabbit[M].Beijing:Science and Technology Literature Press,2009.(in Chinese)
[2]陈庆森,高文茹,朱晨晨,等.原料奶中微生物区系ERIC-PCR DNA指纹图谱的建立[J].农业工程学报,2013,29(11):261-269.
CHEN Q S,GAO W R,ZHU C C,et al.Establishment of ERIC-PCR DNA fingerprint on enterobacteriaceae in raw milk[J].TransactionsoftheChineseSocietyofAgriculturalEngineering,2013,29(11):261-269.(in Chinese)
[3]MISHRA R K,PANDEY B K,PATHAK N,et al.BOX-PCR-and ERIC-PCR-based genotyping and phylogenetic correlation among fusarium oxysporum isolates associated with wilt disease inPsidiumguajavaL[J].BiocatAgricBiotechnol,2014,4(1):25-32.
[4]NATH G,MAURYA P,GULATI A K.ERIC PCR and RAPD based fingerprinting of Salmonella Typhi strains isolated over a period of two decades[J].InfectGenetEvol,2010,10(4):530-536.
[5]何力,傅玲琳,冯立芳,等.大肠埃希氏菌ERIC-PCR指纹图谱构建及贝类污染微生物源示踪[J].中国食品学报,2012,12(8):163-169.
HE L,FU L L,FENG L F,et al.ERIC-PCR fingerprinting of fecalEscherichiacoliand microbial source tracking in non-point pollution of the Shellfish culture area of East China sea[J].JournalofChineseInstituteofFoodScienceandTechnology,2012,12(8):163-169.(in Chinese)
[6]KOSEK M,YORI P P,GILMAN R H,et al.Facilitated molecular typing of Shigella isolates using ERIC-PCR[J].AmJTropMedHyg,2012,86(6):1018-1025.
[7]VIGSNAES L K,HOLCK J,MEYER A S,et al.Invitrofermentation of sugar beet arabino-oligosaccharides by fecal microbiota obtained from patients with ulcerative colitis to selectively stimulate the growth of Bifidobacterium spp.and Lactobacillus spp[J].ApplEnvironMicrobiol,2011,77(23):8336-8344.
[8]宫强,关道明,王耀兵,等.大肠杆菌总DNA快速提取方法的比较研究[J].海洋环境科学,2005,24(4):63-66.
GONG Q,GUAN D M,WANG Y B,et al.Methods extractedEscherichiacoligenomic DNA using the fast ways[J].MarineEnvironmentalScience,2005,24(4):63-66.(in Chinese)
[9]王燚,何廷美,钟志军,等.不同季节亚成体大熊猫肠道菌群ERIC-PCR指纹图谱分析[J].中国兽医科学,2011,41(8):778-783.
WANG Y,HE T M,ZHONG Z J,et al.Analysis of the characteristics of intestinal flora with seasonal shift in subadult giant panda by ERIC-PCR fingerprinting[J].ChineseVeterinaryScience,2011,41(8):778-783.(in Chinese)
[10]YIN Y S,LEI F,ZHU L Y,et al.Exposure of different bacterial inocula to newborn chicken affects gut microbiota development and ileum gene expression[J].ISMEJ,2010,4(3):367-376.
[11]乔德才,陈敬,魏桂芳,等.采用DNA指纹图谱技术分析中长跑运动员肠道菌群结构特征[J].中国运动医学杂志,2004,23(5):517-521.
QIAO D C,CHEN J,WEI G F,et al.Bacterial community structural dynamics in intestinal tract of long and middle distance runners by DNA fingerprinting analysis[J].ChineseJournalofSportsMedicine,2004,23(5):517-521.(in Chinese)
[12]AHMED H A,IBRAHIM A F,HUSSEIN M A,et al.ERIC-PCR fingerprinting of some S.Typhimurium isolates from chicken and humans with reference to the microbiological quality of retail chicken meat in dakahlia,egypt[J].GlobalVeterinaria,2014,13(1):95-104.
(编辑郭云雁)
Establishment and Analysis of ERIC-PCR DNA Fingerprint of Bacteria in Rabbit Manure
LI Yun-fei,QIN Cui-li*,WANG Zhong-ze,YANG Ruo-lan,LI Yang
(CollegeofFood&Bioengineering,HenanUniversityofScience&Technology,Luoyang471023,China)
Abstract:This study was designed to apply ERIC-PCR genetic fingerprinting technology to analyze the microbes in rabbit manure,and establish a rapid analysis and detection method of rabbit manure bacterium changes.The ERIC-PCR DNA fingerprint of bacteria in rabbit manure was established by optimizing PCR reaction conditions.The differences between DNA fingerprints of microbiome and dominant strains isolated from rabbit manure was compared,and then the characteristics of the ERIC fingerprint of bacteria in rabbit manure was analyzed.The ERIC-PCR reaction system for bacteria in rabbit manure was established.The characteristic bands forBifidobacteriawere located at 450 bp,the characteristic bands forE.coliwere located at 1 300 and 4 000 bp.Anaerobic fermentation experiment proved that the trends of the bands’ brightness were consistent with the results of the colony count (the correlation coefficient between the relative brightness and lg cfu was 0.967 6).A rapid analysis and detection method of rabbit manure bacterium changes by using ERIC-PCR genetic fingerprinting technology was established.This method could reflect the changes of beneficial bacteria in rabbit manure,thus providing a basis for prevention,diagnosis and treatment of rabbit intestinal diseases.
Key words:rabbit manure;manure bacterium;ERIC-PCR;DNA fingerprints;characteristic bands
doi:10.11843/j.issn.0366-6964.2016.04.011
收稿日期:2015-03-27
基金项目:新乡市泰弘生物有限公司委托项目(2477)
作者简介:李云飞(1991-),男,河南洛阳人,硕士,主要从事畜禽肠道微生物与宿主的关系、益生菌、动物复合微生态饲料添加剂等的研究,E-mail: liyun12fei@163.com *通信作者:秦翠丽,教授,Tel:0379-64282342,E-mail:qincuili308@163.com
中图分类号:S829.1;Q93-311
文献标志码:A
文章编号:0366-6964(2016)04-0716-07
