玉米ZmPP6C基因的克隆及其响应光质和胁迫处理的表达模式分析
2016-03-03原换换孙广华樊晓聪阳孟凡华宋梅芳詹克慧杨青华杨建平
原换换 孙广华 闫 蕾 郭 林 樊晓聪, 肖 阳孟凡华 宋梅芳,4 詹克慧 杨青华,* 杨建平,,*
1河南农业大学农学院 / 河南粮食作物协同创新中心, 河南郑州 450002;2中国农业科学院作物科学研究所, 北京 100081;3中国农业科学院研究生院, 北京 100081;4北京市辐射中心, 北京 100875
玉米ZmPP6C基因的克隆及其响应光质和胁迫处理的表达模式分析
原换换1,2,**孙广华1,2,**闫蕾2郭林2樊晓聪1,2肖阳3孟凡华2宋梅芳2,4詹克慧1杨青华1,*杨建平1,2,*
1河南农业大学农学院 / 河南粮食作物协同创新中心, 河南郑州 450002;2中国农业科学院作物科学研究所, 北京 100081;3中国农业科学院研究生院, 北京 100081;4北京市辐射中心, 北京 100875
摘要:丝氨酸/苏氨酸蛋白磷酸酶6亚基(catalytic subunits of Ser/Thr protein phosphatase 6, PP6C)是PP6全酶的催化亚基。在模式植物拟南芥中的研究表明, PP6C参与生长素极性运输、脱落酸信号转导和光信号转导途径介导的开花调控。为了明确玉米丝氨酸/苏氨酸蛋白磷酸酶6亚基(ZmPP6C)的蛋白结构特征与同源蛋白间的进化关系, 采用
RT-PCR方法克隆了ZmPP6C的全长基因。序列分析表明, ZmPP6C开放阅读框为912个核苷酸, 编码303个氨基酸残基, 包含PP2A的催化亚基PP2Ac结构域; 系统进化树分析表明, PP6C蛋白在进化上较为保守, 并且与高粱的PP6C蛋白相似性更高。对玉米自交系B73的ZmPP6C基因进行器官特异性表达分析表明, 其表达量在成株期叶片中最高, 是根中的7.9倍; ZmPP6C能够响应不同光质处理, 且受远红光和红光的影响较大; 也能响应长日和短日处理,在长日条件下的光照和黑暗阶段各有一个明显的表达高峰, 在短日条件下的光照和黑暗阶段分别有2个和3个表达峰值; 同时, ZmPP6C还响应高渗透、盐渍和淹水等胁迫处理, 出现明显的上调表达。结果表明, ZmPP6C在玉米光信号转导、开花诱导与胁迫应答中发挥重要作用, 其分子与生化机制值得进一步探讨。
关键词:玉米; 丝氨酸/苏氨酸蛋白磷酸酶; 表达模式分析; 光敏色素; 光信号途径; 非生物胁迫
北京市自然科学基金(重点)项目(6151002), 国家转基因生物新品种培育重大专项(2014ZX08010-002), 国家自然科学基金项目(31570268, 31170267)和中国农业科学院科技创新工程项目的资助。
This study was supported by the Natural Science Foundation (6151002), the Major Project of China on New Varieties of GMO Cultivation (2014ZX08010-002), the National Natural Science Foundation of China (31570268, 31170267) and the Agricultural Science and Technology Innovation Program (ASTIP) of CAAS.
第一作者联系方式: 原换换, E-mail: yuanhuanhuan111@126.com, Tel: 010-82105851; 孙广华, E-mail: guanghuanuli@163.comm, 010-82105851
蛋白磷酸酶的作用和蛋白激酶相反, 它是催化已磷酸化的蛋白质去磷酸化反应的一类酶, 与蛋白激酶相对应存在, 共同组成了磷酸化和去磷酸化这一重要的蛋白质活性的开关系统[1,2]。蛋白磷酸酶6(PP6)是一种类似于PP2A的丝氨酸/苏氨酸蛋白磷酸酶[1,2], 参与调节转录、翻译、形态建成和细胞循环诸多过程[3]。PP6全酶包括催化亚基PP6C、调节亚基PP2A和结构蛋白SAPS。而PP2A全酶包括2个调节亚基PP2Aa和PP2Ab, 以及催化亚基PP2Ac,其中催化亚基PP2Ac 类磷酸酶蛋白在逆境中发挥着重要作用[1,4-6]。在模式植物拟南芥中有AtFyPP1 和AtFyPP32个高度同源的PP6C 催化亚基基因; 二者均能与光敏色素磷酸酶2A结合, 调控植物开花[6]。AtFyPP3过量表达株系延迟开花, 而atftpp3缺失突变体则导致开花提前[6]。AtFyPP3的转录受到光照节律的调节, 长日条件下AtFyPP3的表达明显高于短日条件[6]。酵母双杂测试证实豌豆PP6能与燕麦的phyA和拟南芥的phyB互作, 体外Pull-down实验表明豌豆PP6优先结合磷酸化或者Pfr形式的phyA和phyB[7]。以上研究结果表明PP6C可能通过与光敏色素互作调节植物光形态建成。
研究表明拟南芥的2个PP6C同源蛋白AtFyPP1 和AtFyPP3参与生长素(auxin)的极性运输和ABA (abscisic acid, ABA)介导的胁迫响应。生长素主要在植物顶端分生组织中合成, 然后被运输到植物体的各个部位; 生长素的极性运输取决于生长素输出蛋白PIN (PIN-FORMED)的磷酸化水平; 蛋白丝氨酸/苏氨酸激酶PID (PROTEIN SERINE/THREONINE KINASE PINOID)能促进PIN的磷酸化[8-12]。AtFyPP1和AtFyPP3通过负调控PID的激酶活性抑制PIN的磷酸化, 进而阻遏生长素的极性运输, 导致植物生长发育迟缓[13]。激素ABA能够调节植物对生物胁迫和非生物胁迫(干旱、盐、低温和病原菌等)的响应[14-17]。碱性亮氨酸拉链转录因子ABI5 (ABSCISIC ACID INSENSITIVE5)能促进种子萌发以及萌发后的幼苗生长, 是ABA信号途径中重要的抑制因子[18-21]。AtFyPP1和AtFyPP3通过ABI5的脱磷酸化来加速其降解, 从而促进ABA信号转导[4]。
玉米是集口粮、饲料和工业原料为一体的重要的多用途作物[22,23], 其产量受光照、水分和温度等多种环境因素的制约[23-27]。蛋白磷酸酶在植物逆境信号转导、有丝分裂和细胞循环过程中具重要作用[28-30],而玉米中只有一个蛋白磷酸酶6催化亚基(ZmPP6C),其研究未见报道。本研究克隆了ZmPP6C基因, 初步分析了ZmPP6C蛋白功能结构域, 在氨基酸水平对其进行了系统进化分析, 并对该基因器官表达特异性及响应光和胁迫处理的表达模式进行了较系统的分析。ZmPP6C的表达能够响应不同光质处理, 也能响应长日和短日处理; 同时, ZmPP6C还响应高渗透、盐渍和淹水等胁迫处理, 出现明显的上调表达。我们发现ZmPP6C在玉米光信号转导、开花诱导与胁迫应答中发挥重要作用, 该结果将为以后ZmPP6C功能研究及其在玉米分子抗逆育种中的应用奠定基础。
1 材料与方法
1.1试验材料及样品处理
选用玉米自交系B73为研究材料(中国农业科学院作物科学研究所李新海博士惠赠, 杨建平实验室繁育并保存)。(1)器官特异性表达样品的准备: 在北京地区4月下旬于花盆中播种自交系B73的种子、自然条件下生长60 d后分别取成株的叶、茎、根、幼穗、苞叶、花丝、雄花、花柄、叶鞘和叶枕。(2)各种持续光质处理: 将玉米种子消毒杀菌后, 平放在湿润滤纸的培养皿中28℃催芽3 d, 挑选萌动一致的种子播种于装有培养土塑料小盆(长宽高均为8.5 cm) 中, 每小盆播种9颗种子, 分别放于黑暗(Dk)、远红光(FR, 0.5 µmol m–2s–1)、红光(R, 22.5 µmol m–2s–1)、蓝光(B, 13.0 µmol m−2s−1)和白光(WL, 17.0 µmol m–2s–1)玉米苗子均生长在22℃培养箱中生长。(3)黑暗转换各种光质: 黑暗中生长13 d后分别转入以上各种光质条件下0.25、0.5、1、2、4、8、12和24 h, 分别取样。(4)长日和短日处理: 玉米幼苗在22℃长日(LD, 16-h-光照/8-h-黑暗)或短日(SD, 8-h-光照/16-h-黑暗)条件下中生长13 d。(5)盐渍(200 mmol L–1NaCl)和高渗透(20% PEG-6000)处理: 在正常温室条件下
(16-h-光照/8-h-黑暗, 22℃)生长13 d后对幼苗以盐渍(200 mmol L–1NaCl)和高渗透(20% PEG-6000)分别处理0、1、3、6、12、24和48 h。以正常温室条件下生长和未经处理的苗子作为对照。(6)淹水(灌水没根)处理: 将正常温室条件下(22℃)生长到13 d的幼苗,带钵体一同置水盒中, 水层高出钵体2 cm, 淹水7 d,接着恢复正常温室条件, 分别在淹水3、4、5和7 d以及恢复正常温室条件后3 d和7 d取样, 每天取样时间固定在上午10:00。对照样品如(5)。(7)用作基因克隆的植物材料为生长在22℃黑暗条件下13 d的幼苗。将所有样品, 迅速收获并冻存于–80℃液氮备用。
1.2菌株和载体
大肠杆菌DH5α菌株是本实验室保存的, 克隆载体pEASY-Blunt-Simple和pESAY-T1 Cloning Kit购自北京全式金生物技术有限公司。
1.3酶和试剂
实验中使用的酶和试剂及购置公司为: ExTaq酶和PrimeSTAR HS酶, TaKaRa (大连)公司; Revert Aid First Strand cDNA Synthesis Kit, Thermo Scientific公司; 限制性内切酶和T4 DNA连接酶, NEB公司; 凝胶回收试剂盒和质粒提取试剂盒, 天根生化科技有限公司。
1.4RNA提取及cDNA合成
TRIzol (Invitrogen, USA)法提取各种处理的玉米幼苗总RNA, 经DNase I (RNase-free, TaKaRa大连公司)处理后作为模板, 以Oligo-dT18为引物, 利用RevertAid First Strand cDNA Synthesis Kit (Thermo Scientific公司)反转录成cDNA备用。
1.5玉米PP6C的克隆
根据NCBI 中的ZmPP6C (NM_001175500)的序列设计引物(ZmPP6C-F5′-TCTAGAATGGATGTG GATCTGTGGAT-3′)和ZmPP6C-R (5′-GGATCCTAG GAAATATGGGACTGCGG-3′), 用黑暗下13 d幼苗的cDNA为模板, 扩增得到912 bp的片段, 克隆到pEASY-Blunt-Simple载体上, 得到pEASY-ZmPP6C。经PCR和酶切鉴定后测序(苏州金唯智生物技术有限公司北京分公司), 测序正确后备用。
1.6玉米PP6C的基因序列比对、结构预测与进化树分析
采用NCBI-Blast网站(http://blast.ncbi.nlm.nih. gov/Blast.cgi)获得玉米(Zea mays)、拟南芥(Arabidopsis thaliana)、水稻(Oryza sativa)、高粱(Sorghum bicolor)、谷子(Setaria italica)、小麦(Triticum aestivum)、大麦(Hordeum vulgare)、短柄草(Brachypodium distachyom)、油菜(Brassica napus)、大白菜(Brassica rapa)、棉花(Gossypium raimondii)、大豆(Giycine max)同源蛋白的氨基酸序列。使用DNAMAN Version 6软件比对ZmPP6C与其他植物氨基酸水平的序列, 用SMART网站(http://smart.embl-heidelberg.de/)分析ZmPP6C的结构域。
1.7玉米PP6C表达的实时荧光定量RT-PCR (qRT-PCR)分析
以不同处理的“B73”玉米的cDNA第1链为模板,玉米Tubulin基因(NM_001174192)为内参, 测定ZmPP6C的相对表达量。根据ZmPP6C与Tubulin的序列设计qRT-PCR的引物: ZmPP6C_RT-F (5′-TACAAT AGACCATCGGGACAGT-3′)和ZmPP6C_RT-R (5′-GCACGGGAACAAAATCACA-3′); Tubulin_F (5′-AC TTCATGCTTTCGTCCTACGCTCCA-3′)和Tubulin_R (5′-CTGGGAGGCTGGTAGTTGATTC-3′)。按照全式金Trans Start Green qPCR SuperMix试剂盒的说明书操作, 反应体系为20 µL。用罗氏Light Cycler 480 II Real-Time PCR System (Roche, 瑞士)进行qRT-PCR,按照SYBR Green荧光染料相对定量PCR方法检测,参照基因的2–ΔΔCt法[31]计算结果。经3次独立的生物学重复, 并以此计算其标准差。
2 结果与分析
2.1ZmPP6C基因的cDNA的长度为912 bp
根据NCBI中ZmPP6C的mRNA对应cDNA序列(NM_001175500)设计引物, RT-PCR得到912 bp左右的DNA片段(图1-A), 与预计扩增的ZmPP6C的ORF片段大小一致。将扩增片段回收并连接到pEASY Blunt-Simple载体上, 得到重组载体pEASY-ZmPP6C, 经PCR鉴定后的克隆再经Xba I和BamH I双酶切验证,符合要求的克隆经酶切应具有一条912 bp左右的目的基因条带及一条3830 bp左右的载体条带(图1-B)。最后将上述符合要求的克隆测序显示, 我们得到cDNA克隆序列与NCBI中ZmPP6C的mRNA对应cDNA序列(NM_001175500)完全一致。
2.2PP6C基因家族在双子叶植物和单子叶植物具有相同结构域和较高一致性
通过PCR的方法得到ZmPP6C的全长cDNA序列, 其ORF包含912个核苷酸残基, DNAMAN Version 6软件推测其编码303个氨基酸残基、分子量为37.73 kDa的蛋白质, 等电点4.87 (http://web.expasy.org/
protparam)。使用SMART网站(http://smart.emblheidelberg.de/)对ZmPP6C的结构域分析发现ZmPP6C中包含1个PP2Ac结构域(16~287, 丝氨酸/苏氨酸蛋白磷酸酶2A的催化域)(图2), 预测其还可能包括Fmp27_SW (4~76, RNA聚合酶II转录起始保守域)、Cir_N (7~34, CBF1互作共抑制因子CIR的N末端保守域)、Metallophos (43~238, 具有水解酶活性域)、Kelch (60~109, 蛋白结合活性域)、ZnF_UBR1 (137~ 193, 锌指蛋白N末端底物识别域)和 Costars domain (192~254, 结合肌动蛋白并调节细胞骨架和细胞运动)。同时运用DNAMAN Version 6软件将ZmPP6C与水稻、高粱和拟南芥的PP6C进行了氨基酸水平的序列比对, ZmPP6C与拟南芥AtPyPP1和AtPyPP3的一致性均高达91.09%, 与水稻的一致性达96.04%, 与高粱的一致性更高达99.67%。利用 DNAMAN Version 6软件将该蛋白与来自拟南芥、水稻、高粱、谷子、小麦、大麦、短柄草、油菜、大白菜、棉花、大豆等不同种属的PP6C蛋白进行氨基酸序列水平的系统发育树分析表明, PP6C在玉米和高粱中具有高度一致性, 且无论是单子叶还是双子叶植物, 其PP6C蛋白一致性均较高(图3)。PP6C基因家族在植物进化中的高度一致性, 暗示其功能的高度保守。
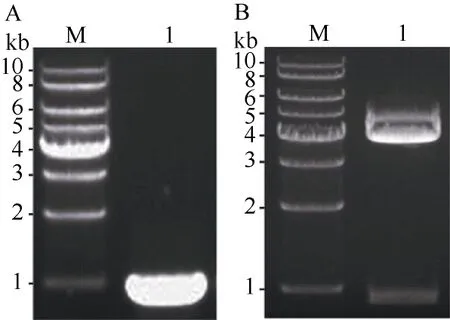
图1 ZmPP6C的ORF的cDNA片段的RT-PCR扩增及其克隆pEASY-ZmPP6C的双酶切鉴定Fig. 1 RT-PCR fragment of ZmPP6C and double digestion of pEASY-ZmPP6C using Xba I and BamH I

图2 玉米与拟南芥、水稻和高粱蛋白磷酸酶6亚基的氨基酸序列比对和结构域分析Fig. 2 Multiple sequence alignments and function domains of PP6Cs from Zea mays, Arabidopsis thaliana, Oryza sativa, and Sorghum bicolor

图3 玉米PP6C与其他植物PP6C的氨基酸水平的系统发育分析Fig. 3 Phylogenetic analysis of amino acid sequences of ZmPP6C and other related PP6C
2.3ZmPP6C器官特异表达分析
基因在植物器官中的富集表达部位与其功能密切相关, 为了研究玉米ZmPP6C的功能, 我们采用荧光定量PCR技术检测了B73成株不同器官中ZmPP6C的表达水平, 结果显示该基因在B73的各个器官中均有表达, 苞叶、叶柄和幼穗的相对表达量较高, 叶片中表达量最高, 大约是根中的8倍, 这暗示ZmPP6C可能在玉米叶片中发挥更重要的作用(图4)。
2.4ZmPP6C响应不同光质处理的表达模式分析
叶片不仅是植物光合作用的主要场所, 也是感受不同光质的主要器官。器官特异性表达分析显示ZmPP6C在叶片中表达量最高, 暗示着ZmPP6C很可能参与植物对光的响应。为了明确ZmPP6C对不同光质的响应, 我们检测了玉米自交系B73幼苗在黑暗和远红光(FRc)、红光(Rc)、蓝光(Bc)和白光(WLc)条件下ZmPP6C的表达情况, 结果显示ZmPP6C在红光和远红光条件下表达丰度相似, 均为黑暗条件下的2倍(图5); 其白光和蓝光下的表达丰度分别是黑暗下的3.2倍和1.6倍。这表明ZmPP6C的确能够受到光的调节。
为了进一步了解ZmPP6C基因受光调节表达特性, 我们进行了光质转换, 将黑暗中生长13 d的玉米自交系B73幼苗转到远红光(FR)、红光(R)、蓝光(B)和白光(WL)中, 并于0.25、0.5、1、2、4、8、12 和24 h验证ZmPP6C对各种光的响应能力。在转入远红光、红光和白光后, ZmPP6C表达丰度均表现为大幅度的升高; 其中在转入远红光30 min、红光15 min和白光30 min的表达丰度达到峰值, 分别是黑暗中的15.7、9.0和3.1倍(图6)。在转入白光下1 h 后, ZmPP6C的表达丰度下降到黑暗的13%, 且在24 h内基本稳定在黑暗的13%~26%水平(图6-C)。转入远红光与转入白光类似, 但其表达丰度高于转入白光中(图6-A)。在转到红光下30分钟后, ZmPP6C的表达丰度急剧下降到黑暗的3.9%, 在1 h和4 h时2次小的回升, 分别是黑暗的0.8倍和2.2倍; 从8 h后稳步上升, 直到24 h达到黑暗的7.2倍(图6-B)。从黑暗转蓝光后, ZmPP6C丰度上下波动较小, 推测ZmPP6C响应蓝光的能力不强(图6-D)。
2.5ZmPP6C的表达对长日和短日处理的响应
为了更全面地了解ZmPP6C对光处理的反应能力, 我们检测了ZmPP6C的表达对长日和短日处理的响应。在长日条件下, ZmPP6C的表达在光照和黑暗阶段, 各有一个明显的表达高峰, 分别出现在光下10 h时和转入黑暗3 h时, 且是黑暗结束时的4.8倍和3.1倍。短日处理下, ZmPP6C表达调控模式较为复杂, 在光照和黑暗阶段分别有2个和3个表达峰值(图7)。光照阶段的表达峰值出现在光照后2 h 和8 h, 分别达到黑暗结束时的1.9倍和5.5倍(图7-A); 黑暗阶段的表达峰值出现在进入黑暗后6、10 和14 h, 分别达到黑暗结束时的2.5、9.9和3.0倍(图7-B)。可见ZmPP6C的表达能响应长日和短日处理。
2.6ZmPP6C的表达对胁迫处理的响应
由于拟南芥中PP6通过ABA信号途径, 来参与对胁迫的调控[4], 为了进一步了解ZmPP6C对胁迫
的响应, 我们检测了ZmPP6C的表达对盐渍(200 mmol L−1NaCl)、高渗透(20% PEG-6000)和淹水(灌水没根)处理的响应。结果表明, 在0~3 h, 无论是否盐渍处理ZmPP6C的表达变化不明显, 维持在处理前的0.9~1.2倍(图8-A); 在6 h, 非盐渍处理样品ZmPP6C的表达水平上升到处理前的2倍, 而盐渍处理的表达仍未见明显变化; 在12 h, 非盐渍处理样品ZmPP6C的表达下降, 而盐渍处理表达上升,二曲线的交叉点出现在处理前的0.8倍; 在24 h和48 h, 非盐渍处理样品ZmPP6C的表达水平继续到处理前的0.4倍, 而盐渍处理表达继续上升1.1倍(24 h)和2.6倍(48 h)。20% PEG-6000处理类似于200 mmol L−1NaCl, 只是20% PEG-6000处理1 h, ZmPP6C的表达量迅速下降, 并且ZmPP6C的较低表达水平(处理前的0.2~0.6倍), 一直维持到24 h; 而20% PEG-6000处理48 h, ZmPP6C的表达水平上升到处理前的4.5倍(图8-B)。淹水(灌水没根)处理3~4 d, ZmPP6C的表达下降到未处理的20%~30%;处理5 d和7 d, ZmPP6C的表达上升到未处理的1.3倍和4.1倍; 随着恢复正常温室生长条件3 d和7 d, ZmPP6C的表达上升到未处理的3.0倍和6.9倍(图8-C)。可见, 与非胁迫处理相比, 盐渍和高渗透处理抑制6 h ZmPP6C的表达; ZmPP6C的表达对200 mmol L−1NaCl处理的响应早于对20% PEG-6000处理; 盐渍、高渗透和淹水处理均导致ZmPP6C的表达显著上调。

图4 ZmPP6C的器官特异性表达的定量RT-PCR分析Fig. 4 qRT-PCR assays of ZmPP6C in different organs of maize
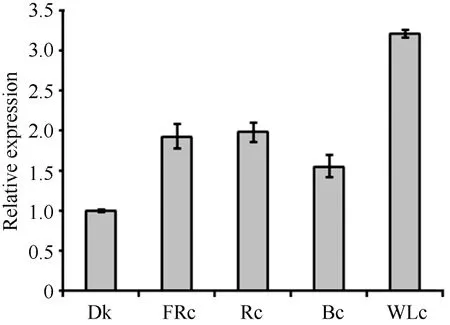
图5 各种持续光质条件下ZmPP6C转录表达的定量RT-PCR分析Fig. 5 qRT-PCR assays of ZmPP6C expression under different continuous light conditions
3 讨论
PP6C是光敏色素蛋白丝氨酸/苏氨酸磷酸酶的催化亚基[22,32], 催化磷酸化的蛋白质分子去磷酸化反应, 在光信号转导中发挥着重要作用[1,2,7]。光是影响植物生长发育最重要的环境因素之一, 首先光是能量物质, 光提供了植物光合作用的能量[33-35], 光合作用是植物生命活动过程中的重要组成部分, 是作物产量和品质的基础; 缺乏光照会导致植株生长
受阻、株高增加、叶面积减少、叶片变黄[36-37]。再者, 光是信号物质, 植物通过光线提供的信息(光照节律、光照强度和光质比例等)来及时调控其生长和发育进程, 包括种子萌发、茎节伸长、向光性、避荫性(shade avoidance)和开花期, 还包括作物的产量和品质等[38-41]。本研究表明ZmPP6C在玉米的叶片中表达量最高(图4), 同时ZmPP6C的表达响应不同光质处理(图5)、黑暗到光的转换(图6), 以及长日和短日处理(图7)等。ZmPP6C的表达响应远红光和红光处理, 但受两种光的表达模式差异较大, 转入远红光30分钟达到峰值, 然后迅速下降, 直到24 h一直保持较低的水平(图6-A); 转入红光15 min达到峰值, 迅速下降后表现迂回上升, 直到24 h达到黑暗的7.2倍(图6-B)。拟南芥、水稻和玉米中的研究表明, 光敏色素B (phyB)是红光的最主要受体, 而光敏色素A (phyA)主要是负责植物对远红光的反应,它们与植物光形态建成、避荫性以及生物产量等性状密切相关[38,41]。另外, PHYA的过量表达或者PHYB缺失突变体能显著地引起提早开花[34]; PHYA和PHYB转基因的植株导致节间缩短、株高降低、株型紧凑、光合作用提高、促进增产[41-46]。研究表明豌豆PP6能够与拟南芥中光敏色素A (phyA)和光敏色素B (phyB)互作; 豌豆PP6基因在拟南芥中异源转基因导致开花延迟, 而拟南芥的2个PP6C同源基因AtFyPP1和AtFyPP3表达的下调却引起开花提前[2]。尽管对光敏色素的激酶活性还没有完全了解, 但业已证明它们磷酸化与其活性密切相关[38]; 很可能AtFyPP1和AtFyPP3通过与phyA和phyB的互作,导致它们磷酸化的丧失, 从而影响其活性。由于玉米仅有一个ZmPP6C基因, 而玉米PHYA和PHYB基因各有2个拷贝, ZmPP6C与ZmphyA1、ZmphyA2, ZmphyB1和ZmphyB2的互作模式, 以及在互作与它们活性之间的关系有待进一步验证。
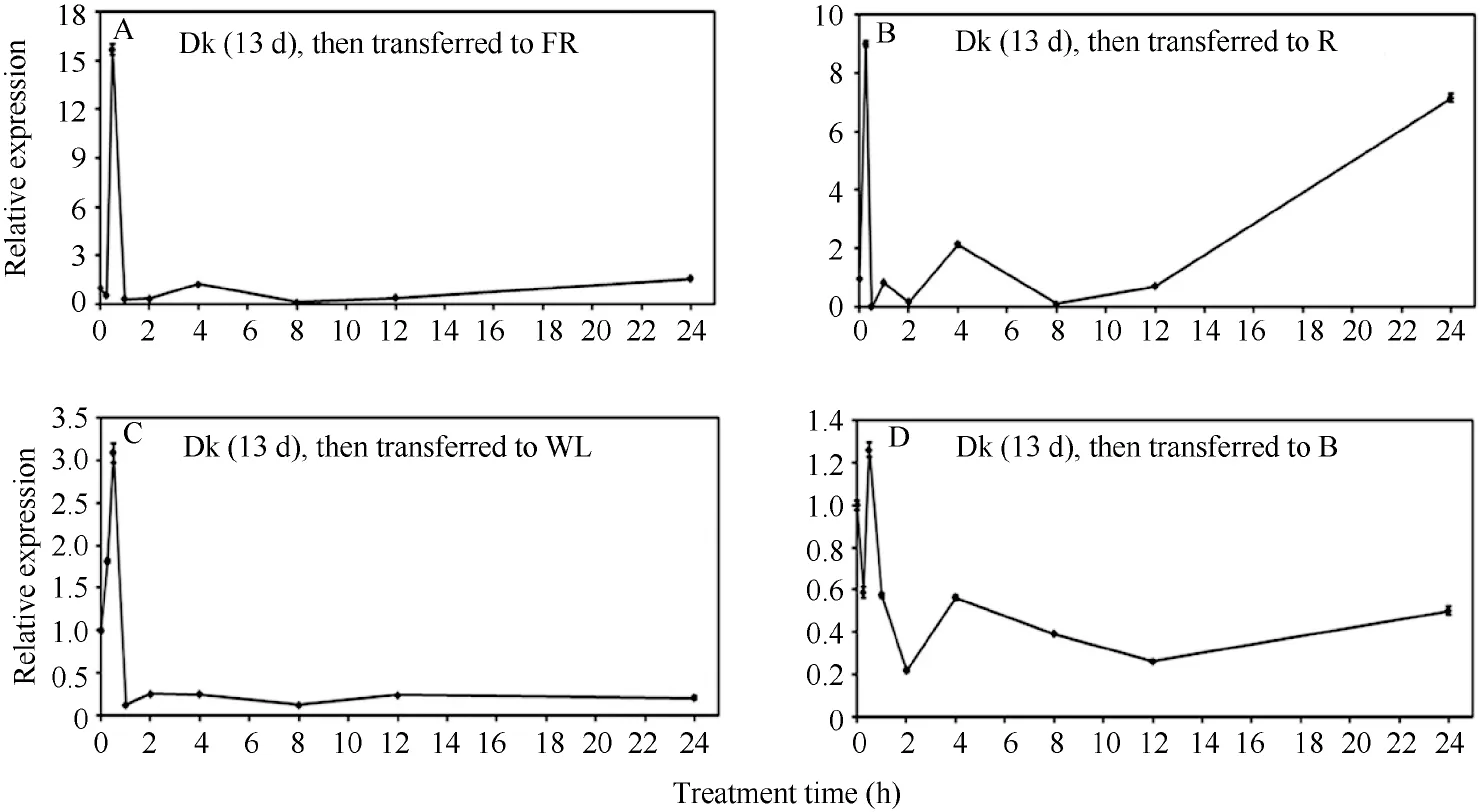
图6 黑暗转入不同光质条件下ZmPP6C转录表达的定量RT-PCR分析Fig. 6 qRT-PCR assays of ZmPP6C expression during transition from darkness to different light conditions
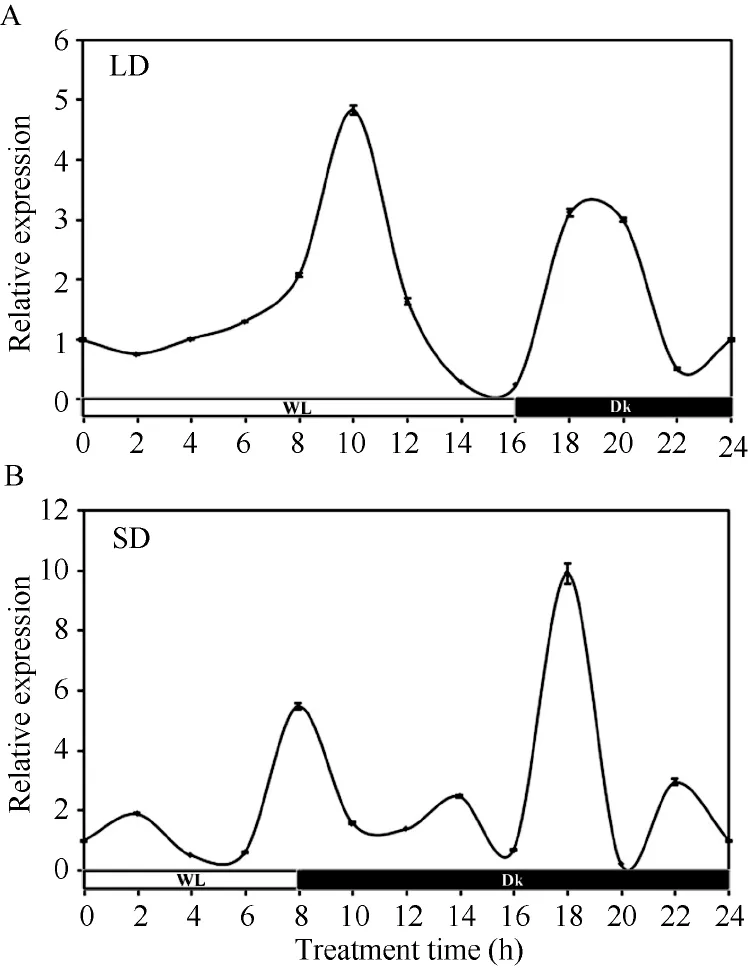
图7 长日和短日条件下ZmPP6C转录表达的定量RT-PCR分析Fig. 7 qRT-PCR assays of ZmPP6C under long day and short day conditions
模式植物拟南芥中的研究表明, 高等植物开花的光周期调控是由光信号转导途径的基因与开花相关的生物钟途径(circadian clock)相关基因如GI (GIGANTEA)、CO (CONSTANS)和FT (FLOWERING LOCUS T)共同完成的[47-50]。在光周期敏感自交系CML288中, CO的同源基因ZmCOL的表达在长短日照条件下均有2个峰值[51,52]。本研究表明玉米ZmPP6C的表达响应长日和短日处理(图7), ZmPP6C在长日下的第1个高峰是光照后10 h, 低谷是进入黑暗之前(图7), 这与光周期敏感自交系CML288的ZmCOL完全一致[51,52]; 我们还发现, 在短日照下ZmPP6C表达高峰发生的时间都提前于长日照, 前人对ZmGI研究也有着同样的现象[51,52]。ZmPP6C与开花调控基因ZmGI、ZmCOL和ZmFT之间是否存在关联, 能否利用RNAi或转基因技术修饰ZmPP6C基因进行开花性状的改良, 值得探讨。此外, 水稻基因组中含有5个PP2Ac类磷酸酶基因, 在盐敏感水稻品种9311中OsPP2Ac-2、OsPP2Ac-3和OsPP2Ac-5的表达上调、OsPP2Ac-4的表达下调; 在耐盐水稻品种“兰胜”中OsPP2Ac-4的表达上调[5,53]; 并且小麦TaPP2Ac-1基因的表达受高盐、ABA和低温诱导[25]。本研究表明ZmPP6C不但包含1个PP2Ac结构域,同时ZmPP6C的表达响应盐渍、高渗透和淹水胁迫处理, 表明玉米ZmPP6C参与了植株对胁迫的应答,但其作用机制需要进一步的验证。
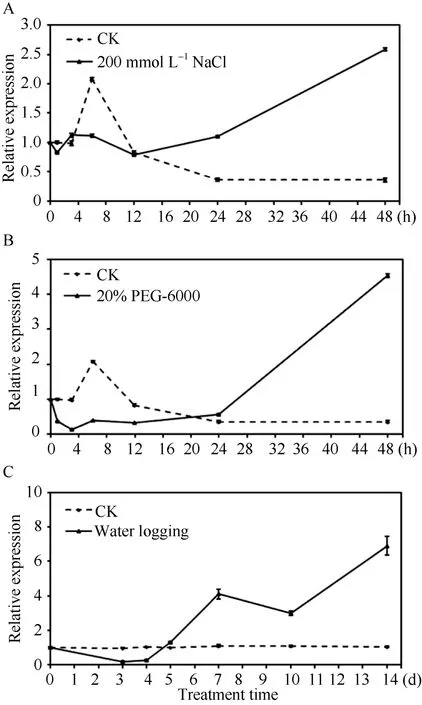
图8 非生物胁迫条件下ZmPP6C转录表达的定量RT-PCR分析Fig. 8 qRT-PCR assays of ZmPP6C under different abiotic stress conditions
4 结论
ZmPP6C与高粱和拟南芥的PP6C具有高度同源性, 具有PP2Ac结构域; ZmPP6C在玉米叶片中高表达, 能受到不同光质的影响, 特别响应远红光和红光处理; 也对长日和短日处理响应, 其表达在长日条件下的光照和黑暗阶段各有1个明显的表达高峰, 在短日条件下的光照和黑暗阶段分别有2个和3个表达峰值; 同时, ZmPP6C还响应高渗透、盐渍和淹水等胁迫处理, 出现明显的上调表达。ZmPP6C在玉米光信号转导、开花与胁迫应答方面发挥重要作用, 其分子与生化机制值得进一步探讨。
References
[1] Terol J, Bargues M, Carrasco P, Pérez-Alonso M, Paricio N. Molecular characterization and evolution of the protein phosphatase 2A B’ regulatory subunit family in plants. Plant Physiol, 2002, 129: 808–822
[2] Moorhead G B, Trinkle-Mulcahy L, Ulke-Lemée A. Emerging roles of nuclear protein phosphatases. Nat Rev Mol Cell Biol, 2007, 8: 234–244
[3] Cohen P T. Novel protein serine/threonine phosphatases: variety is the spice of life. Trends Biochem Sci, 1997, 22: 245–251
[4] Dai M, Xue Q, Mccray T, Margavage K, Chen F, Lee J H, Nezames C D, Guo L, Terzaghi W, Wan J, Deng X W, Wang H. The PP6 phosphatase regulates ABI5 phosphorylation and abscisic acid signaling in Arabidopsis. Plant Cell, 2013, 25: 517–534
[5] 刘钊, 贾霖, 贾盟, 关明俐, 曹英豪, 刘丽娟, 曹振伟, 李莉云,刘国振. 水稻PP2Ac类磷酸酶蛋白质在盐胁迫下的表达. 中国农业科学, 2012, 45: 2339–2345
Liu Z, Jia L, Jia M, Guan M L, Cao Y H, Liu L J, Cao Z W, Li L Y, Liu G Z. Expression on profiling of rice PP2Ac type phosphatase proteins in seedlings under salt stressed conditions. Sci Agric Sin, 2012, 45: 2339–2345 (in Chinese with English abstract)
[6] Kim D H, Kang J G, Yang S S, Chung K S, Song P S, Park C M. A phytochrome-associated protein phosphatase 2A modulates light signals in flowering time control in Arabidopsis. Plant Cell, 2002, 14: 3043–3056
[7] Farkas I, Dombrádi V, Miskei M, Szabados L, Koncz C. Arabidopsis PPP family of serine /threonine phosphatases. Trends Plant Sci, 2007, 12: 169–176
[8] Gälweiler L, Guan C, Müller A, Wisman E, Mendgen K, Yephremov A, Palme K. Regulation of polar auxin transport by AtPIN1 in Arabidopsis vascular tissue. Science, 1998, 282: 2226–2230
[9] Chen R, Hilson P, Sedbrook J, Rosen E, Caspar T, Masson P H. The Arabidopsis thaliana AGRAVITROPIC 1 gene encodes a component of the polar-auxin-transport efflux carrier. Proc Natl Acad Sci USA, 1998, 95: 15112–15117
[10] Müller A, Guan C, Gälweiler L, Tänzler P, Huijser P, Marchant A, Parry G, Bennett M, Wisman E, Palme K. AtPIN2 defines a locus of Arabidopsis for root gravitropism control. EMBO J, 1998, 17: 6903–6911
[11] Friml J, Benková E, Blilou I, Wisniewska J, Hamann T, Ljung K, Woody S, Sandberg G, Scheres B, Jürgens G, Palme K. AtPIN4 mediates sink-driven auxin gradients and root patterning in Arabidopsis. Cell, 2002, 108: 661–673
[12] Petrásek J, Mravec J, Bouchard R, Blakeslee J J, Abas M, Seifertová D, Wisniewska J, Tadele Z, Kubes M, Covanová M, Dhonukshe P, Skupa P, Benková E, Perry L, Krecek P, Lee O R, Fink G R, Geisler M, Murphy A S, Luschnig C, Zazímalová E, Friml J. PIN proteins perform a rate-limiting function in cellular auxin efflux. Science, 2006, 312: 914–918
[13] Dai M, Zhang C, Kania U, Chen F, Xue Q, McCray T, Li G, Qin G, Wakeley M, Terzaghi W, Wan J, Zhao Y, Xu J, Friml J, Deng X W, Wang H. A PP6-type phosphatase holoenzyme directly regulates PIN phosphorylation and auxin efflux in Arabidopsis. Plant Cell, 2012, 24: 2497–2514
[14] Mauch-Mani B, Mauch F. The role of abscisic acid in plant-pathogen interactions. Curr Opin Plant Biol, 2005, 8: 409–414
[15] Fujita Y, Fujita M, Satoh R, Maruyama K, Parvez M M, Seki M, Hiratsu K, Ohme-Takagi M, Shinozaki K, Yamaguchi-Shinozaki K. AREB1 is a transcription activator of novel ABRE-dependent ABA signaling that enhances drought stress tolerance in Arabidopsis. Plant Cell, 2005, 17: 3470–3488
[16] Fujita Y, Fujita M, Shinozaki K, Yamaguchi-Shinozaki K. ABA-mediated transcriptional regulation in response to osmotic stress in plants. J Plant Res, 2011, 124: 509–525
[17] Hauser F, Waadt R, Schroeder J I. Evolution of abscisic acid synthesis and signaling mechanisms. Curr Biol, 2011, 21: R346–355
[18] Hattori T, Totsuka M, Hobo T, Kagaya Y, Yamamoto-Toyoda A. Experimentally determined sequence requirement of ACGT-containing abscisic acid response element. Plant Cell Physiol, 2002, 43: 136–140
[19] Busk P K, Pagès M. Regulation of abscisic acid induced transcription. Plant Mol Biol, 1998, 37: 425–435
[20] Finkelstein R R, Lynch T J. The Arabidopsis abscisic acid response gene ABI5 encodes a basic leucine zipper transcription factor. Plant Cell, 2000, 12: 599–609
[21] Lopez-Molina L, Chua N H. A null mutation in a bZIP factor confers ABA-insensitivity in Arabidopsis thaliana. Plant Cell Physiol, 2000, 41: 541–547
[22] Liu X B, Zhang X Y, Wang Y X, Sui Y Y, Zhang S L, Herbert S J, Ding G. Soil degradation: a problem threatening the sustainable development of agriculture in Northeast China. Plant Soil Environ, 2010, 56: 87–97
[23] Gao Y, Jiang W, Dai Y, Xiao N, Zhang C, Li H, Lu Y, Wu M, Tao X, Deng D, Chen J. A maize phytochrome interacting factor 3 improves drought and salt stress tolerance in rice. Plant Mol Biol, 2015, 87: 413–428
[24] Xu Z S, Ni Z Y, Li Z Y, Li L C, Chen M, Gao D Y, Yu X D, Liu P, Ma Y Z. Isolation and functional characterization of HvDREB1—a gene encoding a dehydration-responsive element binding protein in Hordeum vulgare. J Plant Res, 2009, 122: 121–130
[25] Tian S J, Mao X G, Zhang H Y, Chen S S, Zhai C C, Yang S M, Jing R L. Cloning and characterization of TaSnRK2.3, a novel SnRK2 gene in common wheat. J Exp Bot, 2013, 64: 2063–2080
[26] Liu Z J, Yang X G, Hubbard K G, Lin X M. Maize potential yields and yield gaps in the changing climate of northeast China. Global Change Biol, 2012, 18: 3441–3454
[27] Yang X, Lin E D, Ma S M, Ju H, Guo L P, Xiong W, Li Y, Xu Y L. Adaptation of agriculture to warming in Northeast China. Clim Change, 2007, 84: 45–58
[28] Sun H, Tonks N K. The coordinated action of protein tyrosine phosphatases and kinases in cell signaling. Trends Biochem Sci, 1994, 19: 480–485
[29] Hanada M, Ninomiya-Tsuji J, Komaki K, Ohnishi M, Katsura K, Kanamaru R, Matsumoto K, Tamura S. Regulation of the TAK1 signaling pathway by protein phosphatase 2C. J Biol Chem, 2001, 276: 5753–5759
[30] 翁华, 冉亮, 魏群. 植物蛋白磷酸酶及其在植物抗逆中的作用.植物学通报, 2003, 20: 609–615
Weng H, Ran L, Wei Q. Protein phosphatases and their functions in plant response to environmental stress. Chin Bull Bot, 2003, 20: 609–615 (in Chinese with English abstract)
[31] Rajeevan M S, Ranamukhaarachi D G, Vernon S D, Unger E R. Use of real-time quantitative PCR to validate the results of cDNA array and differential display PCR technologies. Methods, 2001, 25: 443–451
[32] Shi Y. Serine/threonine phosphatases: mechanism through structure. Cell, 2009, 139: 468–484
[33] Fankhauser C, Chory J. Light control of plant development. Annu Rev Cell Dev Biol, 1997, 13: 203–229
[34] Neff M M, Fankhauser C, Chory J. Light: an indicator of time and place. Genes Dev, 2000, 14: 257–271
[35] Ma L, Li J, Qu L, Hager J, Chen Z, Zhao H, Deng X W. Light control of Arabidopsis development entails coordinated regulation of genome expression and cellular pathways. Plant Cell, 2001, 13: 2589–2607
[36] 李潮海, 刘奎. 不同产量水平玉米杂交种生育后期光合效率比较分析. 作物学报, 2002, 28: 379–383
Li C H, Liu K. Analysis of photosynthesis efficiency of maize hybrids with different yield in the later growth stage. Acta Agron Sin, 2002, 28: 379–383 (in Chinese with English abstract)
[37] Smith H. Phytochrome transgenics: functional, ecological and biotechnological applications. Semin Cell Biol. 1994, 5: 315–325 [38] Bae G, Choi G. Decoding of light signals by plant phytochromes and their interacting proteins. Annu Rev Plant Biol, 2008, 59: 281–311
[39] Li J, Li G, Wang H, Deng X W. Phytochrome signaling mechanisms. America: American Society of Plant Biologists, 2011. pp 1–26
[40] Quail P H. Phytochrome photosensory signalling networks. Nat Rev Mol Cell Biol, 2002, 3: 85–93
[41] 詹克慧, 李志勇, 侯佩, 习雨琳, 肖阳, 孟凡华, 杨建平. 利用修饰光敏色素信号途径进行品种改良的可行性. 中国农业科学, 2012, 45: 3249−3255
Zhan K H, Li Z Y, Hou P, Xi Y L, Xiao Y, Meng F H, Yang J P. A new strategy for crop improvement through modification of phytochrome signaling pathways. Sci Agric Sin, 2012, 45: 3249−3255 (in Chinese with English abstract)
[42] Boylan M T, Quil P H. Oat phytochrome is biologically active in transgenic tomatoes. Plant Cell, 1989, 1: 765−773
[43] Nagatani A, Kay S A, Deak M, Chua N H, Furuya M. Rice type I phytochrome regulates hypocotyl elongation in transgenic tobacco seedlings. Proc Natl Acad Sci USA, 1991, 88: 5207−5211
[44] Garg A K, Sawers R J, Wang H, Kim J K, Walker J M, Brutnell T P, Parthasarathy M V, Vierstra R D, Wu R J. Light-regulated overexpression of an Arabidopsis phytochrome A gene in rice alters plant architecture and increases grain yield. Planta, 2006, 223: 627−636
[45] Thiele A, Herold M, Lenk I, Quail P H, Gatz C. Heterologous expression of Arabidopsis phytochrome B in transgenic potato influences photosynthetic performance and tuber development. Plant Physiol, 1999, 120: 73−82
[46] Putterill J, Robson F, Lee K, Simon R, Coupland G. The CONSTANS gene of Arabidopsis promotes flowering and encodes a protein showing similarities to zinc finger transcription factors. Cell, 1995, 80: 847–857
[47] Suárez-López P, Wheatley K, Robson F, Onouchi H, Valverde F, Coupland G. CONSTANS mediates between the circadian clock and the control of flowering in Arabidopsis. Nature, 2001, 410: 1116–1120
[48] Guo H, Yang H, Mockder T C, Lin C. Regulation of flowering time by Arabidopsis photoreceptors. Science, 1998, 279: 1360–1363
[49] Pineiro R P, Coupland G. The control of flowering time and floral identity in Arabidopsis. Plant Physiol, 1998, 17: 1–8
[50] Samach A, Onouchi H, Gold S E, Ditta G S, Schwarz-Sommer Z, Yanofsky M F, Coupland G. Distinct roles of CONSTANS target genes in reproductive development in Arabidopsis. Science, 2000, 288: 1613–1616
[51] 王翠玲, 程芳芳, 孙朝晖, 库丽霞, 陈晓, 陈彦惠. 玉米光周期敏感性的遗转特性及相关基因的研究进展. 玉米科学, 2008, 16: 11–14
Wang C L, Cheng F F, Sun Z H, Ku L X, Chen X, Chen Y H. Advances in genetic research and related genes of photoperiod sensitivity in maize. J Maize Sci, 2008, 16: 11–14 (in Chinese with English abstract)
[52] 李思远, 陈晓, 王新涛, 陈彦惠. 玉米光周期敏感类Hd6基因的克隆和实时定量表达分析. 作物学报, 2008, 34: 713−717
Li S Y, Chen X, Wang X T, Chen Y H. Clone and quantitative analysis by real-time RT-PCR of photoperiod sensitive gene Hd6-like in maize. Acta Agron Sin, 2008, 34: 713−717 (in Chinese with English abstract)
[53] Yu R M, Zhou Y, Xu Z F, Chye M L, Kong R Y. Two genes encoding protein phosphatase 2A catalytic subunits are differentially expressed in rice. Plant Mol Biol, 2003, 51: 295–311
URL: http://www.cnki.net/kcms/detail/11.1809.S.20151207.1121.022.html
Molecular Cloning of ZmPP6C Gene and Its Expression Patterns in Response to Light and Stress Treatments in Maize (Zea mays L.)
YUAN Huan-Huan1,2,**, SUN Guang-Hua1,2,**, YAN Lei2, GUO Lin2, FAN Xiao-Cong1,2, XIAO Yang3, MENG Fan-Hua2, SONG Mei-Fang2,4, ZHAN Ke-Hui1, YANG Qing-Hua1,*, and YANG Jian-Ping1,2,*
1College of Agronomy, Henan Agricultural University / Collaborative Innovation Center of Henan Grain Crops, Zhengzhou 450002, China;2Institute of Crop Science, Chinese Academy of Agricultural Sciences, Beijing 100081, China;3Graduate School, Chinese Academy of Agricultural Sciences, Beijing 100081, China;4Beijing Radiation Center, Beijing 100875, China
Abstract:PP6C is the catalytic subunits of Ser/Thr protein phosphatase 6 (PP6) gene, which plays important roles in auxin transport polarity, ABA (abscisic acid) signal transduction, flowering time control though light signaling pathway. To clarify structural characteristics of PP6C protein and the evolution relationships among plant PP6C homologs, we cloned ZmPP6C gene by RT-PCR. The open reading frame (ORF) of ZmPP6C possesses 912 nucleotides and encodes 303 amino acid residues with one PP2Ac domain (the catalytic subunits of Ser/Thr protein phosphatase 2A). Phylogenetic analysis indicated that ZmPP6C belongs to the same branch with the PP6C of Sorghum bicolor, and shows high similarity to all PP6C proteins from other monocotyledons and dicotyledons. Further quantitative RT-PCR (qRT-PCR) assays indicated that ZmPP6C was highly expressed in leaf and lowly
in stem, stamen, pulvinus, sheath, and pedical. ZmPP6C transcription abundances could respond to different light and circadian treatments (both long-day and short-day conditions), especially to the light transitions from the dark to far-red or red light condition. In addition, ZmPP6C transcription abundances were up-regulated by high osmosis, high salt and water logging. Our results suggested that ZmPP6C may be involved in light signaling pathway, flowering time control, and abiotic stress response in maize, and its roles in crop improvement are worthy of more exploration in the future.
Keywords:Zea mays; ZmPP6C; Expression patterns; Phytochrome; Light signaling pathway; Abiotic stress
收稿日期Received(): 2015-08-27; Accepted(接受日期): 2015-11-20; Published online(网络出版日期): 2015-12-07.
通讯作者*(Corresponding authors): 杨青华, E-mail: yangqh2000@163.com, Tel: 0371-63555778; 杨建平, E-mail: yangjianping02@caas.cn, Tel: 010-82105859**同等贡献(Contributed equally to this work)
DOI:10.3724/SP.J.1006.2016.00170
