基于单因素试验的大豆CDDPPCR反应体系优化
2015-10-21李强高聚林苏二虎廉博牛敏邵志壮谢田
李强 高聚林 苏二虎 廉博 牛敏 邵志壮 谢田

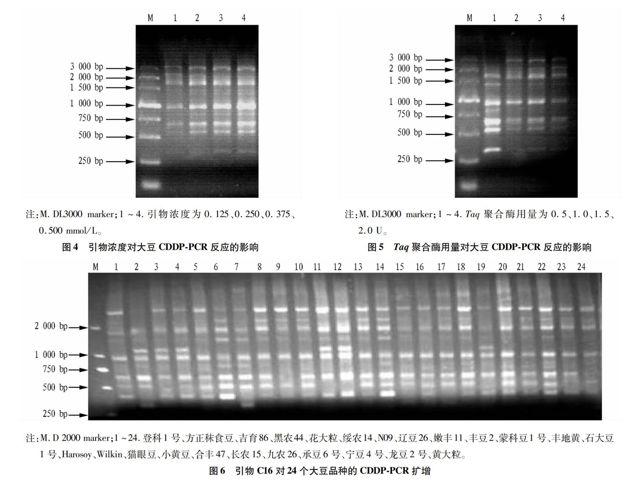
摘要 [目的] 优化大豆CDDP-PCR反应体系。[方法]采用单因素试验方法对影响大豆CDDPPCR反应的Mg2+浓度、Taq DNA 聚合酶用量、引物浓度、dNTPs浓度和模板DNA用量等因素进行优化。[结果]优化后的大豆CDDPPCR体系为:Mg2+浓度2.0 mmol/L、Taq聚合酶用量1.5 U、引物浓度0.375 μmol/L、dNTPs浓度0.3 mmol/L、DNA模板用量40 ng。运用24个大豆品种验证了该反应体系稳定、可靠。[结论]该反应体系的建立为大豆种质遗传多样性分析、遗传连锁图谱构建及分子标记辅助育种奠定了基础。
关键词 大豆;CDDP;单因素试验;体系优化
中图分类号 S565.1 文献标识码 A 文章编号 0517-6611(2015)03-032-03
Optimization of CDDPPCR System by Single factor Experiment Design in Soybean
LI Qiang1, GAO Julin2, SU Erhu1et al
(1.Inner Mongolia Academy of Agricultural and Animal Husbandry Sciences, Huhhot, Inner 010031;2. College of Agronomy, Inner Mongolia Agricultural University, Huhhot, Inner 010019)
Abstract [Objective] The aim was to optimize CDDPPCR system by single factor experiment design in soybean. [Method] Single factor experiment design were applied to optimize CDDPPCR amplification system of soybean in five factors such as Mg2+, Taq DNA polymerase, primer, dNTPs and template DNA. [Result]The suitable reaction system was obtained, that is 20 μl containing 2.0 mmol/L Mg2+, 1.5 U Taq polymerase, 0.375 μmol/L primer, 0.3 mmol/L dNTPs, 40ng DNA template. The optimized CDDPPCR system was tested on 24 soybeans, and the result was stable and reliable. [Conclusion]The system provides the basis for evaluation of genetic diversity, construction of genetic linkage map and molecular marker assisted breeding for the soybean.
Key words Soybean; CDDP; Single factor experiment; System Optimization
基金項目 内蒙古自治区科技创新引导奖励资金项目(20111707);内蒙古农牧业科学院青年创新基金项目(2014QNJJN01)。
作者简介 李强(1982- ),男,内蒙古巴彦淖尔人,助理研究员,博士,从事大豆育种、栽培研究。
*通讯作者,研究员,从大豆育种、栽培研究。
收稿日期 20141215
大豆是世界上重要的粮食、油料、饲料兼用作物,是人类食用油和植物蛋白的主要来源[1]。大豆起源于我国,具有悠久的栽培历史,经过长期自然与人工选择,产生了丰富的遗传变异类型。但品种退化、品种混乱严重给种质区分和大豆育种工作带来了诸多困难,而分子标记技术可有效地进行品种鉴定、遗传多样性研究,并能指导育种实践[2]。前人已先后应用RAPD[3]、AFLP[4]、SSR[5]、ISSR[6]、SRAP[7]等分子标记技术对大豆种质遗传多样性、亲缘关系鉴定及杂交后代鉴定等方面进行了研究。但这些标记技术都是传统意义上的随机DNA分子标记,随着植物基因组学和大规模测序技术的深入发展,NCBI、SOYBASE、GDB等公共基因组、蛋白组数据库信息爆炸式增长,研究者应用这些DNA/ cDNA相关序列信息设计、开发了一大批目的基因分子标记,这类标记与目标基因关联密切,针对性强、效率高,且该类技术在不同物种间可以通用[8]。目的基因分子标记技术有利于实现对植物特定性状进行标记,对重要性状QTL定位研究也具有特殊价值。因此,建立一套简单、有效、实用的目的基因分子标记技术对大豆亲缘关系鉴定、遗传多样性及分子标记辅助育种具有重要意义[9]。
来源保守DNA序列多态性(Conserved DNA-derived polymorphism,CDDP)标记是2009年Collard等[10]提出的一种基于DNA保守序列的新型目的基因分子标记方法。其原理是根据一些功能得到验证的重要基因或基因家族中的保守氨基酸序列对应的DNA保守序列在不同物种间也是相当保守的,利用多重比较软件(CLUSTAL W、Genedoc、DNAman等)对来自不同物种的功能基因序列或功能蛋白质序列进行多重比较,找出不同物种中同时存在的保守DNA或蛋白质序列,为引物的设计提供锚定位点。目前国内外CDDP分子标记应用很少,仅在水稻[10]、欧白英[11]、牡丹[12]、菊花[13]等作物上成功应用,利用正交设计建立了大豆CDDP-PCR反应体系[14],笔者以大豆为材料,采用单因素试验方法进一步优化大豆CDDP-PCR反应体系,对正交设计试验进行验证,建立一套稳定、有效的标记体系,为研究大豆的遗传多样性、遗传图谱构建及分子标记辅助育种提供新的技术手段。
