原发性肝癌的表观遗传学及其治疗
2015-10-13孙凌云李星逾孙志为
孙凌云,李星逾,孙志为
1.昆明理工大学医学院,昆明 650500;
2.昆明理工大学附属医院肝胆外科,昆明 650032
原发性肝癌的表观遗传学及其治疗
孙凌云1,2,李星逾2,孙志为2
1.昆明理工大学医学院,昆明 650500;
2.昆明理工大学附属医院肝胆外科,昆明 650032
肝癌是一种严重危害人类健康的恶性疾病,在全世界患癌人群中,肝癌的发生率排第五,死亡率排第二。原发性肝癌(Hepatocellular carcinoma,HCC)是最普遍的肝癌组织学亚型,属于异质性疾病,对其治疗涉及遗传学、基因组学、环境毒理学等多个领域。尽管许多分子靶向治疗药物如索拉菲尼等已经进入临床应用并证明有效,但细胞毒性等负效应不容忽视,目前迫切需要新的治疗靶点和药物高效并选择性的杀伤肝癌细胞。大量证据表明,肝脏肿瘤的发生和发展与表观遗传学密切相关,DNA甲基化、组蛋白修饰、miRNA表达的异常及表观遗传相关基因表达的异常都是HCC中显著的表观遗传异常现象。表观治疗药物可能会逆转异常基因的表达,从而使HCC的发生和发展得以控制。文章综述了HCC表观遗传学治疗方面的研究进展,展望了未来利用类似的疗法治疗肝癌的潜力。
原发性肝癌;表观遗传学;DNA甲基化;组蛋白修饰;miRNA;lncRNA
肝癌是一种严重危害人类健康的恶性疾病,在全世界患癌人群中,肝癌的发生率排第五,死亡率排第二。肝癌在南亚和东南亚尤其流行,确诊病例占全球50%以上。此外,全球受影响最严重的5个地区还包括非洲中部、西部和南部[1]。原发性肝癌(Hepatocellular carcinoma,HCC)是最普遍的肝癌组织学亚型。在过去的10年间,美国HCC的发病率已经增至3倍,这种增长与高频率丙型肝炎病毒(Hepatitis C virus,HCV)感染密切相关[2,3]。患HCC的危险因素主要包括慢性乙型肝炎病毒(Hepatitis B virus,HBV)感染、丙型肝炎病毒感染、过量饮酒、糖尿病、非酒精性脂肪肝以及饮食摄入黄曲霉素[4]等。
1 HCC治疗所面临的挑战
HCC是一种异质性疾病,对其治疗涉及遗传学、基因组学、环境毒理学等多个领域。临床上面临的主要挑战之一是在早期无法检测到HCC的发生,患者经常在晚期才被检查出患病而确诊,延误了早期治疗的最佳时期,从而限制了治疗方法的选择并且导致预后较差、治疗效果不理想[5]。目前,HCC的治疗方法主要包括:(1)外科手术切除肿瘤和肝移植;(2)利用射频消融术或冷冻消融术进行最小程度的侵入性外科手术;(3)直接向肝内注射药物进行肝动脉栓塞化疗[6]。然而,由于存在以下原因,如:缺乏器官捐赠者,仅有少部分的患者适合进行手术切除,术后复发率高以及存在诸如肝硬化、HBV和HCV感染的潜在并发症[7]等,限制了外科手术以及肝动脉栓塞化疗的应用。
到目前为止,只有部分基于分子水平的疗法在HCC的临床治疗上是可行的。2007年,索拉菲尼获美国食品和药物管理局批准用于晚期HCC的治疗。这种多酪氨酸激酶抑制剂通过干扰血管内皮生长因子信号通路最终阻断肿瘤血管的生成。临床试验表明,该药使患者中位存活期延长了3个月[8]。亚非地区的一组患者经索拉菲尼治疗后,中位总生存期从4.2个月延长到6.5个月[9]。进行中的临床试验正在结合其他药物测试索拉菲尼的疗效[10]。表皮生长因子受体、肝细胞生长因子及其受体c-Met、血小板生长因子受体、哺乳动物类雷帕霉素靶蛋白均参与了与生长因子有关的分子通路[11],针对这些靶点的其他药物也在研究过程中,但这些药物对HCC的治疗效果都不太稳定,或引起严重的不良反应。
2 表观遗传学与肝癌的关系
表观遗传学是研究在不改变DNA序列的前提下,通过某些机制引起可遗传的基因表达或细胞表型变化的学科。表观遗传学包括DNA甲基化(DNA methylation)、基因组印记(Genomic imprinting)、母体效应(Maternal effects)、基因沉默(Gene silencing)、核仁显性、休眠转座子激活和RNA编辑(RNA editing)等。表观遗传机制包括基因组DNA的修饰(DNA中胞嘧啶碱基的甲基化)、组蛋白尾部的化学修饰以及非编码miRNA的调控。在细胞分裂过程中,这些表观遗传修饰保守传递给子代细胞从而使“细胞的记忆”得以维持[12]。
DNA甲基转移酶(DNAmethyltransferases,DNMTs)催化甲基基团(CH3)加入到5'端胞嘧啶核苷酸上。基因启动子区的CpG岛一般处于非甲基化状态,当其表达量异常增高时,会造成某些基因沉默,这些基因包括DNA修复基因、抑癌基因、细胞凋亡基因、细胞周期调节基因等,其中抑癌基因表达沉默是诱发肿瘤的重要因素之一。DNA甲基化导致转录后基因沉默的机制有两种:(1)位于CpG岛上的甲基化在空间上阻碍转录因子与它们各自基因启动子的同源结合位点相结合[13];(2)发生甲基化后,甲基化DNA结合蛋白可以和该段甲基化DNA相结合,从而抑制转录[14]。研究发现,在许多癌症中都存在由DNA甲基化介导的基因沉默。正常的DNA甲基化能够维持基因的稳定性,减少基因组中因同源重组引起的染色体缺失、重复出现等。所以,基因组出现低甲基化状态时也可诱发肿瘤。癌症的发生通常伴随有基因组整体低甲基化以及抑癌基因启动子的高甲基化[15]。一系列的研究表明,表观遗传学改变与肝癌发生和进展密切相关[16]。
近期多个研究报道了肝癌中启动子甲基化的基因,基因组甲基化图谱研究证实:与癌旁正常肝细胞相比,HCC肿瘤细胞呈现出特殊的甲基化模式。Hernandez-Vargas等[17]利用基因芯片检测了30位患有HBV或HCV感染引起的HCC的患者体内1505个CpG位点,发现HCC肿瘤细胞呈现出特殊的DNA甲基化模式,并且DNA甲基化状态与主要感染因素和肿瘤发展进程密切相关。尤其是HCC细胞中某些基因(如APC、RASSFIA、CDKN2A和FZD7)启动子区甲基化状态与癌旁正常肝脏组织明显不同,特别是DNMT1启动子甲基化与肿瘤的低分化密切相关。Song等[18]测定了27位HCC患者肿瘤组织以及癌旁正常肝脏组织的DNA甲基化水平,结果发现启动子区CpG岛DNA甲基化位点显著富集,这些位点在细胞生长、基因表达、细胞凋亡以及癌变的信号通路中发挥着重要作用,其中BMP4、CDKN2A、GSTP1和 NFATC1基因启动子区CpG岛DNA甲基化水平最高。Shen等[19]对部分HBV阳性感染的HCC患者血浆进行了全基因组甲基化研究,发现DAB2IP、BMP4、ZFP41、SPDY1和CDKN2A为5大高甲基化基因,而CCL20、ATK3、SCGB1D1、WFDC6和PAX4为5大低甲基化基因。
目前,对于肝癌组蛋白修饰的研究集中于组蛋白中赖氨酸残基的乙酰化以及甲基化。组蛋白乙酰化通常与转录激活正相关,但甲基化可能伴有转录激活或转录抑制,这取决于组蛋白修饰的位点。研究发现,在基因的表达过程中,组蛋白乙酰化和去乙酰化之间的平衡十分重要,组蛋白去乙酰化和转录受阻之间的失衡与肿瘤的发生或抑制直接相关;目前对于HCC中的组蛋白甲基化状态的评估仅局限于HCC临床病理特征的相关研究,主要运用蛋白质的半定量检测方法如免疫组化或者蛋白质印迹法。HCC肿瘤中组蛋白H3的第4位赖氨酸的高甲基化与患者总生存期短以及预后差密切相关[20]。另有研究表明,组蛋白H3的第27位赖氨酸的高甲基化导致预后更差,并且增强了肿瘤的侵袭特征,包括血管生成、肿瘤增大、多样癌变和肿瘤低分化[21]。肝癌细胞发生各种类型的组蛋白修饰异常,其中一个重要原因是组蛋白修饰酶表达失调。这些修饰酶包括组蛋白去乙酰化酶(Histone deacetylases,HDACs)I类亚型HDACl、HDAC2、HDAC3和SIRTl,III类HDAC和组蛋白甲基转移酶(Histone methyltransferases, HMTs)SMYD3、RIZl和EZH2等。组蛋白去乙酰化酶能够清除靶蛋白赖氨酸残基乙酰基基团,并且在通过染色质重塑协调细胞内部信号转导途径相互作用的过程中扮演着重要角色。所以,通过抑制组蛋白修饰酶表达失调来阻断肝癌发生有望成为肝癌治疗的新途径。
此外,对于肿瘤发生、发展、侵袭和转移的分子机制的探索已进入一个新的里程,microRNA (miRNA)与肿瘤关系的研究是其中的一个热点。miRNA是一种能够调控基因表达的短链非编码RNA,短发夹RNA(Short hairpin RNA,shRNA)可被核酸酶剪切形成miRNA或产生能够进行RNA干扰的siRNA。研究发现,越来越多的miRNA参与编码基因的表达调控,这些基因参与发育、细胞分化、代谢调控、信号转导、细胞增殖和凋亡等一系列生物学过程,这些生物学过程的异常最终导致肿瘤的发生和发展。某些miRNA参与了抵抗病毒HCV和PFV-1的抗病毒防御机制,说明miRNA在病毒感染宿主的过程中发挥着重要的作用[22,23]。Lagos-Quintana等[24]在miRNA文库筛选中,发现miRNA抑制了HepG2细胞中HBV病毒的表达和复制。miRNA在肝脏中表达量最高的是miR-122[25],其在HCC细胞中普遍下调[26,27],因此可以考虑通过恢复miR-122的表达实现其作为抑癌基因[28]的作用。相反,也有报道称miR-1的过表达增强了HBV病毒的复制,而且HBx蛋白抑制了P53介导的miRNA-148a的表达,最终导致人造血相关的PBX相互作用蛋白质(Hematopoietic PBX—interacting protein,HPIP)的表达下降[29]。这些研究均表明,miRNA表达水平的改变在HCC发展过程中具有重要意义。
长链非编码RNA(Long non-coding RNA,lncRNA)是长度在200~100000 nt之间的RNA分子,属于非编码RNA的亚群,通过不同的机制从分子水平发挥调控功能[30]。越来越多的证据表明,lncRNA与很多细胞功能相关并且在许多肿瘤癌变的过程中发挥重要作用[31]。已有研究表明,20%的lncRNA与PRC2有关,lncRNA通过PRC2募集和引导染色质修饰复合物与特定的基因组区域结合从而调控基因转录[32]。lncRNA类似于转录活化子/抑制子能够直接与各种配体相结合。例如lncRNA TERRA可以直接与人的端粒酶结合并抑制端粒酶活性[33]。此外,lncRNA也可作为诱饵与miRNA竞争结合位点从而调节靶基因的表达[34]。lncRNA HULC是最先被检测到在HCC患者血液中高度上调的lncRNA[35],通过下调抑癌基因p18促使肝癌细胞大量增殖[36]。研究还发现,HBx下调一种被称为lncRNA-Dreh的lncRNA,这种抑癌因子在体内和体外均可抑制HCC细胞的增殖和代谢[37]。大量HCC患者体内的lncRNA MALAT-1[38]和lncRNA HOTAIR[39]高度上调,特别是 lncRNA HOTAIR常被作为肝脏移植后肿瘤复发的预后诊断标记物。HCC患者体内高表达的lncRNA MVIH[40]和lncRNA HEIH[41]分别与血管生成和肿瘤复发密切相关,其中lncRNA HEIH能够与EZH2结合,从而下调抑癌基因p16的表达[41]。
3 治疗肝癌的表观遗传学药物
传统的分子靶向治疗方法主要致力于抑制失控基因表达和切断错误的信号通路,相比之下,表观遗传学药物通过逆转表达异常基因的甲基化状态以及组蛋白修饰状态进行特异性治疗[42]。目前共有4种经FDA批准的表观遗传学药物,其中包括两种甲基转移酶(DNMT)抑制剂:5-氮杂胞苷和地西他滨,以及两种组蛋白去乙酰化酶(HDAC)抑制剂:伏立诺他和丙戊酸(Valporic acid)。这些药物已经成功用于治疗血液性肿瘤,特别是治疗一种骨髓失去造血功能的血癌:骨髓增生异常综合征[43]。低剂量的5-氮杂胞苷和地西他滨对于早期白血病细胞[44,45]以及从早期管腔型乳腺癌组织[44]中分离得到的细胞表现出抗肿瘤效应。CD34+过高是肿瘤复发和产生药物抗性的起因,然而5-氮杂胞苷和地西他滨能降低白血病细胞和形成性非黏附性乳腺球群细胞中CD34+的表达[46]。针对HCC中表观遗传学改变的药物见表1。
3.1 治疗HCC的DNA甲基化抑制剂
利用离体细胞[47]和临床前期小鼠模型[48]进行的研究取得了令人瞩目的成果,可为HCC的治疗和处理提供新的途径。Andersen等[48]研究表明,DNMT抑制剂折布拉林(Zebularine)能够有效抑制异种移植小鼠模型中肿瘤的生长。然而对Zebularine产生耐药性的细胞株则表现出癌基因的上调进而促进了肝癌的发生发展,这些发现表明该药仅对部分HCC患者有效。另外,Zebularine也可以抑制癌细胞扩散并诱导HepG2细胞的凋亡。然而,尽管抗凋亡蛋白BCL-2下调,但抑癌基因P53和P21的甲基化水平并不会受到Zebularine的影响,这表明存在某个单独的DNA甲基化调控通路提高了P53和P21蛋白水平[47]。细胞水平的研究表明,5-脱氧氮杂胞苷也具有抗肿瘤作用,主要通过抑制端粒酶的活性同时重新激活P16和c-Myc(原癌基因)的表达从而产生抗肿瘤效果[49]。
DNA甲基化抑制剂能够使异常基因恢复到正常状态[50],适用于表观遗传学治疗癌症[51],但同时也面临着诸多挑战。相对来说,DNA甲基化抑制剂只在癌症早期效果较为显著,这是因为DNA甲基转移酶在恶性肿瘤中的表达量比良性肿瘤高60%,DNA甲基转移酶的表达水平越高,DNA甲基化抑制剂的有效剂量也就越高[52]。5-脱氧氮杂胞苷具有细胞毒性,高浓度的5-脱氧氮杂胞苷会干扰DNA的合成或引起DNA损伤[53]。此外,5-脱氧氮杂胞苷的稳定性较差,极易溶于水,且在胞苷脱氨酶的作用下易脱氨;有限的代谢活性也使其无法作为单一药剂应用于临床[54]。除了使抑癌基因去甲基化,DNA甲基化抑制剂可能对正常的甲基化序列如反转录因子或者其他的转座子也有去甲基化作用[55]。5-脱氧氮杂胞苷使LINE-1元件中的L1-ASP序列发生去甲基化,该序列位于c-MET基因上,因而最终诱导一种不正常的融合转录本表达,该转录本位于内含子LINE-1元件与原癌基因c-MET之间。这种不正常转录本的表观活化可能是人类肿瘤细胞系中药物介导的低甲基化造成的常见后果,在接受去甲基化治疗的患者中也可能存在[55]。此外,5-脱氧氮杂胞苷治疗肿瘤还会产生以下副作用:骨髓抑制、恶心、呕吐以及腹泻,这可能限制了其在临床中特别是对肿瘤的化学预防中的应用[56,57]。与5-脱氧氮杂胞苷相比,折布拉林(Zebularine)细胞毒性低、稳定性高,但其效价低,因此治疗时所需剂量较高[58]。
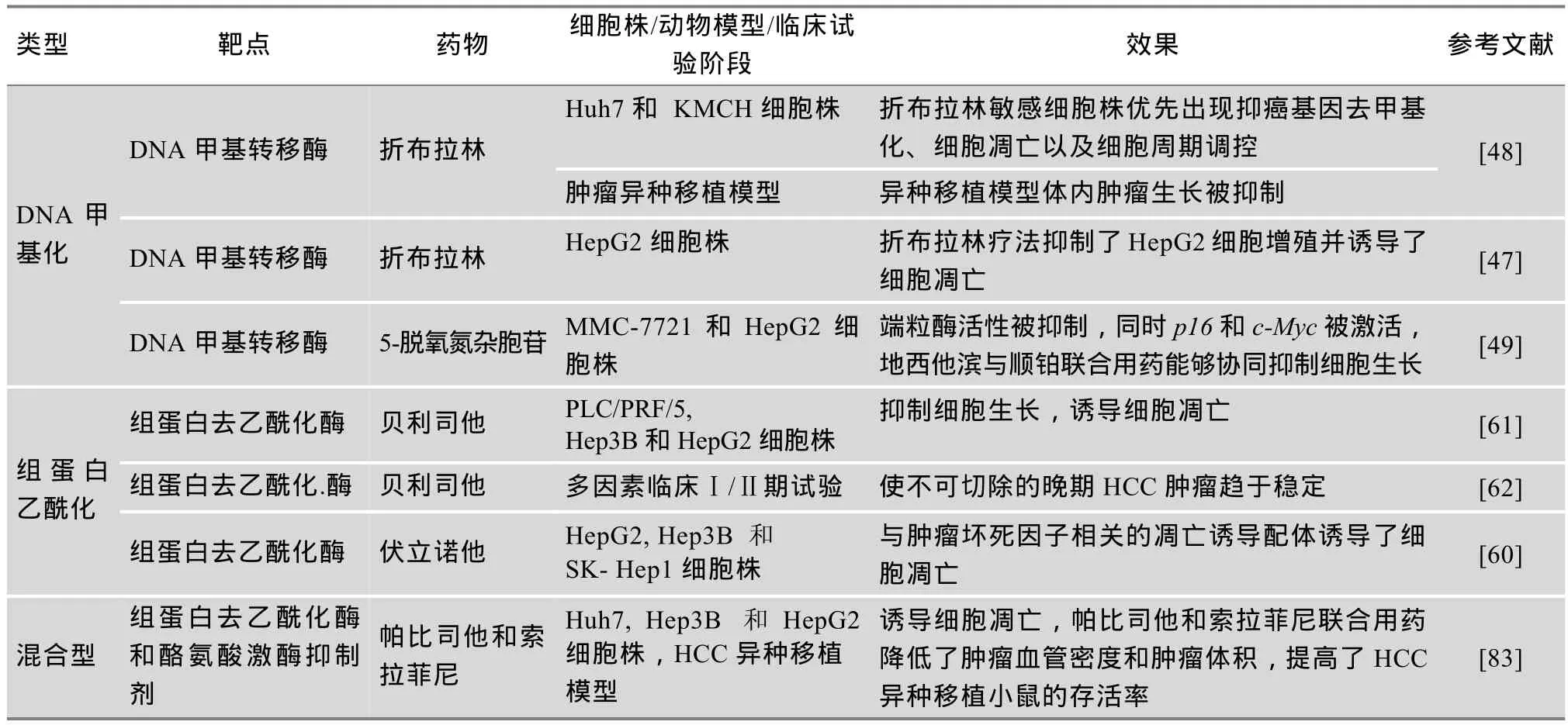
表1 肝癌治疗中针对表观遗传修饰的靶向药物
3.2 治疗HCC的组蛋白去乙酰化酶(HDAC)抑制剂
在临床前期和临床阶段都曾开展过组蛋白去乙酰化酶(HDAC)抑制剂的效应研究。在临床前期研究阶段,伏立诺他(Suberoylanilide hydroxamic acid)使HCC细胞对P53乙酰化[59]以及与肿瘤坏死因子相关的凋亡诱导配体(TNF-related apoptosis-inducing ligand,TRAIL)诱导的细胞凋亡[60]更加敏感,贝利司他(Belinostat)(一种组蛋白去乙酰化酶抑制剂)同样能够抑制HCC细胞株中细胞生长[61]。在一个多因素的临床Ⅰ/Ⅱ期试验中,研究人员发现贝利司他能够使不可进行外科切除手术的晚期HCC患者病情趋于稳定[62]。这一临床试验的另一个重要成果是,发现HR23B可以作为一个潜在的生物标记用于预测人体对于贝利司他的响应[62]。伏立诺他与二氢青蒿素(Dihydroartemisinin)联合用药明显抑制了肝癌移植瘤的生长[63]。值得注意的是,研究人员发现HDAC抑制剂能够诱导细胞调亡同时激活促使肿瘤恶化的基因[64]。这些研究结果表明,想要进一步了解人体对于表观遗传学药物响应的决定性因素,还需要对表观遗传学机制进行深入探究。
最新发现的GSK-1属于表观遗传学药物家族,是第一个化学合成的抑制剂,以组蛋白去甲基化酶JMJD3/UTX(KDM6A/KDM6B)作为靶点,具备特异性和效应性[65]。JMJD3在脂多糖刺激的基础上经巨噬细胞中专有的核因子κB诱导产生,并在炎症反应和表观遗传重编程之间建立重要的联系[66]。这些组蛋白去甲基化酶(JMJD3/UTX)催化H3K27中三甲基标记物的消除。GSK-1能特异性的抑制脂多糖介导的促炎反应,该过程是通过下调肿瘤坏死因子-α(TNF-α)基因的表达实现的,具体的下调机制为GSK-1对TNF-α基因上H3K27的抑制作用以及阻碍转录过程中RNA聚合酶Ⅱ的募集[65]。由HBV和HCV感染引起的慢性炎症反应,或者肥胖,均会导致肝损伤进而缓慢促进HCC的发生发展,HCC患者因此产生高水平的促炎细胞因子TNF-α和白介素(Interleukin,IL)-6[67,68]。由于能够抑制TNF-α的表达,GSK-1作为治疗HCC的特效药具有很大潜能。
3.3 基于miRNA治疗HCC
不同类型的miRNA在肝脏肿瘤中出现调控异常[69],该过程涉及某些细胞内信号通路[70]。另一方面,有研究已经提出利用miRNA作为生物标志物[71]和有效治疗靶点的可能性。基于miRNA作为治疗靶点的药物包括:(1)抗 miRNA的反义寡核苷酸(Anti-miRNAoligonucleotides,AMOS)[72];(2)RNA拮抗剂[73];(3)miRNA类似物[71];(4)特定锁核酸(Locked nucleic acid,LNA)修饰的寡核苷酸(LNA介导的miRNA抑制剂)[74]。
在HCC中miRNA具有抑制肿瘤的作用,如miR-26a,作为抗肿瘤试剂是可行的。有报道称在人类原发性肝癌中存在miR-26a表达水平下降的现象[75],然而在肝脏原癌基因转基因小鼠模型中通过腺病毒介导的基因治疗实现了miR-26a的系统性修复,从而抑制了肿瘤细胞的增殖[76]。miR-26a直接通过靶向下调周期蛋白D2和E2诱导细胞周期停滞进而产生抗肿瘤的作用。重要的是,这种miRNA在主要器官中的细胞毒性是最小的。此外,在利用化学致癌剂二乙基亚硝铵诱导肝癌的小鼠模型中,通过系统性的基因治疗实现了miR-124修复,显著降低了肝脏肿瘤的大小[77],在关键脏器中同样未检测到细胞毒性。
3.4 基于lncRNA治疗HCC
鉴于lncRNAs在癌症中具有重要的调控功能,并且大量研究表明lncRNAs在肝脏肿瘤中表达失调,以lncRNAs为靶点的药物为肝癌治疗提供了新的机遇。原则上,lncRNA的靶向治疗可以通过以下的方法实现:(1)siRNA介导的基因沉默;(2)功能缺失。利用小分子或寡核苷酸抑制剂阻止lncRNAs与蛋白质(如PRC2)的相互作用;(3)结构干扰。利用小分子或寡核苷酸抑制剂来改变或者模拟lncRNAs的二级结构从而竞争其结合位点。但是针对lcnRNAs的药物研究仍处于起步阶段,目前还没有上市[78]。
研究表明,lncRNA MALAT-1[38]和 lncRNA HOTAIR(HOX基因的反义RNA)[39]在大量的HCC患者体内上调,lncRNA HOTAIR可被作为肝脏移植后肿瘤复发的生物标志物进行预后。这两项研究都表明,siRNA介导的 lncRNA MALAT-1和 lncRNA HOTAIR的下调抑制了细胞活性和细胞浸润,促进了TNF-α诱导的细胞凋亡,提高了肿瘤细胞对顺铂和多柔比星的化疗敏感性[39]。
3.5 具有表观遗传特性的天然生物活性化合物
膳食营养素和天然植物产品具有抗肿瘤特性,几年来已应用于癌症治疗。然而近年来诸多研究表明,天然生物活性化合物能够通过表观遗传途径的调控抑制癌症发展[79]。事实上,最近的数据已经清楚表明,各种天然化合物对不同肿瘤细胞系(例如结肠、前列腺、乳腺)中的表观遗传学相关酶和基因(如DNMTs、类HDAC和miRNA)具备特异性调控功能,因此作为治疗性药物和预防药剂具有巨大潜力[80],诸如绿茶多酚EGCG、水飞蓟宾(Silibinin)、喜树碱(CamptothecinL, CPT) 、 双 氢 青 蒿 素(Dihydroartemisinin,DHA)等等[81]。
4 表观遗传学药物与现存治疗方式相结合
在HCC患者体内已经检测到某些HDAC的异常表达以及HDAC3和HDAC5基因拷贝数的增加[82],这就为利用HDAC抑制剂治疗HCC提供了理论依据。在潜伏期肝脏肿瘤模型中,与单独用药相比,泛HDAC抑制剂帕比司他(Panobinostat)和索拉菲尼(Sorafenib)联合用药显著抑制了肿瘤生长[83],并且提高了肿瘤移植小鼠的存活率。具体的分子机制包括细胞凋亡的诱导、组蛋白3的乙酰化、BIRC5基因的下调以及CDH1的上调。该研究结果意味着这种联合用药可能对HCC患者产生良好的治疗效果。
表观遗传学药物除了能在病态条件下改变异常的表观遗传修饰状态,还能通过促进抗原递呈提高宿主免疫原性。DNMT抑制剂5-脱氧氮杂胞苷的全身性用药诱导肿瘤/睾丸抗原(Cancer-testis Antigen, CTA)生成,并且通过提高肿瘤化疗敏感性增强了化疗的效果[84]。在恶性黑色素瘤的炎症微环境中,生黑色素细胞表现出细胞可塑性,即通过肿瘤坏死因子α(TNF-α)引起细胞在已分化型与未分化型状态间进行转换,细胞会逐渐对化疗药物产生抗性[85]。5-脱氧氮杂胞苷在体内的免疫调节活动暗示人们可以将其运用于临床,为黑色素瘤患者设计新的基于CTA的化疗方法[86]。由于HCC通常是在慢性HBV或者HCV感染导致的炎症后发生,并且已检测到在HCC患者体内IL-6[87]和TNF-α[68]的水平升高,因此上述设想可以拓展到HCC的治疗中。
5 展 望
同其他类型的肿瘤相似,HCC与多基因突变和表观遗传畸变有关。然而与基因突变有关的HCC不易被治愈,因此在HCC患者中频繁出现的表观遗传畸变成为新的治疗靶点[88]。迄今为止,全基因组的低甲基化、启动子高甲基化、miRNA的异常表达及其他表观遗传基因(例如EZH2)的异常表达都是HCC中显著的表观遗传异常现象。这些基因在细胞凋亡和细胞周期检查点处发挥着重要的作用,然而表观遗传学药物可能会逆转异常基因的表达,从而使HCC的发生和发展得以控制。令人担忧的是,由于表观遗传药物具有非目标效应,有相反作用的基因可能被激活,因此还需要进一步研究表观遗传药物在多种类型肿瘤中的详细作用机理。目前,多种肿瘤靶向治疗药物已进入临床试验阶段,进一步的研究应该致力于如何利用临床设备更好地筛查得到能最大程度受益于指定治疗方案的患者群体,如根
据病程以及疾病背景进行筛查,同时还要确定使用哪种药物能够取得最佳疗效以及何时给药。值得一提的是,有效的生物标记能够为筛查对药物产生最优反应并且复发可能性低的患者提供有效指导。此外,以往大多数表观遗传药物主要用于癌症晚期,这些药物是否可以被用作预防性药物以及是否可以用于高危患者促使其接受进一步的治疗仍然值得探究。
[1]Jemal A,Bray F,Center MM,Ferlay J,Ward E,Forman D. Global cancer statistics.CA Cancer J Clin,2011,61(2): 69–90.
[2]El-Serag HB.Hepatocellular carcinoma.N Engl J Med, 2011,365(12):1118–1127.
[3]Altekruse SF, McGlynn KA, Reichman ME. Hepatocellular carcinoma incidence, mortality, and survival trends in the United States from 1975 to 2005.J Clin Oncol,2009,27(9):1485–1491.
[4]El-Serag HB,Rudolph KL.Hepatocellular carcinoma: epidemiology and molecular carcinogenesis. Gastroenterology,2007,132(7):2557–2576.
[5]Gish RG. Hepatocellular carcinoma: overcoming challengesin diseasemanagement.Clin Gastroenterol Hepatol,2006,4(3):252–261.
[6]Maluccio M,Covey A.Recent progress in understanding, diagnosing,and treating hepatocellular carcinoma.CA Cancer J Clin,2012,62(6):394–399.
[7]Tsim NC,Frampton AE,Habib NA,Jiao LR.Surgical treatment for liver cancer.World J Gastroenterol,2010, 16(8):927–933.
[8]Llovet JM,Ricci S,Mazzaferro V,Hilgard P,Gane E, Blanc JF,de Oliveira AC,Santoro A,Raoul JL,Forner A, Schwartz M,Porta C,Zeuzem S,Bolondi L,Greten TF, Galle PR,Seitz JF,Borbath I,Häussinger D,Giannaris T, Shan M,Moscovici M,Voliotis D,Bruix J.Sorafenib in advanced hepatocellular carcinoma.N Engl J Med,2008, 359(4):378–390.
[9]Cheng AL,Kang YK,Chen Z,Tsao CJ,Qin S,Kim JS, Luo R,Feng J,Ye S,Yang TS,Xu J,Sun Y,Liang H,Liu J, Wang J,Tak WY,Pan H,Burock K,Zou J,Voliotis D, Guan Z.Efficacy and safety of sorafenib in patients in the Asia-Pacific region with advanced hepatocellular carcinoma: a phase III randomised, double-blind,placebo-controlled trial.Lancet Oncol,2009, 10(1):25–34.
[10]Kelley RK,Nimeiri HS,Munster PN,Vergo MT,Huang Y, Li CM,Hwang J,Mulcahy MF,Yeh BM,Kuhn P,Luttgen MS,Grabowsky JA,Stucky-Marshall L,Korn WM,Ko AH, Bergsland EK, Benson AB, Venook AP. Temsirolimus combined with sorafenib in hepatocellular carcinoma:a phase I dose-finding trial with pharmacokinetic and biomarker correlates.Ann Oncol, 2013,24(7):1900–1907.
[11]Chan SL, Yeo W. Targeted therapy of hepatocellular carcinoma: present and future. J Gastroenterol Hepatol,2012,27(5):862–872.
[12]Pogribny IP,Rusyn I.Role of epigenetic aberrations in the development and progression of human hepatocellularcarcinoma.Cancer Lett,2014,342(2): 223–230.
[13]Hlady RA,Tiedemann RL,Puszyk W,Zendejas I,Roberts LR,Choi JH,Liu C,Robertson KD.Epigenetic signatures of alcohol abuse and hepatitis infection during human hepatocarcinogenesis. Oncotarget, 2014, 5(19): 9425–9443.
[14]Gao XD,Qu JH,Chang XJ,Lu YY,Bai WL,Wang H,Xu ZX,An LJ,Wang CP,Zeng Z,Yang YP.Hypomethylation oflong interspersed nuclear element-1 promoter is associated with poor outcomes for curative resected hepatocellular carcinoma. Liver Int,2014, 34(1):136–146.
[15]Hinrichsen I,Kemp M,Peveling-Oberhag J,Passmann S, Plotz G,Zeuzem S,Brieger A.Promoter methylation of MLH1,PMS2,MSH2 and p16 is a phenomenon of advanced-stage HCCs.PLoS One,2014,9(1):e84453.
[16]Herceg Z, Paliwal A. Epigenetic mechanisms in hepatocellularcarcinoma:how environmentalfactors influence the epigenome.Mutat Res,2011,727(3):55–61.
[17]Hernandez-Vargas H,Lambert MP,Le Calvez-Kelm F, Gouysse G,McKay-Chopin S,Tavtigian SV,Scoazec JY, Herceg Z.Hepatocellularcarcinomadisplays distinct DNA methylation signatures with potential as clinical predictors.PLoS One,2010,5(3):e9749.
[18]Song MA,Tiirikainen M,Kwee S,Okimoto G,Yu H, Wong LL.Elucidating the landscape of aberrant DNA methylation in hepatocellular carcinoma.PLoS One,2013, 8(2):e55761.
[19]Shen J,Wang S,Zhang YJ,Kappil M,Wu HC,Kibriya MG,Wang Q,Jasmine F,Ahsan H,Lee PH,Yu MW,Chen CJ,Santella RM.Genome-wide DNA methylation profiles in hepatocellular carcinoma.Hepatology,2012,55(6): 1799–1808.
[20]He CC,Xu JY,Zhang JL,Xie D,Ye H,Xiao ZY,Cai MY, Xu K,Zeng YJ,Li HG,Wang J.High expression of trimethylated histone H3 lysine 4 is associated with poor prognosis in hepatocellular carcinoma.Hum Pathol,2012, 43(9):1425–1435.
[21]Cai MY,Hou JH,Rao HL,Luo RZ,Li M,Pei XQ,Lin MC, Guan XY,Kung HF,Zeng YX,Xie D.High expression of H3K27me3 in human hepatocellular carcinomas correlates closely with vascular invasion and predicts worse prognosis in patients.Mol Med,2011,17(1–2):12–20.
[22]Pedersen IM,Cheng G,Wieland S,Volinia S,Croce CM, Chisari FV,David M.Interferon modulation of cellular microRNAs as an antiviral mechanism.Nature,2007, 449(7164):919–922.
[23]LecellierCH,DunoyerP,ArarK,Lehmann-CheJ, Eyquem S,Himber C,Saïb A,Voinnet O.A cellular microRNA mediates antiviral defense in human cells. Science,2005,308(5721):557–560.
[24]Hu W,Wang XJ,Ding XR,Li Y,Zhang XJ,Xie PW,Yang J,Wang SQ.MicroRNA-141 represses HBV replication by targeting PPARA.PLoS One,2012,7(3):e34165.
[25]Lagos-QuintanaM,RauhutR,YalcinA,MeyerJ, Lendeckel W,Tuschl T.Identification of tissue-specific microRNAs from mouse. Curr Biol, 2002, 12(9):735–739.
[26]Kutay H,Bai SM,Datta J,Motiwala T,Pogribny I, Frankel W,Jacob ST,Ghoshal K.Downregulation of miR-122 in the rodent and human hepatocellular carcinomas.J Cell Biochem,2006,99(3): 671–678.
[27]Murakami Y,Yasuda T,Saigo K,Urashima T,Toyoda H, Okanoue T,Shimotohno K.Comprehensive analysis of microRNA expression patterns in hepatocellular carcinoma and non-tumorous tissues. Oncogene,2006,25(17):2537–2545.
[28]Tsai WC,Hsu PW,Lai TC,Chau GY,Lin CW,Chen CM, Lin CD,Liao YL,Wang JL,Chau YP,Hsu MT,Hsiao M, Huang HD,Tsou AP.MicroRNA-122,a tumor suppressor microRNA thatregulates intrahepatic metastasis of hepatocellular carcinoma. Hepatology, 2009, 49(5): 1571–1582.
[29]Xu XJ,Fan ZY,Kang L,Han JQ,Jiang CY,Zheng XF, Zhu ZM,Jiao HB,Lin J,Jiang K,Ding LH,Zhang H, Cheng L,Fu HJ,Song Y,Jiang Y,Liu JH,Wang RF,Du N, Ye QN. Hepatitis B virus X protein represses miRNA-148a to enhance tumorigenesis.J Clin Invest, 2013,123(2):630–645.
[30]Cheetham SW,Gruhl F,Mattick JS,Dinger ME.Long noncoding RNAs and the genetics of cancer.Br J Cancer, 2013,108(12):2419–2425.
[31]Prensner JR,Chinnaiyan AM.The emergence of lncRNAs in cancer biology.Cancer Discov,2011,1(5):391–407.
[32]Khalil AM,Guttman M,Huarte M,Garber M,Raj A, Rivea Morales D,Thomas K,Presser A,Bernstein BE,van Oudenaarden A,Regev A,Lander ES,Rinn JL.Many human large intergenic noncoding RNAs associate with chromatin-modifying complexes and affect gene expression.Proc Natl Acad Sci USA,2009,106(28): 11667–11672.
[33]Redon S,Reichenbach P,Lingner J.The non-coding RNA TERRA is a natural ligand and direct inhibitor of human telomerase.Nucleic Acids Res,2010,38(17):5797–5806.
[34]Saito Y,Hibino S,Saito H.Alterations of epigenetics and microRNA in hepatocellularcarcinoma.HepatolRes, 2014,44(1):31–42.
[35]Panzitt K,Tschernatsch MMO,Guelly C,Moustafa T, Stradner M,Strohmaier HM,Buck CR,Denk H,Schroeder R,Trauner M,Zatloukal K.Characterization of HULC,a novel gene with striking up-regulation in hepatocellular carcinoma, as noncoding RNA. Gastroenterology,2007,132(1):330–342.
[36]Du YM,Kong GY,You XN,Zhang S,Zhang T,Gao YE, Ye LH,Zhang XD.Elevation of highly up-regulated in liver cancer(HULC)by hepatitis B virus X protein promotes hepatoma cell proliferation via down-regulating p18.J Biol Chem,2012,287(31):26302–26311.
[37]Huang JF,Guo YJ,Zhao CX,Yuan SX,Wang Y,Tang GN, Zhou WP,Sun SH.Hepatitis B virus X protein (HBx)-related long noncoding RNA(lncRNA)down-r egulated expression by HBx (Dreh) inhibits hepatocellular carcinoma metastasis by targeting the intermediate filament protein vimentin. Hepatology, 2013,57(5):1882–1892.
[38]Lai MC,Yang Z,Zhou L,Zhu QQ,Xie HY,Zhang F,Wu LM,ChenLM,ZhengSS.Longnon-codingRNA MALAT-1 overexpression predicts tumor recurrence of hepatocellular carcinoma after liver transplantation.Med Oncol,2012,29(3):1810–1816.
[39]Yang Z,Zhou L,Wu LM,Lai MC,Xie HY,Zhang F, Zheng SS.Overexpression of long non-coding RNA HOTAIR predicts tumor recurrence in hepatocellular carcinoma patients following liver transplantation.Ann Surg Oncol,2011,18(5):1243–1250.
[40]Yuan SX,Yang F,Yang Y,Tao QF,Zhang J,Huang G, Yang Y,Wang RY,Yang S,Huo XS,Zhang L,Wang F,Sun SH,Zhou WP.Long noncoding RNA associated with microvascular invasion in hepatocellular carcinoma promotes angiogenesis and serves as a predictor for hepatocellular carcinoma patients’poor recurrence-free survivalafterhepatectomy.Hepatology,2012,56(6): 2231–2241.
[41]Yang F,Zhang L,Huo XS,Yuan JH,Xu D,Yuan SX,Zhu N,Zhou WP,Yang GS,Wang YZ,Shang JL,Gao CF, Zhang FR,Wang F,Sun SH.Long noncoding RNA high expression in hepatocellular carcinoma facilitates tumor growth through enhancer of zeste homolog 2 in humans. Hepatology,2011,54(5):1679–1689.
[42]Yoo CB,Jones PA.Epigenetic therapy of cancer:past,present and future.Nat Rev Drug Discov,2006,5(1):37–50.
[43]Fenaux P,Mufti GJ,Hellstrom-Lindberg E,Santini V, Finelli C,Giagounidis A,Schoch R,Gattermann N,Sanz G,List A,Gore SD,Seymour JF,Bennett JM,Byrd J, Backstrom J,Zimmerman L,McKenzie D,Beach C, Silverman LR.Efficacy of azacitidine compared with that ofconventionalcare regimens in the treatmentof higher-risk myelodysplastic syndromes:a randomised, open-label,phase III study.Lancet Oncol,2009,10(3): 223–232.
[44]Issa JPJ,Garcia-Manero G,Giles FJ,Mannari R,Thomas D,Faderl S,Bayar E,Lyons J,Rosenfeld CS,Cortes J, Kantarjian HM.Phase 1 study of low-dose prolonged exposure schedules of the hypomethylating agent 5-aza-2’- deoxycytidine (decitabine) in hematopoietic malignancies. Blood, 2004, 103(5): 1635–1640.
[45]Issa JPJ,Gharibyan V,Cortes J,Jelinek J,Morris G, Verstovsek S,Talpaz M,Garcia-Manero G,Kantarjian HM. Phase II study of low-dose decitabine in patients with chronic myelogenous leukemia resistant to imatinib mesylate.J Clin Oncol,2005,23(17):3948–3956.
[46]Tsai HC,Li HL,Van Neste L,Cai Y,Robert C,Rassool FV, Shin JJ,Harbom KM,Beaty R,Pappou E,Harris J,Yen RW,Ahuja N,Brock MV,Stearns V,Feller-Kopman D, Yarmus LB,Lin YC,Welm AL,Issa JP,Minn I,Matsui W, Jang YY,Sharkis SJ,Baylin SB,Zahnow CA.Transient low doses of DNA-demethylating agents exert durable antitumor effects on hematological and epithelial tumor cells.Cancer Cell,2012,21(3):430–446.
[47]NakamuraK,AizawaK,NakabayashiK,Kato N, Yamauchi J,Hata K,Tanoue A.DNA methyltransferase inhibitor zebularine inhibits human hepatic carcinoma cells proliferation and induces apoptosis.PLoS One,2013,8(1):e54036.
[48]Andersen JB,Factor VM,Marquardt JU,Raggi C,Lee YH, Seo D,Conner EA,Thorgeirsson SS.An integrated genomic and epigenomic approach predicts therapeutic response to zebularine in human liver cancer.Sci Transl Med,2010,2(54):54ra77.
[49]Tao SF,Zhang CS,Guo XL,Xu Y,Zhang SS,Song JR,Li R, Wu MC, Wei LX. Anti-tumor effect of 5-aza-2’-deoxycytidine by inhibiting telomerase activity in hepatocellular carcinoma cells.World J Gastroenterol,2012,18(19):2334–2343.
[50]Siedlecki P, Zielenkiewicz P. Mammalian DNA methyltransferases.Acta Biochim Pol,2006,53(2): 245–256.
[51]Robert MF,Morin S,Beaulieu N,Gauthier F,Chute IC, Barsalou A,MacLeod AR.DNMT1 is required to maintain CpG methylation and aberrant gene silencing in human cancer cells.Nat Genet,2002,33(1):61–65.
[52]Stefanska B, Salame P, Bednarek A, Fabianowska-Majewska K.Comparative effects of retinoic acid,vitamin D and resveratrol alone and in combination with adenosine analogues on methylation and expression of phosphatase and tensin homologue tumour suppressor gene in breast cancer cells. Brit J Nut, 2012, 107(6):781–790.
[53]Qin TC,Jelinek J,Si JL,Shu JM,Issa JP.Mechanisms of resistance to 5-aza-2’-deoxy-cytidine in human cancer cell lines.Blood,2009,113(3):659–667.
[54]Beumer JH,Eiseman JL,Parise RA,Joseph E,Holleran JL, Covey JM,Egorin MJ.Pharmacokinetics,metabolism, andoral bioavailability ofthe DNA methyltransferase inhibitor 5-fluoro-2’-deoxycytidine in mice.Clin Cancer Res,2006,12(24):7483–7491.
[55]WeberB,KimhiS,HowardG,Eden A,LykoF. Hypomethylation of a LINE-1 antisense promoter in the cMet locus impairs Met signalling through induction of illegitimate transcription. Oncogene, 2010, 29(43):5775–5784.
[56]Kantarjian H,Oki Y,Garcia-Manero G,Huang XL, O’Brien S,Cortes J,Faderl S,Bueso-Ramos C,Ravandi F, Estrov Z,Ferrajoli A,Wierda W,Shan JQ,Davis J,Giles F, Saba HI,Issa JP.Results of a randomized study of 3 schedules of low-dose decitabine in higher-risk myelodysplastic syndrome and chronic myelomonocytic leukemia.Blood,2007,109(1):52–57.
[57]Shen Y,Takahashi M,Byun H,Link A,Sharma N, Balaguer F,Leung H,Boland C,Goel A.Boswellic acid induces epigenetic alterations by modulating DNA methylation in colorectal cancer cells.Cancer Biol Ther, 2012,13(7):542–552.
[58]Foubister V.Drug reactivates genes to inhibit cancer.Drug Discov Today,2003,8(10):430–431.
[59]Carlisi D,Vassallo B,Lauricella M,Emanuele S,D’Anneo A,Di Leonardo E,Di Fazio P,Vento R,Tesoriere G. Histone deacetylase inhibitors induce in human hepatoma HepG2 cells acetylation of p53 and histones in correlation with apoptotic effects.Int J Oncol,2008,32(1):177–184.
[60]Carlisi D,Lauricella M,D’Anneo A,Emanuele S,Angileri L,Di Fazio P,Santulli A,Vento R,Tesoriere G.The histone deacetylase inhibitor suberoylanilide hydroxamic acid sensitises human hepatocellular carcinoma cells to TRAIL-induced apoptosis by TRAIL-DISC activation. Eur J Cancer,2009,45(13):2425–2438.
[61]Ma BBY,Sung F,Tao Q,Poon FF,Lui VW,Yeo W,Chan SL,Chan ATC.The preclinical activity of the histone deacetylase inhibitor PXD101 (belinostat) in hepatocellular carcinoma cell lines.Invest New Drugs,2010, 28(2):107–114.
[62]Yeo W,Chung HC,Chan SL,Wang LZ,Lim R,Picus J, Boyer M,Mo FKF,Koh J,Rha SY,Hui EP,Jeung HC, Roh JK,Yu SC,To KF,Tao Q,Ma BB,Chan AWH,Tong JHM,Erlichman C,Chan ATC,Goh BC.Epigenetic therapy using belinostat for patients with unresectable hepatocellular carcinoma:a multicenter phase I/II study with biomarker and pharmacokinetic analysis of tumors from patients in the Mayo Phase II Consortium and the Cancer Therapeutics Research Group.J Clin Oncol,2012, 30(27):3361–3367.
[63]Zhang CZ,Pan YH,Cao Y,Lai PBS,Liu LL,Chen GG, Yun JP. Histone deacetylase inhibitors facilitate dihydroartemisinininduced apoptosis in liver cancer in vitro and in vivo.PLoS One,2012,7(6):e39870.
[64]Lin KT,Wang YW,Chen CT,Ho CM,Su WH,Jou YS. HDAC inhibitors augmented cell migration and metastasis through induction of PKCs leading to identification of low toxicity modalities for combination cancer therapy.Clin Cancer Res,2012,18(17):4691–4701.
[65]Kruidenier L,Chung CW,Cheng Z,Liddle J,Che K, Joberty G,Bantscheff M,Bountra C,Bridges A,Diallo H, Eberhard D,Hutchinson S,Jones E,Katso R,Leveridge M, Mander PK,Mosley J,Ramirez-Molina C,Rowland P, Schofield CJ,Shep-pard RJ,Smith JE,Swales C,Tanner R, Thomas P,Tumber A,Drewes G,Oppermann U,Patel DJ, Lee K,Wilson DM.A selective jumonjiH3K27 demethylase inhibitormodulatesthe proinflammatory macrophage response.Nature,2012,488(7411):404–408.
[66]Raggi C,Factor VM,Seo D,Holczbauer A,Gillen MC, Marquardt JU,Andersen JB,Durkin M,Thorgeirsson SS. Epigenetic reprogramming modulates malignant properties of human liver cancer.Hepatology,2014,59(6):2251–2262.
[67]Naugler WE,Sakurai T,Kim S,Maeda S,Kim K, Elsharkawy AM,Karin M.Gender disparity in liver cancer due to sex differences in MyD88-dependentIL-6 production.Science,2007,317(5834):121–124.
[68]Park EJ,Lee JH,Yu GY,He GB,Ali SR,Holzer RG, Osterreicher CH,Takahashi H,Karin M.Dietary and genetic obesity promote liver inflammation and tumorigenesis by enhancing IL-6 and TNF expression. Cell,2010,140(2):197–208.
[69]Gramantieri L,Fornari F,Callegari E,Sabbioni S,Lanza G,CroceCM,BolondiL,NegriniM.MicroRNA involvement in hepatocellular carcinoma.J Cell Mol Med, 2008,12(6A):2189–2204.
[70]Borel F,Konstantinova P,Jansen PLM.Diagnostic and therapeutic potential of miRNA signatures in patients with hepatocellular carcinoma. J Hepatol, 2012, 56(6): 1371–1383.
[71]Henry JC, Azevedo-Pouly ACP, Schmittgen TD. MicroRNA replacement therapy for cancer.Pharm Res, 2011,28(12):3030–3042.
[72]Weiler J,Hunziker J,Hall J.Anti-miRNA oligonucleotides (AMOs):ammunition to target miRNAs implicated in human disease?Gene Ther,2006,13(6):496–502.
[73]Krützfeldt J,Rajewsky N,Braich R,Rajeev KG,Tuschl T, Manoharan M,Stoffel M.Silencing of microRNAs in vivo with‘antagomirs’.Nature,2005,438(7068):685–689.
[74]Elmén J,Lindow M,Schütz S,Lawrence M,Petri A,Obad S,Lindholm M,Hedtjärn M,Hansen HF,Berger U, Gullans S,Kearney P,Sarnow P,Straarup EM,Kauppinen S.LNA-mediated microRNA silencing in non-human primates.Nature,2008,452(7189):896–899.
[75]Ji JF,Shi J,Budhu A,Yu ZP,Forgues M,Roessler S,Ambs S,Chen YD,Meltzer PS,Croce CM,Qin LX,Man K,Lo CM,Lee J,Ng IO,Fan J,Tang ZY,Sun HC,Wang XW. MicroRNA expression, survival, and response to interferon in liver cancer.N Engl J Med,2009,361(15): 1437–1447.
[76]Kota J,Chivukula RR,O’Donnell KA,Wentzel EA, Montgomery CL,Hwang HW,Chang TC,Vivekanandan P, Torbenson M,Clark KR,MendellJR,MendellJT. Therapeutic microRNA delivery suppresses tumorigenesis in a murine liver cancer model.Cell,2009,137(6): 1005–1017.
[77]Hatziapostolou M,Polytarchou C,Aggelidou E,Drakaki A, Poultsides GA,Jaeger SA,Ogata H,Karin M,Struhl K, Hadzopoulou-Cladaras M,Iliopoulos D.An HNF4α-miRNA inflammatory feedback circuit regulates hepatocellular oncogenesis.Cell,2011,147(6):1233–1247.
[78][78] Sánchez Y, Huarte M. Long non-coding RNAs:challenges for diagnosis and therapies.Nucleic Acid Ther,2013,23(1):15–20.
[79]Stefanska B,Karlic H,Varga F,Fabianowska-Majewska K, Haslberger AG.Epigenetic mechanisms in anti-cancer actions of bioactive food components—the implications in cancer prevention.Br J Pharmacol,2012,167(2): 279–297.
[80]Hardy TM,Tollefsbol TO.Epigenetic diet:impact on the epigenome and cancer.Epigenomics,2011,3(4):503–518.
[81]Anestopoulos I,Voulgaridou GP,Georgakilas AG,Franco R,Pappa A,Panayiotidis MI.Epigenetic therapy as a novel approach in hepatocellular carcinoma.Pharmacol Ther,2015,145:103–119.
[82]Lachenmayer A,Toffanin S,CabellosL,Alsinet C, HoshidaY,Villanueva A,Minguez B,Tsai HW,Ward SC, Thung S,Friedman SL,Llovet JM.Combination therapy for hepatocellular carcinoma:additive preclinical efficacy of the HDAC inhibitor panobinostat with sorafenib.J Hepatol,2012,56(6):1343–1350.
[83]Li GM,Wang YG,Pan Q,Wang J,Fan JG,Sun C.RNAi screening with shRNAs against histone methylation-related genes reveals determinants of sorafenib sensitivity in hepatocellular carcinoma cells.Int J Clin Exp Pathol, 2014,7(3):1085–1092.
[84]Guo ZS,Hong JA,Irvine KR,Chen GA,Spiess PJ,Liu Y, ZengG,WunderlichJR,NguyenDM,RestifoNP, Schrump DS.De novo induction ofa cancer/testis antigen by 5-aza-2’-deoxycytidine augments adoptive immunotherapy in a murine tumor model.Cancer Res, 2006,66(2):1105–1113.
[85]Landsberg J,Kohlmeyer J,Renn M,Bald T,Rogava M, Cron M,Fatho M,Lennerz V,Wölfel T,Hölzel M,Tüting T. Melanomas resist T-cell therapy through inflammation-induced reversible dedifferentiation.Nature, 2012,490(7420):412–416.
[86]Coral S,Sigalotti L,Colizzi F,Spessotto A,Nardi G, Cortini E,Pezzani L,Fratta E,Fonsatti E,Di Giacomo AM,Nicotra MR,Natali PG,Altomonte M,Maio M. Phenotypic and functional changes of human melanoma xenografts induced by DNA hypomethylation: immunotherapeutic implications.J Cell Physiol,2006, 207(1):58–66.
[87]Porta C,De Amici M,Quaglini S,Paglino C,Tagliani F, Boncimino A,MorattiR,Corazza GR.Circulating interleukin-6 as a tumor marker for hepatocellular carcinoma. Ann Oncol, 2008, 19(2): 353–358.
[88]Ozen C,Yildiz G,Dagcan AT,Cevik D,Ors A,Keles U, TopelH,Ozturk M.Genetics and epigenetics of liver cancer.N Biotechnol,2013,30(4):381–384.
(责任编委:朱卫国)
Progress of epigenetics and its therapeutic application in hepatocellular carcinoma
Lingyun Sun1,2,Xingyu Li2,Zhiwei Sun2
1.Medical College,Kunming University of Science and Technology,Kunming 650500,China;
2.Hepatobiliary Surgery,Affiliated Hospital of Kunming University of Science and Technology,Kunming 650032,China
Liver cancer is a severe harmful disease.It is the fifth most frequently diagnosed cancer and second most frequent cause of cancer deaths worldwide.As the most popular histologic subtype of hepatocellular carcinoma (HCC),primary HCC is a heterogeneous disease whose management requires a multidisciplinary approach combining genetics,genomics and environmental toxicology.Although many molecular targeted therapies such as sorafenib have entered clinical application and proven effective,the cytotoxicity and other negative effects cannot be ignored.There is an urgent need to identify new therapeutic targets and drugs,which can kill HCC cells with high efficiency and specificity.Plenty of evidence suggests that occurrence and development of HCC is closely related with epigenetics. DNA methylation,histone modification,aberrant expression of miRNAs and dysregulated expression of many epigenetic regulatory genes are significantly altered in HCC.Epigenetic therapeutic drugs may reverse abnormal gene expression,thus controlling the occurrence and development of HCC.In this review,we summarize the latest research progresses in epigenetics and its therapeutic application in HCC,and the potential treatments to be used in the future.
hepatocellular carcinoma;epigenetics;DNA methylation;histone modification;miRNA;LncRNA
2014-12-15;
2015-03-24
云南省应用基础研究计划项目(编号:2012WS007)资助
孙凌云,硕士研究生,专业方向:分子靶向治疗肝细胞癌的临床及实验。E-mail:sly84@qq.com
孙志为,教授,研究方向:肝胆外科、微创外科、移植外科、消化道肿瘤。E-mail:2833570686@qq.com
10.16288/j.yczz.14-443
时间:2015-5-13 13:58:26
URL:http://www.cnki.net/kcms/detail/11.1913.R.20150513.1358.002.html
