5种石斑鱼核糖体DNA ITS1序列及其分子系统进化关系
2015-04-11周爱国谢少林陈金涛陈言峰邹记兴
周爱国 , 谢少林 , 陈金涛 , 陈言峰, 邹记兴
(1.华南农业大学 动物科学学院, 广东 广州 510642; 2.清远市北江水产科学研究所, 广东 清远 511510;3.佛山科学技术学院 生命科学学院, 广东 佛山 528231)
核糖体DNA ITS1(The first internal transcribed spacer of ribosomal DNA, rDNA)基因是位于18S与5.8S rDNA的间隔区, 进化速率中等, 是研究近源种、复合种及种内生物型间亲缘关系和系统发育的良好分子标记, 对于种间分化时间短, 亲缘关系较近的石斑鱼物种, 该标记可以很好揭示其物种间的亲缘关系[1]。然而, 目前关于 ITS1序列的研究主要集中在无脊椎动物和植物[2-6]。在鱼类研究上也有相应的报道, Pleyte 等[7]采用ITS1标记对鲑属鱼类7个种研究, 发现其系统进化关系分析结果与形态学、同工酶等研究结果一致。而邱凡等[8]利用ITS1 部分序列研究鲭科(Scombrida)鱼类时发现其系统进化关系与形态学上的分类结果存在分歧, 并对鲭科鱼类分类关系进行了澄清; 国外学者也将 ITS1 部分序列成功应用到鱼类系统发育和进化分析中, 解决了一些物种长期存在争议的分类问题[9-10]。张源真等[11]利用 ITS-1序列分析部分鳚亚目鱼类的分子系统进化关系时发现研究结果与形态学分类一致, 种间具有高度变异性, 可以作为物种分类及鉴别的分子依据。而利用ITS1序列作为分子遗传标记对石斑鱼进行系统发育关系的研究相对较少。Guo等[12]研究云纹石斑鱼(Epinephelus moara)和褐石斑鱼(E.bruneus)的亲缘关系时发现核糖体DNA ITS1 序列是一种稳定可靠的分子标记。
本研究旨在利用rDNA ITS1序列作为分子鉴定手段以区别形态上较为相似的 5种石斑鱼(青石斑鱼(E.awoara)、点带石斑鱼(E.malabaricus)、六带石斑鱼(E.sexfasciatus)、宝石石斑鱼(E.areolatus)、巨石斑鱼(E.tauvina)种类, 并以丽鱼科(Cichlidae),罗非鱼属(Oreochromis)的尼罗罗非鱼(Oreochromis niloticus)为外群, 构建 3种分子系统树, 以期从分子水平获得这 5种石斑鱼的系统进化关系, 为生产上的良种选育和杂交选配提供分子生物学方面的依据。
1 材料与方法
1.1 材料
试验材料的种类及来源见表1, 其中外群尼罗罗非鱼的序列来自GenBank。

表1 研究材料种类及其来源Tab.1 The species and sources of materials
1.2 试验方法
1.2.1 总DNA提取
取适量尾鳍或肌肉组织, STE洗涤3次, 离心条件为3 000 r/min, 每次离心10 min; 倒去上清, 将组织转移到灭菌的匀浆器中, 加入 1 mL STE, 匀浆;将匀浆后的组织悬液转移至5 mL EP管, 加入3 mL细胞裂解液, 摇匀直至出现黏状物; 加入质量浓度为10 mg/mL的RNase A至终质量浓度20 µg/mL, 于37℃消化3 h; 加入质量浓度为20 mg/mL的蛋白酶K 至终质量浓度 100 µg/mL, 50℃消化过夜; 次日以酚/酚-氯仿/异戊醇-氯仿/异戊醇各抽提 1次; 冰乙醇沉淀, 用吸头挑出DNA沉淀, –20℃70%乙醇洗1次, 置 4℃冰箱中使之缓慢干燥; 干燥后溶于适量TE中, 用紫外分光光度计测定其纯度及浓度。
1.2.2 PCR扩增
PCR扩增所用引物为自行设计: 正向引物:5’-GTTCCCCTTGAACGAGGA ATTC-3反向引物:5’-CGCATTTCGCTGCGTTCTTC-3’, 它们分别为18SrDNA的5’端和5.8SrDNA的3’端的保守序列, 由上海生物工程公司合成。PCR扩增反应参照Williams等[13]、Welsh[14]的方法。每一样品的反应总体积50 µL, 其中包括6 µL PCR缓冲液(10 mmol/L Tris-HCl, pH9.0, 50 mmol/L KCl, 22.5 mmol/L MgCl2,0.01%的明胶), 2 μL dNTP混合液(每种 dNTP 0.1 mmol/L), 4 μL 的引物(0.2 μmol/L), 4μL 基因组DNA(50 ng/uL), 2 μL Taq酶(2单位), 32 μL 的蒸馏水。扩增反应条件为: 95℃ 预变性10 min, 94℃ 变性60 s, 50℃退火30 s, 72℃延伸1.5 min, 共计40个循环, 最后72℃延伸10 min。
1.2.3 DNA序列测定
扩增产物经 2.0%的低熔点琼脂糖回收纯化, 克隆于M13噬菌体载体, 具体方法参见金冬雁等[15]。样品委托上海联合基因公司进行双向测序, 序列测定仪器为3700型Bigdye-Terminator全自动序列分析仪。
1.2.4 DNA序列的数据处理
用BioEdit软件[16]对测序结果进行编辑, 然后用CLUSTL X软件[17]对尼罗罗非鱼(O.niloticus)与本文所得 5种石斑鱼序列进行序列重排和同源比较。手工校正后, 通过PAUP 4.0软件[18]对序列结果进行统计分析, 分别用最大简约法(Maximum parsimony method, MP法)和邻接法(Neighbor-Joining mehtod,NJ法)构建分子系统树; 通过Pullze软件[19]用最大似然法(Maximum Likelihood method, ML法)构建分子系统树。用“Bootstrap” 1000次检验各系统树分支的置信度。用Phylip 5.72软件[20]中的DNAdist.exe程序分别计算基于 Kimura-2双参数模型和 Jukes-Cantor单参数模型的遗传距离。
2 结果
2.1 PCR结果
5种石斑鱼样品(青石斑鱼、点带石斑鱼、六带石斑鱼、宝石石斑鱼、巨石斑鱼)经PCR扩增都分别得到约 800 bp(包括引物)的目的条带(图1), 测序获得的序列于NCBI进行在线比对分析, 证明是rDNA ITS1区序列。

图1 5种石斑鱼样品rDNA ITS1区序列PCR扩增检测结果Fig.1 PCR results of rDNA ITS1 regions of 5 kinds of groupers
2.2 序列组成分析和遗传距离
测出的 5种石斑鱼 ITS1区序列长度范围为805~814 bp, 其中 18S rDNA长度为 214 bp, 5.8S rDNA长度为63 bp, 这两类rDNA序列在被测的5种石斑鱼上完全一致, 产生序列变异的ITS1长度分别为: 青石斑鱼536 bp、点带石斑鱼537 bp、六带石斑鱼528 bp、宝石石斑鱼532 bp、巨石斑鱼535 bp。
所分析的5个石斑鱼样品rDNA ITS1序列的碱基组成比较接近(表2), 整个碱基组成中A+T的含量低于 C+G的含量, 其中, A+T含量最低为宝石石斑鱼(37.6%), 最高为六带石斑鱼 41.5%; 单个碱基组成中, A的含量较低, C的含量较高, A含量最低为宝石石斑鱼(17.3%), C含量最高为点带石斑鱼和青石斑鱼, 均为33%。

表2 5种石斑鱼rDNA ITS1序列的碱基组成Tab.2 Base composition of rDNA ITS1 sequences of 5 Kinds of groupers
利用Kimura-2双参数模型计算的5种石斑鱼之间的遗传距离在 0.0000~0.3002, 其中, 宝石石斑鱼与青石斑鱼的遗传距离最大(0.3002), 而点带石斑鱼与青石斑鱼的遗传距离几乎为 0.0000; 用 Jukes-Cantor单参数模型计算的 5种石斑鱼的遗传距离在0.0000~0.2970, 也是宝石石斑鱼与青石斑鱼的遗传距离最大(0.2970), 而点带石斑鱼与青石斑鱼的遗传距离几乎为0.0000。从表3可以看出2种遗传模型计算的结果基本一致, 说明这5种石斑鱼rDNA ITS1序列的碱基平均转换数与颠换数之比约为1.5。
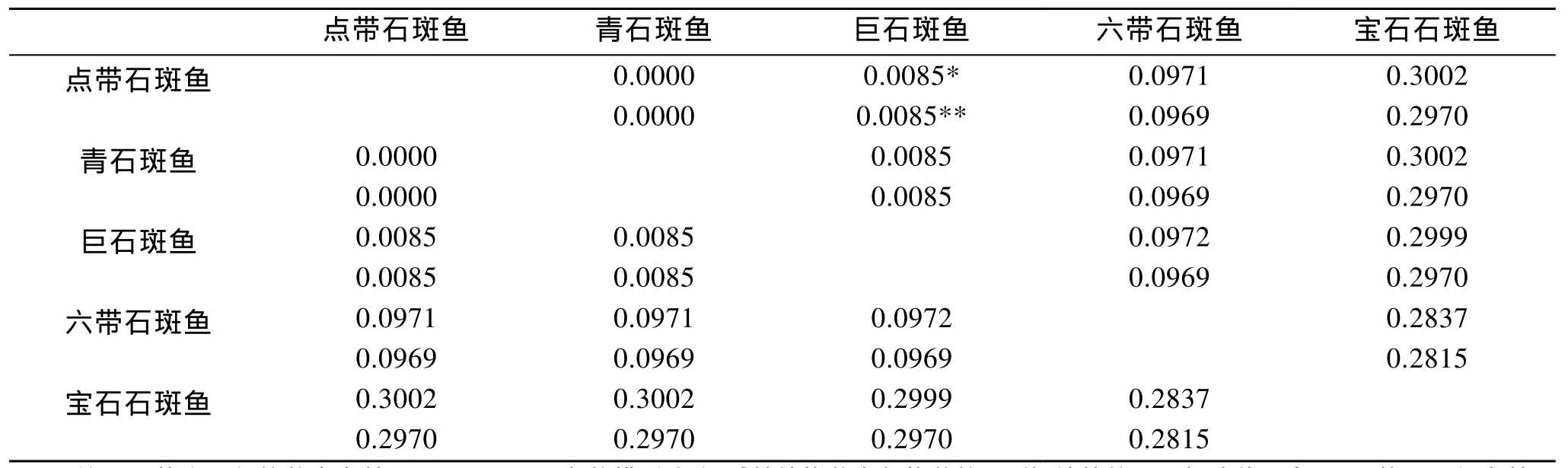
表3 基于两种参数模型5种石斑鱼各种间rDNA ITS1序列的遗传距离Tab.3 Genetic distances of rDNA ITS1 sequences among 5 kinds of groupers based on two parameter models.
2.3 分子系统进化分析
所测定的 5种石斑鱼 rDNA ITS1序列经CLUSTL X软件[12]进行序列重排后, 总位点593个,其中可变位点324个, 信息位点45个, 缺失位点56~65个(图2)。运用PAUP 4.0软件以丽鱼科(Cichlidae)、罗非鱼属的尼罗罗非鱼作外群, 基于 MP法构建rDNA ITS1序列的分子系统树(图3)。5种石斑鱼逐级聚类: 点带石斑鱼与形态最为相似的青石斑鱼首先聚到一起, 然后与形态相似的巨石斑鱼和六带石斑鱼分别先后聚到一起, 最后与形态差异稍大的宝石石斑鱼聚到一起, 置信度为 76%~100%, 而这 5种石斑鱼与外群尼罗罗非鱼聚合的置信率不到 5%, 所以在树上不显示; 根据上述位点信息, 设定 rDNA ITS1序列不同位点的进化是独立事件, 生成数据矩阵, 用 ML法, 似然步数为 1000, 构建 rDNA ITS1序列的分子系统树(图4), 与依据Kimura 2-parameter model估算遗传距离, 用NJ法构建的分子系统树(图5)分支结构完全一致, 而且这两种树与用MP法构建的分子系统树也一致, 同样, 外群无法显示, 说明它与这 5种石斑鱼明显不是一类, 结果是可靠的。MP法、ML法和NJ法构建的3种分子系统树5种石斑鱼聚类结果一致, 充分地反映了 5种石斑鱼种间分子系统进化关系。

图2 排序后的5种石斑鱼与外群尼罗罗非鱼rDNA ITS1序列(黑体为特征序列或碱基)Fig.2 rDNA ITS1 sequences of 5 species of grouper and the outgroup O.niloticus after alignment(The black words denote the characteristic regions or bases)

图3 基于rDNA ITS1序列利用MP法构建5种石斑鱼的分子系统树Fig.3 The molecular phylogenetic tree of 5 kinds of groupers based on rDNA ITS1 sequences constructed using MP method

图4 基于rDNA ITS1序列利用ML法构建5种石斑鱼的分子系统树Fig.4 The molecular phylogenetic tree of 5 kinds of groupers based on rDNA ITS1 sequence constructed using ML method

图5 基于rDNA ITS1序列利用NJ法构建5种石斑鱼的分子系统树Fig.5 The molecular phylogenetic tree of 5 kinds of groupers based on rDNA ITS1 sequences constructed using NJ method

3 讨论与结论
3.1 rDNA ITS区序列特点及5种石斑鱼的ITS1序列及其遗传距离比较
核糖体转录间隔区(rDNA ITS)序列是国际上公认的种属以下的分类、种质鉴别与系统关系分析上是比较稳定和理想的分子遗传标记[3,21]。ITS区序列包括 ITS1、ITS2, 由于 ITS区序列不加入成熟核糖体, 所以受到的选择压力较小, 进化速率较快, 可从较短的序列中获得较多的系统发育信息, 对鱼类种间、亚种和种群水平上的区分鉴定比较实用[22]。已有研究表明ITS1是rDNA中变异较大的区域[23], 在无脊椎动物的系统分类和分子系统进化分析上得到广泛应用[21,24-26]。在鱼类研究方面, 徐晖等[27]在分析6种舌鳎亚科鱼类ITS1序列长度多态性及其系统发育分析时发现舌鳎亚科鱼类ITS1区具有明显的长度多态性。孙玉华等[28]在亚口鱼科鱼类核 DNA 18S-ITS1-5.8 S序列比较分析中也得到了这一结论。郭奕惠等[29]对 5种罗非鱼的 ITS1及其两侧的18sRNA和5.8sRNA部分序列特征进行了分析, 结果发现罗非鱼ITS1序列多态性较高。Sajdak等[30]利用ITS-1序列构建了较大范围的白鲑种类的系统发育树。钟立强等[31]利用 ITS-1 序列有效的分析了4个鲤鱼种群的遗传变异。尽管ITS1做为分子系统进化具有很多优点, 但在鱼类研究相对比较局限。
5种石斑鱼ITS1序列的长度在528 bp ~537 bp,点带石斑鱼只比青石斑鱼多了一个碱基A, 青石斑鱼比巨石斑鱼多1个碱基, 是由于青石斑鱼存在CAG3个碱基, 巨石斑鱼在相应的地方缺失, 巨石斑鱼存在TT两个碱基而青石斑鱼相应的地方缺失所造成。六带石斑鱼的 ITS1序列(528bp)和宝石石斑鱼的 ITS1序列(532 bp)相对较短, 但可以找到相应的特征性序列以区别于其他种石斑鱼。六带石斑鱼的特征性序列为: 5’-ATAATGATGATGATGATGATGT-3’, 22bp;宝石石斑鱼的特征性序列为: 5’-TTGCGGCGGGGTCCTCCAACCCCTCCTT-3’, 28 bp。5 种石斑鱼这一序列特征可在其遗传距离上得到反映: 点带石斑鱼与青石斑鱼只有 1个碱基的差别, 两种鱼的遗传距离趋近于0.0000; 巨石斑鱼与青石斑鱼和点带石斑鱼分别只有1和2个碱基的差别, 与这两种鱼的遗传距离都只有0.0085。六带石斑鱼与上述3种石斑鱼的遗传距离都小于0.1000, 而宝石石斑鱼与其他4种石斑鱼的遗传距离都大于 0.2800。由此可将 5种石斑鱼分成3类: 形态与点带石斑鱼很相似的有青石斑鱼、巨石斑鱼; 形态与点带石斑鱼相似的有六带石斑鱼; 形态与点带石斑鱼有些相似的为宝石石斑鱼。对于形态难以区分的种类可用其特征序列进行识别。
3.2 5种石斑鱼的分子系统进化关系
依据体色、花纹等形态特征对石斑鱼进行分类的难度是从所周知的事实。本研究选择的 5种石斑鱼, 除宝石石斑鱼成鱼阶段可依据形态鉴别外, 其他4种形态均十分相似(表4), 尤其是俗称“青斑”的3种, 虽序列上可以看出它们的区别, 但苗种生产需要了解石斑鱼间的系统关系, 即遗传背景, 以便为良种选配和遗传保护提供依据。

表4 5种石斑鱼的主要形态特征Tab.4 Major morphological characters of five species of groupers
研究的 5种石斑鱼主要分布于太平洋西部和印度洋北部的广泛海域, 在中国主要分布于南海(表1)。利用MP、ML、NJ 3种方法构建的分子系统树与遗传距离和形态分析的结果高度一致, 这说明系统树聚类的正确性, 同时表明点带石斑鱼在进化上与青石斑鱼分化较晚, 而宝石石斑鱼在进化上与点带石斑鱼和青石斑鱼的分化相对较早。在遗传育种研究及良种培育生产上, 应避免点带石斑鱼与形态相似的青石斑鱼和巨石斑鱼间进行交配, 由此导致遗传多样性的丢失, 影响物种的生存力。研究的5种石斑鱼rDNA ITS1 序列分别与它们的形态呈同步进化的关系, 而在石斑鱼 mtDNA Cytb序列的研究发现, mtDNA Cytb序列的进化与形态有些不一致的现象[32-33], 说明mtDNA Cytb序列的进化与rDNA ITS1序列的进化, 可能采取的是两种不同的进化轨迹。比较而言, rDNA ITS1 序列作为研究石斑鱼分子系统进化的遗传标记, 比mtDNA Cytb序列更为理想, 遗憾的是没有更多的序列可供比较。但就 5种石斑鱼而言, 俗称“青斑”的3种石斑鱼分化时间较晚, 在系统进化上具有高度的一致性, 应避免它们之间的彼此混交, 而这正是我们生产中所要注意到的问题。
[1] Ding S X, Zhuang X, Guo F, et al.Molecular phylogebetic rclationships of China seas groupers based on cytochrome b gene gragment sequences[J].Science in China: (Series C ), 2006, 49(3): 235-242.
[2] 唐伯平, 周开亚, 宋大祥.核 rDNA ITS区序列在无脊椎动物分子系统学研究中的应用[J].动物学杂志,2002, 37(4): 67-73.
[3] 缪炜, 张锡元, 余育和, 等.利用三种分子标记研究缘毛类纤毛虫的系统发育地位[J].动物学研究, 2003,1: 1-10.
[4] 王薇.虫草ITS序列分子进化分析及其复合营营养液的体外细胞作用研究[D].成都: 西南交通大学, 2012:94.
[5] 林子杰.三种绿藻的ITS和18SrDNA序列及系统发育分析[D].苏州: 苏州大学, 2011: 53.
[6] 王赟, 漆一鸣.两种眼蚤rDNA ITS1序列的克隆与分析[J].贵州科学, 2011, 4: 29-32.
[7] Pleyte K A, Duncan S D, Phillips R B.Evolutionary relationships of the salmonid fish genus Salvelinus inferred from DNA sequences of the first internal transcribed spacer (ITS 1) of ribosomal DNA[J].Molecular Phylogenetics and Evolution, 1992, 1(3):223-230.
[8] 邱凡, 苏永全, 傅蒙娜, 等.基于线粒体Cytb和ITS1部分序列分析鲭科鱼类分子系统进化关系[J].中国水产科学, 2010, 2: 201-211.
[9] Presa P, Pardo B G, Martínez P, et al.Phylogeographic congruence between mtDNA and rDNA ITS markers in brown trout [J].Mol Biol Evol, 2002, 9: 2161- 2175.
[10] Chow S, Nakagawa T, Suzuki N, et al.Phylogenetic relationships amongThunnusspecies inferred from rDNA ITS1 sequence [J].J Fish Biol, 2006, 68: 24-35.
[11] 张源真, 王伟, 姜志强.基于 ITS-1 序列的部分 鳚 亚目鱼类的分子系统进化关系研究[J].现代农业科技,2012, 8: 318-320.
[12] Guo M L, Su Y Q, Zhang Z W, et al.Differentiation ofEpinephelus moarafromE.bruneusby improved nest-tetra-primer-specific PCR[J].Progress in Natural Science, 2009, 19: 1221-1226.
[13] Williams J G K, Kubilik A R, Liavk K J.DNA polymorphism amplified by arbitrary primers is useful as genetic markers[J].Nacl Acids Res, 1990, 18:6531-6535.
[14] Welsh J K.Fingerprinting genoms using PCR with arbitrary primers[J].Nacl Acids Res, 1990, 18:7213-7218.
[15] 金冬雁, 黎孟枫.分子克隆实验指南(第二版)[M].北京: 科学出版社, 1992: 15-66.
[16] Hall T.BIOEDIT: Biological sequence alignment editor for windows[D].North Carolina, USA: Carolina State University, 1998.
[17] Thompson J D, Gibson T J, Plewniak F, et al.The CLUSTAL_X windows interface: flexible strategies for multiple sequence alignment aided by quality analysis tools[J].Nucleic Acids Res, 1997, 25: 4876-4882.
[18] Swofford D L.PAUP: Phylogenetic analysis using parsimony (and other methods), version 4.0[M].Sunderland, Massachusetts: Sinauer Associates, 2001:128.
[19] Strimmer K, von Haeseler A.Quartet puzzling: a quartet maximum-likelihood method for reconstructing tree topologies[J].Mol Biol Evol, 1996, 13: 964-969.
[20] Felsenstein J.Phylogeny inference package (PHYLIP).Version 3.5[R].Washington, Seattle: Department of Genetics, University of Washington, Seattle, 1993.
[21] 缪炜, 余育和, 沈韫芬, 等.6种累枝虫(Epistylis)rRNA基因18S-ITS1序列及其分子系统发育关系[J].中国科学C辑, 2002, 45(3): 280-288.
[22] 魏晓华.栉孔扇贝和海湾扇贝的遗传多样性研究及扇贝科几种贝类的分子系统学研究[D].青岛: 中国海洋大学, 2004.
[23] Jousson O, Bartoli P, Zaninetti L, et al.Use of the ITS rDNA for elucidation of some life cycles of Mesometridae (Trematoda, Digenea)[J].International Journal for Parasitology, 1988, 28: 1403-1411.
[24] Schilthuizen M, Gittenberger E, Gultyaev A P.Phylogenetic relationships inferred from the sequence and secondary structure of ITS1 rRNA inAlbinariaand putativeIsabellariaspecies (Gastropoda, Pulmonata,Clausiliidae) [J].Mol Phylogenet Evol, 1995, 4(4):457-462.
[25] Lindsay D S, Upton S J, Owens D S, et al.Cryptosporidium andersonin.sp.(Apicomplexa: Crytosporiidae)from Cattle, Bostaurus[J].J Eukaryot Microbiol, 2000,47(1): 91-95.
[26] Dover G A.Molecular drive: a cohesive mode of species evolution[J].Nature, 1982, 299: 111-117.
[27] 徐晖, 李军, 孔晓喻.等, 6种舌鳎亚科鱼类 ITS1序列长度多态性及系统分析[J].海洋与湖沼, 2008,39(1): 35-41.
[28] 孙玉华, 谢从新, 刘思阳.亚口鱼科鱼类核 DNA 18S-ITS1-5.8S序列比较分析[J].水生生物学报, 2006,3: 367-370.
[29] 郭奕惠, 喻达辉, 黄桂菊, 等.细胞核 rDNA 序列分析 5种罗非鱼的亲缘关系[J].安徽农业科学, 2009,37(8): 3444-3447.
[30] Sajdak S L, Phillips R B.Phylogenetic relationships amongCoregonusspecies in ferred from the DNA sequence of the first in ternaltranscribed spacer (ITS1)of ribosomal DNA[J].Can J Fish Aquat Sci, 2003, 54(7) : 1494-1503.
[31] 钟立强, 张成锋, 周凯, 等.四个鲤鱼种群 ITS-1 序列的遗传变异分析[J].湖泊科学, 2011, 2: 271-276.
[32] 朱世华, 杨迎春, 郑文娟, 等.从细胞色素 b 部分序列探讨石斑鱼属的分子系统发育关系[J].水生生物学报, 2006, 30(4): 432-438.
[33] 陈艺燕, 章群, 任岗, 等.10 种石斑鱼系统发育的线粒体细胞色素 b 基因序列分析[J].海洋科学, 2006,30(6): 12-15.
