小麦TaXRCC1基因的克隆及生物信息学分析
2015-04-07张蕾于永昂杨天佑
张蕾,于永昂,杨天佑
(1.河南科技学院,河南新乡453003;2.现代生物育种河南省协同创新中心,河南新乡453003;3.西北农林科技大学,陕西杨凌712100)
小麦TaXRCC1基因的克隆及生物信息学分析
张蕾1,2,于永昂3,杨天佑1,2
(1.河南科技学院,河南新乡453003;2.现代生物育种河南省协同创新中心,河南新乡453003;3.西北农林科技大学,陕西杨凌712100)
为了研究X线修复交叉互补基因1(X-ray repair cross complementing 1,XRCC1)的结构和功能,利用RT-PCR方法克隆了小麦XRCC1基因,命名为TaXRCC1(GenBank登录号:KP772259),并对其进行生物信息学分析.序列分析结果显示,TaXRCC1基因开放阅读框长1 033 bp,编码350个氨基酸;TaXRCC1蛋白相对分子质量为38 040,pI为5.37.该蛋白序列无信号肽,是亲水性蛋白,二级结构中以无规则卷曲(41.71%)和α-螺旋(40.0%)为主.聚类分析结果显示,TaXRCC1基因与水稻和玉米XRCC1基因亲缘关系最近.为进一步研究XRCC1基因的功能奠定了基础.
小麦;X线修复交叉互补基因1(XRCC1);基因克隆;生物信息学分析
在正常条件下,所有生物的基因组是稳定的,其DNA在细胞分裂过程中通过复制把所有遗传物质传递给子代,从而确保生物正常的新陈代谢和繁殖.然而,植物在自然界中经常会受到干旱、冷害、病菌和高盐等生物和非生物胁迫,使DNA出现损伤,从而影响植物正常的生长和发育[1].DNA损伤是指逆境胁迫引起的DNA碱基改变,脱碱基位点的产生,碱基错配、插入或缺失,嘧啶联合,DNA加合物,DNA链断裂等现象[2].受损的DNA可能对植物细胞产生遗传毒性或细胞毒性[3].如果受到损伤的DNA不能及时修复,在一个细胞周期后,DNA损伤就有可能成为突变,由此引起的染色体不稳定将对细胞和生物体产生致命的影响.
植物在长期的进化过程中,为了维持基因组的稳定性,针对不同类型的DNA损伤反应,形成了碱基切除修复(base excision repair,BER)、核苷酸切除修复(nucleotide excision repair,NER)和错配修复(mismatch repair,MMR)等修复途径[4].其中,碱基切除修复是指切除和替换有内源性化学物作用产生的DNA碱基损伤.X线修复交叉互补基因1(X-ray repair cross complementing group 1,XRCC1)是一种重要的DNA修复基因,它编码的蛋白质能够与DNA连接酶Ⅲ相互作用,参与碱基切除修复[5].目前已经发现XRCC1基因的编码区有3个单核苷酸多态位点,导致其蛋白质相应的氨基酸改变,分别是C26304T (Arg194Trp)、G27466A(Arg280His)和G28152A(Arg399Gln)[6].流行病学调查和功能研究已经确定XRCC1在肿瘤发生中起到重要作用.然而,对于植物XRCC1基因的研究相对较少,Yukinobu等[4]研究表明在博来霉素对水稻幼苗造成的DNA损伤中,XRCC1能够与细胞核增殖抗原(proliferatingcell nuclear antigen, PCNA)基因相互作用参与碱基修复途径.迄今为止,在小麦上XRCC1基因的相关研究还未见报道.因此,本研究从小麦新麦26叶片中克隆XRCC1基因,并对其编码的XRCC1蛋白进行生物信息学分析,以期为小麦DNA损伤修复机制提供分子生物学依据.
1 材料与方法
1.1 试验材料
小麦品种为新麦26.将种子用质量分数为0.1%的氯化汞消毒10 min后,种植于铺有无菌滤纸的培养皿中,光照培养箱中培养至三叶期时,取新鲜叶片液氮速冻于-70℃,保存待用.
大肠杆菌DH5α感受态细胞、pGM-Tvector、DL2000plusMarker、Trizol试剂、琼脂糖凝胶回收试剂盒购自北京天根生化科技有限公司;Taq DNA聚合酶、dNTPs、反转录试剂PrimScriptTMRT-ReagentKit购自Takara公司;引物的合成和DNA序列测定由上海生工生物工程有限公司完成;其他试剂均为国产分析纯.
1.2 试验方法
1.2.1 小麦总RNA的提取及反转录利用Trizol试剂提取小麦新麦26叶片的总RNA,经1%琼脂糖凝胶电泳检测RNA质量后,依照反转录试剂盒PrimeScriptTMRT Reagent Kit产品说明书进行反转录试验.
1.2.2 TaXRCC1基因的克隆及测序根据水稻OsXRCC1基因(GenBank登录号:AB292807)的cDNA序列,搜索小麦的EST数据库,拼接获得一个包含完整开发阅读框(ORF)的小麦XRCC1基因cDNA序列,利用Primer 5.0设计PCR扩增引物:
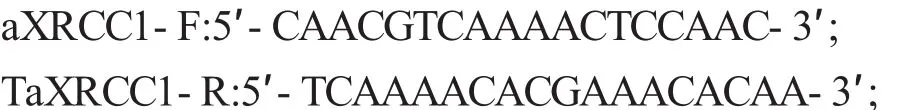
以反转录合成的cDNA第一链为模板进行PCR反应,扩增体系为:1 μL cDNA,2.5 μL10×Taq PCR buffer,1.5 μL 25 mmol/L MgCl2,1 μL 10 mmol/L dNTP,1 μL 10 μmol/L的上下游引物,0.25 μL Taq DNA聚合酶和16.75 μL ddH2O.反应条件为:94℃3 min,94℃30 s,55℃30 s,72℃40 s,共35个循环;最后72℃延伸5 min.扩增产物经1.2%琼脂糖凝胶电泳检测后,用DNA纯化回收试剂盒回收目的条带并连接pGM-T载体,连接产物转化至大肠杆菌DH5α,进行Amp+抗性蓝白斑筛选,挑取阳性菌落提取质粒.经PCR鉴定后送上海生工生物工程有限公司测序.
1.2.3 TaXRCC1基因的生物信息学分析利用ORF finder(http://www.ncbi.nlm.nih.gov/)在线分析软件对测序结果进行开放阅读框的预测;利用ProtParam(http://web.expasy.org/protparam/)在线工具进行蛋白基本理化性质分析.利用DNAMAN 6.0软件进行氨基酸序列多重比对;用Mega 5.0软件,以N-J算法,重复抽样1 000次来构建系统进化树.
2 结果与分析
2.1 RNA质量检测
将小麦叶片RNA样品进行琼脂糖凝胶电泳分析,结果见图1.从电泳结果中可以看出提取的RNA28S、18S、5S条带清晰,表明所提取总RNA完整性较好,没有降解.A260/280值经核酸蛋白检测仪检测处于1.8~2.0之间,同时A260/230值检测后大于2.0,说明RNA没有被碳水化合物、盐类或有机溶剂污染,能够满足下一步基因克隆的要求.

图1 总RNA的提取Fig.1 Extraction oftotal RNA
2.2 TaXRCC1基因cDNA克隆
根据水稻OaXRCC1基因序列,借助生物信息学和GenBank数据库,采用DNAMAN 6.0软件对高度同源的小麦EST序列进行拼接,获得1个含有完整编码框的小麦GST基因的cDNA序列.根据拼接序列设计引物,以小麦cDNA为模板,采用RT-PCR方法扩增获得一个大约1 000 bp的目的条带(见图2),与预期片段大小相符,测序结果显示该序列长1 095 bp,将其命名为TaXRCC1,并将此序列提交到GenBank,获得登录号:KP772259.根据NCBI的ORF Finder分析发现,该序列包含一个1 053 bp的开放阅读框,编码350个氨基酸,起始密码子为ATG,终止密码子为TAG(见图3).
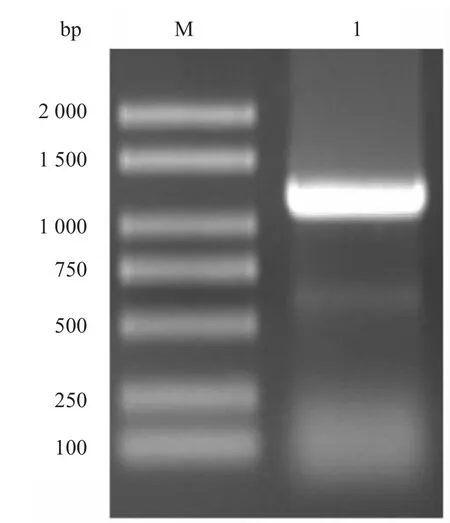
图2 小麦TaXRCC1基因扩增产物Fig.2 RT-PCR products ofTaXRCC1

图3 TaXRCC1基因的核苷酸序列及推测的氨基酸序列Fig.3 Nucleotide sequence and deduced aminoacid sequences of TaXRCC1 gene
2.3 TaXRCC1蛋白理化性质分析
通过Protparam分析该基因编码蛋白的理化性质,推测TaXRCC1蛋白的分子式为C1645H2591N473O541S12,蛋白相对分子质量为38 040,等电点为5.37;N-端氨基酸为蛋氨酸(Met,M),半衰期大约30 h,不稳定系数为48.68,为不稳定蛋白(标准:40以下为稳定蛋白).总的带负电荷的残基(Asp+Glu)为54,总的带正电荷的残基(Arg+Lys)为44.亲水性平均数为-0.798,预测该蛋白为亲水性蛋白,其脂肪指数为59.46.
蛋白二级结构分析显示,肽链中含有α-螺旋40.0%,β-折叠9.71%,无规则卷曲41.71%(见图4). TMHMM-2.0(http://www.cbs.dtu.dk/services/TMHMM-2.0)对TaXRCC1蛋白氨基酸序列进行分析,没有跨膜结构域,推测该蛋白不是跨膜蛋白.SignalP 4.1分析该基因没有信号肽,是非分泌蛋白.

图4 TaXRCC1二级结构预测图Fig.4 Secondarystructure ofTaXRCC1
2.4 TaXRCC1基因的进化分析
利用DNAMAN6.0对不同物种XRCC1氨基酸序列进行多重比对.结果表明:小麦TaXRCC1与水稻、玉米XRCC1蛋白序列相似性最高,分别为71.43%和63.11%(见图5).
通过MEGA 5.0中的Clustal W比对,采用Neighbor-joining方法,构建了不同生物XRCC1基因之间的进化关系.结果表明:小麦TaXRCC1与水稻、玉米XRCC1蛋白处于进化树的同一分支,与其植物的亲缘关系相符(见图6).图6中各物种XRCC1基因信息为:小麦Triticum aestivum(TaXRCC1,KP772259),水稻Oryza sativa(OsXRCC1,AB292807),玉米Zea mays(ZmXRCC1,NP_001149899),烟草Nicotiana tomentosiformis(NtXRCC1,XP_009623792),野生大豆Glycine soja(GsXRCC1,KHN44135),拟南芥Arabidopsis thaliana(AtXRCC1,NM_180654),番茄Solanum lycopersicum(SlXRCC1,XP_004241724),胡杨Populus euphratica(PeXRCC1,XP_011002718),蓖麻Ricinus communis(RcXRCC1,XP_002517520),苹果Malus domestica(MdXRCC1,XP_008342730),葡萄Vitis vinifera(VvXRCC1,XM_002280415),桑树Morus notabilis(MnXRCC1,XP_010110299),棉花Gossypium arboretum(GaXRCC1,KHG18866),芝麻Sesamum indicum(SiXRCC1,XP_011072285).

图5 小麦XRCC1氨基酸序列与其他物种XRCC1氨基酸序列比对Fig.5 Multiple alignment ofTaXRCC1 with the other plant XRCC1s

图6 不同物种XRCC1基因系统进化树分析Fig.6 Phylogenic tree analyses ofXRCC1 fromvarious organismspecies
3 讨论
DNA损伤在生物体内经常发生,对植物的生长和发育造成严重的影响.因此,DNA损伤修复系统在生物体内具有重要的保护功能,为受损的DNA进行修复,减少损伤的致死性或避免突变、重组或损伤的DNA进入子代细胞[7].XRCC1属于主要修复氧化损伤、电离辐射及甲基化等导致DNA链断裂的BER通路.XRCC1在动物细胞的碱基切除修复途径中起到关键的作用[8-10].敲除小鼠的XRCC1基因,会导致突变小鼠胚胎死亡,XRCC1缺陷型细胞对于DNA损伤诱导剂产生的单链损伤敏感性增强[8].
本研究根据水稻的XRCC1基因的cDNA序列在小麦的EST数据库进行检索,获得了多个小麦XRCC1基因同源序列,并对这些序列进行拼接,通过RT-PCR方法克隆得到小麦XRCC1基因序列,该序列长1095bp,开放阅读框1053bp,编码350个氨基酸.生物信息学分析发现,该蛋白不存在信号肽和跨膜结构域,系统进化树分析结果显示该基因编码的蛋白质与水稻OsXRCC1基因的亲缘关系最近.本研究为进一步研究小麦TaXRCC1基因的结构和功能奠定了基础.
[1]杜秀敏,殷文璇,赵彦修,等.植物中活性氧的产生及清除机制[J].生物工程学报,2001,17(2):121-125.
[2]刘宛,李培军,周启星,等.环境污染条件下生物体内DNA损伤的生物标记物研究进展[J].应用于环境生物学报,2005,11(2): 251-255.
[3]聂志刚,王艳,李韶山.重金属诱导拟南芥原生质体DNA损伤的单细胞凝胶电泳检测[J].植物学报,2009,44(1):117-123.
[4]Yokinobu U,YukoS,KengoS.Characterization ofplant XRCC1 and its interaction with proliferatingcell nuclear antigen[J].Planta, 2008,227(6):1233-1241.
[5]Kubota Y,Nashi R A,Klungland A,et al.Reconstitution of DNA base excision-repair with purified human proteins:interaction between DNApolymerasebetaand theXRCC1 protein[J].TheEMBOJournal,1996,15(23):6662-6670.
[6]Shen MR,Jones I M,Mohrenweiser H.Nonconservative aminoacid substitution variants exist at polymorphic freguency in DNA repair genes in healthyhumans[J].Cancer Research,1998,58(4):604-608.
[7]吴家睿.细胞周期检查点的分子机制[J].生物工程进展,1999,19(4):7-10.
[8]Tebbs R S,Flannery M L,Meneses J J,et al.Requirement for the Xrcc1 DNA base excision repair gene during early mouse development[J].Developmental Biolgy,1999,208(2):513-529.
[9]Dogliotti E,Fortini P,Pascussi B,et al.The mechanism of switching among multiple BER pathways[J].Progress in Nucleic Acid Research and Molecular Biology,2001,68:3-27.
[10]Caldecott KW.XRCC1 and strand break repair[J].DNARepair,2003,2(9):955-969.
(责任编辑:邓天福)
Cloning and bioinformatic analysis ofTaXRCC1 gene in wheat
ZHANG Lei1,2,YU Yongang3,YANG Tianyou1,2
(1.Henan Institute ofScience and Technology,XinXiang453003,China;2.Collaborative Innovation Center ofModern Biological BreedingofHenan Province,XinXiang453003,China;3.Northwest A&F University,Yangling712100,China)
In order to learn about the structure and function of XRCC1(X-ray repair cross complementing 1)gene,a gene designated as TaXRCC1(GenBank NO.KP772259)was cloned from young leaves of Triticum aestivum by RTPCR technology and its bioinformatic analysis was conducted.The protein sequence had no signal peptide,belonged to hydrophilic protein.Its secondary structures were mainly the random coil(41.71%)and α-helix(40.0%).The results of clustering analysis showed that the genetic relationship of TaXRCC1,Oryza sativa XRCC1 and Zea mays XRCC1 was closer.The research laid a foundation to further study the XRCC1 function in Triticum aestivum.
Triticum aestivum L.;X-ray repair cross complementing 1(XRCC1);gene cloning;bioinformatic analysis
S512
A
1008-7516(2015)06-0001-07
10.3969/j.issn.1008-7516.2015.06.001
2015-10-19
国家自然科学基金项目(1305047);河南省教育厅科学技术研究重点项目(12A180011);河南省高等学校青年骨干教师资助计划项目(2014GGJS-100)
张蕾(1987―),女,河南卫辉人,硕士,助理实验师.主要从事植物分子生物学研究.
