不同种源辽东冷杉rDNA ITS序列及其亲缘关系1)
2015-03-07腰政懋徐程扬李乐
腰政懋 徐程扬 李乐
(省部共建森林培育与保护教育部重点实验室(北京林业大学),北京,100083)
责任编辑:潘 华。
核糖体DNA(ribosomal DNA,rDNA)内转录间隔区(Internal Transcribed Spacer,ITS)中的ITS1(介于18S 和5.8S 之间)和ITS2(介于5.8S 和26S 之间)序列作为非编码区(Non-Coding Region),承受的选择压力较小,进化速率较快[1],且与植物生活型呈相关性[2],可以提供丰富的遗传分析所需要的序列及位点信息。rDNA ITS 序列分析已被广泛应用于植物种间或种内遗传多样性和系统发育关系的研究[3-6],但对冷杉属(Abies)植物进行rDNA ITS 序列分析的报道仍然较少,仅有向巧萍等[7]曾对rDNA ITS 序列在28种冷杉属植物中的长度多态性进行了研究。
辽东冷杉(Abies holophylla)为松科冷杉属常绿乔木,别名沙松,原产于我国东北牡丹江流域山区、长白山区及辽宁东部山区的气候寒冷湿润地带,生长较快、材质优良、抗病虫害能力强,是温带针阔混交林中的主要用材树种之一。目前,天然林中辽东冷杉资源急剧减少,林相残缺破碎[8],生境片断化严重,亟需得到保护。目前有关辽东冷杉遗传多样性的研究集中在形态学水平[9-10],DNA 分子水平的遗传多样性研究未见报道。
本研究采用PCR 扩增产物直接测序的方法,对6 个种源辽东冷杉的rDNA ITS 序列进行测定与分析,首次从DNA 分子水平上比较辽东冷杉种源间的差异并分析其亲缘关系,旨在探讨辽东冷杉种内变异程度及遗传多样性,为辽东冷杉种质资源的保护和栽培利用提供科学依据。
1 材料与方法
1.1 材料的采集和保存
试验材料来自我国东北地区的黑龙江省宁安市,吉林省敦化市、和龙市、抚松县,辽宁省清原满族自治县和宽甸满族自治县,基本覆盖了辽东冷杉的整个天然分布区,共计6 个种源90 个样本。各种源地的具体情况详见表1。采样过程中按照均匀分布、随机取样的原则进行采样,保证采样母树的间距在50 m 以上。采样时所选叶片均为新鲜幼嫩叶片,采后用变色硅胶干燥法保存。带回到实验室后置于超低温冰箱-80 ℃冷冻保存备用。
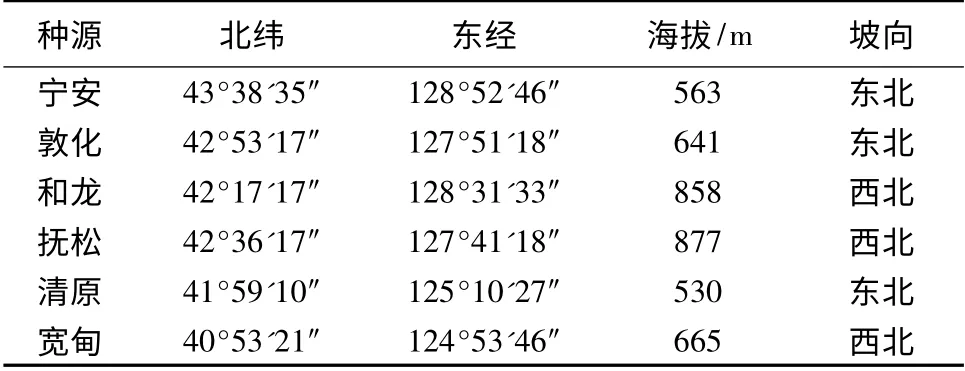
表1 东北地区辽东冷杉6 个种源地的基本情况
1.2 基因组DNA 的提取
采用Ezup 柱式植物基因组DNA 抽提试剂盒(生工生物工程(上海)股份有限公司)提取各干燥叶片样品的总DNA,按试剂盒操作指南进行。提取后用0.8%的琼脂糖凝胶电泳和紫外分光光度计检测其浓度与纯度,统一稀释到50 mg·L-1,置于冰箱-20 ℃保存备用。
1.3 PCR 扩增
使用真核生物rDNA ITS 序列的通用引物,上游引物ITS-1 的序列为:5’-TCCGTAGGTGAACCTGCGG-3’,位于18S 上;下游引物ITS-4 的序列为:5’-TCCTCCGCTTATTGATATGC-3’,位于26S 上。引物均由生工生物工程(上海)股份有限公司合成。
PCR 扩增反应在ABI 2720 Thermal Cycler(Applied Biosystems Inc.,USA)中进行。以5 个种源辽东冷杉的rDNA 为模板进行ITS1-5.8S-ITS2 整段扩增,PCR 反应体系总体积为25 μL,其中包括50 mg·L-1模板DNA 0.5 μL,10 μmol·L-1引物各0.5 μL,2.5 mmol·L-1dNTPs 1 μL,5 ×PCR 缓冲液2.5 μL,2 U·μL-1Taq DNA 聚合酶0.2 μL,加入dH2O 补足。上述试剂均购自生工生物工程(上海)股份有限公司。PCR 扩增程序为94 ℃预变性4 min;94 ℃变性45 s,55 ℃退火45 s,72 ℃延伸1 min,30 个循环;72 ℃再延伸10 min。扩增产物于4 ℃下保存。
反应结束后,以DNA Ladder Mix maker(生工生物工程(上海)股份有限公司)作为分子量标记,用1%的琼脂糖凝胶电泳(电压150 V,电流100 mA,时间20 min)检测PCR 扩增产物,电泳结果在FR980型凝胶成像仪(上海复日科技仪器有限公司)中观察和拍照。
1.4 PCR 扩增产物的纯化和测序
使用SanPrep 柱式DNA 胶回收试剂盒(生工生物工程(上海)股份有限公司)对扩增的rDNA ITS片段进行纯化,按试剂盒操作指南进行。将纯化后的PCR 产物作为测序反应的模板,用PCR 反应的引物作为测序引物直接测序。序列测定在ABI 3730xl DNA Analyzer(Applied Biosystems Inc.,USA)中完成。
1.5 序列分析方法
所得序列使用NCBI(National Center for Biotechnology Information,USA)的在线BLAST 程序进行同源检索;使用Clustal X(Version 2.0)[11]软件进行对位排列和多重比对,并辅以人工校对;使用分子进化遗传分析软件MEGA(Version 6.0)[12]软件分析碱基组成、(G+C)含量、DNA 序列差异百分率和碱基置换数,并用Kimura 2-Parameter 模型(K2P Model)构建序列遗传距离矩阵,NJ 法(Neighbor- Joining Method)构建系统发生树。系统发生树各分支的置信度用自觉检验法(Bootstrap Test)检验,共进行1 000次循环,以评价各分支的系统学意义及可靠性。
2 结果与分析
2.1 辽东冷杉rDNA ITS 序列PCR 扩增产物的电泳分析
以6 个种源辽东冷杉基因组DNA 为模板进行rDNA ITS 序列的PCR 扩增反应,PCR 扩增产物的电泳结果显示,6 个种源辽东冷杉的rDNA ITS 序列的PCR 扩增产物均在1 200~1 500 bp 处有1 条清晰的特异性条带。
2.2 辽东冷杉rDNA ITS 序列的长度
将辽东冷杉在NCBI 中进行在线Blast 比对,发现GenBank 中尚没有辽东冷杉rDNA ITS 序列的登录,因此参考冷杉属其他树种如秦岭冷杉(Abies chensiensis)(登录号EF057702.1)、资源冷杉(Abies ziyuanensis)(登录号EF057704.1)和臭冷杉(Abies nephrolepis)(登录号EF057712.1)的rDNA ITS 序列,分析确定了辽东冷杉rDNA ITS 序列中ITS1 和ITS2 与3 个编码区18S、5.8S、26S 的界限,并使用NCBI 在线工具BankIt 向GenBank 提交了辽东冷杉的rDNA ITS 序列(登录号KT164592)。
6 个种源辽东冷杉rDNA ITS 全序列长度均为1 355 bp,其中ITS1 序列长度为1 162 bp,5.8S 编码区序列长度为162 bp,ITS2 序列长度仅为71 bp,较短。各区在种源间均未发生长度变异。
2.3 辽东冷杉rDNA ITS 序列的碱基变异
6 个种源辽东冷杉的rDNA ITS 序列的碱基变异较少,只有8 个位点发生了变异,均位于ITS1 序列上。其中6 个位点发生了A—G 或C—T 碱基转换,2 个位点G—C 或G—T 碱基颠换,无插入或缺失变异(表2)。5.8S 编码区序列和ITS2 序列均非常保守,没有变异位点。清原和宽甸种源的rDNA ITS 序列完全一致,宁安和敦化种源均各有1 个位点变异,和龙和抚松种源变异位点较多,均为4 个。

表2 6 个种源辽东冷杉rDNA ITS 序列的碱基变异位点
2.4 辽东冷杉rDNA ITS 序列的碱基含量
6 个种源辽东冷杉的ITS1 序列中各碱基的含量存在明显差异(表3),其中鸟嘌呤G 含量最高,腺嘌呤A 含量最低,(G + C)含量在60.24%~60.59%,基本相等;ITS2 序列中各碱基的含量也存在明显差异,从16.13%~38.71%不等,其中胞嘧啶C含量最高,胸腺嘧啶T 含量最低,(G +C)含量均为58.06%。ITS1 和ITS2 序列中(G +C)含量均明显高于(A+T)含量。不同物种的(G +C)含量不同,物种间的亲缘关系越近,(G + C)含量差异越小[13-14]。种源间(G +C)含量差异极小,也说明辽东冷杉种源间亲缘关系非常近。
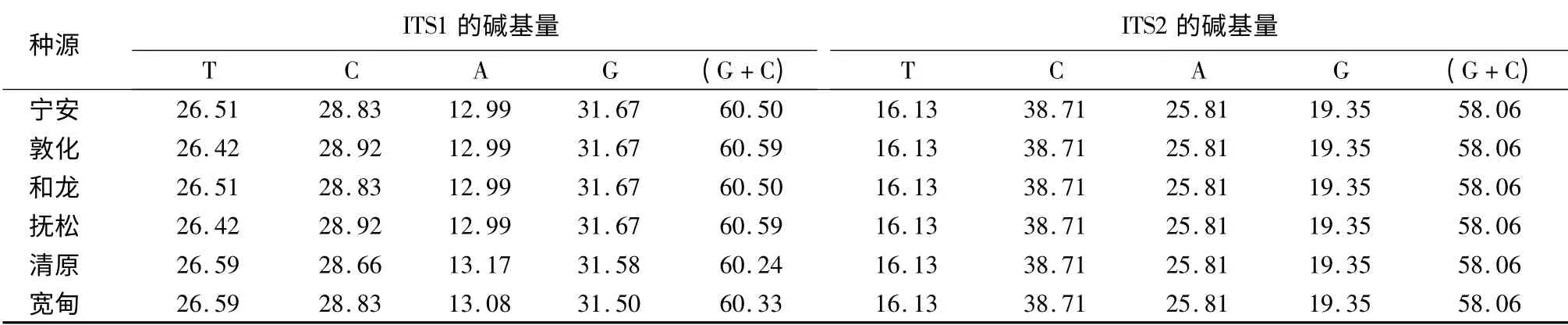
表3 6 个种源辽东冷杉rDNA ITS 序列的碱基含量 %
2.5 辽东冷杉rDNA ITS 序列间的遗传距离
根据Kimura 2-parameter 模型(K2P model)构建的辽东冷杉rDNA ITS 序列种源间遗传距离矩阵(表4)显示,清原种源与宽甸种源的遗传距离最小,为0,表明它们的序列同源性最高,亲缘关系极近;清原和宽甸种源均与和龙和抚松种源间的遗传距离最大,为0.003 7,亲缘关系最远;其它种源间遗传距离也在0.000 7~0.003 0;遗传距离平均值为0.002 3,表明辽东冷杉种源间亲缘关系较近,遗传变异程度较低。
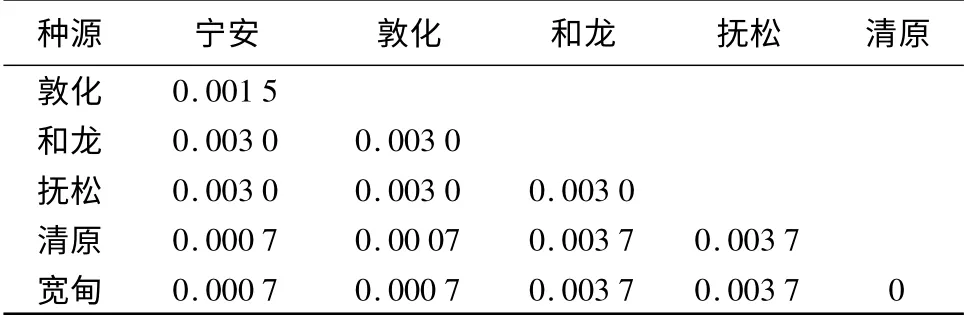
表4 6 个种源辽东冷杉rDNA ITS 序列间的遗传距离
2.6 辽东冷杉rDNA ITS 序列的系统发生树
基于6 个种源辽东冷杉rDNA ITS 序列的系统发生树(图1)表明,地理距离相近的种源聚在一起。位于黑龙江省东南部的宁安种源与相邻的位于吉林省东北部的敦化种源即牡丹江流域的两个种源聚在一起,位于长白山区的吉林省的和龙和抚松种源也聚在一起,并与上述两个种源共同聚为一个大类;位于辽宁省东部的清原和宽甸种源聚在一起,单独聚为一个大类。这与遗传距离分析所得的结果一致,表明辽东冷杉rDNA ITS 序列的差异与其地理分布相关性较高,地理距离相近则遗传距离也较近,表现出一定的分子地域性差异。
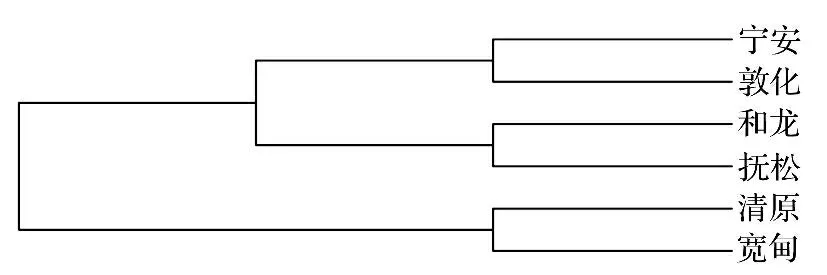
图1 基于6 个种源辽东冷杉rDNA ITS 序列的系统发生树
3 结论与讨论
rDNA 在不同物种间存在丰富变异,核苷酸序列变化大,可以提供详尽的遗传学信息[15],相对于线粒体DNA(Mitochondrial DNA,mtDNA)和叶绿体DNA(Chloroplast DNA,cpDNA),其受到细胞核保护机制的保护,进化过程稳定,且其ITS1、ITS2 序列的间隔区(18S、5.8S 和26S rDNA)极为保守,能够用通用引物进行扩增及直接测序[16-17]。因此,非常适用于种以上水平的系统发育和分类鉴定研究,在裸子植物中,已先后应用于铁杉属(Tsuga)[18]、松属(Pinus)[19]、落叶松属(Larix)[20]、黄杉属(Pseudotsuga)[20]、冷杉属(Abies)[7]和云杉属(Picea)[21]等。但在鉴别种内差异方面,其应用价值则因种而异[22-24]。
裸子植物的rDNA ITS 序列长度(包括ITS1、ITS2 和5.8S rDNA)介于975~3 125 bp 之间,平均长度约为1 500 bp[25],明显大于被子植物的565~700 bp[26]。向巧萍等[7]对28 种冷杉属植物rDNA ITS 序列长度的研究发现分布于欧亚大陆和其它分布于北美的冷杉属植物的rDNA ITS 全序列长度约为1 700 bp。本研究结果认为6 个种源辽东冷杉rDNA ITS 序列总长度均为1 355 bp,与裸子植物rDNA ITS 序列的平均长度相近,但稍短于1 700 bp,这可能与物种差异及测序引物不同有关。
本研究发现不同种源辽东冷杉rDNA ITS 序列碱基变异位点较少,仅有8 个,均位于ITS1 序列上,而且变异类型也不丰富,仅有碱基置换(包括碱基转换和颠换)这一种类型,没有碱基插入或缺失变异,甚至清原种源和宽甸种源的rDNA ITS 序列完全一致,且序列长度分析表明ITS1 序列、ITS2 序列和5.8S rDNA 序列在6 个种源间均未发生长度变异,说明辽东冷杉rDNA ITS 序列整体具有较高的保守性。这不利于使用rDNA ITS 序列分析的方法对辽东冷杉进行精准的种源鉴定,也提示rDNA ITS 序列在种内差异鉴别方面的应用有一定的局限性[27]。
辽东冷杉rDNA ITS 序列种源间(G+C)含量差异极小和遗传距离平均值仅为0.002 3,均表明辽东冷杉种源间亲缘关系较近,遗传变异程度较低。由于冷杉植物的生长对于温度有着较严格的要求,因此第四纪温度的变化尤其是末次盛冰期对世界现代冷杉属植物的分布格局的形成起着决定性的作用[28]。在冰期,大多数高纬度地区物种必须退缩到冰期避难所或者分布范围原地急速收缩,在冰期结束后,这些物种在当地扩张范围或回迁到它们从前的分布区域,伴随该过程的奠基者效应和瓶颈效应不可避免地降低种群内和物种内的多样性[29],辽东冷杉作为分布在我国东北高纬度地区的树种,不可避免地经历了这一分布“收缩—扩张”的过程,导致其种源间遗传多样性较低。
植物种群遗传变异的分布情形与该种的地理分布情形和生态特征密切相关[30]。在对植物使用rDNA ITS 序列分析进行的遗传多样性研究中,种源间遗传距离与地理距离间是否存在显著的相关性并没有统一的结论[31-32]。根据NJ 法构建的辽东冷杉系统发生树基本上将分布区地理距离较近的种源聚在一起,辽东冷杉6 个种源呈现出明显的地域分布规律。这表明辽东冷杉种源遗传距离与地理空间距离有明显的相关性,即如果种源间的地理距离较近,其遗传距离也相对较小。
[1] 李喜凤,邱天宝,张红梅,等.不同种群蒲公英的内转录间隔区(ITS)序列及其亲缘关系分析[J].中国药学杂志,2013,48(9):687-690.
[2] 严寒静,房志坚,余世孝.不同种源何首乌的ITS 序列分析及其亲缘关系研究[J].西北植物学报,2008,28(5):922-927.
[3] Kress W J,Liu A Z,Newman M,et al.The molecular phylogeny of Alpinia (Zingiberaceae):a complex and polyphyletic genus of gingers[J].American Journal of Botany,2005,92(1):167-178.
[4] Yamaji H,Fukuda T,Yokoyama J,et al.Reticulate evolution and phylogeography in Asarum sect.Asiasarum (Aristolochiaceae)documented in internal transcribed spacer sequences (ITS)of nuclear ribosomal DNA[J].Molecular Phylogenetics & Evolution,2007,44(2):863-884.
[5] 徐纲,于超,阳勇,等.不同居群栽培牡丹rDNA ITS 区序列分析及鉴别[J].天然产物研究与开发,2009,21(2):225-230.
[6] 王晓锋,刘娜娜,季孔庶.黄杨属植物ITS 序列分子进化特点分析[J].分子植物育种,2011,9(4):506-513.
[7] 向巧萍,向秋云,Liston A,等.ITS(nrDNA)片段在冷杉属植物中的长度多态性及其在松科的系统与演化研究中的应用[J].植物学报:英文版,2000,42(9):946-951.
[8] 张有富,刘高义,陈文山,等.黑龙江省沙松资源评价[J].防护林科技,2002(1):56-60.
[9] 祝旭加,孙岳胤.沙松球果表型性状地理变异规律的研究[J].防护林科技,2013(9):12-15.
[10] 腰政懋,徐程扬,柴源,等.辽宁省东部不同种源沙松冷杉种子品质的差异[J].东北林业大学学报,2014,42(8):15-19.
[11] Larkin M A,Blackshields G,Brown N P,et al.Clustal W and Clustal X version 2.0[J].Bioinformatics,2007,23(21):2947-2948.
[12] Tamura K,Stecher G,Peterson D,et al.MEGA6:molecular evolutionary genetics analysis version 6.0[J].Molecular Biology& Evolution,2013,30(12):2725-2729.
[13] Rodríguez Trelles F,Tarrío R,Ayala F J.Evidence for a high ancestral GC content in Drosophila[J].Molecular Biology & Evolution,2000,17(11):1710-1717.
[14] 陈妍,高燕会,廖望仪,等.乳白石蒜rDNA-ITS 序列分析及种内系统发育研究[J].植物资源与环境学报,2009,18(3):25-31.
[15] Schmidt G J,Schilling E E.Phylogeny and biogeography of Eupatorium (Asteraceae:Eupatorieae)based on nuclear ITS sequence data[J].American Journal of Botany,2000,87(5):716-726.
[16] Ainouche M L,Bayer R J.On the origins of the tetraploid Bromus species (section Bromus,Poaceae):insights from internal transcribed spacer sequences of nuclear ribosomal DNA[J].Genome,1997,40(5):730-743.
[17] Baraket G,Saddoud O,Chatti K,et al.Sequence analysis of the internal transcribed spacers (ITSs)region of the nuclear ribosomal DNA (nrDNA)in fig cultivars (Ficus carica L.)[J].Scientia Horticulturae,2009,120(1):34-40.
[18] Vining T F,Campbell C S.Phylogenetic signal in sequence repeats within nuclear ribosomal DNA internal transcribed spacer 1 in Tsuga[J].American Journal of Botany,1997,84(S1):702.
[19] Liston A,Robinson W A,Piriero D,et al.Phylogenetics of Pinus (Pinaceae)based on nuclear ribosomal DNA internal transcribed spacer region sequences[J].Molecular Phylogenetics &Evolution,1999,11(1):95-109.
[20] Gernandt D S,Liston A.Internal transcribed spacer region evolution in Larix and Pseudotsuga (Pinaceae)[J].American Journal of Botany,1999,86(5):711-723.
[21] Campbell C S,Wright W A,Cox M,et al.Nuclear ribosomal DNA internal transcribed spacer 1 (ITS1)in Picea (Pinaceae):sequence divergence and structure[J].Molecular Phylogenetics& Evolution,2005,35(1):165-185.
[22] Francisco Ortega J,Santos Guerra A,Hines A,et al.Molecular evidence for a mediterranean origin of the macaronesian endemic genus Argyranthemum (Asteraceae)[J].American Journal of Botany,1997,84(11):1595-1613.
[23] 孟娜,周守标,蒋继宏.五种大戟属植物nrDNA 的ITS 序列分析及其叶的比较解剖学研究[J].广西植物,2006,26(1):18-21.
[24] 王洁,杨旭,杨志玲.不同产区厚朴nrDNAITS 序列分析及亲缘关系鉴定[J].广西植物,2013,33(1):35-41.
[25] Liston A,Robinson W A,Oliphant J M,et al.Length variation in the nuclear ribosomal DNA internal transcribed spacer region of non-flowering seed plants[J].Systematic Botany,1996,21(2):109-120.
[26] Baldwin B G.Molecular phylogenetics of Calycadenia (Compositae)based on ITS sequences of nuclear ribosomal DNA:chromosomal and morphological evolution reexamined[J].American Journal of Botany,1993,80(2):222-238.
[27] 张君毅,郭巧生,吴丽伟,等.我国不同地区半夏rDNA 序列分析[J].中国中药杂志,2006,31(21):1768-1772.
[28] 向巧萍.中国的几种珍稀濒危冷杉属植物及其地理分布成因的探讨[J].广西植物,2001,21(2):113-117.
[29] Hewitt G.The genetic legacy of the quaternary ice ages[J].Nature,2000,405:907-913.
[30] Loveless M D,Hamrick J L.Ecological determinants of genetic structure in plant populations[J].Annual Review of Ecology and Systematics,1984,15(4):65-95.
[31] 沈洁,丁小余,张卫明,等.花椒及其混淆品的rDNA ITS 区序列分析与鉴别[J].药学学报,2005,40(1):80-86.
[32] 陈建梅,王钊宇,张立娜,等.基于ITS 条形码技术分析北京云蒙山杜鹃花属遗传多样性[J].中国农学通报,2014,30(25):43-48.
