苯二酚内酯合成途径中的聚酮合酶组合表达研究
2015-02-21徐玉泉
白 净, 陆 伟, 徐玉泉, 陈 明
中国农业科学院生物技术研究所, 北京 100081
研究论文
苯二酚内酯合成途径中的聚酮合酶组合表达研究
白 净, 陆 伟, 徐玉泉, 陈 明*
中国农业科学院生物技术研究所, 北京 100081
由真菌聚酮合酶合成的苯二酚内酯类次生代谢产物结构和功能多样,在医药和农业上具有广泛的用途。苯二酚内酯由一对还原型聚酮合酶和非还原型聚酮合酶协同生物催化合成。还原型聚酮合酶和非还原型聚酮合酶由多功能结构域组成,每个结构域在生物合成的过程中程序化地执行特定的功能。通过交换不同真菌苯二酚内酯合成途径中非还原型聚酮合酶的起始物酰基转移酶结构域,在酿酒酵母中与相应的还原型聚酮合酶组合表达,合成了“非天然”的苯二酚内酯聚酮产物,并初步讨论了起始物酰基转移酶结构域的识别规律。
聚酮合酶;真菌;苯二酚内酯;生物合成
真菌苯二酚内酯聚酮化合物是一类结构和生物活性多样的次级代谢产物,对真菌自身生长、发育等产生重要影响。该类化合物在医药和农业生产中具有广泛的应用,例如,脱氢弯孢霉素(dehydrocurvularin)具有免疫抑制剂的活性[1],根赤壳菌素(radicicol)能够抑制Hsp90分子伴侣蛋白的合成,具有抗肿瘤的活性[2]。
大多数真菌聚酮类化合物是由多结构域的聚酮合酶(polyketide synthase,PKSs)通过克莱森(Claisen)缩合反应将小分子羧基酸单体催化合成长链化合物。聚酮合酶的催化结构域共价相连,其中的3个基本功能结构域分别为:酮缩酶结构域(KS)、酰基转移酶结构域(AT)和酰基载体蛋白结构域(ACP)[3]。2001年,Nicholson等[4,5]根据基因中编码的结构域将真菌PKSs进一步分为3种不同类型:不能还原β酮基的非还原型聚酮合酶(non-reduced PKS,NR PKS),部分还原型聚酮合酶(parcially reduced PKS,PR PKS)和完全还原型聚酮合酶(highly reduced PKS,HR PKS)。非还原型聚酮合酶含有起始物酰基转移酶结构域(SAT)、产物模板结构域(PT)和硫酯酶结构域(TE)。在某些情况下,还包含转甲基酶结构域(CM)和还原酶结构域(R)。部分或完全还原β酮基的还原型聚酮合酶,不含有SAT、PT、TE结构域,但含有β酮脂酰还原酶结构域(KR)、脱水酶结构域(DH)和烯酰还原酶结构域(ER)。
近几年对真菌聚酮合酶各个结构域功能的研究为天然聚酮类化合物的程序化合成机制奠定了重要理论基础。研究发现KS结构域控制了最终聚酮化合物的链长,并不是延伸次数。KS结构域与产物甲基化也有着一定的关系[6]。丙二酰AT结构域在转移丙二酰辅酶A基团时对底物有特异性[7]。硫酯酶结构域(TE)不仅影响聚酮产物的结构,而且还影响聚酮产物的产量[8],并决定了产物最后的释放。PT结构域在聚酮化合物的产生中是必不可少的,并且它控制了产物的F和S环化模式[6,9]。PT结构域通过特定醛的环化和芳构化将可变的前体转变为稳定的形式,因而增强了产物的稳定性[10]。最近对PksA PT结构域单体的晶体学研究表明PT催化活性中心由3部分构成:入口端的磷酸泛酰巯基乙胺(PPT)结合区域、催化口袋底端的疏水乙基结合结构域和两者之间的环化腔。从而在结构上解释了分子环化的机制。SAT结构域特异地选择起始单元,通过转移起始单元到ACP结构域来起始聚酮化合物的合成[11]。
利用基因敲除和异源表达等方法,已证明真菌苯二酚内酯聚酮代谢物的生物合成是由一对还原型和非还原型的聚酮合酶共同催化完成。还原型聚酮合酶合成聚酮链(最终形成苯二酚内酯的大环部分)作为起始单元被转移到非还原型聚酮合酶上,通过非还原型聚酮合酶得到延伸,延伸的聚酮链部分不再被进一步还原。随后,非还原型聚酮合酶逐步催化形成苯二酚环和大环内酯环。在苯二酚内酯中,二羟基苯甲酸内酯(RALs)在C2~C7形成苯环,而二羟基苯乙酸内酯(DALs)则在C3~C8形成苯环[12]。天然RALs内酯环一般为12或14环(RAL12或RAL14),例如Monocillin Ⅱ。而DALs内酯环为10或12环(DAL10或DAL12)。但对于聚酮链是如何起始、延长、折叠以及环化仍有待进一步研究。
通过将不同真菌来源的还原型聚酮合酶和非还原型聚酮合酶随机重组能够实现一系列“非天然”苯二酚内酯类聚酮产物的一步合成[3]。本研究将通过交换不同真菌苯二酚内酯合成途径中非还原型聚酮合酶的起始物酰基转移酶结构域(SAT),并在酵母中与相应的还原型聚酮合酶组合表达,以期合成新的聚酮化合物和发现起始物酰基转移酶结构域的识别规律。
1 材料与方法
1.1 菌株与载体
高频转化大肠杆菌DH10B和酿酒酵母BJ5464为本实验室保存。大肠杆菌-酿酒酵母兼容性表达载体YEpADH2-Trp-FLAGLink和YEpADH2-Ura-FLAGLink为本实验室保存。pJET1.2/blunt高拷贝克隆质粒购买于Fermentas公司,具有Amp抗性。
1.2 培养基及培养条件
LB培养基:酵母提取物5 g/L、NaCl 10 g/L、胰蛋白胨10 g/L,固体培养基配制时加入琼脂15 g/L。
YPD培养基:酵母膏10 g/L、蛋白胨20 g/L、D-葡萄糖20 g/L。
SC二缺培养基:不含氨基酸的酵母基本氮源6.7 g/L,Trp-Ura缺失氨基酸混合物0.72 g/L,D-葡萄糖20 g/L。固体培养基配制时加入琼脂20 g/L。
大肠杆菌在LB培养基中37℃培养。酿酒酵母在YPD培养基30℃培养。酵母发酵使用含1%葡萄糖的YPD培养基;酵母筛选选用合成缺陷型培养基SC基本培养基(Trp-Ura二缺培养基)。
1.3 主要试剂与仪器
DNA聚合酶Phusion Hot Start ⅡHigh-Fidelity DNA Polymerase Kit购自Finnzymes公司;质粒提取试剂盒EZ-10 Spin Column plasmid DNA miniPreps Kit购自BIO BASIC INC. 公司;DNA胶回收试剂盒GeneJETTM Gel Extraction Kit、DNA连接试剂盒CloneJETTM PCR Cloning Kit购自Thermo公司,限制性内切酶购于NEB公司和Thermo公司。酵母感受态细胞制备及酵母转化试剂盒Frozen EZ Yeast Transformation Ⅱ(TZ001) 购自ZYMO RESEARCH公司。引物由Bioneer公司合成。高压液相色谱仪(Hp Hewlett-Packard Series 1050)为Agilent/Hewlett-Packard公司产品。
1.4 基因克隆
以Genbank中已注释信息的真菌聚酮合酶基因为参照,利用生物信息学软件FGENESH和手工相结合的方法预测选定基因的内含子和外显子区域。每个基因都被设计为翻译起始位点含有NdeⅠ(或AseⅠ)酶切位点,终止子后含有PmeⅠ酶切位点,利用融合PCR的方法将预测的外显子组装在一起合成完整的无内含子的基因。融合PCR扩增程序为:98℃预变性30 s;98℃变性10 s,62℃退火20 s,72℃延伸,延伸时间依产物而定,2 kb/min,30个循环;72℃延伸10 min。
1.5 酵母表达载体构建
YEpADH2-Trp-FLAGLink和YEpADH2-Ura-FLAGLink为兼容性酿酒酵母表达载体,其启动子为乙醇脱氢酶诱导启动子,可以起始真菌聚酮合酶基因的表达。将获得的选定基因连接到pJET1.2/blunt大肠杆菌高拷贝克隆质粒上,测序正确后,使用NdeⅠ(或AseⅠ)和PmeⅠ双酶切pJET1.2/blunt质粒及酿酒酵母表达载体,将切胶回收的非还原型聚酮合酶基因片段与YEpADH2-Trp-FLAGLink使用T4 DNA连接酶连接,还原型聚酮合酶基因片段与YEpADH2-Ura-FLAGLink载体连接。经酶切验证连接正确后用于酿酒酵母的转化。
1.6 SAT结构域的交换
通过融合PCR的方法,将乙醇脱氢酶诱导型启动子ADH2p和终止子ADH2t分别克隆到pXW06和pXK30F上,还原型聚酮合酶基因和非还原型聚酮合酶基因的载体分别命名为pXK30F-nr PKS和pXW06-Red PKS。在实验室已成功构建好的聚酮化合物10,11-dehydrocurvularin和resorcylide 非还原型聚酮合酶AtCurS2和AzResS2表达质粒的基础上,通过融合PCR的方法将asperfuranone非还原型聚酮合酶AfoE与AtCurS2和AzResS2的SAT结构域进行交换。融合PCR扩增程序见1.2.1。
1.7 酵母转化
使用Frozen EZ Yeast Transformation Ⅱ(TZ001)试剂盒将构建的载体转入到酿酒酵母中,该酵母的染色体上整合有构巢曲霉磷酸泛酰巯基乙胺基转移酶,使其表达的聚酮合酶具有活性。在Trp和Ura二缺SC培养基上筛选双质粒酵母转化菌落。
1.8 发酵及产物萃取
分别挑取Trp和Ura二缺SC培养基平板上的2个菌落,接种到SC二缺液体培养基中至OD600nm=0.8,加入等体积的酵母丰富培养基1%葡萄糖的YPD,发酵72 h,调节菌液的pH至5.0,用等体积的乙酸乙酯萃取发酵产物,真空低温蒸馏,用少量的甲醇溶解,14 000 r/min离心10 min,取20 μL用于HPLC分析。
1.9 产物鉴定
采用高效液相色谱法(HPLC)测定发酵产物。采用Kromasil C18色谱柱(250 mm×4.6 mm, 5 μm,SUPELCO Inc.)。HPLC条件为:采用水(0.1%TFA)/乙腈(0.1%TFA)为流动相。0 min:水-乙腈(95∶5);5 min:水-乙腈(95∶5);15 min:水-乙腈(5∶95);20 min:水-乙腈(5∶95);25 min:水-乙腈(95∶5)。柱温为室温;进样量为15 μL;检测波长300 nm;流速0.8 mL/min。
2 结果与分析
2.1 真菌苯二酚内酯聚酮合酶相似性分析
真菌苯二酚内酯由一对多功能结构域组成的模块化还原型聚酮合酶(HR PKS)和非还原型聚酮合酶 (NR PKS)协同催化合成。在组成上还原型聚酮合酶各结构域的排列方式为KS-AT-DH-KR-ER-ACP,非还原型聚酮合酶各结构域的排列方式为SAT-KS-AT-PT-ACP-TE(图1)。不同真菌的HR PKS和NR PKS具有较高的直向同源关系。以合成苯二酚内酯radicicol、dehydro-curvularin、lasiodiplodin和resorcylide的聚酮合酶为例,还原型聚酮合酶氨基酸的一致性为54%~59%,非还原型聚酮合酶氨基酸的一致性为60%~68%。

图1 苯二酚内酯聚酮合酶的结构Fig.1 The structure of fungal benzenediol lactones polyketide synthase.
在构巢曲霉Aspergillusnidulans中,聚酮asperfuranone和苯二酚内酯具有相似的生物合成途径,其核心骨架由还原型聚酮合酶AfoG和非还原型聚酮合酶AfoE协同合成。 AfoE由8 285个氨基酸组成,疏水性氨基酸占20.64%。将AfoE与参与合成上述苯二酚内酯的4个非还原型聚酮合酶(CcRadS2、AtCurS2、LtLasS2、AzResS2)进行序列比对。结果表明:AfoE与上述4种苯二酚内酯非还原型聚酮合酶的序列相似性为50%~51%,其他4个聚酮合酶之间的序列相似性都达到60%以上(表1)。AfoE与上述四种苯二酚内酯聚酮合酶的差异,有可能通过合成途径或结构域的重组合成新的聚酮化合物。
2.2 AfoE SAT结构域的互换研究
在苯二酚内酯聚酮的生物合成中,利用不同来源的还原型聚酮合酶和非还原型的聚酮合酶重组,或者替换非还原型聚酮合酶的起始物酰基转移酶结构域SAT可合成“非天然”的聚酮化合物。
本研究采取了体外克隆丝状真菌构巢曲霉asperfuranone非还原型聚酮合酶afoE基因,并对AfoE的SAT结构域与10,11-dehydrocurvularin和resorcylide 非还原型聚酮合酶AtCurS2和AzResS2的SAT结构域在体外进行了交换,形成了新的杂合非还原型聚酮合酶AfoE-SATAtCurS2、AfoE-SATAzResS2、AtCurS2-SATAfoE和AzResS2-SATAfoE。通过重组酵母,分离发酵产物,发现土曲霉AtCurS2及AzResS2的SAT结构域不能识别asperfuranone还原型聚酮合酶AfoG合成的产物,因此没有新的产物合成。而构巢曲霉非还原型聚酮合酶的SAT结构域却可以识别10,11-dehydrocurvularin和resorcylide还原型聚酮合酶AtCurS1和AzResS1合成的产物,异源合成新的化合物1~5(图2)。

表1 真菌苯二酚内酯聚酮合酶相似性分析Table 1 Similarity analysis of fungal benzenediol lactones polyketide synthase.
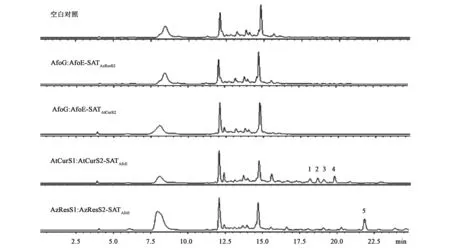
图2 重组发酵代谢产物变化Fig.2 Biosynthetic products of S.cerevisiae BJ5464-NpgA coexpressing nrPKS and hrPKS.
这些产物具有苯二酚内酯类聚酮典型的紫外吸收光谱。以上结果表明,除了SAT结构域,非还原型聚酮合酶的其他结构域也对还原型聚酮合酶的产物有选择利用的能力。鉴于真菌聚酮合酶基因的多结构域的复杂特性,对于其程序化合成机制仍需进一步研究。
3 讨论
在基因组中,合成苯二酚内酯的聚酮合酶HR PKS和NR PKS 编码基因位于同一基因簇上。在构巢曲霉中,有一对符合苯二酚内酯聚酮合酶特征的基因,非还原型聚酮合酶基因afoE及还原型聚酮合酶基因afoG,但在不同的培养条件下均分离不到聚酮类次生代谢产物,将afoE缺失突变,代谢谱也没有变化,说明这对基因在培养条件下是沉默的。将这对基因上游转录因子的启动子更换为可诱导的乙醇脱氢酶启动子alcA(P)后,在诱导条件下可以激活整个基因簇的表达,而且能够检测到新的聚酮类化合物,命名为asperfuranone[9],结构鉴定表明其结构中含有一个高度还原的侧链[7]。本实验进一步证明更换基因启动子可以用于激活沉默基因的表达,并且体外构建表达质粒可以缩短产物生产周期,用于大量次级代谢产物的组合生物合成中。
非还原型聚酮合酶SAT结构域特异识别底物,开启链的延伸。研究表明SAT结构域可以识别与天然底物不同的起始单元。Xu等[1]通过交换不同真菌苯二酚内酯非还原型聚酮合酶的SAT结构域,能够产生新的聚酮化合物或提高产物的产量。Wang等[2]也使用相同策略通过交换丝状真菌构巢曲霉非还原型聚酮合酶AfoE(asperfuranone biosynthesis)和杂色曲霉素聚酮合酶(sterigmatocystin biosynthesis,StcA)的SAT结构域得到一个StcA-AfoE杂合聚酮合酶,能够催化产生一种新的主要次级代谢产物。产物结构鉴定表明非还原型聚酮合酶(NR-PKSs)的SAT结构域可以识别长度与天然底物不同的起始单元。本研究中,土曲霉AtCurS2及AzResS2的SAT结构域不能识别asperfuranone还原型聚酮合酶AfoG合成的产物,这可能是由于起始单元太短,不能识别新的SAT结构域,因此不能产生代谢产物,或者是由于这些次级代谢产物虽然能够产生,但由于产量太低,不易检测到。另一种可能是结构域之间不兼容,因而形成的嵌合体没有活性。同时在构巢曲霉中,聚酮化合物中间体的甲基化作用也会影响分子的空间结构。具体原因仍需进一步实验验证。通过苯二酚内酯基因的人工合成、异源表达以及结构域的交换为人工合成“非天然”次级代谢产物提供了一种新的方法。这种“即插即用”模块化方法可应用于结构多样的全新化学物质的组合生物合成,为揭示天然聚酮类化合物的合成机制奠定了重要的理论基础。本研究对进一步开发新型聚酮类生物活性物质、预测真菌聚酮合酶基因的功能和提高真菌基因组注释精度具有重要的参考价值。
[1] Xu Y,Espinosa-Artiles P,Schubert V,etal.. Characterization of the biosynthetic genes for 10,11-dehydrocurvularin,a heat shock response-modulating anticancer fungal polyketide fromAspergillusterreus[J]. Appl. Environ. Microbiol.,2013,79(6):2038-2047.
[2] Wang S,Xu Y,Maine E A,etal.. Functional characterization of the biosynthesis of radicicol, an Hsp90 inhibitor resorcylic acid lactone fromChaetomiumchiversii. [J]. Chem Biol.,2008,15(12):1328-1338.
[3] Xu Y, Zhou T, Zhang S,etal.. Diversity-oriented combinatorial biosynthesis of benzenediol lactone scaffolds by subunit shuffling of fungal polyketide synthases.[J]. Proc. Nat. Acad. Sci. USA,2014,111(34):12354-12359.
[4] Austin M B,Bowman M E,Ferrer J L,etal.. An aldol switch discovered in stilbene synthases mediates cyclization specificity of type Ⅲ polyketide synthases[J]. Chem. biol.,2004,11(9):1179-1194.
[5] Cheng Y Q,Tang G L,Shen B. Type I polyketide synthase requiring a discrete acyltransferase for polyketide biosynthesis[J]. Proc. Nat. Acad. Sci. USA,2003,100(6):3149-3154.
[6] Liu T,Sanchez J F,Chiang Y M,etal.. Rational domain swaps reveal insights about chain length control by ketosynthase domains in fungal nonreducing polyketide synthases[J]. Org. Lett.,2014,16(6):1676-1679.
[7] Ma Y, Smith L H, Cox R J,etal.. Catalytic relationships between type I and type Ⅱ iterative polyketide synthases:TheAspergillusparasiticusnorsolorinic acid synthase[J]. ChemBioChem,2006,7(12):1951-1958.
[8] Xu Y,Zhou T,Zhang S,etal.. Thioesterase domains of fungal nonreducing polyketide synthases act as decision gates during combinatorial biosynthesis[J]. J. Am. Chem. Soc.,2013,135(29):10783-10791.
[9] Liu T,Chiang Y M,Somoza A D,etal.. Engineering of an “unnatural” natural product by swapping polyketide synthase domains inAspergillusnidulans[J]. J. Am. Chem. Soc.,2011,133(34):13314-13316.
[10] Li Y,Chooi Y H,Sheng Y,etal.. Comparative characterization of fungal anthracenone and naphthacenedione biosynthetic pathways reveals an α-hydroxylation-dependent claisen-like cyclization catalyzed by a dimanganese thioesterase[J]. J. Am. Chem. Soc.,2011,133(39):15773-15785.
[11] Crawford J M,Thomas P M,Scheerer J R,etal.. Deconstruction of iterative multidomain polyketide synthase function[J]. Science,2008,320(5873):243-246.
[12] Xu Y,Zhou T,Zhou Z,etal.. Rational reprogramming of fungal polyketide first-ring cyclization[J]. Proc. Nat. Acad. Sci. USA, 2013,110(14):5398-5403.
Combinatorial Biosynthesis of Benzenediol Lactones with Domain Swaps
BAI Jing, LU Wei, XU Yu-quan, CHEN Ming*
BiotechnologyResearchInstitute,ChineseAcademyofAgriculturalSciences,Beijing100081,China
Fungal benzenediol lactones, biosynthesized by a pair of highly reduced polyketide synthases and non-reduced polyketide synthases, have diverse structures and varied biological activities. Fungal polyketide synthases contain multiple functional domains which catalyze the specific structure of final products. We found that unnatural polyketides could be synthesized in the yeastSaccharomycescerevisiaeco-expressing different reduced fungal polyketide synthases and non-reduced polyketide synthases with functional domain swaps. Furthermore, identification pattern of acyltransferase domain was studied and discussed.
polyketide synthases; fungi; benzenediol lactones; biosynthesis
2014-12-16; 接受日期:2014-12-30
转基因生物新品种培育科技重大专项(2014ZX08003-001);国家863计划项目(2012AA02A703);农业部公益性行业科研专项(201103007)资助。
白净,硕士研究生,主要从事次生代谢产物的组合生物合成研究。E-mail:baijing19901028@163.com。*通信作者:陈明,研究员,博士,主要分子微生物生物学研究。E-mail:chenming01@caas.cn
10.3969/j.issn.2095-2341.2015.01.08
