铜胁迫对农田土壤酶活性、细菌和古菌数量的影响
2014-09-27王斐黄益宗王小玲高柱余发新徐峰保琼莉胡莹乔敏金姝兰黄永春李季向猛
王斐,黄益宗,王小玲,高柱,余发新,徐峰,保琼莉,胡莹,乔敏,金姝兰,黄永春,李季,向猛
1. 中国科学院生态环境研究中心, 北京 1000852. 农业部环境保护科研监测所, 天津3001913. 江西省科学院生物资源研究所, 南昌 3300964. 上饶师范学院, 上饶 334000
铜胁迫对农田土壤酶活性、细菌和古菌数量的影响
王斐1,黄益宗2,*,王小玲3,高柱3,余发新3,徐峰1,保琼莉1,胡莹1,乔敏1,金姝兰4,黄永春2,李季1,向猛1
1. 中国科学院生态环境研究中心, 北京 1000852. 农业部环境保护科研监测所, 天津3001913. 江西省科学院生物资源研究所, 南昌 3300964. 上饶师范学院, 上饶 334000
为了研究铜矿周边农田土壤中不同Cu含量对土壤酶活性、细菌和古菌基因拷贝数的影响,在铜矿周边农田土壤中采集了8个不同Cu含量的土壤,测定土壤的酶活性、细菌和古菌基因拷贝数,结果表明,土壤总Cu含量为28.30~1 019.27 mg·kg-1,酸可提取态Cu含量为3.35~415.11 mg·kg-1,细菌16S rRNA基因拷贝数在每克干土3.10×1010~1.84×1011个之间,古菌16S rRNA基因拷贝数在每克干土7.37×108~6.82×109个之间。土壤总Cu和酸可提取态Cu含量对土壤脲酶活性影响较大,但对土壤脱氢酶和转化酶活性影响不显著。土壤细菌基因拷贝数均与总Cu含量、各形态Cu含量呈极显著的负相关关系,同样土壤古菌基因拷贝数与总Cu含量、各形态Cu含量也均呈极显著的负相关关系,说明Cu胁迫对土壤细菌和古菌活性具有较大的抑制作用。
real-time PCR;铜;细菌;古菌;脲酶;脱氢酶;转化酶
世界矿产资源年评报道,我国Cu储量占全球储量的4%,居世界第6位,随着工业的发展,在2010年,我国对铜矿的需求占到了全球的41%[1-2]。Cu需求量的增加使铜矿的开采和冶炼活动不断加强,导致大量的Cu进入到矿区周边的农田土壤,给土壤的生态平衡带来了巨大的破坏。Cu是生物所需的微量元素,但作为毒性最强的重金属元素之一,过量的Cu会对植物的正常生长造成负面的影响,比如会造成植物根的褐化、降低植物叶片的酶活性等[3-4]。土壤酶来自于植物和土壤微生物,且可以将大分子物质分解成小分子物质而为植物和微生物所用,因此土壤酶是参与土壤中物质能量代谢的重要物质,有学者将土壤酶作为重要指示物来反映土壤环境质量的变化[5]。由于脲酶对重金属非常敏感,人们常把脲酶作为监测土壤重金属污染状况的指示物[6-7]。Cu污染可以对土壤微生物的生长和繁殖产生较大的影响[8-10],一定浓度范围的Cu可造成土壤微生物群落结构的改变[11-12]。细菌和古菌是土壤中广泛存在的微生物,其种群数量可以直观地反映出土壤环境的变化,尤其是细菌数量常被用来反映生态系统整体是否稳定的指标之一[13-14]。
矿区周边农田土壤中不同Cu含量对土壤酶活性和微生物活性的影响报道较少,尤其是对古菌活性的影响还未见报道。本文在矿区周边农田土壤中采集不同Cu含量的土壤,主要研究土壤中的细菌和古菌对Cu长期胁迫的响应,利用Real-time PCR技术分析土壤中细菌和古菌的基因拷贝数与受Cu长期胁迫的土壤中Cu全量、各形态Cu含量的相关关系,同时研究Cu胁迫对土壤酶活性的影响,以期为Cu污染土壤监测和修复提供科学依据。
1 材料和方法(Materials and methods)
1.1 土壤采集与测定
在江西某铜矿冶炼厂周边根据不同Cu污染状况采集农田土壤样品8个,每个土壤样品由5~8个蛇形取样的0~20 cm表层土壤样品混合而来。土壤样品装入自封袋带回实验室,一部分在室温下风干,过100目筛后储存在聚乙烯塑料瓶内,进行理化性质的测定;一部分直接贮存在-80℃冰箱里,以备土壤DNA的提取、细菌和古菌定量PCR的测定。土壤基本理化性质及土壤酶活性参考《土壤农业化学分析方法》进行分析测定[15],其中,总Cu含量采用王水-高氯酸法进行消解后用ICP-OES和ICP-MS进行测定;pH值采用水土比2.5:1法处理后用DELTA 320 pH仪进行测定。土壤CEC(阳离子交换量)采用乙酸铵法、有机质采用低温外热重铬酸钾比色法、土壤总碳、总氮、总硫和碳氮比采用元素分析仪(Vario ELIII, Elementar company, Germany )进行测定。土壤的基本理化性质见表1。
1.2 土壤Cu的形态提取方法
采用Rauret等[16]改进的BCR分级提取法提取土壤中Cu的化学形态。将Cu分级提取的各形态含量与总量相比计算回收率,Cu的回收率在95%~110%之间,BCR具体提取步骤见表2。
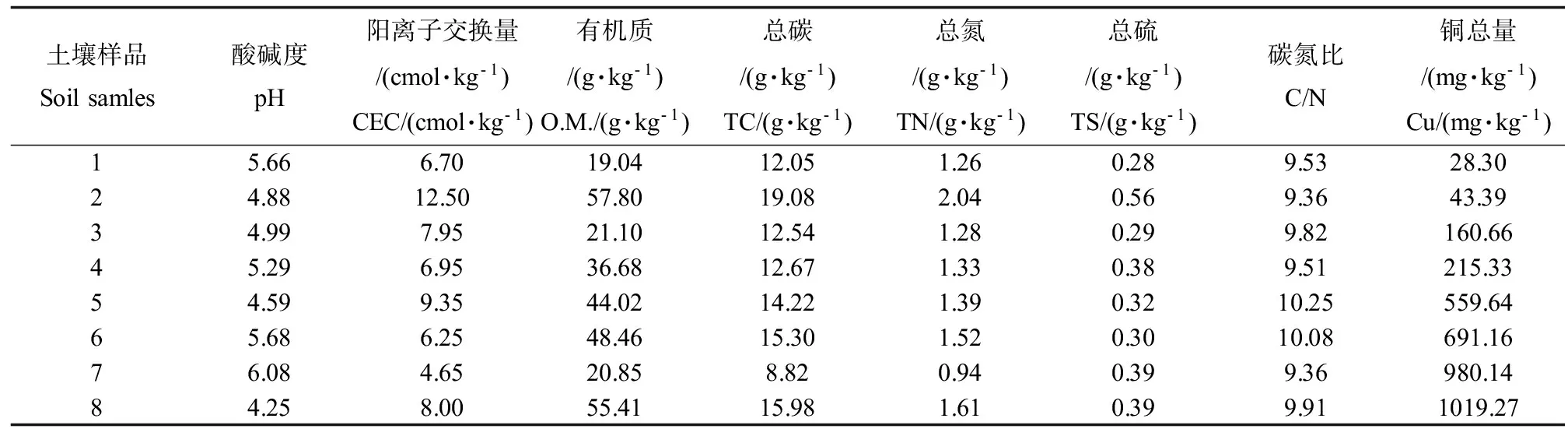
表1 土壤的基本理化性质Table 1 The basic properties of the soil samples
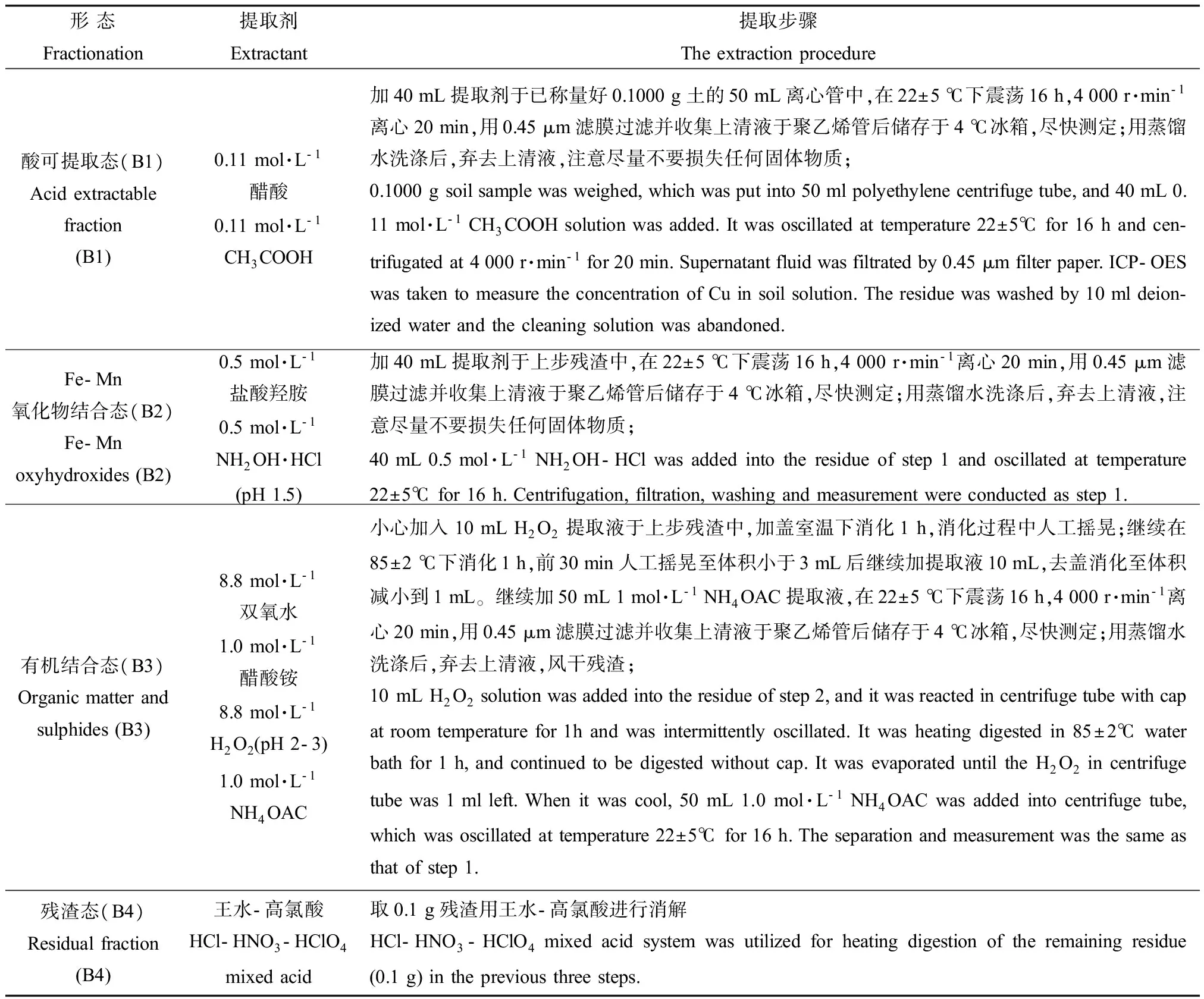
表2 BCR提取步骤Table 2 Sequential extraction procedure of BCR
1.3 土壤DNA的提取和测定
土壤样品均采用FastDNA SPIN Kit(Bio 101, Inc., USA)试剂盒提取其总DNA,提取过程按试剂盒说明书进行。待土壤总DNA提取完成后,用浓度为1%的琼脂糖凝胶电泳配合marker检验DNA的片段大小,并用ND-1000 Spectrophotometer(NanoDrop)分析仪器测定所提取DNA的浓度和OD260/OD280值,保证所提取DNA的质量。
土壤中细菌和古菌的16S rRNA基因,所用引物由英潍捷基(上海)有限公司合成,引物详情及其反应条件见表3。用Eppendorf PCR仪进行PCR预实验,摸索基因的反应条件和体系。Real time PCR反应在型号为iQ5(Bio-Rad, USA)定量PCR仪上运行,以测定土壤中细菌和古菌的基因拷贝数。细菌的基因扩增反应使用TaKaRa公司的PremixExTagTM(Probe qPCR) 试剂盒,古菌的基因扩增反应采用TaKaRa公司SYBR®PremixExTaqTM(Perfect Real Time)试剂盒进行。
1.4 定量所用标准曲线
用于细菌和古菌基因定量的标线分别采用供试土壤样品的克隆子。具体的方法是:(1)提取土壤总DNA(Fast DNA®SPIN Kit for soil);(2)以土壤DNA为模板,应用特异性引物扩增目标片段;(3)所得PCR产物经切胶纯化后连接在pGEM-T Easy载体(Promega, Corp., Madison, Wis., USA),然后转化到大肠杆菌JM109感受态细胞(TaKaRa)中,筛选获取的白色菌斑采用T7/SP6通用引物鉴定阳性克隆值并测序验证;(4)阳性克隆子在LB+Ampicillin(氨苄
青霉素)培养基上27℃过夜培养,采用MiniBEST质粒纯化试剂盒(TaKaRa)提取质粒DNA,并用Nanodrop测定其浓度;(5)将质粒DNA进行10倍梯度系列稀释制作标准样品和待测样品一起扩增,根据所得标准曲线计算出样品中的基因拷贝数,最后以基因拷贝数每克干土为单位进行分析。
1.5 数据统计分析
实验中定量所得数据分析利用iCyclery软件(Version 1.0.1384.0 CR)进行,土壤中细菌和古菌的数量动态可分别从基因拷贝数变化反映出来。采用SPSS 19.0对数据进行ANOVA(单因素方差分析)及相关性分析。绘图采用Sigma Plot 12.5完成。
2 结果(Results)
2.1 不同样点土壤Cu的赋存形态
样品3~样品8的Cu的总量均超出了国家土壤环境质量二级标准[17]。采用改进的BCR法提取的各土壤样品的Cu的含量见表4,从表中可以看出,不同样品Cu的各提取态含量及百分比差异较大,同时Cu总量较大的样品其酸可提取态Cu含量也较多。样品3-样品8的酸可提取态Cu含量较大而样品1和样品2的酸可提取态Cu含量较小,这是因为样品3-样品8采自靠近铜冶炼厂周边的农田,该土壤受到了Cu的严重污染。
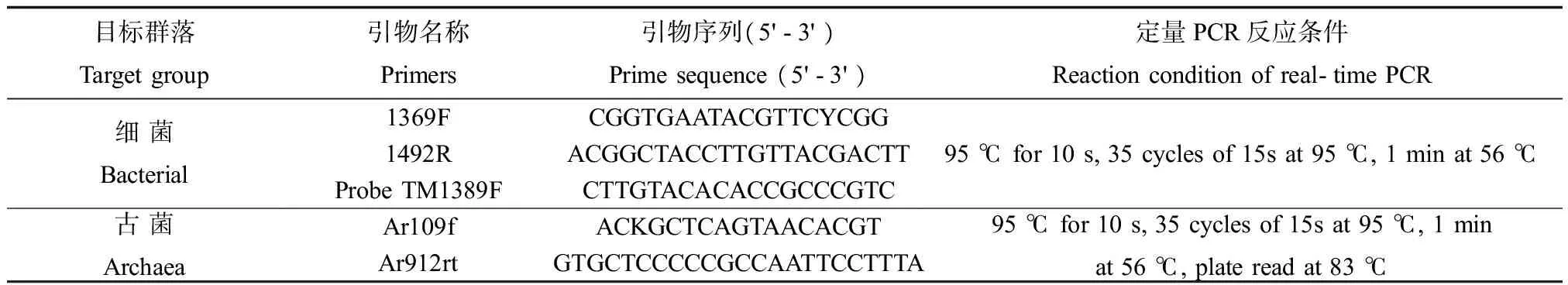
表3 实时荧光定量PCR实验中使用的引物及PCR条件Table 3 Primers and PCR conditions used in the real-time PCR
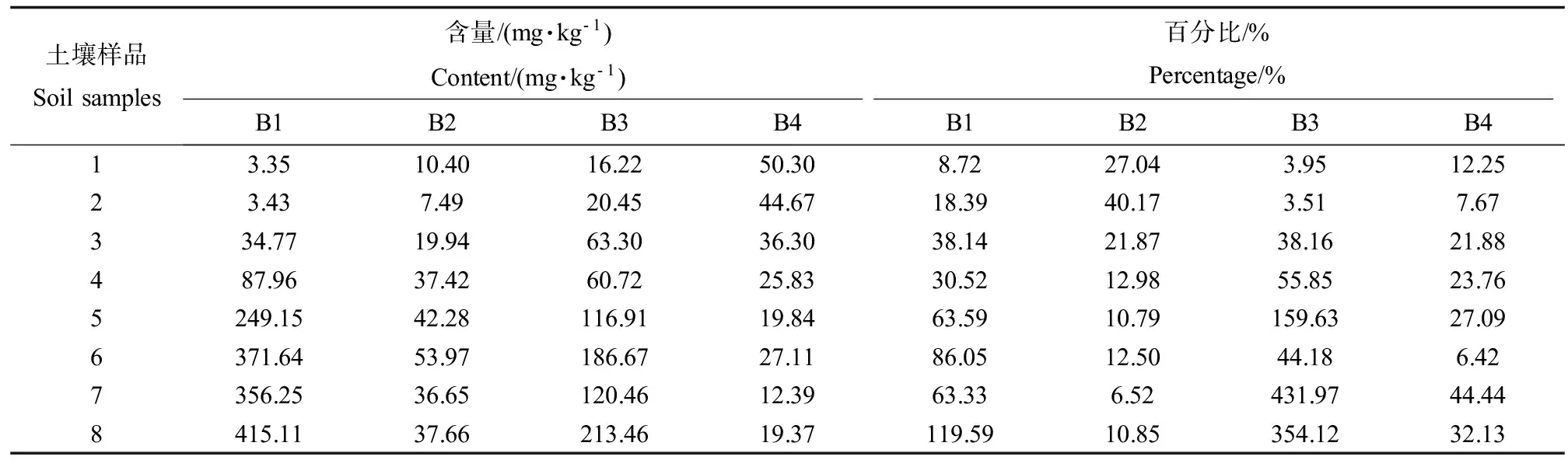
表4 土壤样品中Cu的形态含量及其百分比Table 4 The concentration of extractable Cu by BCR and the fractionation of Cu in different soils
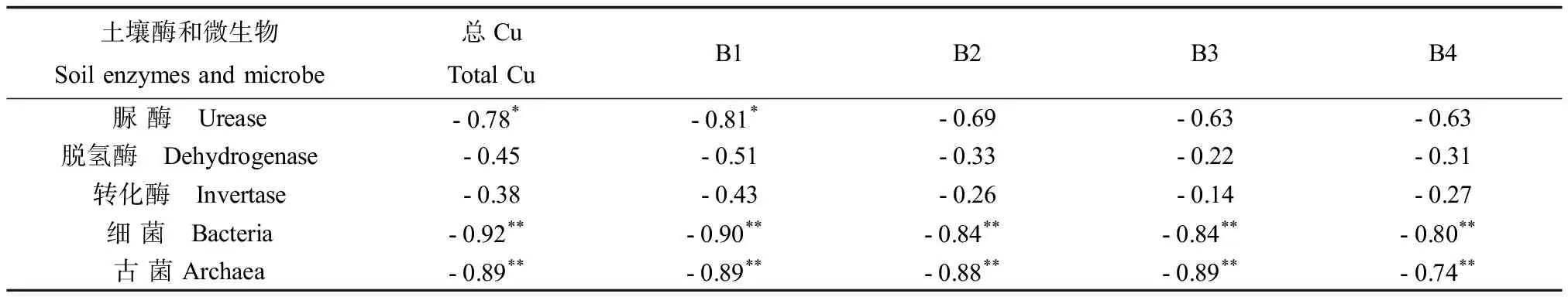
表5 土壤酶活性、细菌和古菌基因拷贝数与总Cu、不同形态Cu之间的相关性关系Table 5 Correlation between the concentrations of Cu and soil enzymes, the gene copies of bacterial and archaea
注:*表示在0.05水平上显著相关,**表示在0.01水平上极显著相关。
Note: * was used to describe significantly correlated at the 0.05 level, **was used to describe significantly correlated at the 0.01 level.
2.2 不同Cu含量对土壤酶活性的影响
图1是不同土壤样品的脲酶活性变化规律,图中发现土壤脲酶活性在8.12~47.08 mg NH3-N·g-1dry soil·d-1范围之间,样品2和样品3的脲酶活性较高,而样品4-样品8的脲酶活性较低,它们对应的土壤Cu含量也是较高的。对不同土壤样品的脲酶活性与Cu总量、Cu形态进行相关分析(表5),发现脲酶活性与土壤Cu总量、酸可提取态Cu含量均呈显著的负相关关系,说明Cu胁迫对土壤脲酶活性影响较大。
图2为不同土壤样品的脱氢酶活性变化规律,从图中看出,土壤脱氢酶活性变化在9.24~29.81 mg TPF·g-1dry soil·d-1范围之间。样品2和样品3的脱氢酶活性最大,样品5~样品7脱氢酶活性较小。尽管样品8的Cu含量最大,但其脱氢酶活性也比较大,仅次于样品2和样品3。土壤脲酶活性与Cu总量、Cu各个形态相关性不显著(表5),说明Cu胁迫不是影响土壤脱氢酶活性的主要因素。
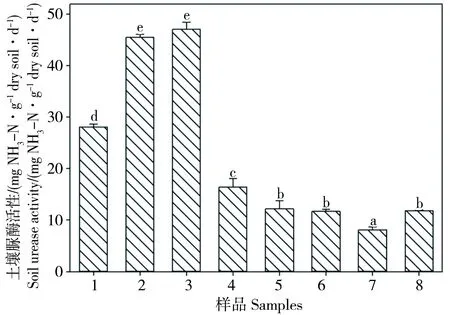
图1 不同土壤样品的脲酶活性变化规律Fig. 1 Soil urease activity in different samples

图2 不同土壤样品的脱氢酶活性变化规律Fig. 2 Soil dehydrogenase activity in different samples
不同土壤样品的转化酶活性变化规律见图3,从图中看出转化酶活性在0.37-4.63 mg glucose·g-1dry soil·d-1范围之间。土壤转化酶活性的变化规律与脲酶、脱氢酶变化不一致,样品2和样品3的转化酶活性最大,样品6和样品7的转化酶活性最小。尽管样品5和样品8的Cu含量较高,但是其转化酶活性也比较大,Cu含量也不是影响土壤转化酶活性的主要因素。相关性分析结果表明(表5),土壤Cu含量与脱氢酶活性不存在显著的相关关系,因此推断土壤转化酶活性可能受Cu含量影响较小。
2.3 不同Cu含量对细菌基因拷贝数的影响
不同土壤样品的细菌基因拷贝数含量情况见图4,从图中看出细菌基因拷贝数在每克干土3.10×1010~1.84×1011个范围之间。样品1~样品3的细菌基因拷贝数含量较高,而样品7、样品8的细菌基因拷贝数含量较低。对不同土壤样品的细菌基因拷贝数与Cu总量、Cu形态进行相关分析(表5),结果显示,细菌基因拷贝数与土壤Cu总量呈极显著的负相关关系,相关系数为-0.92(p<0.0001);细菌基因拷贝数与土壤酸可提取态Cu含量呈极显著的负相关关系,相关系数为-0.90(p<0.001);细菌基因拷贝数与土壤Fe-Mn氧化物结合态Cu、有机结合态Cu和残渣态Cu含量也均呈极显著负相关关系,这表明,Cu胁迫对土壤细菌活动具有较大抑制作用。
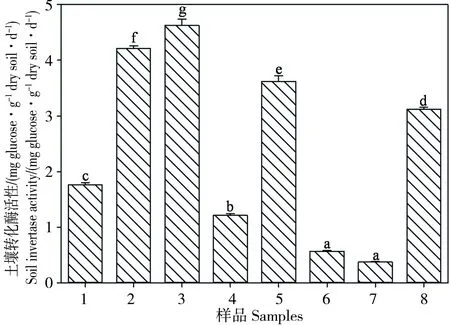
图3 不同土壤样品的转化酶活性变化规律Fig. 3 Soil invertase activity in different samples

图4 不同土壤样品的细菌基因拷贝数变化规律Fig. 4 The quantification of 16S rRNA gene copies of bacteria in different soils
2.4 不同Cu含量对古菌基因拷贝数的影响
不同土壤样品的古菌基因拷贝数含量见图5,图中所示古菌基因拷贝数在每克干土7.37×108~6.82×109个之间。不同土壤样品古菌基因拷贝数的变化规律与细菌基因拷贝数变化相一致,即在Cu含量较高的土壤样品中古菌基因拷贝数较少,反之则较多。利用SPSS软件对古菌基因拷贝数和土壤Cu总量、Cu形态含量进行相关分析(表5),发现土壤古菌基因拷贝数与Cu总量呈显著的负相关关系,相关系数为-0.89(p<0.001);土壤古菌基因拷贝数与酸可提取态Cu含量也呈显著的负相关关系,相关系数为-0.89(p<0.001);古菌基因拷贝数与土壤Fe-Mn氧化物结合态Cu、有机结合态Cu和残渣态Cu含量也均呈极显著的负相关关系,说明Cu胁迫对土壤古菌活动也有抑制作用。
3 讨论(Discussion)
土壤脲酶是与土壤氮循环相关的酶,分为胞内和胞外两种,胞内脲酶在进入土壤后可以迅速地被土壤中的蛋白酶消化,而胞外脲酶由于被固定在土壤固体中因此可以长时间地发挥作用[18]。自Rotini在1935年研究了土壤脲酶活性之后,脲酶受到了人们的广泛关注[19]。Ge等[20]研究Cu污染土壤中脲酶活性的变化,发现土壤总Cu和土壤可溶性Cu均与脲酶呈现出显著的负相关关系。我们的结果与Ge等的结果相一致。Hu等[21]研究Cu或Se对土壤脲酶的影响,结果表明,当Cu的浓度为200、400和800 mg·kg-1时,脲酶活性比对照处理分别下降31.5%、38.1%和59.6%。本文的研究结果表明,当Cu的含量为980.14 mg·kg-1时土壤脲酶活性最低,但是当Cu的含量继续增加时,土壤脲酶活性出现了一定的回升,这可能是因为土壤类型及周围环境对脲酶活性影响较大。土壤脱氢酶常被用作土壤环境变化的指示性物质。Bartowiak和Lemanowicz以磷酸酶和脱氢酶作为快速指示物来探讨重金属对乌尼斯瓦夫盆地土壤质量的影响,发现土壤中Zn含量对脱氢酶有显著影响,原因可能是Zn作为酶的重要组成部分,在一定范围内可以促进酶的活性,但超过一定的阈值后又会显著地抑制酶的活性[22-23]。本研究中,脱氢酶活性并没有和土壤中的Cu含量呈现出明显的相关关系,但是在一些土壤样品中Cu显著地抑制了脱氢酶的活性。土壤转化酶与土壤碳循环相关,是一种水解酶,其可以将土壤中高分子量的蔗糖水解成小分子而被生物所利用[24]。因此,转化酶活性的大小可以反映出土壤中有机物质的转化规律。本研究中,土壤转化酶活性和Cu含量无显著的相关性,但在一些土壤样品中Cu显著地抑制了转化酶的活性。
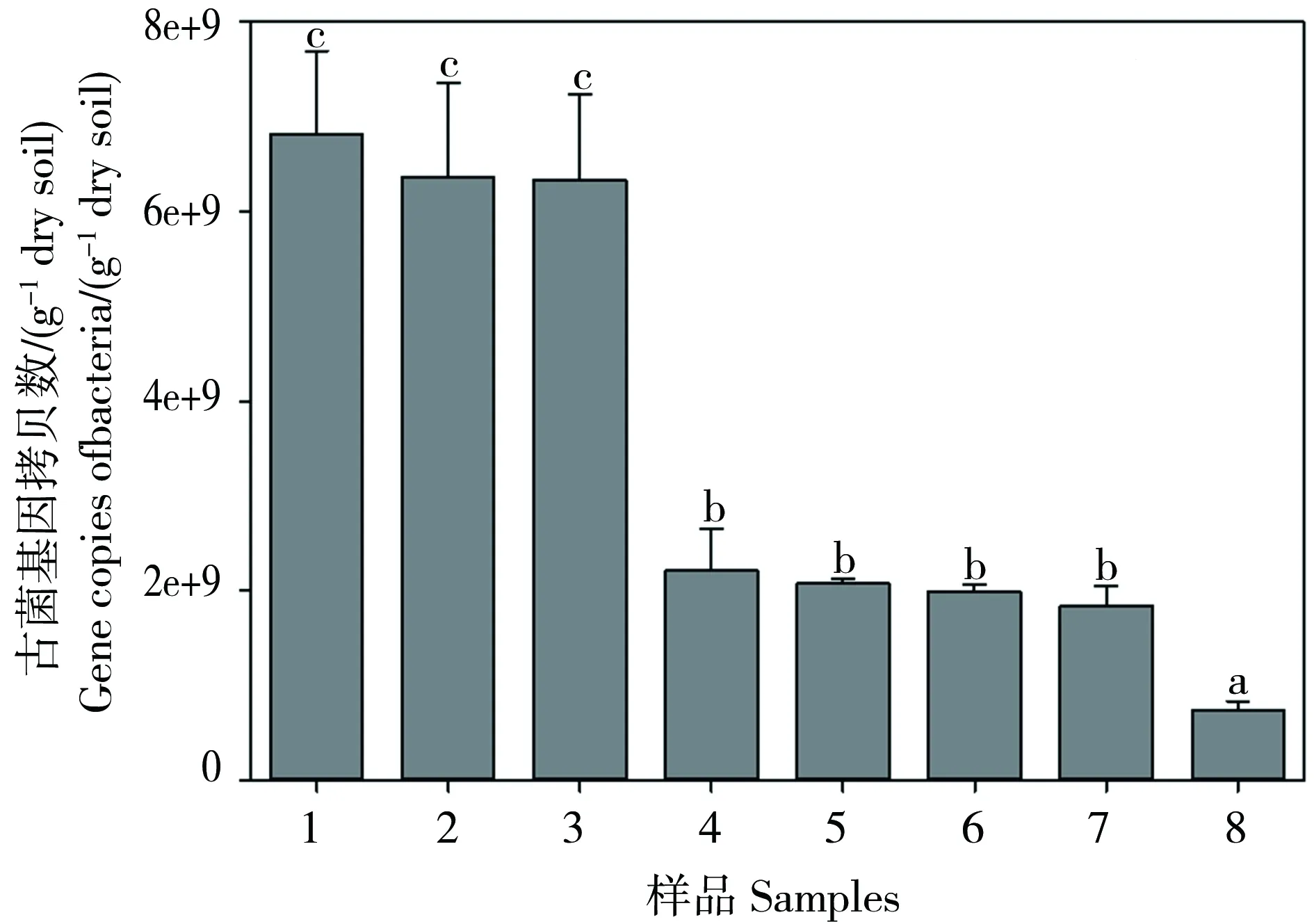
图5 不同土壤样品的古菌基因拷贝数变化规律Fig. 5 The quantification of 16S rRNA gene copies of Archaea in different soils
微生物在土壤新陈代谢和土壤结构的维持中起着重要的作用,微生物的种群和数量可以指示土壤的质量且微生物数量的改变是最简单、最直接的反映土壤质量变化的指标。重金属含量对土壤微生物数量有一定的影响。细菌是土壤系统中广泛存在且所占比例较高的微生物,所以,很多学者通过研究细菌群落数量和结构的变化来反映土壤的质量[25]。Li等[26]研究了不同Cu浓度(0、50、100、200、400、800、1 600和3 200 mg·kg-1)的短期或长期暴露对细菌种群数量变化的影响,结果表明,在短期暴露实验中,细菌基因拷贝数呈整体下降趋势,而在长时间Cu的胁迫下,细菌表现出对环境(3 200 mg·kg-1)的适应性,细菌基因拷贝数呈现出增长趋势。本研究所采集土样已长期受到周边冶炼厂Cu污染的影响,细菌受Cu胁迫的影响结果趋于稳定,即细菌基因拷贝数随Cu总量的增加而减少。Chen等利用PCR-DGGE指纹图谱技术研究了提取态Cu含量和细菌多样性的关系,发现细菌多样性与提取Cu含量在一定范围内呈显著的负相关[27]。本研究利用BCR法对土壤中Cu的不同形态进行了提取,发现土壤酸可提取态Cu含量与细菌基因拷贝数呈显著的负相关,这与Chen等的研究结果相一致。Brajesh等[28]利用焦磷酸测序和T-RFLP(Terminal-restriction fragment length polymorphism)方法对重金属污染土壤的微生物群落结构进行研究,发现重金属胁迫不仅改变了土壤中微生物的数量,也改变了土壤微生物的群落结构,表现为明显的多样性降低。微生物群落结构可以进一步反映土壤生态系统的变化,而基因拷贝数仅仅从表面上反映了受胁迫后土壤微生物的响应,因此,群落结构变化应作为今后土壤微生物受胁迫响应的研究重点,以此来了解核心微生物或功能微生物群体[28]。本文只从细菌基因拷贝数的变化来研究土壤Cu胁迫的影响,今后还需借助T-RFLP或DGGE技术来研究哪一类细菌或功能微生物受到土壤Cu胁迫的影响。
土壤中的古菌在生态系统中起着重要的作用:产甲烷古菌可通过影响厌氧过程来影响碳的循环,而氨氧化古菌可通过硝化作用来影响氮的循环。Tarah等[29]报道,古菌的群落组成与土壤提取态Cu含量呈显著的正相关关系(r=0.61,p<0.001),而我们的结果与其相反,古菌基因拷贝数与土壤Cu总量、酸可提取态Cu含量均呈显著的负相关关系。Xia等的研究也表明,可利用态Cu含量的增加将明显地降低土壤氨氧化古菌的基因拷贝数,这是由于Cu2+、H+、及其它离子对土壤硝化过程的抑制而造成的[8]。Orell等研究Cu对古菌群落的影响,发现Sulfolobusmetallicus(金属硫化叶菌)和Ferroplasmaacidarmanus(嗜酸乳杆菌)均表现出对高浓度Cu(312 mmol·L-1)的适应性。Besaury等的研究表明,Cu虽然降低了古菌群落的丰度,但却提高了它们的活性[30],这是因为Cu浓度的增加诱导了古菌的一些抗氧化基因的表达[31]。为了更加深入地了解Cu胁迫对土壤古菌群落的影响,有必要利用最新的分子生物学手段来确定Cu胁迫下的古菌优势种群,以期为Cu污染土壤修复提供理论依据。
综上所述,Cu胁迫显著地抑制土壤脲酶活性,对土壤脱氢酶和转化酶活性也有一定的影响。土壤细菌基因拷贝数与Cu总量、酸可提取态Cu含量均呈极显著的负相关关系,同样土壤古菌基因拷贝数与Cu总量、酸可提取态Cu含量也呈极显著的负相关关系,说明Cu胁迫对土壤细菌、古菌活动均有明显的抑制作用。
[1] 王 威, 李以科, 封 宁. 全球铜矿资源格局分析[J]. 资源与产业, 2013, 5: 27-32
Wang W, Li Y K, Feng N. Global cooper resource pattern [J]. Resources and Industires, 2013, 15(5): 27-32
[2] 张 波, 管静怡, 李 孟. 中国铜矿企业对外投资现状、风险因素与策略选择[J]. 对外经贸实, 2013, 3: 81-84
Zhang B, Guan J Y, Li M. Status of foreign investment, risk factors and strategies for Chinese copper enterprise [J]. Practice in Foreign Economic Relations and Trade, 2013, 3: 81-84 (in Chinese)
[3] Harmanescu M, Alda L M, Bordean D M, et al. Heavy metals health risk assessment for population via consumption of vegetables grown in old mining area; a case study: Banat County, Romania [J]. Chemistry Central Journal, 2011, 5(1): 1-10
[4] Guo P, Wang T, Liu Y, et al. Phytostabilization potential of evening primrose (Oenotheraglazioviana) for copper-contaminated sites [J]. Environmental Science and Pollution Research, 2014, 21(1): 631-640
[5] Zhan J, Sun Q. Development of microbial properties and enzyme activities in copper mine wasteland during natural restoration [J]. CATENA, 2014, 116: 86-94
[6] Ramesh R, Aarthy M, Gowthaman M K, et al. Screening and production of a potent extracellular Arthrobacter creatinolyticus urease for determination of heavy metal ions. Journal of Basic Microbiology, 2014, 54(4): 285-295
[7] Wang Y P, Shi J Y, Wang H, et al. The influence of soil heavy metals pollution on soil microbial biomass, enzyme activity, and community composition near a copper smelter. Ecotoxicology and Environmental Safety, 2007, 67(1): 75-81
[8] Li X, Zhu Y G, Cavagnaro T R, et al. Do ammonia-oxidizing archaea respond to soil Cu contamination similarly asammonia-oxidizing bacteria? [J]. Plant and Soil, 2009, 324(1-2): 209-217
[9] Kunito T, Nagaoka K, Tada N, et al. Characterization of Cu-resistant bacterial communities in Cu-contaminated soils [J]. Soil science and plant nutrition, 1997, 43(3): 709-717
[10] Li X F, Yin H B, Su J Q. An attempt to quantify Cu-resistant microorganisms in a paddy soil from Jiaxing, China [J]. Pedosphere, 2012, 22(2): 201-205
[11] Wakelin S A, Chu G, Lardner R, et al. A single application of Cu to field soil has long-term effects on bacterial community structure, diversity, and soil processes [J]. Pedobiologia, 2010, 53(2): 149-158
[12] Kimball B E. Biogeochemical cycling of copper in acid mine drainage [D]. The Pennsylvania State University, 2009
[13] Ghosh S, Moitra M, Woolverton C J, et al. Effects of remediation on the bacterial community of an acid mine drainage impacted stream [J]. Canadian journal of microbiology, 2012, 58(11): 1316-1326
[14] Zhan J, Sun Q. Development of microbial properties and enzyme activities in copper mine wasteland during natural restoration [J]. CATENA, 2014, 116: 86-94
[15] 鲁如坤. 土壤农业化学分析方法[M]. 北京: 中国农业科技出版社, 2000
Lu R K. Methods for Soils Agricultural Chemical Analysis [M]. Beijing: Chinese Agricultural Science and Technology Press, 2000 (in Chinese)
[16] Rauret G, López-Sánchez J F, Sahuquillo A, et al. Application of a modified BCR sequential extraction (three-step) procedure for the determination of extractable trace metal contents in a sewage sludge amended soil reference material (CRM 483), complemented by a three-year stability study of acetic acid and EDTA extractable metal content [J]. Journal of Environmental Monitoring, 2000, 2(3): 228-233
[17] G B 15618-1995. 土壤环境质量标准[S]. 1995
GB 15618-1995. Environmental Quality Standard for Soils [S]. 1995 (in Chinese).
[18] Ramesh R, Aarthy M, Gowthaman M K, et al. Screening and production of a potent extracellular Arthrobacter creatinolyticus urease for determination of heavy metal ions [J]. Journal of basic microbiology, 2013
[19] Makoi J H J R, Ndakidemi P A. Selected soil enzymes: Examples of their potential roles in the ecosystem [J]. African Journal of Biotechnology, 2008, 7(3): 181-191
[20] Ge C R, Zhang Q C. Microbial community structure and enzyme activities in a sequence of copper-polluted soils [J]. Pedosphere, 2011, 21(2): 164-169.
[21] Hu B, Liang D, Liu J, et al. Ecotoxicological effects of copper and selenium combined pollution on soil enzyme activities in planted and unplanted soils [J]. Environmental Toxicology and Chemistry, 2013, 32(5): 1109-1116
[22] Bartkowiak A, Lemanowicz J. Application of biochemical tests to evaluate the pollution of the Unislaw basin soils with heavy metals [J]. International Journal of Environmental Research, 2014, 8(1): 93-100
[23] Arriagada C, Almonacid L, Cornejo P, et al. Influence of an organic amendment comprising saprophytic and mycorrhizal fungi on soil quality and growth of Eucalyptusglobulusin the presence of sewage sludge contaminated with aluminium [J]. Archives of Agronomy and Soil Science, 2013, 60(9): 1229-1248
[24] Song Y, Song C, Tao B, et al. Short-term responses of soil enzyme activities and carbon mineralization to added nitrogen and litter in a freshwater marsh of Northeast China [J]. European Journal of Soil Biology, 2014, 61: 72-79
[25] Behera B C, Mishra R R, Patra J K, et al. Impact of heavy metals on bacterial communities from mangrove soils of the Mahanadi Delta (India) [J]. Chemistry and Ecology, 2013, 29(7): 604-619
[26] Li J, Zheng Y M, Liu Y R, et al. Initial copper stress strengthens the resistance of soil microorganisms to a subsequent copper stress [J]. Microbial Ecology, 2014, 67(4): 931-941
[27] Chen Y X, Wang Y P, Wu W X, et al. Impacts of chelate-assisted phytoremediation on microbial community composition in the rhizosphere of a copper accumulator and non-accumulator [J]. Science of the Total Environment, 2006, 356(1): 247-255
[28] Singh B K, Quince C, Macdonald C A, et al. Loss of microbial diversity in soils is coincident with reductions in some specialized functions [J]. Environmental Microbiology. doi: 10.1111/1462-2920.12353.
[29] Sullivan T S, McBride M B, Thies J E. Soil bacterial and archaeal community composition reflects high spatial heterogeneity of pH, bioavailable Zn, and Cu in a metalliferous peat soil [J]. Soil Biology and Biochemistry, 2013, 66: 102-109
[30] Besaury L, Ghiglione J F, Quillet L. Abundance, activity, and diversity of archaeal and bacterial communities in both uncontaminated and highly copper-contaminated marine sediments [J]. Marine Biotechnology, 2013: 1-13
[31] Orell A, Navarro C A, Arancibia R, et al. Life in blue: Copper resistance mechanisms of bacteria and archaea used in industrial biomining of minerals [J]. Biotechnology Advances, 2010, 28(6): 839-848
◆
EffectsofCuStressonEnzymeActivity,BacteriaandArchaeaQuantityinSoils
Wang Fei1,Huang Yizong2,*,Wang Xiaoling3,Gao Zhu3,Yu Faxin3,Xu Feng1,Bao Qiongli1,Hu Ying1,Qiao Min1,Jin Shulan4,Huang Yongchun2,Li Ji1,Xiang Meng1
1. Research Center for Eco-Environmental Sciences, Chinese Academy of Sciences, Beijing 100085, China2. Agro-Environmental Protection Institute, Ministry of Agriculture, Tianjin 300191, China3. Institute of Biological Resources, Jiangxi Academy of Sciences, Nanchang 330096, China4. Shangrao Normal University, Shangrao 334000, China
12 May 2014accepted1 July 2014
To study different Cu contents in surrounding soils of a copper smelting on soil enzyme activity, bacterial and archaeal quantity, eight soil samples were collected, and enzyme activity, bacterial and archaeal quantity in soils were determined. The results showed that the concentrations of total Cu and acid extractable Cu were in the ranges of 28.3-1 019.27 mg·kg-1and 3.35-415.11 mg·kg-1, respectively. The ranges of gene copies numbers of bacteria and archaea were 3.10×1010-1.84×1011and 7.37×108-6.82×109copies per gram of dry soil, respectively. The soil urease activity was significantly affected by total Cu and acid extractable Cu in soils, while soil dehyrogenase and incertase activity were not affected by them. There was significant negative relationship between gene copies of bacteria (or archaea) and total Cu (or different forms Cu), suggesting that the activities of bacteria and archaea could be inhibited by Cu stress.
real-time PCR; copper; bacteria; archaea; urease; dehydrogenase; invertase
2014-05-12录用日期:2014-07-01
1673-5897(2014)4-707-08
: X171.5
: A
黄益宗(1970—),男,博士,研究员,主要从事重金属在土壤-植物系统中的迁移、转化和积累研究,以及污染土壤修复技术、污染生态学等方面的研究,近年来在国内外核心刊物发表论文130多篇。
国家自然科学基金面上项目(21377152)和江西省科学院省级重点实验室开放基金项目(2012-KLB-1和2013-KLB-08)资助
王斐(1988-),女,硕士,研究方向为土壤生态学,E-mail: xiaofeilx.2008@163.com;
*通讯作者(Corresponding author),E-mail: hyz@rcees.ac.cn
10.7524/AJE.1673-5897.20140512002
王 斐,黄益宗,王小玲, 等. 铜胁迫对农田土壤酶活性、细菌和古菌数量的影响[J]. 生态毒理学报, 2014, 9(4): 707-714
Wang F, Huang Y Z, Wang X, et al. Effects of Cu stress on enzyme activity, bacteria and Archaea quantity in soils [J]. Asian Journal of Ecotoxicology, 2014, 9(4): 707-714 (in Chinese)
