利用TAIL-PCR分离申克氏孢子丝菌T-DNA插入突变体侧翼序列的研究
2014-08-25徐廷滔白忠义龙朋朋于保东
徐廷滔,白忠义,龙朋朋,张 攀,于保东
(1.吉林大学植物科学学院,吉林 长春130062;2.吉林大学中日联谊医院,吉林 长春130033)
申克氏孢子丝菌(Sporothrixschenckii)是双相型病原真菌,在自然条件下表现为菌丝相,而在体内和37℃为酵母相。申克氏孢子丝菌引起人和动物的皮肤、皮下组织及其附近淋巴系统的慢性感染,称之为孢子丝菌病(Sporotrichosis)[1]。该病常常与皮肤的轻微外伤后接触被病原菌污染的物质有关。临床上孢子丝菌病分为淋巴管型、固定型及播散型。其中最常见的是淋巴管型,引起皮肤渗出、化脓,甚至溃烂。其次是固定型,常引起面部皮疹,多见于儿童。虽然播散型孢子丝菌病发生率不高,但该病如不及时治疗,可引起死亡[2]。近年来,随着抗肿瘤药物、抗生素等的广泛应用,以及恶性血液病和艾滋病等免疫受损人群的不断扩大,播散型孢子丝菌病呈上升趋势,严重威胁人类健康[3]。
随着后基因组时代的到来,迫切需要了解大量未知基因的功能。农杆菌介导的T-DNA插入突变技术是通过反向遗传学研究基因功能的重要方法,在多种植物及真菌中已获得大量T-DNA插入突变体[4-6]。而T-DNA插入位点侧翼序列的克隆是鉴定突变基因的关键步骤。热不对称交错PCR(thermal asymmetric interlaced PCR,TAIL-PCR)由Liu和Whittier首先研究并报道[6]。TAIL-PCR以基因组DNA为模板,利用低退火温度的简并引物和高退火温度的特异性嵌套引物,通过低特异性PCR和高特异性PCR交替的三轮温度不对称循环,扩增获得特异性产物。由于TAIL-PCR具有操作简单、快速、特异性强、重复性好、成本较低等优点,成为克隆已知序列侧翼的常用方法。
目前,对申克氏孢子丝菌分子生物学研究刚刚起步,要深入了解其致病机制,需要对相关基因进行分离和分析。本研究以申克氏孢子丝菌T-DNA插入突变株为材料,利用TAIL-PCR方法分离突变菌株T-DNA插入位点侧翼序列,为进一步挖掘申克氏孢子丝菌的功能基因,在分子水平上探讨相关机制奠定基础。
1 材料
1.1 菌株
申克氏孢子丝菌(S.schenckii)野生型及T-DNA插入突变菌株Ss1-Ss5由吉林大学中日联谊医院提供。
1.2 引物
本研究所用引物见表1,其中T-DNA-R和T-DNA-F为扩增T-DNA(left border -trpC-hph-right border)引物;LB1,LB2,LB3为TAIL-PCR左边界嵌套引物;RB1,RB2,RB3为TAIL-PCR右边界嵌套引物;AD1,AD2,AD3,AD4为随机引物,上述引物由上海生物工程技术服务有限公司合成。

表1 本研究所用引物
2 方法
2.1 申克氏孢子丝菌基因组DNA的提取
将申克氏孢子丝菌野生型和突变菌株(Ssl-Ss5)接种于5 ml PDB(potato dextrose broth)培养基,于28℃,100 r/min振荡培养5 d。挑取培养的菌丝体,转移至1.5 ml离心管内,10 000 r/min 离心1 min,收集菌丝体,参照文献提供的方法提取孢子丝菌基因组DNA[8,9]。
2.2 T-DNA插入突变体的PCR 验证
利用提取的基因组DNA,以野生型申克氏孢子丝菌为阴性对照,PCR扩增突变菌株(Ssl-Ss5)的T-DNA。PCR反应程序为95℃,2 min后进入循环程序:94℃,30 s;53℃,30 s;72℃,1 min,共30个循环,最后,于72℃延伸10 min[10]。
2.3 TAIL-PCR分离突变体T-DNA侧翼序列
根据T-DNA插入载体pBHt1上的T-DNA序列设计左边界和右边界两套TAIL-PCR特异性嵌套引物LB1、LB2、LB3和RB1、RB2、RB3,同时设计四条随机简并引物AD1~AD4,TAIL-PCR扩增体系及扩增条件见表2和表3。TAIL-PCR是基于简并引物和特异引物的热不对称循环,分为三轮反应:第一轮PCR包括5次高特异性循环、1次低特异性循环、10次较低特异性循环和15次热不对称循环;第二轮PCR是以第一轮PCR反应产物稀释50倍作为模板,通过15次热不对称循环,使特异产物选择性放大,而非特异性的产物降到极低的浓度;第三轮PCR又将第二轮PCR反应产物稀释50倍作为模板,再通过30次热不对称循环,使特异产物进一步得到放大。通过三轮PCR反应即可获得T-DNA邻近的侧翼序列。TAIL-PCR产物经1%琼脂糖凝胶电泳进行检测。TAIL-PCR产物回收后,以RB-3或LB-3作为引物,送北京英骏生物技术有限公司进行测序。
表2 TAIL-PCR反应体系的组成
3 结果
3.1 T-DNA插入突变株Ss1-Ss5分子鉴定
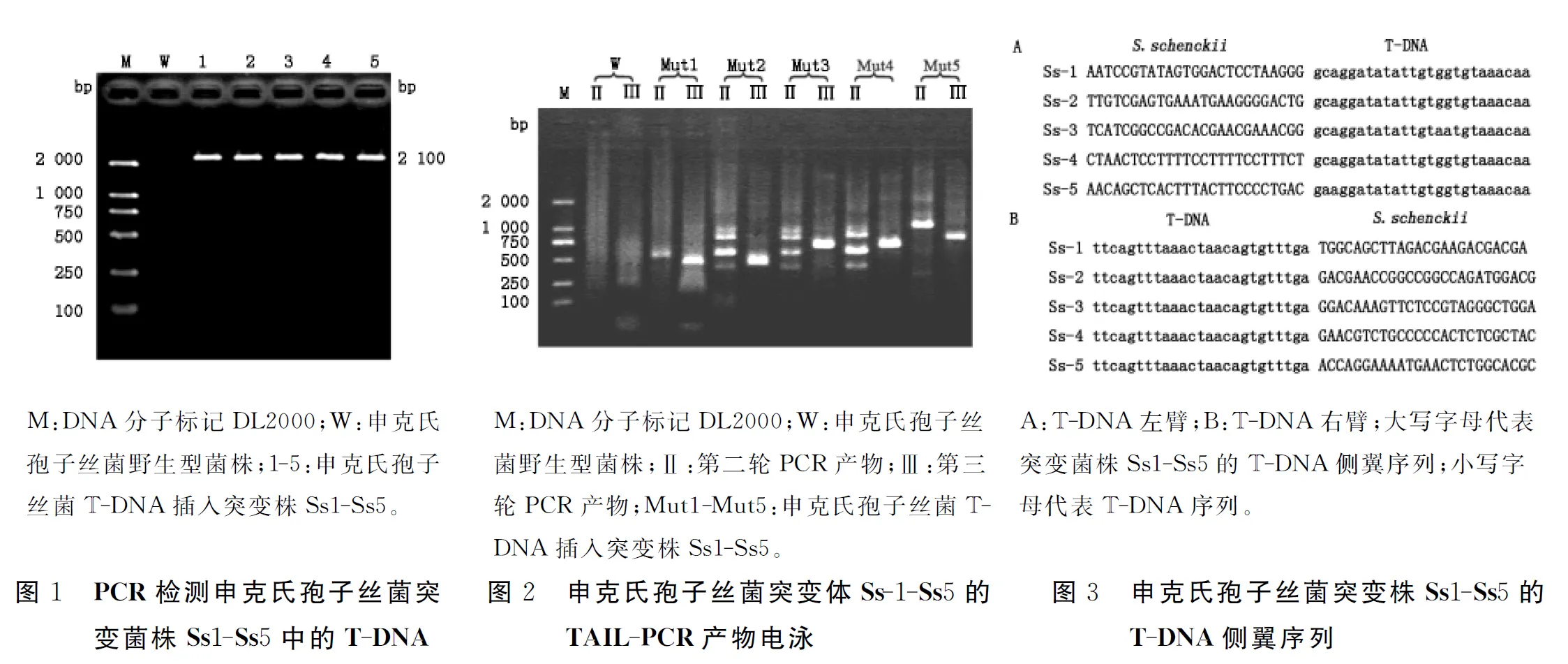
根据申克氏孢子丝菌突变菌株Ss1-Ss5中T-DNA(left border -trpC-hph- right border)左臂、右臂设计特异性引物T-DNA-F和T-DNA-R(两者之间片段大小为2 100 bp),对突变菌株进行PCR鉴定。结果从这5株突变体中均扩增出一条大小约2 100 bp的条带,而野生菌株无特异性条带,表明T-DNA已整合到申克氏孢子丝菌Ss1-Ss5基因组中(见图1)。

表3 TAIL-PCR反应程序
*,**,***和****分别代表5,10,15和30个循环。
3.2 T-DNA插入突变株侧翼序列的分离
取TAIL-PCR的第二轮和第三轮PCR扩增产物,进行琼脂糖凝胶电泳,结果这五株突变体,在第二轮扩增时均获得2-3条PCR产物条带,特异性较差;经过第三轮扩增后,获得大小为300 bp左右的特异条带,且第三轮PCR产物比第二轮PCR产物小100 bp左右(见图2)。产物经凝胶回收试剂盒纯化后进行测序。序列分析表明,利用TAIL-PCR从五株突变株中均获得申克氏孢子丝菌T-DNA插入位点侧翼序列,且侧翼序列之间无同源性(见图3)。
4 讨论
T-DNA插入位点侧翼序列的克隆是分离功能基因的关键步骤。目前已建立了几种用于克隆T-DNA插入位点侧翼序列的方法,如接头连接介导PCR、反向PCR、质粒营救及TAIL-PCR等[11,12]。而由Whitter和Liu等首先研究并报道的TAIL-PCR是利用嵌套的特异引物和简并引物组合,以基
因组DNA作为模板,利用引物长度和特异性的差异设计不对称的温度循环,然后通过三轮反应来扩增特异性的目标产物,获得已知序列的侧翼序列[7]。
利用TAIL-PCR扩增侧翼序列与其它方法相比具有如下优点[13]:首先TAIL-PCR直接以基因组DNA为模板筛选目标序列,可快速获得目标片段。操作简单、快速,省去了酶切、连接、加尾等繁琐步骤。其次,TAIL-PCR技术不涉及连接反应,实验结果较稳定,重复性好,准确可靠。另外,接头连接介导PCR、反向PCR等方法均需要DNA连接酶、限制性内切酶及对引物进行特殊修饰,而TAIL-PCR只需特异引物和简并引物,降低实验成本。
本实验采用TAIL-PCR技术,从5株申克氏孢子丝菌突变体中克隆到T-DNA插入位点的侧翼序列,该实验结果表明TAIL-PCR是简单、快速、高效分离申克氏孢子丝菌T-DNA侧翼序列的有效方法,为深入挖掘申克氏孢子丝菌的功能基因,探讨双相型原真菌的分子机制奠定基础。
参考文献:
[1] Alexandro Bonifaz,Denisse Vázquez-González.Diagnosis and Treatment of Lymphocutaneous Sporotrichosis:What Are the Options[J].Current Fungal Infection Reports,2013,7(3):252.
[2]Luisa HM Miranda,Fátima Conceião-Silva,Leonardo P.Quintella,et al.Feline sporotrichosis:Histopathological profile of cutaneous lesions and their correlation with clinical presentation[J].Microbiology and Infectious Diseases,2013,36(4):425.
[3]Silva-Vergara ML,Maneira FR,De Oliveira RM,et al.Multifocal sporotrichosis with meningeal involvement in a patient with AIDS[J].Med Mycol,2005,43,187.
[4]Maruthachalam K,Klosterman SJ,Kang S,et al.Identification of Pathogenicity-Related Genes in the Vascular Wilt Fungus Verticillium dahliae by Agrobacterium tumefaciens-Mediated T-DNA Insertional Mutagenesis[J].Molecular Biotechnology,2011,49(3):209.
[5]Huser A,Takahara H,Schmalenbach,et al.Discovery of pathogenicity genes in the crucifer anthracnose fungus Colletotrichum higginsianum,using random insertional mutagenesis[J].Mol Plant Microbe Interact,2009,22,143.
[6] Zhang Y, Li G,He D,et al.Efficient insertional mutagenesis system for the dimorphic pathogenic fungus Sporothrix schenckii using Agrobacterium tumefaciens[J].J Microbiol Methods,2011,84,418.
[7] Liu YG,Robert F Whitter.Thermal asymmetric interlaced PCR:automatable amplification and sequencing of insert end fragment from P1 and YAC clones for chromosome walking[J].Genomics,1995,25:674.
[8]Zhang YJ,Zhang S,Liu XZ,et al.A simple method of genomic DNA extraction suitable for analysis of bulk fungal strains[J].Letters in applied microbiology,2010,51(1):114.
[9]Xiaoyan Qu,Baodong Yu,Jinliang Liu,et al.MADS-Box Transcription Factor SsMADS Is Involved in Regulating Growth and Virulence in Sclerotinia sclerotiorum[J].Int J Mol Sci,2014,15:8049.
[10]Sun L,Yan M,Ding Z,et al.Improved dominant selection markers and co-culturing conditions for efficient Agrobacterium tumefaciens-mediated transformation of Ustilago scitaminea[J].Biotechnology letters,2014,1:6.
[11] Erster O,Liscovitch M.A modified inverse PCR procedure for insertion,deletion,or replacement of a DNA fragment in a target sequence and its application in the ligand interaction scan method for generation of ligand-regulated proteins[J].Methods Mol Biol,2010,634:157.
[12]O'Malley RC,Alonso JM,Kim CJ,et al.An adapter ligation-mediated PCR method for high-throughput mapping of T-DNA inserts in the Arabidopsis genome[J].Nat Protoc.2007,2:2910.
[13] Reimer L C,Spura J,Schmidt-Hohagen K,et al.High-Throughput Screening of a Corynebacterium glutamicum Mutant Library on Genomic and Metabolic Level[J].PloS one,2014,9(2):e86799.
