山鹧鸪属鸟类线粒体基因组的比较及系统发育研究
2014-05-25李雪娟黄原雷富民
李雪娟,黄原,雷富民
1. 陕西师范大学生命科学学院,西安 710062;
2. 中国科学院动物研究所动物进化与系统学院重点实验室,北京 100101
山鹧鸪属鸟类线粒体基因组的比较及系统发育研究
李雪娟1,黄原1,雷富民2
1. 陕西师范大学生命科学学院,西安 710062;
2. 中国科学院动物研究所动物进化与系统学院重点实验室,北京 100101
海南山鹧鸪(Arborophila ardens)对生境选择比较严格,种群数量稀少,属于濒危物种。为进一步研究山鹧鸪属的进化和系统发育关系,文章利用Illumina Hiseq2000高通量测序技术获得了海南山鹧鸪线粒体全基因组序列,从比较基因组学角度分析了4种山鹧鸪鸟类的线粒体基因组特征,并探讨了山鹧鸪属鸟类的系统发育地位。研究结果表明:(1) 海南山鹧鸪线粒体基因组长度为16 730 bp,编码13个蛋白质编码基因、2个核糖体RNA基因、22个转运RNA基因以及1个控制区;(2) 山鹧鸪属物种受到了纯化选择的作用,且在进化过程中积累了更多的非同义替换;(3) 山鹧鸪属位于雉科鸟类系统树的基部位置,其中白眉山鹧鸪与红喉山鹧鸪互为姐妹群,海南山鹧鸪位于山鹧鸪属的基部位置,与其他3种山鹧鸪鸟类的亲缘关系较远。
海南山鹧鸪;线粒体基因组;比较基因组学;系统发育
山鹧鸪属(Arborophila)鸟类隶属鸡形目(Galliformes)、雉科(Phasianidae)[1],分布于我国西藏、四川、云南、广西、广东、福建、海南和台湾等地,国外见于印度、缅甸、泰国及东南亚地区。山鹧鸪属鸟类全世界共有 18种,其中 10种分布于我国,4种为我国特有种。海南山鹧鸪(Arborophila ardens)是我国的特有种,仅分布于海南岛,生活环境与白鹇(Lophura nycthemera whiteheadi)及灰孔雀雉(Polyplectron bicalcaratum katsumatae)相似[2]。海南山鹧鸪对生境选择比较严格,主要栖息于海南热带雨林和山地常绿林[2],包括尖峰岭、霸王岭、五指山和吊罗山等地[3]。该物种种群数量稀少,目前已处于濒危状态[2],是国家Ⅰ级重点保护动物,被列入中国濒危动物红皮书[4]和世界濒危鸟类红皮书[5]。国际自然保护同盟(IUCN)物种生存委员会(SSC)把海南山鹧鸪列入2000~2004年的保护行动计划,并建议作为海南物种保护的“旗舰种”[6]。
迄今为止,人们对于海南山鹧鸪的研究还仅局限于换羽[7]、活动区和生境利用[8,9]等方面。为深入研究山鹧鸪属物种的进化和系统发育关系,本研究测定了海南山鹧鸪(A. ardens)线粒体全基因组序列,结合GenBank数据库中已公开的另外3种山鹧鸪属鸟类的线粒体基因组序列——白眉山鹧鸪(A. gingica, FJ752425)、红喉山鹧鸪(A. rufogularis, FJ752424)、四川山鹧鸪(A. rufipectus, FJ194942),对山鹧鸪属鸟类线粒体基因组序列的结构特征进行了比较分析,并联合数据库已公布的线粒体基因组序列构建了雉科鸟类的系统发育树,探讨了山鹧鸪属鸟类的系统发育关系。
1 材料和方法
1.1 材料
海南山鹧鸪标本于2008年采自海南岛,浸泡于无水乙醇中,并保存于-20℃冰箱。相关凭证标本现保存于中国科学院动物研究所动物进化与系统学院重点实验室鸟类标本馆。
1.2 方法
1.2.1 DNA提取、引物设计、PCR扩增和测序
采用传统的酚-氯仿-异戊醇抽提法提取总DNA[10]。参考Sorenson等[11]提供的引物位置和序列,结合GenBank数据库中相关鸡形目鸟类的线粒体基因组序列,本研究最终确定了10对PCR扩增引物,并且使用Oligo 6.0软件对引物进行了评价(附表1)。
PCR反应程序为:93℃预变性2 min;92℃变性10 s,58~53℃复性30 s,68℃延伸10 min,共20个循环;92℃变性10 s,53℃复性30 s,68℃延伸10 min,且延伸步骤每个循环增加20 s,共20个循环;68℃终延伸7 min;4℃保存。PCR扩增体系为15 µL,包括: 10×LA PCR Buffer 1.5 µL,MgCl2(25 mmol/L) 1.5 µL,dNTP(2.5 mmol/L) 2.4 µL,上下游引物(10 µmol/L)各2.1 µL,LA TaqDNA聚合酶(5 U/µL) 0.18 µL,总DNA(模板)1~2 µL,然后加ddH2O补足。
等浓度混合纯化所得质量较好的片段,构建约500 bp的测序文库,采用Illumina Hiseq2000进行双末端测序,reads长度为100 bp。拼接过程中产生的缺口(gap)使用Sorenson等[11]提供的邻近引物以Sanger测序法补全(附表1中的补充引物)。测序工作由深圳华大基因科技有限公司完成。
1.2.2 序列拼接和注释
海南山鹧鸪线粒体基因组序列采用 SOAP-denovo软件进行拼接。线粒体基因组序列用在线软件 tRNAscan-SE1.21[12]预测 tRNA基因的位置及二级结构,必要时需根据典型三叶草形结构特点辅以人工校正,最终得到了除 tRNAVal和 tRNASer(AGY)基因之外20个tRNA的二级结构。参考其他已发表的山鹧鸪属物种线粒体基因组注释信息,本研究进而确定tRNAVal和tRNASer(AGY)基因、蛋白质编码基因、rRNA基因及 CR的位置。参考 RNA数据库(http://www.rna.ccbb.utexas.edu/)中红原鸡(Gallus gallus)、绿头鸭(Anas platyrhynchos)和非洲爪蟾(Xenopus laevis),以及地山雀(Pseudopodoces humilis)[13]、黑尾地鸦(Podoces hendersoni)[14]和黄牛(Bos taurus)[15]的 rRNA二级结构,预测海南山鹧鸪 12S和16S rRNA二级结构。利用MEGA4.1[16]软件分析线粒体基因组核苷酸组成、蛋白质编码基因氨基酸组成等信息。
1.2.3 同义替换率和非同义替换率
本研究将4种山鹧鸪属物种分为6组:海南山鹧鸪-白眉山鹧鸪、海南山鹧鸪-红喉山鹧鸪、海南山鹧鸪-四川山鹧鸪、红喉山鹧鸪-白眉山鹧鸪、红喉山鹧鸪-四川山鹧鸪以及白眉山鹧鸪-四川山鹧鸪,利用 Kaks_calculator2.0软件分别计算 13个蛋白质编码基因的同义替换率(Synonymous substitution rate, Ks)、非同义替换率(Nonsynonymous substitution rate, Ka)以及非同义替换率与同义替换率的比值(Ka/Ks)[17]。
1.2.4 系统发育分析
结合从GenBank数据库下载的数据,本研究共选取雉科7亚科21属24个物种及1个外群(Alectura lathami, NC_007227)的线粒体基因组序列,从线粒体基因组水平探讨了山鹧鸪属鸟类的系统发育关系。将比对好的单个线粒体基因连接成一个联合数据集(mitogenome, PCGs+rRNAs+tRNAs+CR),分别采用最大简约法(Maximum parsimony, MP)、最大似然法(Maximum likelihood, ML)及贝叶斯推论法(Bayesian inference, BI)构建系统树。利用软件MrModelTest 2.2为联合数据集选择最优模型。利用PAUP*4.0 b10软件以启发式搜索构建 MP树(自举 1000次);利用RAxML 7.0.3软件以最优模型(GTR+I+G)构建 ML树(自举1000次);利用MrBayes 3.1.2软件以最优模型(GTR+I+G)构建BI树,采用马尔科夫链的蒙特卡洛(Markov Chain Monte Carlo, MCMC)方法,共运行100万代,舍去前1000个老化样本。
2 结果与分析
2.1 海南山鹧鸪线粒体全基因组序列结构特征
海南山鹧鸪线粒体全基因组高通量测序最终组装的有效数据为126.52 Mb,测序深度为7 668.06 X。海南山鹧鸪线粒体全基因组序列长度为 16 730 bp (GenBank登录号:KJ716444),编码13个蛋白质编码基因、22个tRNA基因、2个rRNA基因(12S和16S rRNA)以及1个控制区(CR)。海南山鹧鸪线粒体基因组结构紧凑,基因排列顺序与鸟类线粒体基因典型的排列方式一致[18](附表2)。
2.1.1 蛋白质编码基因
13个蛋白质编码基因全长11 355 bp(不包括终止密码子),共编码3785个氨基酸,除ND6基因定位在N链上外,其余均定位在J链上。ND3基因第174位置存在胞嘧啶插入。起始密码子除COI基因为GTG、ND5为ATA外,其余均为标准的ATG;有1个基因的终止密码子为AGG(COI),1个为TAG (ND6),3个为不完全终止密码子T(ND2、COIII和ND4),其余基因均为标准的TAA。在所编码的3785个氨基酸中,使用最频繁的氨基酸为 Leu,占所有氨基酸的17.78%。
2.1.2 rRNA和tRNA基因
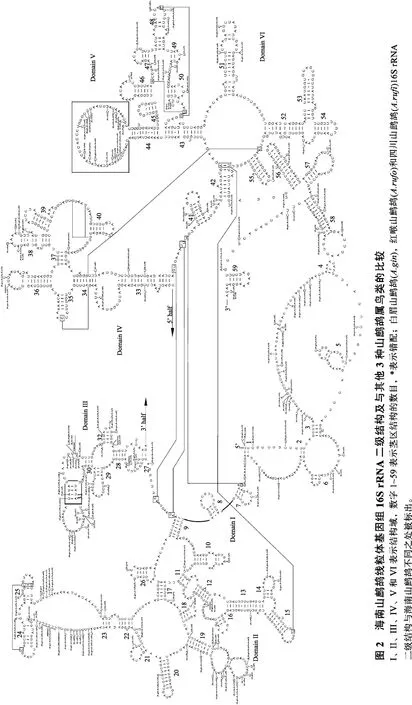
12S和 16S rRNA 基因分别位于 tRNAPhe和tRNAVal以及 tRNAVal和 tRNALeu(UUR)之间,基因长度分别为972 bp和1629 bp。预测的12S rRNA二级结构包含 3个结构域,46个茎环结构(图 1);16S rRNA二级结构含有6个结构域,59个茎环结构(图2)。22个tRNA基因的长度为66~77 bp,其中14个由J链编码,8个由N链编码。除tRNASer(AGY)(缺失DHU臂)之外,其余21种tRNA均可形成典型的三叶草二级结构。所有tRNA基因的二级结构中共存在31处碱基错配现象,其中G-U错配出现的频率最高。
2.1.3 控制区
控制区长度为1178bp,位于tRNAGlu和tRNAPhe之间,其中包含3个结构域:ETAS(extended termination-associated sequence)结构域I (nt 1~315),中央保守结构域II (nt 316~785)和CSB结构域III (nt 786~1178)[19~21](附图1)。ETAS结构域I包括类似于雪雁(Anas caerulescens)“goose hairpin”的序列(36-64)[22]、ETAS1(64~126)和 ETAS2(124~163)保守区。中央保守结构域II中含有F box、E box、D box、C box和Bird similarity box[23]保守序列。CSB结构域III包含类似哺乳动物重链复制起始位点的 poly(C)序列(OH,nt 786~797),且含有翻译的双向启动子(LSP和HSP,nt 998~1020)。
2.2 山鹧鸪属鸟类线粒体基因组比较
4种山鹧鸪属鸟类线粒体基因组的结构和组成类似。除四川山鹧鸪之外,其他 3种山鹧鸪属鸟类ND3基因第 174位置均存在胞嘧啶插入。利用mitogenome联合数据集计算的未校正P距离中,白眉山鹧鸪与红喉山鹧鸪之间的距离最小(0.04),四川山鹧鸪和海南山鹧鸪之间的距离最大(0.088)。
2.2.1 A+T含量和核苷酸偏向性

图1 海南山鹧鸪线粒体基因组12S rRNA二级结构及与其他3种山鹧鸪属鸟类的比较I、II和III表示结构域,数字1~46表示茎区结构的数目,*表示错配;白眉山鹧鸪(A.gin)、红喉山鹧鸪(A.rufo)和四川山鹧鸪(A.rufi)12S rRNA二级结构与海南山鹧鸪不同之处被标出。
对4种山鹧鸪鸟类线粒体全基因组、13个蛋白质编码基因、核糖体RNA基因、转运RNA基因及控制区等区域的碱基组成和核苷酸偏向性分析结果见附图 2。本研究结果表明,线粒体基因组不同区域的A+T含量、AT和GC偏向性在山鹧鸪属鸟类中是类似的。山鹧鸪属鸟类线粒体全基因组A+T含量略大于G+C含量,具有明显的GC偏向性(C含量远大于G)。线粒体基因组4种不同类型的划分中,A+T含量大致为:CR>tRNA>PCG>rRNA,其中,tRNA二级结构环区序列(tRNA-loop)表现出较高的 A+T含量;AT偏向性在蛋白质编码基因密码子第二位点(PCG-2nd,T含量远大于A)和第三位点(PCG-3rd,A含量远大于T)较为明显;蛋白质编码基因密码子第三位点(PCG-3rd)表现出明显的GC偏向性,C含量远大于G。
2.2.2 RNA二级结构比较
山鹧鸪属鸟类线粒体rRNA二级结构中多数茎区序列较为保守,而环区变化较大(图1和图2),例如16S rRNA二级结构茎区44上方环区中含有多处替换。山鹧鸪属鸟类 tRNA二级结构的茎区序列较为保守,保守位点(Conserved sites, C)比例高达96%,环区变化较大。tRNA茎区中存在大量G-U错配,这种配对方式仍然能够形成双螺旋结构,且在维系tRNA二级结构稳定性方面发挥着重要作用。tRNA二级结构中,tRNALeu(UCN)较为保守,而 tRNAPhe和tRNALys变化相对较大。
2.2.3 蛋白质编码基因密码子及氨基酸使用频率
山鹧鸪属鸟类中绝大多数蛋白质编码基因的起始和终止密码子较为保守。起始密码子一般为ATG,但COI基因以GTG为起始密码子。终止密码子共有4种类型(TAA、AGG、TAG和不完全终止密码子T),AGG仅存在于COI中;TAG仅出现在山鹧鸪属鸟类ND6基因及四川山鹧鸪Cytb基因中;ND2、COIII和ND4基因以不完全密码子T为终止;其他基因均以TAA为终止密码子。PCG数据集中,UCA、CUA和CGA密码子的使用频率较高;PCG数据集翻译成的氨基酸序列中,Leu使用频率最高,Cys最低。
2.3 同义替换率和非同义替换率
山鹧鸪属鸟类线粒体基因组蛋白质编码基因的Ks值明显高于Ka(附图3)。6个分组中,ATP8基因Ka值普遍较高,COI基因Ka值则较低。红喉山鹧鸪-白眉山鹧鸪中大多数蛋白质编码基因的Ka和Ks数值小于其他5个分组。山鹧鸪属鸟类Ka/Ks值均小于 1,这表明山鹧鸪属物种蛋白质编码基因受到了纯化选择的作用。ATP8基因的Ka/Ks数值较高,ND系列基因数值处于中等,CO系列、ATP6和Cytb基因的数值较低。此外,大多数基因的P值(来自于Fisher精确检验)均明显小于0.001,表明各组的差异达到了显著水平。本研究进一步对上述 6个分组的PCG数据集分别统计序列的转换(Ts)和颠换(Tv),结果表明:碱基转换数目远高于颠换数目;PCG-3rd中转换与颠换的数目明显大于PCG-1st和PCG-2nd;白眉山鹧鸪-红喉山鹧鸪中 PCG、PCG-1st和PCG-3rd的Ts/Tv数值均高于其他5个组。
2.4 山鹧鸪属系统发育关系
本研究基于线粒体基因组联合数据集构建的ML和BI树具有一致的拓扑结构(图3)。白眉山鹧鸪与红喉山鹧鸪互为姐妹群,而后再与四川山鹧鸪聚在一起,海南山鹧鸪则位于山鹧鸪属的基部位置;山鹧鸪属的单系性得到了良好地支持,且山鹧鸪属位于所有雉科鸟类的基部。
3 讨 论
3.1 线粒体基因组特征
山鹧鸪属鸟类线粒体基因组中多个蛋白质编码基因使用不完全终止密码子 T,这类密码子在脊椎动物中较为常见[24]。通常情况下,不完整的终止密码子与相邻的tRNA之间存在重叠序列,该终止密码子能够利用转录后加工过程将其补充为完整的TAA,有些学者认为 tRNA在此过程中起主要作用[25,26]。除四川山鹧鸪外,其他3种山鹧鸪属物种ND3基因174位置存在胞嘧啶的插入现象,在转录翻译过程中,这个额外插入的碱基被RNA通过自我修复剪切掉,从而有效地避免了因移码突变导致基因转录提前终止[27]。四川山鹧鸪 ND3基因中不存在胞嘧啶插入,这可能是鸟类快速辐射进化在线粒体基因组中的遗留物,也可能是自然选择的结果[28]。tRNA-loop的A+T含量较高,而PCG-1st和tRNA-stem的A+T含量较低,线粒体基因组不同分区的AT趋势可能是转录耦合的修复和脱氨基作用的结果[29]。山鹧鸪属鸟类rRNA二级结构环区变化较大,茎区较为保守[30,31],这可能是因为两者所受的选择压力不同。tRNASer(AGY)缺少DHU臂,这种现象在脊椎动物中较常见[32],而缺失DHU臂后的tRNASer(AGY)仍可形成倒L型三级结构来维持CCA接受臂与反密码子间的距离[33]。
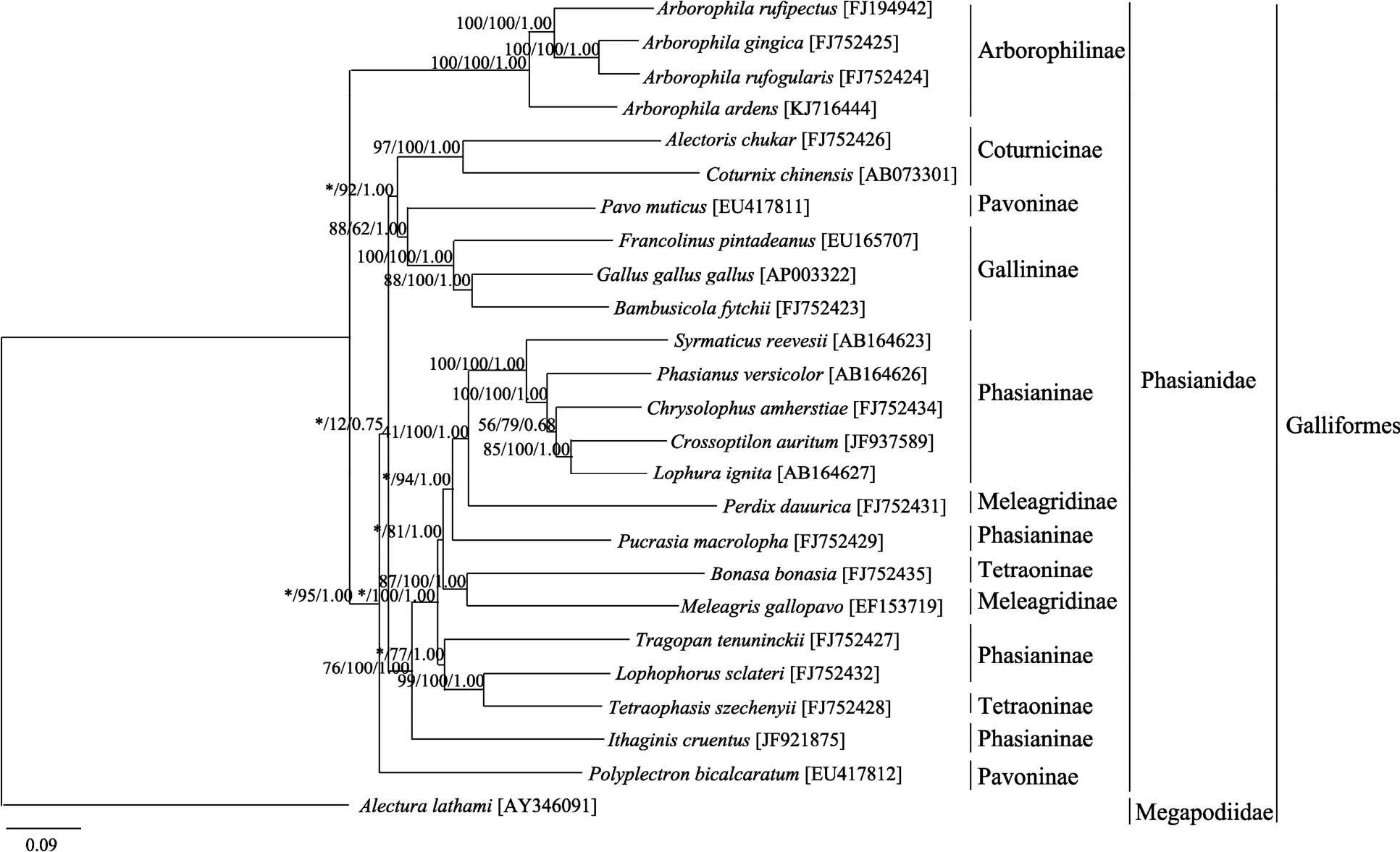
图3 mitogenome数据集构建的系统发育树分支支持度依次为MP/ML/BI,其中*表示MP建树结果与ML/BI不同。
3.2 同义替换率和非同义替换率
山鹧鸪属鸟类蛋白质编码基因处于纯化选择作用下,这种现象普遍存在于其他物种中,例如蝽[34]、长棘海星属[35]、真鲨目和鳐形目[36]、翼手目[37]、绒螯蟹属和青蟹属[38]、庸鲽属和星鲽属及舌鳎属[39]、桡足动物[40]。山鹧鸪属鸟类线粒体基因组蛋白质编码基因中,ATP8基因Ka/Ks值较大,COI基因Ka/Ks值较小,这与其他学者的研究类似[34,35,37,38,40~42],表明ATP8和COI基因分别经历了较弱和较强的选择压力。此外,Li等[34]的研究表明,蛋白质编码基因的Ka/Ks值与其GC含量之间存在负相关;且GC含量的变化可能会导致基因间出现不同的进化模式[34,43],但在本研究中,蛋白质编码基因的 Ka/Ks值与其相应的GC含量之间未观察到明显的负相关性。
飞行能力是鸟类的重要特征,飞行过程中所需的绝大部分能量都是通过线粒体中的氧化磷酸化过程产生的,飞行速度快的鸟类应该比飞行速度慢或不能飞行的鸟类具有更高效的能量代谢。对于善于飞行的鸟类而言,非同义突变大部分是有害的,一旦发生非同义替换,就会影响氧化磷酸化过程中能量的产生,也就不能满足飞行的需求,所以这种有害的非同义突变就会被纯化选择消除。本研究中,山鹧鸪属鸟类蛋白质编码基因Ka/Ks平均值为0.0513,与Shen等[44]研究中不善飞行组鸟类的Ka/Ks值(平均 0.05)接近,且高于其研究中善于飞行组鸟类的Ka/Ks值(平均0.04),这表明不善飞行的山鹧鸪属物种在进化历史中积累了更多的非同义替换。
3.3 山鹧鸪属系统发育关系
4种山鹧鸪属物种具有共同的生物学特征,例如上体呈橄榄褐色,繁殖期在4~6月份。相比而言,红喉山鹧鸪和白眉山鹧鸪的形态学特征更为相似,例如,眼周裸出的皮肤为红色,嘴黑色,腿、脚红色,而四川山鹧鸪的腿、脚赭褐色,海南山鹧鸪的嘴灰色,脚深粉红色,这与上述红喉山鹧鸪和白眉山鹧鸪具有较明显的差别。本研究的系统发育结果表明:白眉山鹧鸪与红喉山鹧鸪互为姐妹群,显示出较近的亲缘关系,这与上述形态学特征及未校正P距离结果是一致的;海南山鹧鸪位于山鹧鸪属的基部位置,与其他3种山鹧鸪属鸟类的亲缘关系较远,与 Wang等[45]的研究结果一致。山鹧鸪属位于雉科的基部位置,与其他雉科鸟类形成姐妹群关系,这种系统发育关系与Crowe等[46]、李喜凤[47]的研究结果类似。
附录:附表及附图见WWW.Chinagene.cn。
[1] 郑光美. 中国鸟类分类与分布名录. 北京: 科学出版社, 2005.
[2] 高育仁. 海南山鹧鸪. 见卢汰春主编: 中国濒危野生鸡类. 福州: 福建科学技术出版社, 1991: 149-153.
[3] 李湘涛. 中国鸡形目鸟类种与亚种的分布和生存现状(英文). 动物学报, 1996, 42(增刊): 25-30.
[4] 汪松, 郑光美, 王歧山. 中国濒危动物红皮书: 鸟类.北京: 科学出版社, 1998.
[5] IUCN 2013. The IUCN Red List of Threatened Species. Version 2013. 2. <http://www.iucnredlist.org>
[6] 梁伟, 史海涛, 王力军. 对海南省设立省鸟的探讨. 海南师范学院学报(自然科学版), 2002, 15(2): 67-70.
[7] 高育仁. 海南山鹧鸪的换羽. 中国动物科学研究——中国动物学会第十四届会员代表大会及中国动物学会 65周年年会论文集, 1999: 498-501.
[8] 韦锋. 海南山鹧鸪的活动区和夜栖地利用[学位论文].海南师范大学, 2008.
[9] 杨灿朝. 海南鹦哥岭自然保护区海南山鹧鸪(Arborophila ardens)的领域、活动区和生境利用[学位论文]. 海南师范大学, 2007.
[10] 党江鹏, 刘念, 叶伟, 黄原. 云斑车蝗线粒体基因组全序列测定与分析. 昆虫学报, 2008, 51(7): 671-680.
[11] Sorenson MD. Avian mtDNA primers. 2003. <http:// people.bu.edu/msoren/ Primers.html>
[12] Lowe TM, Eddy SR. tRNAscan-SE: A program for improved detection of transfer RNA genes in genomic sequence. Nucleic Acids Res, 1997, 25(5): 955-964.
[13] 杨超, 雷富民, 黄原. 地山雀线粒体基因组全序列的测定与分析. 动物学研究, 2010, 31(4): 333-344.
[14] 柯杨, 黄原, 雷富民. 黑尾地鸦线粒体基因组序列测定与分析. 遗传, 2010, 32(9): 951-960.
[15] Burk A, Douzery EJP, Springer MS. The secondary structure of mammalian mitochondrial 16S rRNA molecules: refinements based on a comparative phylogenetic approach. J Mam Evol, 2002, 9(3): 225-252.
[16] Kumar S, Tamura K, Nei M. MEGA: molecular evolutionary genetics analysis software for microcomputers. Comput Appl Biosci, 1994, 10(2): 189-191.
[17] Zhang Z, Li J, Zhao XQ, Wang J, Wong GKS, Yu J. KaKs-Calculator: calculating Ka and Ks through model selection and model averaging. Genomics Proteomics Bioinformatics, 2006, 4(4): 259-263.
[18] Mindell DP, Sorenson MD, Dimcheff DE. Multiple independent origins of mitochondrial gene order in birds. Proc Natl Acad Sci USA, 1998, 95(18): 10693-10697.
[19] Brown GG, Gadaleta G, Pepe G, Saccone C, Sbisà E. Structural conservation and variation in the D-loop-containing region of vertebrate mitochondrial DNA. J Mol Biol, 1986, 192(3): 503-511.
[20] Randi E, Lucchini V. Organization and evolution of the mitochondrial DNA control region in the avian genus Alectoris. J Mol Evol, 1998, 47(4): 449-462.
[21] Saccone C, Pesole G, Sbisá E. The main regulatory region of mammalian mitochondrial DNA: structure-function model and evolutionary pattern. J Mol Evol, 1991, 33(1): 83-91.
[22] Quinn TW. The genetic legacy of Mother Goose-phylogeographic patterns of lesser snow goose Chen caerulescens caerulescens maternal lineages. Mol Ecol, 1992, 1(2): 105-117.
[23] Ruokonen M, Kvist L. Structure and evolution of the avian mitochondrial control region. Mol Phyl Evol, 2002, 23(3): 422-432.
[24] Cui ZX, Liu Y, Li CP, You F, Chu KH. The complete mitochondrial genome of the large yellow croaker, Larimichthys crocea (Perciformes, Sciaenidae): unusual features of its control region and the phylogenetic position of the Sciaenidae. Gene, 2009, 432(1-2): 33-43.
[25] Quinn TW. Molecular evolution of the mitochondrial genome. In: Mindell DP. Avian Molecular Evolution and Systematics. San Diego: Academic Press, 1997: 3-28.
[26] Ojala D, Montoya J, Attardi G. tRNA punctuation model of RNA processing in human mitochondria. Nature, 1981, 290(5806): 470-474.
[27] Mindell DP, Sorenson MD, Dimcheff DE. An extra nucleotide is not translated in mitochondrial ND3 of some birds and turtles. Mol Biol Evol, 1998, 15(11): 1568-1571.
[28] 高英凯, 苗永旺, 苏小茜, 池振奋, 俞贇, 姜枫. 74 种鸟类线粒体基因组碱基组成及特征分析. 云南农业大学学报, 2009, 24(1): 51-58.
[29] Francino MP, Ochman H. Strand asymmetries in DNA evolution. Trends Genet, 1997, 13(6): 240-245.
[30] Woese CR, Magrum LJ, Gupta R, Siegel RB, Stahl DA,Kop J, Crawford N, Brosius J, Gutell R, Hogan JJ, Noller HF. Secondary structure model for bacterial 16S ribosomal RNA: phylogenetic, enzymatic and chemical evidence. Nucleic Acids Res, 1980, 8(10): 2275-2293.
[31] Noller HF. Structure of ribosomal RNA. Annu Rev Biochem, 1984, 53(1): 119-162.
[32] Härlid A, Janke A, Arnason U. The mtDNA sequence of the ostrich and the divergence between paleognathous and neognathous birds. Mol Biol Evol, 1997, 14(7): 754-761.
[33] Hanada T, Suzuki T, Watanabe K. Translation activity of mitochondrial tRNA with unusual secondary structure. Nucl Acids Symp Ser, 2000, 44(1): 249-250.
[34] Li H, Liu H, Shi AM, Štys P, Zhou XG, Cai WZ. The Complete Mitochondrial Genome and Novel Gene Arrangement of the Unique-Headed Bug Stenopirates sp. (Hemiptera: Enicocephalidae). PLoS ONE, 2012, 7(1): e29419.
[35] 田美, 申欣, 孟学平, 程汉良. 7个海星动物线粒体基因组比较及基因变异位点分析. 台湾海峡, 2012, 31(2): 189-194.
[36] 申欣, 田美, 孟学平, 程汉良. 10种软骨鱼线粒体基因组特征分析. 渔业科学进展, 2011, 32(3): 26-32.
[37] Meganathan PR, Pagan HJT, McCulloch ES, Stevens RD, Ray DA. Complete mitochondrial genome sequences of three bats species and whole genome mitochondrial analyses reveal patterns of codon bias and lend support to a basal split in Chiroptera. Gene, 2012, 492(1): 121-129.
[38] 田美, 申欣, 孟学平, 程汉良. 短尾派线粒体基因组特征及基因差异位点分析. 水产科学, 2011, 30(1): 31-37.
[39] 申欣, 田美, 孟学平, 程汉良, 许建和, 阎斌伦. 鲆鲽类线粒体基因排列、特征比较及系统发生分析. 水产科学, 2013, 32(10): 590-596.
[40] Wang MX, Sun S, Li CL, Shen X. Distinctive mitochondrial genome of Calanoid copepod Calanus sinicus with multiple large non-coding regions and reshuffled gene order: Useful molecular markers for phylogenetic and population studies. BMC Genomics, 2011, 12(1): 73.
[41] Helfenbein KG, Fourcade HM, Vanjani RG, Boore JL. The mitochondrial genome of Paraspadella gotoi is highly reduced and reveals that chaetognaths are a sister group to protostomes. Proc Natl Acad Sci USA, 2004, 101(29): 10639-10643.
[42] Papillon D, Perez Y, Caubit X, Le Parco Y. Identification of chaetognaths as protostomes is supported by the analysis of their mitochondrial genome. Mol Biol Evol, 2004, 21(11): 2122-2129.
[43] Hua JM, Li M, Dong PZ, Cui Y, Xie Q, Bu WJ. Comparative and phylogenomic studies on the mitochondrial genomes of Pentatomomorpha (Insecta: Hemiptera: Heteroptera). BMC Genomics, 2008, 9(1): 610.
[44] Shen YY, Shi P, Sun YB, Zhang YP. Relaxation of selective constraints on avian mitochondrial DNA following the degeneration of flight ability. Genome Res, 2009, 19(10): 1760-1765.
[45] Wang N, Kimball RT, Braun EL, Liang B, Zhang ZW. Assessing phylogenetic relationships among galliformes: a multigene phylogeny with expanded taxon sampling in phasianidae. PLoS ONE, 2013, 8(5): e64312.
[46] Crowe TM, Bowie RCK, Bloomer P, Mandiwana TG, Hedderson TAJ, Randi E, Pereira SL, Wakeling J. Phylogenetics, biogeography and classifcation of, and character evolution in, gamebirds (Aves: Galliformes): effects of character exclusion, data partitioning and missing data. Cladistics, 2006, 22(6): 495-532.
[47] 李喜凤. 鸡形目、鹤形目和雀形目七种鸟类线粒体全基因组的测定与分析[学位论文]. 安徽师范大学, 2012.
(责任编委: 李 辉)
Comparative and phylogenomic analyses on mitochondrial genomes of Arborophila species
Xuejuan Li1, Yuan Huang1, Fumin Lei2
1. School of Life Sciences, Shaanxi Normal University, Xi’an 710062, China;
2. Key Laboratory of Zoological Systematics and Evolution, Institute of Zoology, Chinese Academy of Sciences, Beijing 100101, China
Arborophila ardens, an endangered species, has a strict habitat preference and sparse population. In order to further study evolution and phylogenetic relationship of Arborophila, the mitochondrial genome (mitogenome) of A. ardens was obtained by Illumina Hiseq2000 high-throughput sequencing. The comparative genomic analyses were made among four Arborophila species, and phylogenetic relationship of Arborophila was discussed. The results revealed that: (1) the complete mitogenome of A. ardens is 16 730 bp in length, including 13 protein-coding genes (PCGs), two ribosomal RNA genes (rRNAs), 22 transfer RNA genes (tRNAs) and a control region (CR); (2) Arborophila species were affected by purifying selection, and more nonsynonymous substitutions were accumulated in the process of evolution; (3) Arborophila was at the basal position of Phasianidae within the phylogenetic tree, and A. gingica was placed as sister to A. rufogularis, while A. ardens formed the basal position of Arborophila, which indicated a distant genetic relationship with other three species.
Arborophila ardens; mitochondrial genome; comparative genomics; phylogeny

附表1 海南山鹧鸪线粒体基因组PCR扩增引物
2014-03-14;
2014-07-25
国家杰出青年科学基金(编号:30925008)和中国科学院动物进化与系统学重点实验室开放课题(编号:O529YX5105)
李雪娟,博士研究生,研究方向:分子进化生物学。E-mail: lixuejuan456@163.com
黄原,教授,博士生导师,研究方向:分子进化与分子系统学。E-mail: yuanh@snnu.edu.cn
雷富民,研究员,博士生导师,研究方向:鸟类学。E-mail: leifm@ioz.ac.cn
10.3724/SP.J.1005.2014.0912
时间: 2014-8-12 14:16:59
URL: http://www.cnki.net/kcms/detail/11.1913.R.20140812.1416.001.html
