莱茵衣藻线粒体遗传系统及其外源基因表达
2013-11-27胡章立李建成程雪薇
胡章立,李建成,程雪薇
深圳市海洋生物资源与生态环境重点实验室,深圳市海洋藻类开发应用工程实验室,深圳大学生命科学学院,深圳518060
莱茵衣藻(Chlamydomonas reinhardtii)是一种单细胞光合真核藻类,属于绿藻门团藻目衣藻科.它与酵母有许多共同之处,如生长速度快且周期短;既可在固体平板培养基上形成单克隆,也可进行液体培养;以单倍体与二倍体两种形式生长等.藻类是分子生物学、膜生物学和功能基因组学等研究领域的模式生物,其中最为典型的是莱因衣藻.作为单细胞真核绿藻,莱因衣藻具有生长周期短,生长迅速快,光合效率高等特点.它已成为藻类基因组计划研究中的模式植物.因此,莱茵衣藻有“光合酵母”之称[1].此外,莱茵衣藻在光照下能进行光合自养,而在黑暗和醋酸盐条件下能进行异养生长;有两根鞭毛,可在水中自由游动;有眼点,具有感光能力等特征.20世纪50年代以来,莱茵衣藻作为模式生物在分子遗传、鞭毛运动、光合作用、叶绿体起源、线粒体功能、趋光性和生理节律等领域扮演重要角色[2-5].
莱茵衣藻具有细胞核基因组、叶绿体基因组和线粒体基因组3套遗传系统.核基因组和叶绿体基因组可方便有效地进行遗传转化,其表达调控机理也得到较为深入的研究.由于其具有培养条件简单、生长周期短、光合效率高和遗传背景清楚等特点,莱茵衣藻被认为是具有广泛应用前景的转基因生物反应器,至今已有多种外源基因在莱茵衣藻细胞核和叶绿体中获得成功表达[6-9].近年来,基因枪轰击技术的问世,大幅提高了外源基因的遗传转化效率,该技术通过自身携带的动力系统将带有DNA的金属颗粒(一般为钨粒或金粒),以一定速度带入受体细胞内,由于金属颗粒穿透力强,所以不需对受体细胞进行破壁处理即可进入基因组,从而实现高效转化的目的.基因枪轰击技术具有应用面广,方法简单,转化周期短等优点.然而,自Randolph-Anderson等[10]利用基因枪将野生莱茵衣藻完整的线粒体DNA转化到莱茵衣藻呼吸作用缺陷型突变株dum-1,获得具有正常呼吸特性的转基因藻株以来,有关莱茵衣藻线粒体基因组遗传转化的研究很多,但成功进行线粒体遗传转化的报道很少[11-13],而线粒体遗传转化是研究线粒体基因功能、基因表达调控、线粒体与核质间相互作用等生物学过程的重要途径.本课题组Hu等[13]首次成功利用光合生物——莱茵衣藻线粒体遗传系统表达了外源egfp基因,随后又利用该系统成功表达了来自细菌的ble基因[14].本文结合该课题组研究结果,评述莱茵衣藻线粒体遗传转化系统及其外源基因表达的研究进展.
1 莱茵衣藻线粒体基因组的结构
Ryan等[15]从莱茵衣藻线粒体中分离纯化出线粒体DNA.它在氯化铯溶液中的浮力密度为1.706 g/mL,在标准柠檬酸盐溶液中的溶解温度为87.9℃,动力学复杂度为9.78×106Da.电子显微镜观察结果显示,线性分子占99%,开环或闭环分子少于1%.线状或环状的分子长度均在4.0~5.4 μm.每个细胞内线粒体基因的拷贝数约为50个,占细胞内总DNA含量的0.6%.
莱茵衣藻线粒体基因组的测序已经在1993年完成(Genbank序列号:U03843)[16].测序结果显示,该线性 DNA分子全长15 758碱基对(base pair,bp),由13个基因组成(GC含量45%),分别编码细胞色素氧化酶c亚基Ⅰ(复合体Ⅵ)、细胞色素b(复合体Ⅲ)及还原型辅酶(nicotinamide adenine dinucleotide,NADH);辅酶Q还原酶的5个亚基(复合体Ⅰ),一个反转录蛋白,3个tRNA(trnW、trnQ和trnM)及组成大小核糖体亚基的rRNA.其中,rRNA基因的组成是不连续的,它被间隔成4个rrnS分子和8个rrnL分子而分散于线粒体基因组中.有研究表明,正常的rRNA是由这些小RNA分子通过广泛的分子间相互作用而形成的[17].在莱茵衣藻线粒体DNA中也存在非编码序列,但其非编码序列很短且不含内含子.Vahrenholz等[16,18]的研究发现,莱茵衣藻线粒体基因组的末端有长度为531 bp和532 bp的末端反向重复序列,还发现左右两边的3'端各有一段向外延伸的相同序列(长度在39~41个核苷酸之间).末端重复序列每边最外端的86个核苷酸都与中间的一段86个核苷酸的序列相同,这段序列位于L2b基因的3'端,距离基因组的右端674 bp[19],详见图1.
2 莱茵衣藻线粒体遗传转化系统
2.1 莱茵衣藻线粒体DNA的复制、转录与翻译
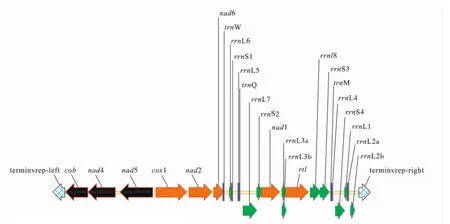
图1 莱茵衣藻线粒体基因组的基因分布[19]Fig.1 Gene organization of C.reinhardtii mitochondrial genome[19]
莱茵衣藻线粒体是具有半自主特性的细胞器,能单独进行DNA复制、转录和蛋白质合成.与细胞核DNA复制一样,线粒体DNA是以半保留方式进行复制,但两条链的复制不同步,这与细胞核DNA准确地在每个细胞分裂期只复制1次不同.Grant等[20]研究指出,莱茵衣藻线粒体基因组上两个长而非互补的单链末端避免了mtDNA自连成环状,而且mtDNA的5'末端可被T4多核苷酸激酶有效标记,这也排除了复制相关蛋白与这些末端通过共价结合而进行复制的可能性.Vahrenholz等[16]也提出了两种莱茵衣藻线粒体DNA复制模型,这两种复制模型都以线粒体基因组中86 bp的内部重复序列的存在为前提.
文献[21]研究发现,在莱茵衣藻线粒体基因组中,13个功能基因分两组排列在线性DNA分子的两侧,cob、nad4和nad5基因位于左边,其转录方向是从 3'到5'端;cox1、nad2、nad6、nad1、rtl、rRNA及tRNA基因位于线粒体DNA右边,其转录方向是从5'到3'端进行.在nad5和cox1基因之间的间隔区有一小段序列起双向启动子的作用,同一转录方向的基因组成一个操纵子,由公共双向启动子共同启动它们的转录.但也有持相反观点,因为对转录图谱的研究发现,有低水平的nad5基因前体RNA的5'端与cox1基因的起始位点相重叠[20].在进行线粒体基因转录时,包括rRNA在内的许多基因转录子的3'末端紧接着另一个转录子的5'末端.在3'末端非翻译区的反向重复序列中包含能折叠成发夹结构的序列,这些序列可能含有转录终止、RNA剪切加工和RNA稳定等信息[22].
莱茵衣藻线粒体与核基因组一样利用通用密码子编码基因,但对密码子具有很强的偏爱性[23].在莱茵衣藻线粒体基因表达过程中,有9种密码子根本未用到,有4种密码子也仅在rtl基因中出现[19].尽管在莱茵衣藻线粒体中缺失某些密码子,但翻译线粒体基因组信息至少需要23个tRNA[17].然而,在莱茵衣藻mtDNA中却只发现3个线粒体的tRNA基因,这说明线粒体内大部分的tRNA很可能是从线粒体外运输进来的[24].
2.2 莱茵衣藻线粒体遗传转化方法
相对细胞核转化,将外源基因从细胞外导入线粒体是非常困难的,需要通过细胞膜和线粒体膜系统.Johnson等[25]利用基因枪法成功地转化了酵母(Saccharomyces cerevisiae)线粒体,将野生株的oxi3导入呼吸缺陷株(947/PA5)整合到线粒体基因组的同源位点,获得具有细胞色素氧化酶活性的转化株.Randolph-Anderson等[10]将基因枪转化技术应用到莱茵衣藻线粒体遗传转化中,成功地将野生株CC-125中包含CYB基因的线粒体DNA左侧片段导入呼吸缺陷突变株CC-2654中,获得具有健全呼吸代谢功能的转基因藻.目前莱茵衣藻线粒体遗传转化方法均采用基因枪法,其转化效率由最初的每微克 DNA 获得 7.3×10-7[10]个转化子提高到现在的100~250个转化子[12].以下介绍本课题组开展莱茵衣藻线粒体遗传转化的实验操作步骤、技术参数和转化效率等情况.
2.2.1 受体藻细胞制备
以莱茵衣藻作为遗传转化受体,采用“基因枪法”对外源基因进行高效表达,必须要保证整个操作过程严格无菌.首先使用去离子水配置新鲜TAP培养基,高温高压灭菌20 min、自然冷却;然后将受体莱茵衣藻接种于TAP液体培养基中,培养至对数生长期(约5×106~6×106mL-1);取25 mL上述藻液于50 mL离心管中离心收集藻细胞(6 000 r/min,20℃,1 min),弃上清后加入约100 μL新鲜TAP液体培养基重悬藻细胞;将藻细胞悬液均匀地涂布于TAP固体平板中间(呈圆形,直径约3 cm),22℃,2 000 Lx下持续光培养1~2 d.
2.2.2 金粉准备与DNA包被
称取30 mg金粉(直径 0.6 μm)置于 1.5 mL离心管中,加入1 mL体积分数为70%的乙醇剧烈振荡3~5 min,浸泡15 min,离心去上清;加入1 mL无菌水涡旋且离心清洗3次,加500 μL体积分数为50%的灭菌甘油,使金粉终质量浓度达60 mg/mL;取50 μL充分混匀的金粉液于1.5 mL离心管中,并依次加入 5 μg 目的DNA(1 μg/μL)、50 μL 2.5 mol/L 的CaCl2和20 μL 0.1 mol/L 亚精胺(现配现用),涡旋2~3 min,静置1 min,短暂离心2 s,小心吸去液体;缓缓地加入140 mL体积分数为70%的乙醇漂洗沉淀,轻轻吸去液体;再缓缓地加入140 mL无水乙醇漂洗沉淀,轻轻吸去液体;最后加入48 mL无水乙醇,轻叩管壁重悬沉淀,低速涡旋2~3 s(使用时每枪涂布10 μL于微弹载体中心).
2.2.3 基因枪主要参数与转化效率
以Biolistic PDS-1000/He Particle Delivery system(Bio-Rad)为例,基于文献[10-12]报道的莱茵衣藻线粒体的转化参数,根据不同的轰击距离、可裂膜规格组合成4组基因枪转化参数(表1).

表1 基因枪转化的主要参数组合*Table1 Summary of the major parameter combinations of microprojectile bombardment
比较4组基因枪转化结果(表2)发现,组合④的转化效率最高,每微克DNA能得到约600个转化株,比Remacle等[12]转化效率高2~3倍.
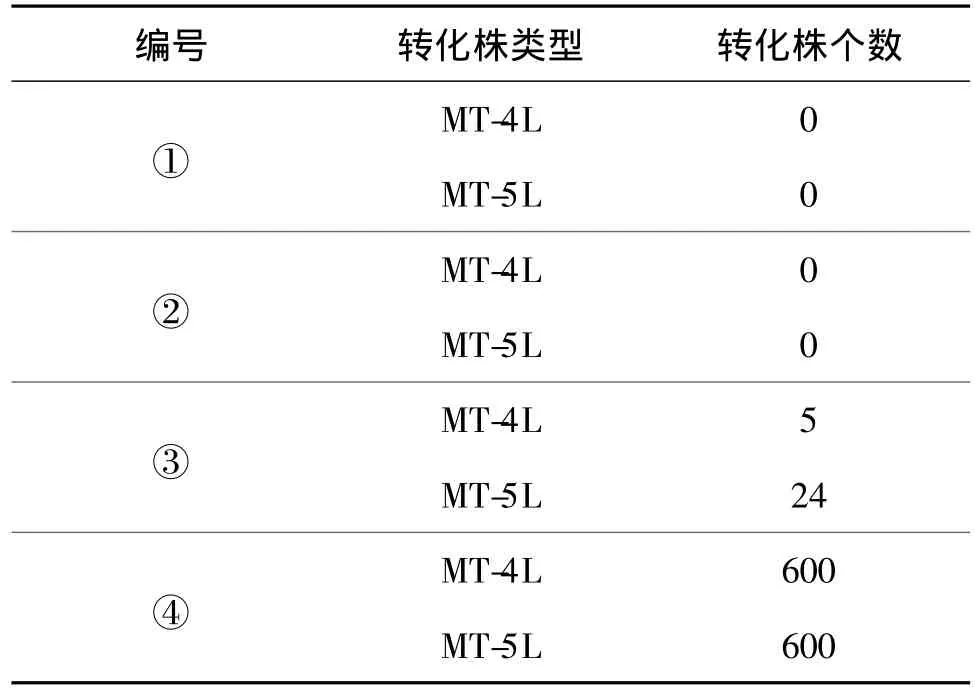
表2 不同参数组合对转化株获得个数的影响Table2 Numbers of transformants obtained by different parameter combinations
2.3 莱茵衣藻线粒体遗传转化的筛选标记基因
利用线粒体基因缺陷突变株进行莱茵衣藻线粒体基因组遗传转化,是目前最主要的遗传转化筛选途径.早期利用化学诱变的方法(吖啶黄和溴已啶染料等),筛选到一系列呼吸缺陷型突变株[26-27].在异养条件下,突变株细胞不能分裂,其生长停滞;在兼养条件下(光照+醋酸盐),突变株细胞的总呼吸仅为野生型的20%~50%,其生长速率下降;在光合自养条件下(光照但无醋酸盐),突变株的生长与野生型的总呼吸率非常接近,生长速率几乎无差别.进一步分子生物学分析发现,这些突变株的基因突变位点来自线粒体,根据基因缺失或突变特性分为24种突变株,其中7种突变株(dum-1、dum-2、dum-3、dum-4、dum-11、dum-14和dum-16)是由于莱茵衣藻线粒体DNA左端不同长度片段缺失,导致cob基因部分或全部缺失,使Complex III活性丧失[27].目前应用于线粒体遗传转化的突变株是莱茵衣藻呼吸缺陷型dum-1 mt-CC-2654,由于其线粒体DNA左端缺失了1.5 kb片段(包括terminvrep-left和cob)[26],在黑暗条件下,它不能利用醋酸钠作为碳源进行异养生长,Randolph-Anderson等[10]将野生型莱茵衣藻和史密斯(Chlamydomonas smithii)mtDNA转入呼吸缺陷型dum-1 mt-CC-2654,在黑暗条件下,分别得到了修复缺失突变的转化子.随后报道的莱茵衣藻线粒体转化研究均是利用导致藻细胞呼吸缺陷的cob基因作为筛选标记[11-13].
Mireau等[28]利用 barstar基因作为筛选标记,成功地进行了酵母(Saccharomyces cerevisiae)线粒体基因组的遗传转化.由于芽孢杆菌RNA酶能够干扰线粒体基因的表达.当通过带有barnase基因的质粒在酵母细胞中表达芽孢杆菌RNA酶时,导入到线粒体内的芽孢杆菌RNA酶能够抑制细胞的呼吸代谢,使酵母细胞在培养基上无法生长.barstar是芽孢杆菌RNA酶的抑制剂,将barstar基因整合到mtDNA,在酵母培养基上得到了转化子.这说明barstar能够作为酵母线粒体转化的筛选标记.这一筛选标记不需要使用呼吸缺陷突变株,为多细胞生物线粒体转化提供了可能的筛选途径,但芽孢杆菌RNA酶作为一种细胞毒素,如何控制其表达量以免对受体生物产生影响是需要解决的问题,这也大大限制了barstar作为线粒体转化筛选标记的应用.
Remacle等[12]曾尝试利用基因枪将两个质粒进行共转化,其中一个质粒携带核选择标记,另一个带有线粒体目的基因.通过核转化筛选标记在光照下进行转化子筛选后,再转到黑暗中进行线粒体转化子筛选,但这一途径未得到预期效果.
本课题组Hu等[4]成功地将egfp基因整合到莱茵衣藻线粒体基因组terminvrep-left和cob基因之间,转基因藻在实验室保持培养两年后,在激光共聚焦显微镜下能够检测到藻细胞线粒体中稳定表达的绿色荧光蛋白.尽管egfp基因的表达量还需要提高,但该研究结果为莱茵衣藻线粒体基因组的遗传转化提供了一种新的筛选标记.随后Hu等[14]成功地将ble基因整合到莱茵衣藻线粒体基因组中,使转基因藻细胞具有Phleomycin的抗性,这使线粒体外源基因转化的筛选问题变得容易,完全可以脱离之前依赖呼吸突变株进行遗传转化筛选的局面.
3 外源基因在莱茵衣藻线粒体中的表达
外源基因遗传转化是在活体内评估体外进行的DNA改造结果和分析基因组内由于新基因出现造成的显型改变的唯一方法.利用线粒体遗传转化系统进行外源基因表达具有重要理论意义和应用价值:一方面是研究线粒体基因表达调控、线粒体与核质间相互作用等生物学过程的重要途径[11,29];另一方面是进行线粒体基因工程操作的前提条件.如许多人类疾病与线粒体DNA突变有关[30],基因治疗是根治线粒体疾病的有效措施.植物细胞质雄性不育(cytoplasmic male sterility,CMS)与mtDNA密切相关[31],针对CMS相关基因进行mtDNA转基因操作,大量培育出杂交育种所需要的细胞质雄性不育系、保持系和恢复系,将从根本上改变目前建立细胞质雄性不育时,繁琐地使用回交育种的方法[31].然而,迄今为止,只有酵母(Saccharomyces cerevisiae)线粒体遗传系统能够进行稳定的外源基因表达[28,33],在高等动物和植物线粒体遗传转化中,只有体外离体线粒体转化的报告[34-35].
莱茵衣藻是唯一可以进行细胞线粒体遗传转化的光合生物.然而,尽管莱茵衣藻核基因组和叶绿体基因组已可方便有效地进行外源基因稳定表达[6-9],发现了很多用于筛选转化株的标记基因[36],但线粒体异源功能基因表达研究进展缓慢.关于莱茵衣藻线粒体遗传转化的研究迄今甚为少见.Randolph-Anderson等[10]最早报道了莱茵衣藻线粒体遗传转化,他们用野生型莱茵衣藻和野生型史密斯衣藻的完整mtDNA,成功转化了呼吸缺陷型莱茵衣藻dum-1突变株,得到能够完全修复突变株呼吸缺陷的转基因藻.在线粒体转化过程中,DNA的整合方式是同源重组.与叶绿体[37](每个处理细胞获得1×10-4个转化株)或核[38](1×10-3)转化相比,线粒体的转化效率是很低的(0.8×10-7~12.5×10-7)[10].Yamasaki等[11]分别构建 4种莱茵衣藻mtDNA片段,包括5.0 kb(含左端反向重复序列、cob、nad4的C端67 bp序列、nad5的N端180 bp序列、双向启动子序列、cox1、rRNA基因、ori和右端重复序列)、3.8 kb(与5.0 kb片段相比,cox1基因N端缺失1.2 kb)、1.8 kb(含左端反向重复序列、cob和nad4的C端67 bp序列)和3.2 kb PCR产物(包含cob和nad4).将4种mtDNA片段分别转化5个莱茵衣藻呼吸缺陷突变株品系(dum-1、dum-14、dum-15、dum-16 和dum-19),结果表明,因mtDNA左端反向重复序列和cob部分或全部缺失导致的呼吸缺陷突变株(dum-1、dum-14和dum-16)均得到转化子(每平板约9~14),而在突变株dum-15(cob的140密码子TCT突变成TAC)和dum-19(在cox1的152密码子-1T)则没有通过同源重组得到转化子.进一步分析发现,莱茵衣藻mtDNA左端末端及反向重复序列缺失可能有利于同源重组的发生,从而提高线粒体遗传转化效率.
Remacle等[12]利用高效基因枪转化法进行莱茵衣藻线粒体基因插入突变株的构建.利用缺失1.2 kb片段(包括左端重复序列和cob基因)的呼吸缺陷突变株dum-11作为受体藻株,将包含左端重复序列和cob基因的不同长度线粒体DNA片段导入衣藻线粒体,得到了较高的转化效率(每微克DNA获得100~250个转化株).进一步分析发现,线性化的mtDNA有利于提高转化效率;mtDNA的端粒区能够提高线粒体遗传转化效率,但并不是必需的因素;当mtDNA的左侧端粒区缺失时,右侧端粒区序列能够通过拷贝来构建左侧端粒序列.另外,通过莱茵衣藻线粒体转化,有害突变和无害突变能够被引入,如通过cob基因的单核苷酸替换筛选到粘噻唑抗性的突变株,含缺失23密码子nad4基因突变株构建等,这些结果为进一步开展线粒体起源、定点突变及反向遗传学提供了参考.
Hu等[13-14]将编码绿色荧光蛋白的egfp和ble基因分别插入莱茵衣藻mtDNA的terminvrep-left和cob基因之间,构建包含terminvrep-left、ble、cob和nad4部分片段的表达载体pBsLPNCG及pBsLPNCB,并利用基因枪将其分别导入莱茵衣藻dum-1突变株CC-2654线粒体中,获得能够有效表达绿色荧光白的转基因藻和具有Phleomycin抗性的转基因藻.据检索了解,这是迄今国际上首次利用光合生物线粒体遗传系统成功表达外源功能基因的报道.
4 存在问题与展望
莱茵衣藻线粒体遗传系统的基因组结构已经非常清楚,但线粒体基因的表达调控及其与细胞核和叶绿体基因组的协同关系等问题,是下一步需要解决的重要问题.外源基因在莱茵衣藻线粒体遗传系统中的稳定表达是研究这些问题的基础.尽管近年已取得重要进展,但仍存在以下问题:
1)遗传转化株的异质性问题.由于莱茵衣藻细胞中线粒体基因组有30~50个拷贝,在转基因操作时并不是每个线粒体基因组中都导入了外源基因,这就出现同一藻细胞不同基因组拷贝中出现含有外源基因和没有整合外源基因的异质体转化株,随着转基因藻的亚克隆筛选,使异质体的基因组分离,最后得到全部包含外源基因的同质化转化株.在莱茵衣藻叶绿体遗传转化过程中,这种同质化过程非常快[38],但依赖呼吸缺陷突变株进行遗传转化,得到的转化子线粒体同质化过程则很缓慢,要经过2~3个月的黑暗培养后,才能检测到异质化转化子的亚克隆株[12].可见,寻找更有效的遗传转化株筛选方法,在短时间内获得全部包含外源基因的同质化转化株,将是莱茵衣藻线粒体遗传转化研究的重点.
2)遗传转化株的高效筛选途径.利用呼吸缺陷突变株作为转基因受体藻株,转化子线粒体基因组因为整合了突变株缺失的cob片段而能在黑暗中生长而得以筛选出来.这种转化子筛选途径在高等多细胞生物中不容易实现,因为呼吸缺陷后这些生物将不可能存活[28].至今,尚未找到高效抗生素抗性基因能够在线粒体中稳定表达,这大大制约了线粒体遗传转化系统的发展.
3)莱茵衣藻线粒体基因表达调控问题.虽然莱茵衣藻线粒体遗传系统表达外源基因已经获得成功,外源基因在转基因藻中的表达水平是比较低的[13],究其原因可能包括:① 在线粒体表达载体中,并没有额外的启动子和其他的增强序列,egfp只能利用mtDNA中的双向启动子跟随cob基因进行本底水平的转录表达;②莱茵衣藻线粒体具有很强的密码子偏爱性,虽然编码egfp基因的密码子大部分都符合莱茵衣藻线粒体的偏爱密码子,但也有少数几个密码子并不常有,这有可能影响egfp基因的表达水平;③ 缺少莱茵衣藻线粒体基因5'UTR和3'UTR也可能导致egfp基因缺少适当的表达调控,5'UTR和3'UTR被认为与基因转录后水平调控密切相关,可能具有翻译起始、维持mRNA的稳定性、mRNA的转录后修饰和保证翻译效率等功能;④在莱茵衣藻mtDNA基因中,有一段公共的序列(5'AAAUUUAU 3')被确定能与核糖体小亚基末端序列互补,这段序列在一般位于ATG起始密码子上游3~7 bp处,缺少这些非编码序列,有可能影响 ble的表达水平[40].
由于基因表达调控极为复杂,莱茵衣藻线粒体外源基因表达调控涉及包括整合位点、转录活性、核质互作和翻译转运等方面的调控,将是今后相当一段时间内需要解决的重点难题.
/References:
[1]Rochaix J D.Chlamydomonas reinhardtii as the photosynthetic yeast[J].Annual Review of Genetics,1995,29:209-230.
[2]Sager R.Mendelian and non-mendelian inheritance of streptomycin resistance in Chlamydomonas reinhardtii[J].Proceedings of the National Academy of Sciences,1954,40:356-363.
[3]Hippler M,Redding K,Rochaix J D.Chlamydomonas genetics,a tool for the studay of bioenergetic pathways[J].Biochimica et Biophysica Acta,1998,1367(1/2/3):1-62.
[4]Rochaix J D.Chlamydomonas,a model system for studying the assembly and dynamics of photosynthetic complexes [J].FEBS Letters,2002,529(1):34-38.
[5]Harris E.Chlamydomonas as a model organism [J].Physiology and Plant Molecular Biology,2001,52:363-406.
[6]Ishikura K,Takaoka Y,Kato K,et al.Expression of a foreign gene in Chlamydomonas reinhardtii chloroplast[J].Journal of Bioscience and Bioengineering,1999,87(3):307-314.
[7]Franklin S,Ngo B,Efuet E,et al.Development of a GFP reporter gene for Chlamydomonas reinhardtii chloroplast[J].The Plant Journal,2002,30(6):733-744.
[8]Wang Chaogang,Hu Zhangli,Hu Wei,et al.Expression and molecular analysis of phbB gene in Chlamydomonas reinhardtii[J].Chinese Bulletin of Science,2004,49(16):1713-1717.
[9]Wang Chaogang,Hu Zhangli.Biosynthesis of poly-3-hydroxybutyrate(PHB)in transgenic green algae Chlamydomonas reinhardtii[J].Journal of Phycology,2010,46(2):396-402.
[10]Randolph-Anderson B L,Boynton J E,Gillham N W,et al.Further characterization of the respiratory deficient dum-1 mutation of Chlamydomonas reinhardtii and its use as a recipient for mitochondrial transformation [J].Molecular and General Genetics,1993,236(2/3):235-244.
[11]Yamasaki T,Kurokawa S,Watanabe K I,et al.Shared molecular characteristics of successfully transformed mitochondrial genomes in Chlamydomonas reinhardtii[J].Plant Molecular Biology,2005,58(4):515-527.
[12]Remacle C,Cardol P,Coosemans N,et al.Highefficiency biolistic transformation of Chlamydomonas mitochondria can be used to insert mutations in complex I genes[J].Proceedings of the National Academy of Sciences of the United States of America,2006,103(12):4771-4776.
[13]Li Jiancheng,Peng Shiqing,Hu Zhangli.Expression and immunological activity of enterotoxin C2 from Staphylococcusaureus in Chlamydomonasreinhardtii[J].Journal of Shenzhen University Science and Engineering,2012,29(2):159-164.(in Chinese)李建成,彭世清,胡章立.SEC2在莱茵衣藻中表达及免疫学活性分析 [J].深圳大学学报理工版,2012,29(2):159-164.
[14]Hu Zhangli,Fan Zhun,Zhao Zhonglin,et al.Stable expression of antibiotic-resistant gene ble from streptoalloteichus hindustanus in themitochondria of Chlamydomonas reinhardtii[J].PLoS ONE,2012,7(4):e35542.
[15]Ryan R,Grant D,Chiang K S,et al.Isolation and charaterization of mitochondrial DNA from Chlamydomonas reinhardtii[J].Proceedings of the National Academy of Sciences,1978,75(7):3268-3272.
[16]Vahrenholz C,riemen G,Pratje E,et al.Mitochondria DNA of Chlamydomonas reinhardtii:the structure of the ends of the linear 15.8-kb genome suggests mechanisms for DNA replication [J].Current Genetics,1993,24(3):241-271.
[17]Boer P H,Gray M W.Scrambled ribosomal RNA gene pieces in Chlamydomonas reinhardtii mitochondrial DNA[J].Cell,1988,55(3):399-411.
[18]Ma D P,King Y T,Kim T Y,et al.Amplification and characterization of an inverted repeat from Chlamydomonas reinhardtii mitochondrial genome [J].Gene,1992,119(2):253-257.
[19]Rochaix J D,Goldschmidt-Clermont M,Merchant S.The Molecular Biology of Chloroplasts and Mitochondia in Chlamydomonas[M].Dordrecht:Kluwer Academic Publishers,1998:661-674.
[20]Grant D,Chiang K S.Physical mapping and characterization of Chlamydomonas mitochondrial DNA molecules:their unique ends,sequence homogeneity,and conservation [J].Plasmid,1980,4(1):82-96.
[21]Boer P H,Gray M W.Genes encoding a subunit of respiratory NADH dehydrogenase(ND1)and a reverse transcriptase-like protein(RTL)are linked to ribosomal RNA gene pieces in Chlamydomonas reinhardtii mitochondrial DNA [J].The EMBO Journal,1988,7(11):3501-3508.
[22]Harris E H,Stern D B,Witman G B.The Chlamydomonas Sourcebook[M].2nd ed.San Diego:Academic Preess,2009:445-461.
[23]Kück U,Neuhaus H.Universal genetic code evidenced in mitochondria of Chlamydomonas reinhardtii[J].Applied Microbiology and Biotechnology,1986,23:462-469.
[24]Schneider A.Import of RNA into mitochondria [J].Trends in Cell Biology,1994,4(8):282-286.
[25]Johnson S A,Anziano P Q,Shark K,et al.Mitochondrial tansformation in yeast by bombardment with microprojectiles [J].Science,1988,240(4858):1538-1541.
[26]Matagne R F,Michel-Wolwertz M R,Munaut C,et al.Induction and characterization of mitochondrial DNA mutants in Chlamydomonas reinhardtii[J].The Journal of Cell Biology,1989,108(4):1221-1226.
[27]Dorthu M P,Remy S,Michel-Wolwertz M R,et al.Biochemical genetic and molecular characterization of new respiratory-deficient mutants in Chlamydomonas reinhardtii[J].Plant Molecular Biology,1992,18(4):759-772.
[28]Mireau H,Arnal N,Fox T D.Expression of barstar as a selectable marker in yeast mitochondria[J].Molecular Genetics and Genomics,2003,270(1):1-8.
[29]Pfeuty A,Dufresne C,Gueride M,et al.Mitochondrial upstream promotersequences modulate in vivo the transcription of a gene in yeast mitochondria[J].Mitochondrion,2006,6(6):289-298.
[30]Helen A L T,Emma L B,Douglass M T,et al.Mitochondrial DNA mutations and human disease[J].Biochimica et Biophysica Acta,2010,1797(2):113-128.
[31]Levings C S,Pring D R.Restriction endonuclease analysis of mitochondrial DNA from normal and texas cytoplasmic male-sterile maize [J].Science,1976,193(4248):158-160.
[32]Sandhu A P,Abdelnoor R V,Mackenzie S A.Transgenic induction of mitochondrial rearrangements for cytoplasmic male sterility in crop plants[J].Proceedings of the National Academy of Sciences,2007,104(6):1766-1770.
[33]Cohen J S,Fox T D.Expression of green fluorescent protein from a recoded gene inserted into Saccharomyces cerevisiae mitochondrial DNA [J].Mitochondrion,2001,1(2):181-189.
[34]Young Geol Yoon1,Michael D Koob.Transformation of isolated mammalian mitochondria by bacterial conjugation[J].Nucleic Acids Research,2005,33(16):e139.
[35]Farré J C,Choury D,Araya A.In organello gene expression and RNA editing studies by electroporation-mediated transformation ofisolated plantmitochondria [J].Methods Enzymol,2007,424:483-500.
[36]Goldschmidt-Clemont M.Transgenicexpressionofaminoglycoside adenine transferase in the chloroplast:a selectable marker of site-directed transformation of Chlamydomonas[J].Nucleic Acids Research,1991,19(15):4083-4089.
[37]Boynton J E,Gillham N W.Chloroplast transformation in Chlamydomonas[J].Methods Enzymol,1993,217(4858):510-536.
[38]Kindle K A.High-frequency nuclear transformation of Chlamydomonas reinhardtii[J].Proceedings of the National Academy of Sciences,1990,87(3):1228-1232.
[39]Newman S M,Boynton J E,Gillham N W,et al.Transformation of chloroplast ribosomal RNA genes in Chlamydomonas:molecular and genetic characterization of integration events[J].Genetics,1990,126(4):875-888.
[40]Colleaux L,Michel-Wolwertz M R,Matagne R F,et al.The apocytochrome b gene of Chlamydomonas smithii contains a mobile intron related to both Saccharomyces and Neurospora introns [J].Molecular and General Genetics,1990,223(2):288-296.
